文章信息
- 杨康, 宋蒙飞, 魏庆镇, 王晶, 陈劲枫, 娄群峰
- YANG Kang, SONG Mengfei, WEI Qingzhen, WANG Jing, CHEN Jinfeng, LOU Qunfeng
- 控制黄瓜白绿色果皮基因w的定位和候选基因预测
- Mapping and prediction of candidate gene w controlling white-green fruit color in cucumber
- 南京农业大学学报, 2018, 41(6): 1003-1008
- Journal of Nanjing Agricultural University, 2018, 41(6): 1003-1008.
- http://dx.doi.org/10.7685/jnau.201801054
-
文章历史
- 收稿日期: 2018-01-31
黄瓜(Cucumis sativus L., 2n=14)属于葫芦科甜瓜属植物, 是世界上重要的蔬菜作物。分子标记的发展、高密度遗传图谱的构建和目标基因的发掘有效促进了黄瓜目标性状机制的解析和黄瓜品种选育的进程[1]。但到目前为止, 仅有少量的黄瓜基因被定位到了染色体上[2]。已克隆的目标性状候选基因为数不多, 主要有Bt(苦味果实)、Bl(苦叶)、Tu(有瘤果实)、M(两性花)[3-5]等, 这显然阻碍了目标性状的解析和分子育种的进程。
果实相关性状是黄瓜品种改良的重要性状, 其中果皮颜色是黄瓜商品果外观品质的重要特征之一。我国的黄瓜种质按相关标准可以分为:墨绿、深绿、绿、浅绿、白绿、黄白和乳白7类[6]。目前与黄瓜果皮颜色相关的基因有5个, 分别是深绿色、绿色、黄绿色、浅绿色和白色(w)果皮基因[7]。孙晓丹等[8]研究表明, 控制嫩果白色果皮的基因(w)对绿色基因(yg)为隐性, 且w与其他修饰基因存在互作, 互作类型为隐性上位。董邵云等[9]利用F2群体, 结合BSA法筛选到14个与w基因连锁的SSR标记, 构建了该基因的SSR连锁群, 将其定位到黄瓜3号染色体SSR23517和SSR23141标记之间, 在该区域中共预测了500个候选基因。Liu等[10]利用BC1和F2群体, 将控制白色果皮的基因w定位到3号染色体SNP标记ASPCR39262和ASPCR39229之间的33 kb区间内。Liu等[11]利用包含9 497株的F2大群体将白果皮基因定位到8.2 kb的区间内, 并预测了候选基因。
本研究以1个果皮颜色为白绿色表型的黄瓜自然突变体为试材, 采用群体混池测序技术, 将控制黄瓜白绿色果皮的基因定位在3号染色体上, 然后通过基因注释和qPCR分析, 筛选出白绿色果皮相关的候选基因Csa 3G904140, 旨在为果实果皮颜色的基础研究和遗传改良提供依据与指导。
1 材料与方法 1.1 试验材料供试材料为黄瓜野生型品种‘长春密刺’和突变体, 均由南京农业大学葫芦科作物遗传与种质创新实验室提供。‘长春密刺’是华北型黄瓜, 嫩瓜青绿色, 棍棒形, 棱不明显, 瘤密, 刺白色, 瓜长30~40 cm, 单瓜质量200 g。2013年在田间发现1株果皮颜色表现为白绿色的突变单株, 多代自交得到性状稳定的自交系, 然后与‘长春密刺’进行杂交得到F1代, 2015年秋季得到160株F2群体。所有材料均种植于南京农业大学葫芦科作物遗传与种质创新实验室的江浦实验基地。
1.2 方法 1.2.1 果皮性状遗传分析对亲本、F1代、F2代单株进行果皮颜色的统计, 观察果实从子房期到成熟期的果色变化。以商品果时期的果色统计数据进行遗传分析, 数据用SPSS 16.0软件处理。
1.2.2 叶绿素含量测定叶绿素含量测定采用李合生[12]的方法。分别从授粉后12 d的绿色和白绿色果实上刮取0.1 g果皮, 将其剪碎并放入20 mL混合提取液(乙醇、丙酮、蒸馏水体积比为4.5 : 4.5 : 1)中, 在黑暗下浸泡提取至果皮碎片完全变为白色为止。以提取液为对照, 在752型分光光度计上测定浸提液的D645和D663值, 计算叶绿素和类胡萝卜素含量。
1.2.3 集群分离分析法(BSA)混池的构建及测序从F2代分离群体中选取果皮颜色为绿色和白绿色2个极端性状的植株各20株, 用CTAB法提取DNA并等量混池, 构成突变池和正常池。利用Illumina Hi-Seq 4000测序平台进行测序。原始数据经过两步处理:第一, 过滤接头, 去污染; 第二, 为了得到更高质量的数据, 如果1条reads低质量的碱基数占整条reads比例的50%及以上, 则把该条reads扔掉。得到的所有reads中质量值大于20的碱基占总reads长度的比例均大于94%。
1.2.4 SNP-index分析将子代混合池的数据与黄瓜品种‘9930’参考基因组(384 Mb)进行比较, 对一致性序列数据位点和SNP位点计算SNP-index值, 计算方法为覆盖该位点的突变基因型的reads占覆盖该位点的总的reads数的比例。例如, 覆盖某一位点的reads数目为20, 而突变型的reads数目为15, 那么SNP-index值为0.75。对原始的SNP集进行过滤, 去除质量值小于20的SNP位点。使用1 Mb的窗口进行滑窗分析, 每次步移10 kb, 以窗口为横坐标, SNP-index为纵坐标, 可以得到SNP-index在基因组上的分布曲线。对所有染色体的SNP-index进行绘图, SNP-index值越大, 表示与性状连锁的可能性也越大。结合SNP-index曲线与χ2分布的结果, 预测控制果皮颜色基因的候选区域, 然后在该区域内筛选SNP位点。
SNP位点按以下条件进行筛选:1)SNP在正常池和突变池中只有2种基因型; 2)在正常池中SNP-index应小于0.3, 在突变池中SNP-index应大于0.9;3)碱基的测序质量值和比对质量值均大于30;4)SNP位点的测序深度3~45;5)对杂合SNP位点, 每一种基因型的测序深度都应大于2。Indel位点按以下条件进行筛选:1)突变位点位于CDS区; 2)去除亲本中发生的Indel位点。
1.2.5 候选基因筛选根据对2个极端池测序结果的比对, 结合参考基因组序列以及SNP的筛选条件, 在目标区段内筛选符合上述条件的SNP位点以及Indel位点。根据黄瓜基因组注释结果, 得到该区段内所有基因, 进行基因预测、注释, 筛选候选基因。
1.2.6 基因表达分析分别从绿色和白绿色果实上选取授粉后4、8、12、16、20和24 d的果实, 用灭过菌的解剖刀刮取大小相同的果皮, 立即放入液氮速冻后保存于-80 ℃冰箱待用。用Trizol法提取RNA, 将利用PrimeScriptTM RT反转录试剂盒(TaKaKa)进行cDNA第1链的合成。利用SYBR Premix Ex TaqTM(TaKaRa)的方法在Bio-Rad CFX96荧光定量PCR仪上进行RT-qPCR。该PCR的总体系为20 μL, 其中cDNA模板1 μL, SYBR Premix Ex Taq Ⅱ 10 μL, 正向引物0.5 μL, 反向引物0.5 μL, 双蒸水4 μL。PCR反应程序为:95 ℃ 30 s, 95 ℃ 5 s, 60 ℃ 30 s, 40个循环; 然后进行熔解曲线分析。引物用Primer Premier 5.0软件设计。选取黄瓜看家基因actin作为内参基因, 计算基因的相对表达量。根据单因素方差分析进行差异显著性分析。
2 结果与分析 2.1 突变体果皮颜色表型特征如图 1所示:白绿色突变体的果皮颜色在子房时期与绿色果皮野生型‘长春密刺’较为接近, 随着生长发育的进行果皮颜色逐渐变白, 在12 d时果皮颜色完全变为白绿色。而‘长春密刺’果实的颜色在生长过程中一直表现为绿色, 到老熟果时果色才逐渐变浅。

|
图 1 突变体(左)和野生型(右)不同生长时期果皮颜色的表型特征 Figure 1 The phenotypic characteristics of mutant(left) and wild type(right)fruits at different growth stages |
为了明确果色的差异是否由色素含量的差异导致, 分别测定了2种材料商品瓜时期(12 d)的叶绿素含量。结果表明, 突变体果皮中的叶绿素a和叶绿素b的含量均明显低于野生型‘长春密刺’的含量。
2.2 突变体果实的遗传分析为了明确突变体白绿色果皮颜色的遗传特性, 将白绿色果皮突变体和绿色果皮野生型‘长春密刺’进行杂交, 得到F1代; 随后自交得到F2代。田间性状统计结果显示F1群体有40株表现为绿色果皮。160株F2群体中, 116株表现为绿色果皮表型, 44株为白绿色果皮, 经χ2测验, 符合孟德尔的3 : 1遗传定律, 说明白绿色果皮为单基因控制的隐性性状。将该基因命名为w基因。
2.3 白绿果实w基因候选基因筛选如图 2所示:对所有染色体的SNP-index进行绘图, Y1为突变池, G4为正常池。同时画出2个极端池在同一染色体同一窗口的Δ(SNP-index)绝对值在染色体上的分布, Δ(SNP-index)绝对值越大, 表示与性状连锁的可能性也越大。图中曲线为置信水平为99%的阈值线, 超过此阈值线的区域被视为差异显著的区域。结合SNP-index曲线与χ2分布的结果, 确定了置信水平为99%时, 推测了2个候选区域, 分别为Chr3的35.50~37.77 Mb和38.21~39.71 Mb。
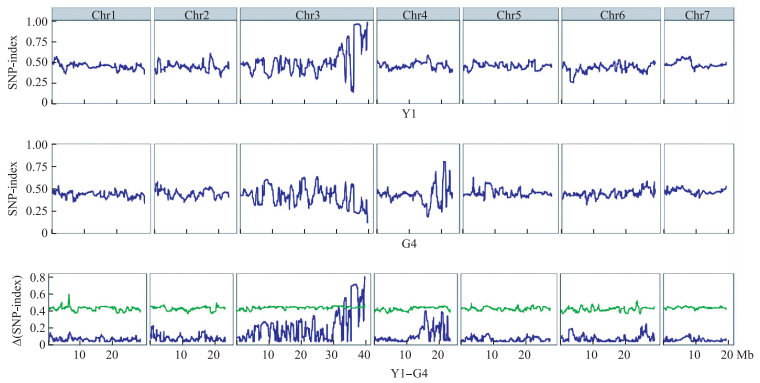
|
图 2 突变体果实染色体的SNP-index分析 Figure 2 SNP-index analysis of the chromosomes of the mutant fruit Y1:突变池Mutant pool; G4:正常池Normal pool. |
该区间内, 符合筛选条件的SNP 621个, 位于CDS区的45个SNP位点, 其中非同义突变有12个, 基因注释表明这12个基因并没有参与叶绿体的代谢途径及果实颜色的调控; 筛选了符合条件的Indel 5个(表 1)。进一步通过基因表达分析发现, 在果实的不同发育时期, Csa 3G904140在2个材料间的表达差异明显, 野生型绿色果实果皮中的表达水平均显著高于突变体白绿色果实果皮(图 3)。Csa 3G904140在白绿色果实果皮和绿色果实果皮中的表达水平都是先逐渐升高然后下降, 在商品瓜(12 d)时表达量最高, 由此推测Csa 3G904140可能是控制黄瓜白绿色果实颜色的候选基因。
| 染色体 Chromosome |
位置 Position |
突变体 Mutant |
野生型 Wild type |
注释 Annotation |
基因 Gene |
| 3 | 37088867 | — | T | 编码区Coding region | Csa3G879520 |
| 3 | 37562736 | — | GC | 编码区Coding region | Csa3G888530 |
| 3 | 38968099 | TTC | — | 编码区Coding region | Csa3G902250 |
| 3 | 39006451 | — | AGC | 编码区Coding region | Csa3G902310 |
| 3 | 39254100 | — | C | 编码区Coding region | Csa3G904140 |
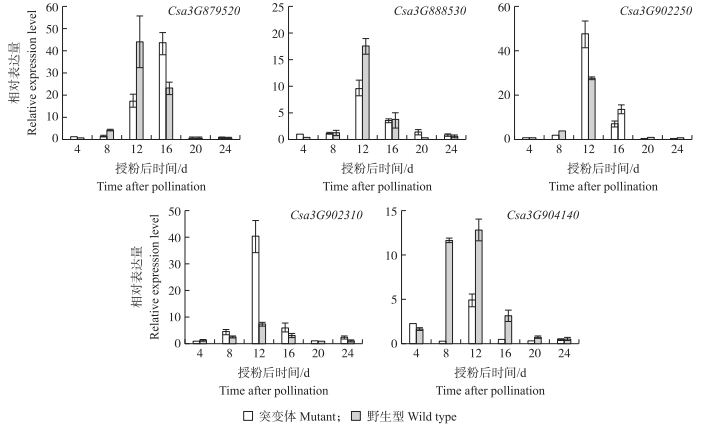
|
图 3 不同时期突变体和野生型果实候选基因的相对表达量 Figure 3 Relative expression level of candidate genes in mutant and wild type fruits at different stages |
对候选基因Csa 3G904140进行了测序以及序列比对分析, 该基因全长4 724 bp, CDS区1 308 bp, 包含12个外显子和11个内含子。如图 4所示:在突变体上检测到1个单核苷酸插入, 插入位点在第9外显子的最后1个核苷酸上。进一步分析其氨基酸序列, 发现了该单核苷酸的插入导致移码突变, 使得终止密码子提前, 从而造成白绿色黄瓜中该蛋白第101个氨基酸残基的缺失。注释结果表明该基因与叶绿体的发育相关。
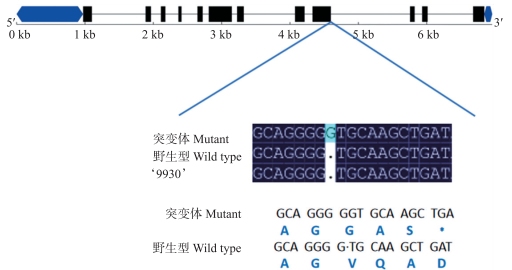
|
图 4 突变体候选基因结构图 Figure 4 Candidate gene structure of mutant |
图位克隆等传统基因定位技术虽然准确性高, 但是需要耗费大量的时间和人力。高通量测序的出现解决了传统基因定位的难题。利用基因组测序则可以直接实现从区间定位到候选基因筛选的工作[13]。集群分离分析法(BSA)是1991年提出的一种快速定位方法[14]。通常在1个分离群体中, 选取在目标性状上具有极端表型的单株构成2个基因池, 对2个池进行变异检测, 利用突变基因与邻近位点共分离的原理, 检测群体中的等位基因频率, 从而缩小定位区域, 识别候选基因。
随着测序成本的降低与通量的提高, 全基因组范围的重测序越来越普及。测序技术与BSA分析结合起来, 大大加快了基因定位研究的周期, 是一种新的技术方法[15]。黄瓜作为蔬菜作物中第1个完成测序的作物, 其基因组信息完善、基因组小, 利用BSA结合重测序(BSA-seq)进行目标性状基因定位较为方便可行[16]。本研究中, 利用F2分离群体, 采用BSA-seq方法成功将控制黄瓜白绿色果皮的基因定位到了3号染色体上, 并通过序列比对、基因表达等综合分析确定Csa 3G904140是控制黄瓜白绿色果实颜色的候选基因。该基因与Liu等[11]利用F2大群体定位到的控制白色果皮的基因APRR2完全一致, 都是由于第9外显子上1个单核苷酸插入造成终止密码子提前, 使101氨基酸残基缺失, 从而导致果实果皮颜色表现为白绿色。但是本研究的果实果皮颜色比Liu等[11]研究的果皮颜色要深, 这也说明该性状可能还被其他因素影响。
R2R3-MYB转录因子是许多作物中常见的控制果实颜色的基因[17-19], 其主要调控花青素生物合成[20-21]。类胡萝卜素是植物中具有重要功能的色素之一, 在叶绿体中高水平积累[22]。叶绿素是负责植物叶片和未成熟果实表现为绿色的物质, 叶绿素代谢是一系列酶催化反应高度协调的过程[23]。拟南芥中有27个编码这类酶的基因[24], 然而, 候选基因w似乎并不参与叶绿素生物合成途径[25]。w同源基因在番茄和甜椒的表达与质体数量的增加及在果实组织中的叶绿体色素积累有关[25-26], 但是拟南芥、番茄或辣椒中w基因的机制仍然不清楚[25], 可能是绿色黄瓜中w基因的表达参与了成熟信号机制以及叶绿素的积累和叶绿体的发育[25, 27]。而w基因编码区的1个单核苷酸插入导致移码位移使终止密码子提前, 从而形成了白绿色黄瓜基因型。因此, 我们推测这种C末端101个氨基酸的缺乏阻碍了成熟信号过程中基因产生生物功能, 从而导致叶绿素含量低以及叶绿体数量少。本研究显示, 绿色果实和白绿色果实的叶绿素含量与w基因的表达水平具有明显的相关性, 这表明w基因对叶绿体发育和叶绿素生物合成, 以及黄瓜果皮外观有重要的调节作用, 但w基因的具体调节机制还需要进一步探究。本研究结果有助于果实颜色形成的基础研究, 同时为黄瓜果实性状改良提供了指导。
| [1] |
Huang S, Li R, Zhang Z, et al. The genome of the cucumber, Cucumis sativus L.[J]. Nature Genetics, 2009, 41(12): 1275-1281. DOI:10.1038/ng.475 |
| [2] |
Song M, Wei Q, Wang J, et al. Fine mapping of CsVYL, conferring virescent leaf through the regulation of chloroplast development in cucumber[J]. Frontiers in Plant Science, 2018, 9: 432. DOI:10.3389/fpls.2018.00432 |
| [3] |
Li Z, Wang S, Tao Q, et al. A putative positive feedback regulation mechanism in CsACS2 expression suggests a modified model for sex determination in cucumber (Cucumis sativus L.)[J]. Journal of Experimental Botany, 2012, 63(12): 4475-4484. DOI:10.1093/jxb/ers123 |
| [4] |
Shang Y, Ma Y, Zhou Y, et al. Biosynthesis, regulation, and domestication of bitterness in cucumber[J]. Science, 2014, 346: 1084. DOI:10.1126/science.1259215 |
| [5] |
Yang X, Zhang W, He H, et al. Tuberculate fruit gene Tu encodes a C2H2 zinc finger protein that is required for the warty fruit phenotype in cucumber (Cucumis sativus L.)[J]. Plant Journal for Cell & Molecular Biology, 2014, 78(6): 1034-1046. |
| [6] |
王建科, 方小雪, 李雪红, 等. 黄瓜嫩果皮颜色的遗传研究[J]. 园艺学报, 2013, 40(3): 479-486. Wang J K, Fang X X, Li X H, et al. Genetic studies on the color of cucumber tender peel[J]. Acta Horticulturae Sinica, 2013, 40(3): 479-486 (in Chinese with English abstract). |
| [7] |
Xie J. Gene list 2001 for cucumber[J]. Cucurbit Genetics Cooperative Report, 2001, 24: 110-136. |
| [8] |
孙晓丹, 商庆梅, 秦智伟. 黄瓜嫩果白色果皮颜色遗传规律及其AFLP标记研究[J]. 北方园艺, 2011(3): 135-140. Sun X D, Shang Q M, Qin Z W. Study on the genetic rule of white fruit color of cucumber and its AFLP markers[J]. Northern Horticulture, 2011(3): 135-140 (in Chinese). |
| [9] |
董邵云, 苗晗, 张圣平, 等. 黄瓜白色果皮基因遗传规律及定位研究[J]. 西北植物学报, 2012, 32(11): 2177-2181. Dong S Y, Miao H, Zhang S P, et al. Genetic rule and location of cucumber white peel gene[J]. Acta Botanica Boreali-Occidentalia Sinica, 2012, 32(11): 2177-2181 (in Chinese with English abstract). DOI:10.3969/j.issn.1000-4025.2012.11.005 |
| [10] |
Liu H, Meng H, Pan Y, et al. Fine genetic mapping of the white immature fruit color gene w, to a 33.0-kb region in cucumber (Cucumis sativus L.)[J]. Theoretical and Applied Genetics, 2015, 128(12): 2375-2385. DOI:10.1007/s00122-015-2592-z |
| [11] |
Liu H, Jiao J, Liang X, et al. Map-based cloning, identification and characterization of the w gene controlling white immature fruit color in cucumber (Cucumis sativus L.)[J]. Theoretical and Applied Genetics, 2016, 129(7): 1247-1256. DOI:10.1007/s00122-016-2700-8 |
| [12] |
李合生. 植物生理生化实验原理和技术[M]. 北京: 高等教育出版社, 2000. Li H S. Principle and Technology of Plant Physiological and Biochemical Experiment[M]. Beijing: Higher Education Press, 2000 (in Chinese). |
| [13] |
Schneeberger K, Ossowski S, Lanz C, et al. SHOREmap:simultaneous mapping and mutation identification by deep sequencing[J]. Nature Methods, 2009, 6(8): 550. DOI:10.1038/nmeth0809-550 |
| [14] |
Michelmore R W, Paran I, Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis:a rapid method to detect markers in specific genomic regions by using segregating populations[J]. Proc Natl Acad Sci USA, 1991, 88(21): 9828-9832. DOI:10.1073/pnas.88.21.9828 |
| [15] |
Abe A, Kosugi S, Yoshida K, et al. Genome sequencing reveals agronomically important loci in rice using MutMap[J]. Nature Biotechnology, 2012, 30(2): 174. DOI:10.1038/nbt.2095 |
| [16] |
白凤虎, 李德芳, 陈安国, 等. 基于BSA分析法的分子标记基因定位技术在农作物中的应用[J]. 中国麻业科学, 2006, 28(6): 282-288. Bai F H, Li D F, Chen A G, et al. The application of the molecular gene localization technique based on BSA in crop[J]. Plant Fiber Sciences in China, 2006, 28(6): 282-288 (in Chinese with English abstract). DOI:10.3969/j.issn.1671-3532.2006.06.002 |
| [17] |
Borevitz J O, Xia Y, Blount J, et al. Activation tagging identifies a conserved MYB regulator of phenylpropanoid biosynthesis[J]. The Plant Cell, 2000, 12(12): 2383-2393. DOI:10.1105/tpc.12.12.2383 |
| [18] |
Feller A, Machemer K, Braun E L, et al. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J]. Plant Journal, 2011, 66(1): 94-116. DOI:10.1111/tpj.2011.66.issue-1 |
| [19] |
Singh R, Low E T L, Ooi C L, et al. The oil palm VIRESCENS gene controls fruit colour and encodes a R2R3-MYB[J]. Nature Communications, 2014, 5: 4106. DOI:10.1038/ncomms5106 |
| [20] |
Han S E, Lee H E, Heo S, et al. Isolation and characterization of genes expressed differently in mature fruits of 'Redfield' and 'Greensleeves' apples[J]. Hort Environ Biotechnol, 2011, 52(4): 413-421. DOI:10.1007/s13580-011-0213-6 |
| [21] |
Kayesh E, Shangguan L, Korir N K, et al. Fruit skin color and the role of anthocyanin[J]. Acta Physiologiae Plantarum, 2013, 35(10): 2879-2890. DOI:10.1007/s11738-013-1332-8 |
| [22] |
Lu S, van Eck J, Zhou X J, et al. The cauliflower Or gene encodes a DnaJ cysteine-rich domain-containing protein that mediates high levels of β-carotene accumulation[J]. The Plant Cell, 2006, 18(12): 1-12. |
| [23] |
Beale S I. Enzymes of chlorophyll biosynthesis[J]. Photosynthesis Research, 1999, 60(1): 43-73. DOI:10.1023/A:1006297731456 |
| [24] |
Beale S I. Green genes gleaned[J]. Trends in Plant Science, 2005, 10(7): 309-312. DOI:10.1016/j.tplants.2005.05.005 |
| [25] |
Pan Y, Bradley G, Pyke K, et al. Network inference analysis identifies an APRR2-like gene linked to pigment accumulation in tomato and pepper fruits[J]. Plant Physiology, 2013, 161(3): 1476-1485. DOI:10.1104/pp.112.212654 |
| [26] |
Brand A, Borovsky Y, Meir S, et al. pc8.1, a major QTL for pigment content in pepper fruit, is associated with variation in plastid compartment size[J]. Planta, 2012, 235(3): 579-588. DOI:10.1007/s00425-011-1530-9 |
| [27] |
Barry C S, Aldridge G M, Herzog G. Altered chloroplast development and delayed fruit ripening caused by mutations in a zinc metalloprotease at the lutescent2 locus of tomato[J]. Plant Physiology, 2012, 159(3): 1086-1098. DOI:10.1104/pp.112.197483 |




