文章信息
- 李纯, 王超龙, 陈赛华, 郑天慧, 鲁健, 崔松, 刘宗凯, 陈亮明, 江玲, 周时荣, 万建民
- LI Chun, WANG Chaolong, CHEN Saihua, ZHENG Tianhui, LU Jian, CUI Song, LIU Zongkai, CHEN Liangming, JIANG Ling, ZHOU Shirong, WAN Jianmin
- 水稻RZ54/南京11 F2群体抽穗期QTL分析
- QTL analysis of heading date in rice RZ54/Nanjing11 F2 population
- 南京农业大学学报, 2018, 41(5): 801-807
- Journal of Nanjing Agricultural University, 2018, 41(5): 801-807.
- http://dx.doi.org/10.7685/jnau.201803003
-
文章历史
- 收稿日期: 2018-03-01
2. 中国农业科学院作物科学研究所, 北京 100081
2. Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China
水稻抽穗期是重要的农艺性状,挖掘抽穗期基因,培育适当早熟的高产品种具有重要价值。许多研究者利用不同的定位群体定位了一批对水稻抽穗期有影响的QTL。Yano等[1-2]定位了5个影响水稻抽穗期的QTL(Hd1—Hd5),其中Hd1和Hd2是主效QTL,其余3个则是微效QTL,这5个QTL可解释水稻抽穗期变异的84%。通过构建含有Hd1、Hd2和Hd3的NIL群体,Yamamoto等[3]精细定位了这3个QTL。Monna等[4]也通过精细定位后发现Hd3a和Hd3b这2个QTL,由于其是紧密连锁的,所以在之前的定位中仅表现为Hd3这1个QTL。Lin等[5]通过近等基因系精细定位了Hd 4和Hd5。郭龙彪等[6]通过对1个籼籼交RIL群体进行抽穗期QTL定位,得到3个QTL位点,其中效应最大的QTL位点QHd7连续2年都被检测到。陈俊宇等[7]利用NIL将1个同时控制抽穗期和产量性状的QTL qHd1定位在95 kb的范围内。这些精细定位的结果为抽穗期QTL的克隆打下了良好的基础。
通过对水稻抽穗期QTL分析,目前已有多个QTL位点被克隆,进一步完善了水稻抽穗期调控的分子遗传机制[8]。Hd1是水稻中首个被克隆的控制开花期的基因[1]。‘Kasalath’中的Hd1相比于‘日本晴’发生了多处突变,导致功能缺失[1-2]。Hd1在短日照条件下促进抽穗,长日照条件下抑制抽穗[9],而Hd1对抽穗期的影响与Hd3a等成花素基因的表达水平密切相关[10]。Hd3a是第1个被克隆的水稻成花素基因,其转录水平直接影响水稻的抽穗期[11]。除了Hd1和Hd3a调控开花的途径,在水稻中还存在1条以Ehd1作为核心的调控抽穗期的通路[12]。Ehd1的QTL已被克隆[13-14]。在诸多调控因子中,Ghd7是直接调控Ehd1的关键基因,它在长日照下通过直接或间接的方式抑制Ehd1的表达而延迟抽穗[15]。Xue等[16]克隆了这一重要调节因子Ghd7,它能同时控制水稻每穗粒数、株高和抽穗期3个性状。DTH8/Ghd8与Ghd7类似,主要在长日照下抑制Ehd1的表达而延迟抽穗[17-18]。Wu等[19]定位并克隆到1个微效抽穗期QTL——DTH2,由此发现了DTH2介导的1条独立于Hd1和Ehd1的新的光周期开花途径。
水稻抽穗期QTL的定位分析,是解析复杂数量性状水稻抽穗期基因位点的重要方法,也是抽穗期调控网络不断完善的重要前提。本研究利用早熟品种‘RZ54’与晚熟品种‘南京11’杂交构建F2群体,进行抽穗期的QTL定位,以期发掘出新的控制早熟且有育种利用价值的QTL位点,为后续重要位点的克隆奠定了基础。
1 材料与方法 1.1 供试材料水稻品种‘RZ54’是在南京农业大学水稻研究所种质资源库中来自日本的早熟粳稻资源,抽穗期比‘南京11’等本地品种明显提前。以‘RZ54’作为母本,‘南京11’作为父本杂交获得F2分离群体,从F2群体中随机选取184个单株进行QTL定位。F2群体及其亲本于2016年5月18日播种,6月18日移栽于南京农业大学土桥水稻育种试验站,生长期间管理同常规大田生产。
1.2 试验方法 1.2.1 抽穗期的调查方法对F2分离群体分单株编号、取样并记载抽穗期,单株以第1穗穗尖露出叶鞘1 cm作为该单株始穗的标准,以播种到始穗的时间(d)作为抽穗期,每隔2 d调查1次。
1.2.2 DNA的提取参照沈子杰等[20]的方法,采用CTAB方法提取DNA。
1.2.3 多态性分子标记的筛选利用本实验室已有的引物,包括SSR(simple sequence repeat,简单序列重复)标记、Indel(insertion-deletion,插入缺失)标记等,从中选出具有多态性的141对引物对F2群体进行分子标记基因型分析。
1.2.4 PCR扩增PCR扩增采用沈子杰等[20]的方法,部分分子标记扩增时对退火温度进行适当调整。PCR产物经80 g · L-1非变性聚丙烯酰胺凝胶检测,银染显色后于灯箱上读取数据。
1.2.5 分子连锁图谱的构建利用QTL IciMapping v3.2软件中的MAP功能进行标记间的连锁分析。根据各标记之间的连锁关系,采用Kosambi函数将重组率转换成遗传距离(centimorgan,cM)并绘图[21]。
1.2.6 数据统计分析利用QTL IciMapping v3.2软件的完备区间作图法(ICIM)在全基因组范围内检测控制抽穗期的位点[21]。PIN设为0.01,每1 cM步长(walking speed)对目的性状进行全基因组扫描,采用模拟运算(permutation)1 000次的方法确定LOD阈值,当某个位置检测到的LOD值大于阈值时,就认为该位置存在1个QTL。在5%显著性水平下,2.5为临界值,即取LOD≥2.5作为判定QTL存在的依据。
2 结果与分析 2.1 亲本及F2群体抽穗期的表现在南京地区夏季种植,‘RZ54’和‘南京11’的平均抽穗期为60.2 d和86.1 d,F2群体的抽穗期为55~130 d,抽穗期性状在F2群体中呈现连续分布和双向超亲分离(图 1),这说明抽穗期性状受多基因控制,在‘RZ54’与‘南京11’之间可能存在多个影响抽穗期的位点。另外,F2群体中大部分单株偏向于迟熟,这可能与籼粳亚种间杂交的“疯狂分离”有关。
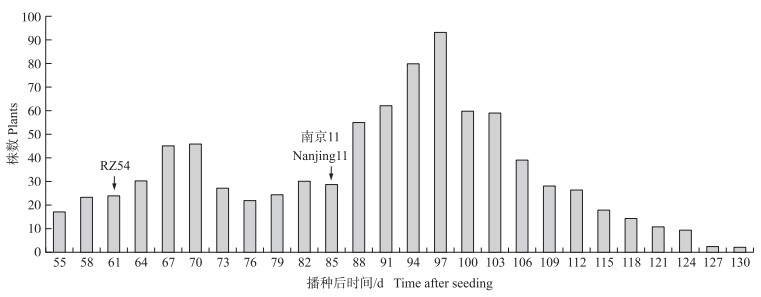
|
图 1 RZ54/南京11 F2群体在南京抽穗期的频率分布 Figure 1 Distribution of heading date of RZ54/Nanjing11 F2 population in Nanjing |
本研究利用包含184个单株的RZ54/南京11 F2群体,构建了1张包含141个分子标记,全长为1 741.4 cM,覆盖水稻全基因组12条染色体的连锁图谱(表 1,图 2)。每条染色体标记数为9~17个,遗传距离为93.3~244.6 cM,标记间平均距离为12.35 cM,符合QTL作图的基本条件。其中第3染色体的标记数最多,第8染色体覆盖距离最长,第11染色体的标记密度最大,标记间的距离及顺序与已发表的分子图谱有较好的一致性,能满足定位的要求。
| 染色体Chromosome | 标记数No.of markers | 覆盖距离/cM Distance | 标记间平均距离/cM Average interval distance |
| 1 | 14 | 194.3 | 13.88 |
| 2 | 10 | 136.6 | 13.66 |
| 3 | 17 | 195.2 | 11.48 |
| 4 | 11 | 118.3 | 10.75 |
| 5 | 10 | 128.8 | 12.88 |
| 6 | 10 | 123.0 | 12.30 |
| 7 | 12 | 124.5 | 10.38 |
| 8 | 13 | 244.6 | 18.81 |
| 9 | 12 | 135.7 | 11.31 |
| 10 | 12 | 105.8 | 8.81 |
| 11 | 11 | 93.3 | 8.48 |
| 12 | 9 | 141.3 | 15.7 |
| 合计/平均Total/Mean | 141 | 1 741.4 | 12.35 |
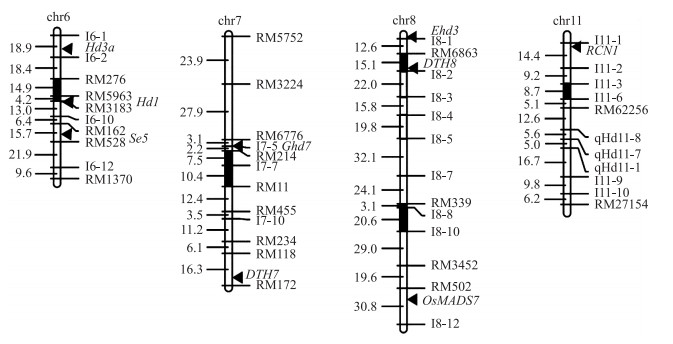
|
图 2 RZ54/南京11 F2群体的遗传图谱及检测的抽穗期QTL Figure 2 Molecular linkage map and QTL controlling heading date of RZ54/Nanjing11 F2 population ■本文检测到的QTL位点QTL detected in this paper;◀已报道的抽穗期位点Reported heading date locus. |
在此基础上对抽穗期性状进行QTL分析,共检测到与抽穗期有关的5个QTL位点(图 2),分布在第6、7、8和11染色体上,分别命名为qHd-6、qHd-7、qHd-8-1、qHd-8-2和qHd-11。
2.3 抽穗期QTL效应位点的分析qHd-6位于第6染色体的第3个标记RM276和第5个标记RM3183之间,贡献率为6.53%,LOD值为4.21,2个标记之间的遗传距离为19.1 cM,该位点的加性效应为-7.16,来自于亲本‘南京11’(表 2)。在第6染色体该效应区段内并未存在已克隆的抽穗期相关基因,但在其下游位置存在1个已报道的抽穗期相关基因Hd1。由于作图群体不同且群体量小、群体遗传背景差异大、计算方法不同等原因,很可能会导致QTL位点区间的偏移,因此不排除该位点的效应是由Hd1 引起的[22-23]。
| 基因座 Locus |
染色体 Chromosome |
标记区间 Interval |
区间距离/cM Distance |
贡献率/% Variance |
LOD值 LOD score |
加性效应 Additive |
| qHd-6 | 6 | RM276—RM3183 | 19.1 | 6.53 | 4.21 | -7.16 |
| qHd-7 | 7 | RM214—RM11 | 17.9 | 15.19 | 8.78 | -9.35 |
| qHd-8-1 | 8 | RM6863—I8-2 | 15.1 | 20.18 | 10.87 | 11.89 |
| qHd-8-2 | 8 | RM339—I8-10 | 23.7 | 7.63 | 4.76 | 6.16 |
| qHd-11 | 11 | I11-3—I11-6 | 8.7 | 5.40 | 3.28 | 2.98 |
qHd-7位于第7染色体的第5个标记RM214和第7个标记RM11之间,贡献率为15.19%,LOD值为8.78,2个标记间的遗传距离为17.9 cM。该位点的加性效应为-9.35,来自于亲本‘南京11’(表 2)。在该效应区段内尚未有已报道的抽穗期相关基因,但已报道的重要抽穗期基因Ghd7位于其上游。QTL是一种对统计概率的判断,在QTL的初步定位中,同一个QTL在不同群体中,用不同的作图方式计算,它的位置往往会产生偏移。因此我们需要进一步考虑这个位置的效应是否与Ghd7有关。
在第8染色体上检测到2个效应位点,qHd-8-1位于第2个标记RM6863和第3个标记I8-2之间,该位点的贡献率比较大,解释表型变异的20.18%,LOD值为10.87,标记之间的遗传距离为15.1 cM,该位点的加性效应为11.89,来自于亲本‘RZ54’。第9个标记I8-8和第10个标记I8-10之间存在1个贡献率为7.63%、LOD值为4.76的位点qHd-8-2 ,标记之间的遗传距离为23.7 cM,该位点的加性效应为6.16,来自于亲本‘RZ54’(表 2)。这2个位点对早熟性有较大的贡献。分析发现在qHd-8-1 区段内存在1个已克隆的抽穗期基因DTH8 ,然而后一个主效位点则没有明确的已报道基因。
第11染色体的第3个标记I11-3和第4个标记I11-6之间存在1个贡献率为5.40%、LOD值为3.28的效应位点qHd-11 ,2个标记之间的遗传距离为8.7 cM,该位点的加性效应为2.98,来自于亲本‘RZ54’,对早熟性有微小贡献(表 2)。在该效应区段内并未发现已报道的抽穗期相关基因。
在效应区段qHd-6、qHd-7附近分别存在已报道的抽穗期相关基因Hd1和Ghd7,而qHd-8-1的效应区段内发现了已克隆的重要的抽穗期相关基因DTH8,为进一步确认或者排除这些效应是否受这些基因的影响,我们分别对亲本‘RZ54’与‘南京11’的这些基因位置进行测序。
在Hd1 这个基因位点上,通过测序发现‘RZ54’与‘南京11’之间仅存在1个单碱基差异,这个变异发生在外显子上,造成1个氨基酸的差异,‘RZ54’中第165位的天冬氨酸在‘南京11’中为赖氨酸(图 3)。

|
图 3 ‘RZ54’与‘南京11’的Hd1蛋白氨基酸序列比对 Figure 3 Amino acid sequence alignment of Hd1 protein between'RZ54'and'Nanjing11' A. RZ54;B.南京11 Nanjing11.The same as follows. |
在Ghd7 这个位点上,‘RZ54’与‘南京11’的差异较大。在基因组上检测到13个单碱基差异和1个碱基的缺失,其中有3个单碱基差异发生在外显子上,造成3个氨基酸的差异,‘RZ54’中第141位的丙氨酸在‘南京11’中为甘氨酸,第166位的甘氨酸变为丝氨酸,而第179位的甲硫氨酸则变为缬氨酸(图 4)。

|
图 4 ‘RZ54’与‘南京11’的Ghd7蛋白氨基酸序列比对 Figure 4 Amino acid sequence alignment of Ghd7 protein between'RZ54'and'Nanjing11' |
我们也对‘RZ54’和‘南京11’的DTH8基因组进行了测序,发现有14个单碱基差异与1段8个碱基的缺失,其中有7个单碱基差异发生在外显子上,导致5个氨基酸的变化,‘RZ54’中第19位的亮氨酸在‘南京11’中为丝氨酸,第25位的谷氨酸变为丙氨酸,第189位的酪氨酸变为丝氨酸,第219位的半胱氨酸变为丙氨酸,而第295位的甲硫氨酸则变为丝氨酸(图 5)。

|
图 5 ‘RZ54’与‘南京11’的DTH8蛋白氨基酸序列比对 Figure 5 Amino acid sequence alignment of DTH8 protein between'RZ54'and'Nanjing11' |
本研究对‘RZ54’和‘南京11’的F2群体进行全基因组扫描,分别在第6、7、8和11染色体上发现5个具有效应的QTL位点。其中,qHd-8-1 是检测到的贡献率(20.18%)最大位点,但是该区段内存在1个已克隆的抽穗期基因DTH8 ,Yan等[17]和Wei等[18]都定位到这一位点,由此可见该区域控制抽穗期的QTL具有较好的重现性。DTH8 编码NF-YB11,属于NF-Y转录因子复合体的1个亚基,在拟南芥中NF-YB中有1个天冬氨酸Asp-55残基,是NF-YB蛋白的功能位点,参与调控种子的胚胎形成[24]。我们研究发现‘RZ54’与‘南京11’在DTH8的氨基酸序列中也存在几个氨基酸的变异,这些氨基酸的改变也有可能影响其功能。Wei等[18]对多个品种中的DTH8 基因进行测序,将其分为8种基因型,经过对比分析,‘RZ54’属于其中的Ⅰ型,是有功能的DTH8 ,而‘南京11’则属于Ⅷ型,是无功能的DTH8,在我们检测到的效应位点中,qHd-8-1是正加性效应,说明该效应来自于‘RZ54’,与Wei等[18]的结论一致。
qHd-6、qHd-7位点附近都分别存在已克隆的抽穗期基因Hd1与Ghd7,不排除这2个QTL的效应受到这些基因的影响。测序发现,在‘RZ54’和‘南京11’之间,Hd1 的编码区有1个碱基的差异从而导致1个氨基酸的变化。魏鑫[25]对多份栽培稻材料进行测序,发现Hd1 在各品种中的变异很大,但大多没有造成Hd1 功能的改变,‘RZ54’中的Hd1 属于其中一种有功能的基因型。对于该位点的分析存在以下2种可能性。其一,在该效应区段很有可能还存在另外一个微效位点影响抽穗期的改变。其二,这个突变位点虽然没有造成Hd1 功能的丧失,但是可能在‘南京11’中Hd1 在长日照下对于抽穗的抑制作用更明显,因此有1个来自于‘南京11’的微效效应。在Hd3a 的定位与克隆中也有类似的情况[4]。由于Hd1 具有短日照促进抽穗、长日照抑制抽穗的双功能性,下一步可以通过回交构建qHd-6 的近等基因系,对其在不同日照情况下的效应进行检测,分析其与Hd1 的关系。对‘RZ54’和‘南京11’中的Ghd7 位点进行测序,发现多个造成氨基酸差异的碱基变异。Xue等[16]对19个品种中的Ghd7进行测序,将其基因型分为Ghd7-1、Ghd7-2、Ghd7-3、Ghd7-0和Ghd7-0a。经过比较分析,‘RZ54’中的Ghd7 属于其中的Ghd7-2型,而‘南京11’中的Ghd7 属于原始的野生型Ghd7-1,研究发现,Ghd7-2比Ghd7-1具有更小的表型影响,这与表型相符合。在我们检测到的效应位点中,qHd-7 是负加性效应,说明‘南京11’在这个位点的效应更大,这也与之前的结论相吻合。
我们得到的qHd-11和qHd-8-2区段内都没有发现已报道的抽穗期基因,这2个位点的贡献率都较小。它们很有可能是一个新的较为微效的控制抽穗期的QTL位点。在水稻育种中大幅度调整抽穗期可能带来减产等负面效应,实践中往往期望对抽穗期的适度调整,因此微效基因在抽穗期的精准改良中具有重要价值。为了研究qHd-11、qHd-8-2这2个新位点,应该利用高代回交的方法将QTL分解成单个性状,从而更好地对这些QTL进行研究。我们的研究结果为后续工作提供了方向。
| [1] | Yano M, Katayose Y, Ashikari M, et al. Hd1, a major photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering time gene CONSTANS[J]. Plant Cell, 2000, 12(12): 2473-2483. DOI: 10.1105/tpc.12.12.2473 |
| [2] | Yano M, Harushima Y, Nagamura Y, et al. Identification of quantitative trait loci controlling heading date in rice using a high-density linkage map[J]. Theoretical and Applied Genetics, 1997, 95(7): 1025-1032. DOI: 10.1007/s001220050658 |
| [3] | Yamamoto T, Kuboki Y, Lin S Y, et al. Fine mapping of quantitative trait loci Hd-1, Hd-2 and Hd-3, controlling heading date of rice, as single Mendelian factors[J]. Theoretical and Applied Genetics, 1998, 97(1/2): 37-44. |
| [4] | Monna L, Lin H X, Kojima S, et al. Genetic dissection of a genomic region for a quantitative trait locus, Hd3, into two loci, Hd3a and Hd3b, controlling heading date in rice[J]. Theoretical and Applied Genetics, 2002, 104(5): 772-778. DOI: 10.1007/s00122-001-0813-0 |
| [5] | Lin H X, Liang Z W, Sasaki T, et al. Fine mapping and characterization of quantitative trait loci Hd4 and Hd5 controlling heading date in rice[J]. Breeding Science, 2003, 53(1): 51-59. DOI: 10.1270/jsbbs.53.51 |
| [6] |
郭龙彪, 罗利军, 邢永忠, 等. 汕优63重组自交系群体重要农艺性状遗传分析和利用[J].
作物学报, 2002, 28(5): 644-649.
Guo L B, Luo L J, Xing Y Z, et al. Genetic analysis and utilization of the important agronomic traits on Zhenshan 97×Minghui 63 recombinant inbred lines(RIL)in rice(Oryza sativa L.)[J]. Acta Agronomica Sinica, 2002, 28(5): 644-649. DOI: 10.3321/j.issn:0496-3490.2002.05.012 (in Chinese with English abstract) |
| [7] |
陈俊宇.水稻抽穗期和产量性状微效QTL qHd1的精细定位[D].北京: 中国农业科学院, 2016.
Chen J Y.Fine mapping of qHd1, a minor QTL having pleiotropic effects for heading date and yield traits in indica rice[D]. Beijing: Chinese Academy of Agricultural Sciences, 2016(in Chinese with English abstract). |
| [8] | Matsubara K, Hori K, Ogiso-Tanaka E, et al. Cloning of quantitative trait genes from rice reveals conservation and divergence of photoperiod flowering pathways in Arabidopsis and rice[J]. Frontiers in Plant Science, 2014, 5: 193. |
| [9] | Tsuji H, Taoka K, Shimamoto K. Florigen in rice:complex gene network for florigen transcription, florigen activation complex, and multiple functions[J]. Current Opinion in Plant Biology, 2013, 16(2): 228-235. DOI: 10.1016/j.pbi.2013.01.005 |
| [10] | Ishikawa R, Aoki M, Kurotani K, et al. Phytochrome B regulates heading date 1(Hd1)-mediated expression of rice florigen Hd3a and critical day length in rice[J]. Molecular Genetics and Genomics, 2011, 285(6): 461-470. DOI: 10.1007/s00438-011-0621-4 |
| [11] | Tamaki S, Tsuji H, Matsumoto A, et al. FT-like proteins induce transposon silencing in the shoot apex during floral induction in rice[J]. Proc Natl Acad Sci USA, 2015, 112(8): E901-E910. DOI: 10.1073/pnas.1417623112 |
| [12] | Naranjo L, Talon M, Domingo C. Diversity of floral regulatory genes of japonica rice cultivated at northern latitudes[J]. BMC Genomics, 2014, 15: 101. DOI: 10.1186/1471-2164-15-101 |
| [13] | Doi K, Izawa T, Fuse T, et al. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1l[J]. Genes and Development, 2004, 18(8): 926-936. DOI: 10.1101/gad.1189604 |
| [14] | Hori K, Ogiso-Tanaka E, Matsubara K, et al. Hd16, a gene for casein kinase I, is involved in the control of rice flowering time by modulating the day-length response[J]. Plant Journal, 2013, 76(1): 36-46. |
| [15] | Weng X, Wang L, Wang J, et al. Grain number, plant height, and heading date7 is a central regulator of growth, development, and stress response[J]. Plant Physiol, 2014, 164(2): 735-747. DOI: 10.1104/pp.113.231308 |
| [16] | Xue W Y, Xing Y Z, Weng X Y, et al. Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice[J]. Nature Genetics, 2008, 40(6): 761-767. DOI: 10.1038/ng.143 |
| [17] | Yan W H, Wang P, Chen H X, et al. A major QTL, Ghd8, plays pleiotropic roles in regulating grain productivity, plant height, and heading date in rice[J]. Molecular Plant, 2011, 4(2): 319-330. DOI: 10.1093/mp/ssq070 |
| [18] | Wei X, Xu J, Guo H, et al. DTH8 suppresses flowering in rice, influencing plant height and yield potential simultaneously[J]. Plant Physiology, 2010, 153(4): 1747-1758. DOI: 10.1104/pp.110.156943 |
| [19] | Wu W X, Zheng X M, Lu G W, et al. Association of functional nucleotide polymorphisms at DTH2 with the northward expansion of rice cultivation in Asia[J]. Proc Natl Acad Sci USA, 2013, 110(8): 2775-2780. DOI: 10.1073/pnas.1213962110 |
| [20] |
沈子杰, 范德佳, 何俊, 等. 籼稻品种'IR13427-45-2-1-2-2-2'抗白背飞虱QTL定位[J].
南京农业大学学报, 2017, 40(6): 957-962.
Shen Z J, Fan D J, He J, et al. Mapping of the quantitative trait locus conferring white-backed planthopper resistance in indica cultivar 'IR13427-45-2-1-2-2-2'[J]. Journal of Nanjing Agricultural University, 2017, 40(6): 957-962. DOI: 10.7685/jnau.201704027 (in Chinese with English abstract) |
| [21] | Li H, Ye G, Wang J. A modified algorithm for the improvement of composite interval mapping[J]. Genetics, 2007, 175(1): 361-374. |
| [22] | Darvasi A, Soller M. A simple method to calculate resolving power and confidence interval of QTL map location[J]. Behavior Genetics, 1997, 27(2): 125-132. DOI: 10.1023/A:1025685324830 |
| [23] |
苏成付, 赵团结, 盖钧镒. 不同统计遗传模型QTL定位方法应用效果的模拟比较[J].
作物学报, 2010, 36(7): 1100-1107.
Sun C F, Zhao T J, Gei J Y. Simulation comparisons of effectiveness among QTL mapping procedures of different statistical genetic models[J]. Acta Agronomica Sinica, 2010, 36(7): 1100-1107. (in Chinese with English abstract) |
| [24] | Lee H S, Fischer R L, Goldberg R B, et al. Arabidopsis LEAFY COTYLEDON1 represents a functionally specialized subunit of the CCAAT binding transcription factor[J]. Proc Natl Acad Sci USA, 2003, 100(4): 2152-2156. DOI: 10.1073/pnas.0437909100 |
| [25] |
魏鑫.基于核苷酸多态性的亚洲栽培稻起源进化研究[D].武汉: 华中农业大学, 2013.
Wei X.Origin and evolution of Oryza sativa revealed by nucleotide polymorphisms[D]. Wuhan: Huazhong Agricultural University, 2013(in Chinese with English abstract). |




