文章信息
- 肖连杰, 黄捷, 曹鹏辉, 牟昌龄, Thanhliem Nguyen, 刘世家, 陈亮明, 江玲
- XIAO Lianjie, HUANG Jie, CAO Penghui, MOU Changling, Thanhliem Nguyen, LIU Shijia, CHEN Liangming, JIANG Ling
- 水稻早衰突变体zs的鉴定与基因定位
- Analysis and gene mapping of rice premature senescence mutant zs
- 南京农业大学学报, 2018, 41(5): 793-800
- Journal of Nanjing Agricultural University, 2018, 41(5): 793-800.
- http://dx.doi.org/10.7685/jnau.201802016
-
文章历史
- 收稿日期: 2018-02-08
在叶片衰老过程中,叶片细胞及结构经历着复杂的代谢变化和顺序退化,如植株叶色逐渐变黄、枯死,光合能力下降,叶绿体结构破坏,脂质、蛋白质、核酸水解,营养物质转移到植物其他部位,细胞死亡,最终显示出叶片的衰老死亡[1]。叶片衰老由一个复杂的内源性分子网络控制,细胞内的细胞器和大分子主动失去平衡,并将营养物质重新分配到发育的组织或储存器官中。正常的叶片衰老在植物发育中需要在严格的分子调控下进行[2]。然而,叶片早衰将严重影响植物的光合作用,进而影响谷物灌浆,最终影响产量[3]。因此,克隆水稻叶片早衰相关基因,研究其作用的分子机制,对培育高产水稻品种具有重要的指导意义。
近年来,对水稻叶片衰老的研究已取得较大进展,虽然科研工作者在水稻中定位了大量与衰老相关的基因(senescence-associated gene,SAG),但是已克隆的能够直接调控衰老的基因却比较少。迄今,水稻中有14个叶片衰老相关基因被克隆,包括SGR[4-5]、SPL28 [6]、SPL29[7]、RLS1[8]、OsNAP[9]、OsSIK2[10]、OsWRKY42 [11]、OsABC1-2[12]、OsAkαGal[13]、OsLMS[14]、nol1[15]、nyc1[16]、NYC3[17]和NYC4[18],并初步阐明了其作用的分子机制。如sgr突变体衰老过程中,叶绿素降解缓慢、叶绿素-蛋白复合物和类囊体膜结构稳定,sgr的突变表型是由于其编码的叶绿体转运肽蛋白中缬氨酸替换成了甲硫氨酸导致的[4-5];spl28突变体苗期正常,分蘖期产生小的红褐色斑点,SPL28编码1个网络型衔接蛋白复合物1的中间亚基μ1(AP1M1)[6];spl29突变体从苗期到成熟期叶片均出现小的深褐色斑点,斑点周围出现枯萎,SPL29编码UAP1蛋白,它参与调控水稻早衰和防卫反应过程[7];RLS1编码1个C端含有ARM结构域、N端含核酸结合位点的蛋白,在暗诱导衰老中表达量上调[8];OsNAP是在ps1-D突变体中发现的,psl-D在4叶期前表型正常,进入分蘖期后叶片开始衰老,灌浆后加速黄化,比野生型早7~10 d衰老,ps1-D是由T-DNA插入引起的显性突变,编码1个植物特异的NAC转录激活因子OsNAP[9];OsSIK2是S结构域受体类激酶,过表达OsSIK2的转基因植株对盐和干旱耐受性增强[10];OsWRKY42为WRKY转录抑制因子,在衰老叶片中高度表达[11];osabc1-2是T-DNA插入突变体,表现出矮化和浅绿色表型,色素含量和光合效率降低,叶片中过氧化物歧化酶活性增强;过表达OsABC1-2增强了对黑暗胁迫的耐受性[12];OsAkαGal在水稻叶片衰老过程的类囊体膜降解中起重要作用,参与双半乳糖甘油二脂的降解[13];OsLMS编码1个含羧基末端域(CTD)磷酸酶结构域、2个双链RNA结合基序,lms在早期生长阶段对冷胁迫敏感,但对稻瘟病的抵抗能力增强[14]。nol是编码叶绿素b还原酶的基因[15],nyc1在叶片衰老过程中使叶片一直保持绿色,nyc1编码叶绿素b氧化还原酶[16],水稻nol突变体与nyc1突变体有相似的表型,即使在叶片衰老后期,叶片仍然保持绿色。NOL和NYC1都编码短链脱氢酶/还原酶,均定位在类囊体膜上。nyc3编码细胞质α/β水解折叠酶蛋白,在黑暗条件下,nyc3的叶绿素a和叶绿素b含量都比野生型更高[17]。NYC4与叶绿素-蛋白复合物的降解有关[18],与sgr、nyc3不同的是,nyc4中光系统Ⅱ的Fv/Fm值在衰老期间仍保持较高水平。这些研究表明叶片衰老的机制非常复杂,仍存在很多不清楚的过程,特别是关于叶片早衰的分子机制和调控网络研究较少,需挖掘控制叶片早衰的基因,为阐明叶片早衰的分子调控网络奠定基础。
在本研究中,我们利用甲基磺酸乙酯(EMS)化学诱变粳稻品种‘宁粳1号’,筛选获得1个叶片早衰突变体zs,对zs的生理特征、农艺性状以及基因定位进行研究,为该基因的克隆和功能分析奠定基础,为解释叶片早衰的分子机制提供理论依据,为水稻高产育种提供指导。
1 材料与方法 1.1 材料粳稻品种‘宁粳1号’经过EMS化学诱变,筛选得到1个遗传稳定的早衰突变体zs。将zs与籼稻品种‘N22’进行杂交、自交,获得F2分离群体。观察F2分离群体幼苗表型,选取具有突变体早衰表型的极端个体进行基因定位。
1.2 农艺性状调查2016年试验在南京农业大学土桥试验基地进行,将野生型和zs种子室温浸种48 h、催芽12 h,然后播种、移栽,株距与行距均为10 cm,正常田间管理。成熟期随机取20株野生型和zs,测定株高、穗长、有效穗数、结实率、粒长、粒宽等农艺性状,利用考种分析仪(万深SC-G)测定野生型与zs的粒长、粒宽以及千粒质量,重复3次。
1.3 光合色素含量的测定取约0.03 g叶片,剪成约0.01 cm的小块,黑暗条件下浸泡在5 mL 95%乙醇溶液中,重复3次,避光,室温放置48 h,期间多次颠倒混匀。避光吸取200 μL的上清液,利用多功能酶标仪M3(Sp470ectra MaxM3 microplate reader)测定上清液在665、649和470 nm波长下的吸光值,根据Lichtenthaler[19]提出的公式,计算叶片单位质量的叶绿素a(Chla)、叶绿素b(Chlb)和类胡萝卜素x(Chlx)的含量:Chla=12.21A665-2.81A649,Chlb=20.13A649-5.03A665,Chlx=(1 000A470-2.05Chla-114Chlb)/245,A代表吸光值。
1.4 叶片过氧化分析参照Wang等[20]方法,从生长14 d的野生型和zs的第2叶叶片上有斑点的部位,切下2 cm的组织,放入2.0 mL的离心管中,加入1 mg · mL-1DAB(3,3-diaminobenzidine)溶液和0.1%NBT(nitroblue tetrazolium)溶液;用锡箔纸包裹离心管,离心1 min,在室温下放置12 h。将染色溶液倒出,然后去除叶绿素以观察染色情况。DAB染料与细胞内所产生的H2O2结合,形成不可溶、肉眼可辨认的红棕色斑点,可以检测叶片中H2O2的积累情况。NBT染料与细胞内所产生的

参照Shan等[21]方法,利用透射电镜(transmission electron microcopy,TEM)进行叶绿体超微结构观察。分别取野生型和zs生长至14 d的第2叶片,沿叶脉切成1 mm的小块。在2.5%戊二醛固定液中,抽真空,室温固定12 h,用蒸馏水冲洗后,用1%锇酸溶液再固定4 h,经不同浓度梯度的乙醇脱水,用环氧树脂SPURR包埋、聚合、修块、切片、醋酸铀染色,经透射电镜(JEM-1230)观察、拍照。
1.6 基因定位根据Kessler等[22]方法加以改进,即将加入SDS的样品继续水浴30 min。加入醋酸钾溶液后,放入-20 ℃冰箱中冷冻30 min,其他步骤不变。提取F2群体极端个体的叶片DNA,利用笔者所在实验室已有的均匀分布在水稻12条染色体上的SSR和InDel标记,筛选在zs和‘N22’两亲本间具有多态性的引物。从zs/N22的F2分离群体中挑选10个早衰极端个体。用有多态的引物扩增两亲本和极端个体的DNA,用聚丙烯酰胺凝胶电泳,染色检测,具有zs的带型记为1,具有‘N22’的带型记为2,两亲本带型都有的记为3。利用Primer Premier 5.0软件设计引物,扩大早衰极端个体群体,进行精细定位。
1.7 叶绿素合成相关基因与质体发育相关基因的表达分析使用RNA PrepPure Plant Kit(Tiangen),对野生型和zs生长14 d的第2叶叶片提取RNA,提取步骤见说明书。Real-time PCR的反应体系20 μL:反转录产物2 μL,10 μmol · L-1的正、反向引物各0.4 μL,SYBR Green混合物(TaKaRa)10 μL,补水至20 μL。反应程序为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环。以水稻的ACTIN基因作为内参基因。每个样品设置3个重复。利用2-ΔΔCT法,计算目的基因的相对表达量。叶绿素合成相关基因和质体发育相关基因的定量引物参照文献[23]。
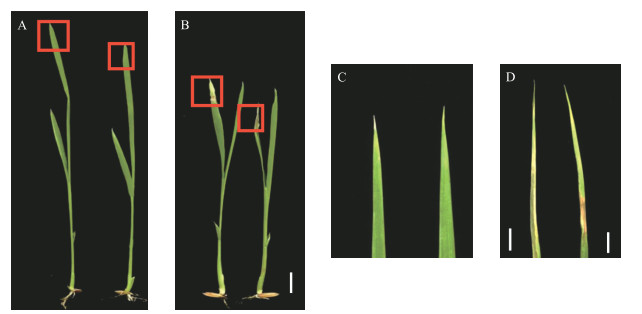
2 结果与分析 2.1 突变体zs的早衰表型及农艺性状表现与野生型相比,zs在28 ℃光照培养箱中生长14 d时,第2叶叶尖开始出现黄褐色斑点,然后发黄加重、出现枯萎、皱缩,并逐渐从叶尖扩张到整张叶片(图 1)。随着植株的生长,早衰的叶片逐渐增多,植株生长迟缓,症状加重。在成熟期,zs的株高、穗长、分蘖数、千粒质量均极显著降低(表 1)。
|
图 1 野生型和zs的苗期表型 Figure 1 Performance of wild type and zs at the seedling stages 在光照培养箱播种14 d后野生型(A)和zs(B)苗期表型;野生型(C)和zs(D)在光照培养箱播种14 d后的叶片局部表型。比例尺均为2 cm。 Seedling performance of the wild type(A)and zs(B)after 14 days sowing in light incubator; The leaf local phenotype of wild type(C)and zs(D)after 14 days sowing in light incubator. Bar=2 cm. |
| 材料 Material |
株高/cm Plant height |
穗长/cm Panicle length |
有效分蘖数 Productive tiller |
结实率/% Seed-setting rate |
千粒质量/g 1 000-grain weight |
粒长/mm Grain length |
粒宽/mm Grain width |
| WT | 92.1±2.9 | 16.0±0.9 | 10±2 | 81.6±6.1 | 29.01±0.42 | 6.00±0.03 | 2.79±0.03 |
| zs | 60.0±2.7** | 12.4±0.8** | 6±2** | 81.0±4.1* | 24.90±0.35** | 5.88±0.01 | 2.76±0.01 |
| Note:* P < 0.05,* *P < 0.01. The same as follows. | |||||||
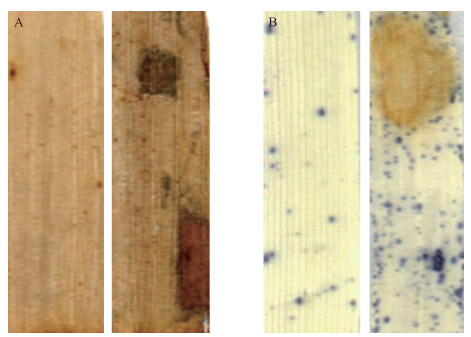
叶片的衰老常伴随着ROS和丙二醛含量的增加,使叶绿体降解,膜结构被破坏[24]。突变体zs的叶片在生长14 d时出现黄褐色的斑点,这是衰老的典型症状。利用DAB和NBT对生长14 d的zs叶片衰老部位及对应的野生型叶片的染色结果表明,突变体zs衰老部位的叶片能被DAB染成棕褐色斑点,野生型的叶片不能被DAB染色;用NBT染色,突变体zs出现了很多深蓝色斑点,而野生型仅出现少量深蓝色斑点(图 2)。这些结果表明zs的叶片细胞有大量的ROS积累,且伴随有H2O2的产生。
|
图 2 野生型和zs的DAB和NBT染色 Figure 2 DAB and NBT staining of wild type and zs A. DAB染色(左:野生型;右:zs);B. NBT染色(左:野生型;右:zs)。 A. DAB staining(left:wild type; right:zs); B. NBT staining(left:wild type; right:zs). |
从图 3可见:生长14 d时,突变体zs的第2叶叶片的叶绿素a、叶绿素b含量均显著低于野生型,突变体zs的光合色素含量显著低于野生型,类胡萝卜素含量仅略有下降。

|
图 3 野生型与zs光合色素含量比较 Figure 3 Comparison of photosynthetic pigment contents between wild type and zs |
利用透射电子显微镜,我们观察了野生型与zs生长至14 d的绿色相同部位叶绿体超微结构。野生型的叶绿体呈纺锤形,类囊体排列整齐,而zs的叶绿体结构较蓬松,呈现不规则椭圆形,类囊体排列也不规则(图 4)。TEM结果提示zs叶片早衰,使叶绿体结构逐渐裂解、丧失,最终导致叶片枯死。

|
图 4 野生型和zs的叶绿体超微结构比较 Figure 4 Comparison of chloroplasts ultrastructures between the wild type and zs A~C.野生型叶绿体超微结构;D~F:.zs叶绿体超微结构。 A-C. TEM images of chloroplast structure in the wild type; D-F. TEM images of chloroplast structure in zs. |
利用正反交试验,将野生型与zs互为父母本,进行杂交,后代F1在自然条件下生长,叶片颜色均表现为正常绿色,其自交后代F2群体中个体的表型出现明显分离,即正常绿色和早衰表型,且正常绿色与早衰表型的植株数经卡方测验符合3 : 1的分离比,说明该性状是由1对隐性核基因控制的(表 2)。
| 组合 Combination |
F2正常株数 Number of green plants |
F2早衰株数 Number of early ageing leaf plants |
总株数 Total number of plants |
χ2(χ0.052=3.84) |
| WT×zs | 274 | 93 | 367 | 2.95:1 |
| zs×WT | 280 | 91 | 371 | 3.08:1 |
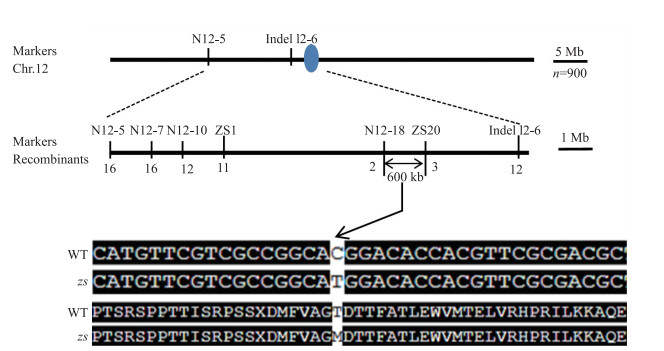
将zs与籼稻品种‘N22’进行杂交,选取其后代F2群体中具有突变体表型的单株共900株。利用InDel和SSR标记,筛选出与zs表型一致的极端个体10个,将基因初定位在第12染色体短臂标记N12-5与Indel12-6之间,遗传距离为10.8 cM(图 5)。为进一步精细定位,我们开发出两亲本之间有多态的引物30对,最终将基因精细定位在标记N12-18与ZS20之间,两者相距600 kb(图 5)。在该区间内共有24个基因,其中有2个为已报道的与叶片衰老相关的基因异黄酮还原酶基因LOC_Os12g1641和细胞色素P450单加氧酶基因LOC_Os12g16720 [25]。测序发现突变体zs与野生型在异黄酮还原酶基因序列上没有差异,但在LOC_Os12g16720基因的第941位碱基C突变成了T,导致氨基酸序列由苏氨酸变成了甲硫氨酸,因此我们将其作为候选基因。
|
图 5 基因定位 Figure 5 Fine mapping of gene |
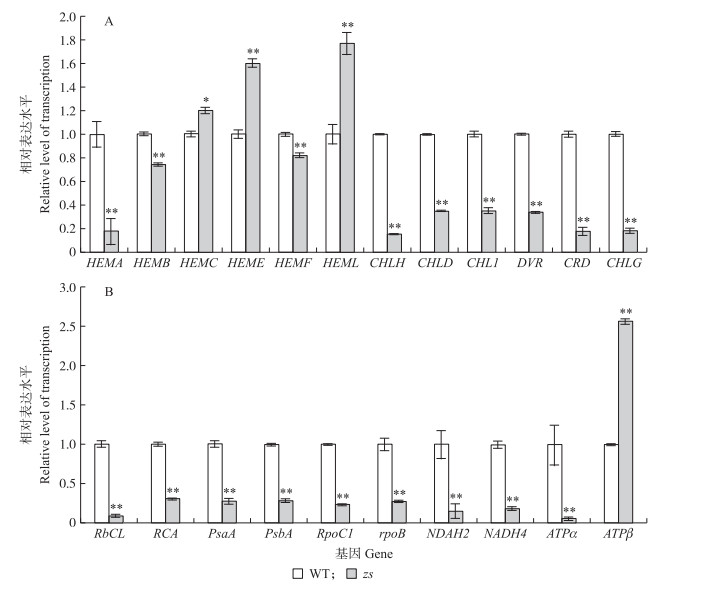
从图 6可见:与野生型相比,zs中羟甲基后胆色素原合酶基因(HEMC)、尿卟啉原脱羧酶基因(HEME)、谷氨酰-1-半醛转氨酶基因(HEML)的表达均上调,谷氨酰-tRNA还原酶基因(HEMA)、胆色素原合酶基因(HEMB)、粪卟啉原氧化脱羧酶基因(HEMF)、镁獒合酶H亚基基因(CHLH)、镁獒合酶D亚基基因(CHLD)、二乙烯还原酶基因(DVR)、镁原卟啉Ⅸ单甲酯环化酶基因(CRD)、叶绿素合成酶基因(CHLG)等与叶绿素合成相关基因的表达都下调。质体发育相关基因只有ATP合酶β亚基基因(ATPβ)表达显著上调,其他基因的表达均下调。表明zs中叶绿素合成和质体发育受到了显著影响。
|
图 6 叶绿素合成(A)和质体发育(B)相关基因的表达分析 Figure 6 Expression of genes associated with chlorophyll biosynthesis(A)and plastid development(B) 野生型基因转录水平设为1。 The gene transcript level of the wild type was set to 1. |
叶片是植物进行光合作用的重要器官,叶片的光合作用能力是调控产量和生物量的主要因子之一[26]。叶片早衰导致光合能力下降,甚至丧失,极大地影响作物的产量。本研究中早衰突变体zs的光合色素含量降低,叶片的光合作用减弱,zs的株高和千粒质量与野生型相比明显降低。因此,若能通过遗传策略控制叶片早衰,则有利于提高和稳定作物的产量。
本研究通过EMS诱变得到的突变体zs,在苗期时与野生型相比就出现明显差异,生长至14 d时叶片开始出现黄褐色斑点,在整个生育期,zs的叶片颜色都始终保持带有黄褐色斑点的绿色,且随着发育进程,黄褐色斑点持续增多,但zs植株直至完全成熟也没有死亡。zs与水稻衰老突变体nyc1[16]、nyc3[17]、nyc4[18]、sgr[4-5]类似,在植株的衰老过程中植株叶片的总体颜色始终是绿色的,叶片并不会表现出衰老枯萎死亡的表型;但zs与水稻早衰突变体sms1[27]、es-t[28]、pgl2[29]、pse(t)[30]有很多不同点。如sms1除了叶片有早衰症状外,稻穗还会出现严重的雄性不育[27]。es-t在幼苗时叶片首先变黄,随后叶片开始衰老,在叶片上还有铁锈色的斑点[28]。pgl2在苗期叶色正常,没有出现衰老表型,抽穗前叶片开始出现衰老,从倒4叶开始逐渐衰老,当植株灌浆结束时剑叶完全衰老死亡[29]。pse(t)在抽穗期时最先在老的叶片上出现黄褐色斑点,之后整个植株叶片出现黄化衰老表型[30]。表明本研究的早衰突变体zs与以往发现的早衰突变体并不相同,提示其可能受其他分子机制调控。
ZS被定位在第12染色体的600 kb的区间内,根据水稻基因表达数据库(http://ricexpro:dna.affr.go.jp/)对600 kb区间内的基因进行预测,在该区间内共有24个基因,没有与衰老相关的基因被报道。结合测序分析,发现突变体zs中仅在基因LOC_Os 12g16720的第941位碱基C突变成了T,导致氨基酸序列由苏氨酸变成了甲硫氨酸,该基因编码细胞色素P450蛋白,因而将其作为候选基因。P450单加氧酶家族是植物系统中最大的酶蛋白家族,其不仅通过多种生物合成途径和解毒途径促进植物生长发育,还保护植物免受胁迫反应。Fujiwara等[25]报道了一个与抗病相关的基因SL,它编码一个细胞色素P450单加氧酶家族的CYP71P1蛋白,该蛋白参与血清素的生物合成,病原菌的激发子几丁质和稻瘟病菌侵染均诱导水稻SL基因的表达,促进植株中血清素水平的提高,而血清素诱导包括防御基因在内的抗病基因的表达和细胞死亡途径,参与植株的抗病反应。SL基因的突变引起的病斑表型与zs的植株表型非常相似。本研究中突变体zs与水稻中的抗病基因SL是否等位,对病原菌是否有抵抗能力仍需要进一步探索。
致谢:
农业农村部长江中下游粳稻生物学与遗传育种重点实验室、江苏省现代作物生产中心、长江流域杂交水稻协同创新中心给予支持,谨至谢意。
| [1] | Gan S S, Amasino R M. Making sense of senescence:molecular genetic regulation and manipulation of leaf senescence[J]. Plant Physiology, 1997, 113(2): 313-319. DOI: 10.1104/pp.113.2.313 |
| [2] | Schippers J H. Transcriptional networks in leaf senescence[J]. Current Opinion in Plant Biology, 2015, 27: 77-83. DOI: 10.1016/j.pbi.2015.06.018 |
| [3] | Yoo S C, Cho S H, Zhang H T, et al. Quantitative trait loci associated with functional stay-green SNU-SG1 in rice[J]. Molecules & Cells, 2007, 24(1): 83-94. |
| [4] | Park S Y, Yu J W, Park J S, et al. The senescence-induced staygreen protein regulates chlorophyll degradation[J]. Plant Cell, 2007, 19(5): 1649-1664. DOI: 10.1105/tpc.106.044891 |
| [5] | Jiang H W, Li M R, Liang N T, et al. Molecular cloning and function analysis of the stay green gene in rice[J]. Plant Journal, 2007, 52(2): 197-209. DOI: 10.1111/tpj.2007.52.issue-2 |
| [6] | Qiao Y L, Jiang W Z, Lee J H, et al. SPL28 encodes a clathrin-associated adaptor protein complex 1, medium subunit μ1(AP1M1) and is responsible for spotted leaf and early senescence in rice(Oryza sativa)[J]. New Phytologist, 2010, 185(1): 258-274. DOI: 10.1111/j.1469-8137.2009.03047.x |
| [7] | Wang Z, Wang Y, Hong X, et al. Functional inactivation of UDP-N-acetylglucosamine pyrophosphorylase 1(UAP1) induces early leaf senescence and defence responses in rice[J]. Journal of Experimental Botany, 2015, 66(3): 973-987. DOI: 10.1093/jxb/eru456 |
| [8] | Jiao B B, Wang J J, Zhu X D, et al. A novel protein RLS1 with NB-ARM domains is involved in chloroplast degradation during leaf senescence in rice[J]. Molecular Plant, 2012, 5(1): 205-217. DOI: 10.1093/mp/ssr081 |
| [9] | Liang C Z, Wang Y Q, Zhu Y N, et al. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice[J]. Proc Natl Acad Sci USA, 2014, 111(27): 10013-10018. DOI: 10.1073/pnas.1321568111 |
| [10] | Chen L J, Wuriyanghan H, Zhang Y Q, et al. An S-domain receptor-like kinase, OsSIK2, confers abiotic stress tolerance and delays dark-induced leaf senescence in rice[J]. Plant Physiology, 2013, 163(4): 1752-1765. DOI: 10.1104/pp.113.224881 |
| [11] | Han M, Kim C Y, Lee J, et al. OsWRKY42 represses OsMT1d and induces reactive oxygen species and leaf senescence in rice[J]. Molecules & Cells, 2014, 37(7): 532-539. |
| [12] | Gao Q S, Yang Z F, Zhou Y, et al. Characterization of an Abc1 kinase family gene OsABC1-2 conferring enhanced tolerance to dark-induced stress in rice[J]. Gene, 2012, 498(2): 155-163. DOI: 10.1016/j.gene.2012.02.017 |
| [13] | Lee R H, Hsu J H, Huang H J, et al. Alkaline α-galactosidase degrades thylakoid membranes in the chloroplast during leaf senescence in rice[J]. New Phytologist, 2009, 184(3): 596-606. DOI: 10.1111/nph.2009.184.issue-3 |
| [14] | Undan J R, Tamiru M, Abe A, et al. Mutation in OsLMS, a gene encoding a protein with two double-stranded RNA binding motifs, causes lesion mimic phenotype and early senescence in rice(Oryza sativa L.)[J]. Genes and Genetic Systems, 2012, 87(3): 169-179. DOI: 10.1266/ggs.87.169 |
| [15] | Sato Y, Morita R, Katsuma S, et al. Two short-chain dehydrogenase/reductases, NON-YELLOW COLORING 1 and NYC1-LIKE, are required for chlorophyll b and light-harvesting complex Ⅱ degradation during senescence in rice[J]. Plant Journal, 2009, 57(1): 120-131. DOI: 10.1111/tpj.2008.57.issue-1 |
| [16] | Kusaba M, Ito H, Morita R, et al. Rice NON-YELLOW COLORING1 is involved in light-harvesting complex Ⅱ and grana degradation during leaf senescence[J]. Plant Cell, 2007, 19(4): 1362-1375. DOI: 10.1105/tpc.106.042911 |
| [17] | Morita R, Sato Y, Masuda Y, et al. Defect in non-yellow coloring 3, an α/β hydrolase-fold family protein, causes a stay-green phenotype during leaf senescence in rice[J]. Plant Journal, 2009, 59(6): 940-952. DOI: 10.1111/tpj.2009.59.issue-6 |
| [18] | Yamatani H, Sato Y, Masuda Y, et al. NYC4, the rice ortholog of Arabidopsis THF1, is involved in the degradation of chlorophyll protein complexes during leaf senescence[J]. Plant Journal, 2013, 74(4): 652-662. DOI: 10.1111/tpj.2013.74.issue-4 |
| [19] | Lichtenthaler H K. Chlorophylls and carotenoids:pigments of photosynthetic biomembranes[J]. Methods in Enzymology, 1987, 148: 350-382. DOI: 10.1016/0076-6879(87)48036-1 |
| [20] | Wang Y C, Zhang Y, Wang Z, et al. A missense mutation in CHS1, a TIR-NB protein, induces chilling sensitivity in Arabidopsis[J]. Plant Journal, 2013, 75(4): 553-565. DOI: 10.1111/tpj.2013.75.issue-4 |
| [21] | Shan X Q, Wang H O, Zhang S Z, et al. Accumulation and uptake of light rare earth elements in a hyperaccumulator Dicropteris dichotoma[J]. Plant Science, 2003, 165(6): 1343-1353. DOI: 10.1016/S0168-9452(03)00361-3 |
| [22] | Kessler B. Isolation, characterization and distribution of a DNA ligase from higher plants[J]. Biochimica et Biophysica Acta, 1971, 240(4): 496-505. DOI: 10.1016/0005-2787(71)90706-4 |
| [23] |
刘喜, 周春雷, 任雅琨, 等. 水稻叶色白化转绿突变体WGL的遗传分析与基因定位[J].
南京农业大学学报, 2015, 38(5): 712-719.
Liu X, Zhou C L, Ren Y K, et al. Genetic analysis and gene mapping of virescent albino leaf mutant WGL in rice[J]. Journal of Nanjing Agricultural University, 2015, 38(5): 712-719. (in Chinese with English abstract) |
| [24] | Tewari R K, Singh P K, Watanabe M. The spatial patterns of oxidative stress indicators co-locate with early signs of natural senescence in maize leaves[J]. Acta Physiologiae Plantarum, 2013, 35(3): 949-957. DOI: 10.1007/s11738-012-1138-0 |
| [25] | Fujiwara T, Maisonneuve S, Isshiki M, et al. Sekiguchi lesion gene encodes a cytochrome P450 monooxygenase that catalyzes conversion of tryptamine to serotonin in rice[J]. The Journal of Biological Chemistry, 2010, 285(15): 11308-11313. DOI: 10.1074/jbc.M109.091371 |
| [26] | Wu X Y, Kuai B K, Jia J Z, et al. Regulation of leaf senescence and crop genetic improvement[J]. Journal of Integrative Plant Biology, 2012, 54(12): 936-952. DOI: 10.1111/jipb.2012.54.issue-12 |
| [27] | Yan W Y, Ye S H, Jin Q S, et al. Characterization and mapping of a novel mutant sms1(senescence and male sterility 1)in rice[J]. Journal of Genetics and Genomics, 2010, 37(1): 47-55. DOI: 10.1016/S1673-8527(09)60024-2 |
| [28] | Yang Y L, Rao Y C, Liu H J, et al. Characterization and fine mapping of an early senescence mutant(es-t)in Oryza sativa L[J]. Chinese Science Bulletin, 2011, 56(23): 2437-2443. DOI: 10.1007/s11434-011-4587-8 |
| [29] |
朱丽, 刘文真, 吴超, 等. 水稻着丝粒附近一个淡绿叶突变相关基因的定位分析[J].
中国水稻科学, 2007, 21(3): 228-234.
Zhu L, Liu W Z, Wu C, et al. Identification and fine mapping of a gene related to pale green leaf phenotype near centromere region in rice(Oryza sativa L.)[J]. Chinese Journal of Rice Science, 2007, 21(3): 228-234. DOI: 10.3321/j.issn:1001-7216.2007.03.002 (in Chinese with English abstract) |
| [30] | Li F Z, Hu G C, Fu Y P, et al. Genetic analysis and high-resolution mapping of a premature senescence gene Pse(t)in rice(Oryza sativa L.)[J]. Genome, 2005, 48(4): 738-746. DOI: 10.1139/g05-030 |




