文章信息
- 吴黎明, 李曦, 伍辉军, 高学文
- WU Liming, LI Xi, WU Huijun, GAO Xuewen
- 芽胞杆菌抗菌二肽溶杆菌素的研究进展
- Research advances on Bacilysin from Bacillus
- 南京农业大学学报, 2018, 41(5): 778-783
- Journal of Nanjing Agricultural University, 2018, 41(5): 778-783.
- http://dx.doi.org/10.7685/jnau.201803047
-
文章历史
- 收稿日期: 2018-03-23
芽胞杆菌(Bacillus)作为典型的根围促生菌,具有良好的防治植物病害和促进植物生长的效果[1]。芽胞杆菌生防机制普遍被认可的主要有3类:营养和空间位点的竞争、诱导抗性和抗菌物质的产生[2]。其中抗菌物质目前研究较为深入并被作为生防芽胞杆菌筛选的重要指标之一。芽胞杆菌抗菌物质种类繁多,按生物合成途径可分为由核糖体途径合成的小分子物质,如细菌素;由非核糖体途径依赖sfp基因合成的多肽类物质,如脂肽类化合物、聚酮类化合物和由非核糖体途径不依赖sfp基因合成的溶杆菌素(Bacilysin)等[3-4]。与其他抗菌物质相比,Bacilysin的独特之处在于其结构简单,仅由2个氨基酸组成;作用靶标6-磷酸葡萄糖胺合成酶分别参与真菌和细菌细胞壁组成成分几丁质和肽聚糖的合成,可导致微生物细胞壁的结构受损,细胞溶解死亡,且对真菌和细菌都具有很强的杀伤活性和抑制作用[5-7]。这些特点使Bacilysin可为农药和医药的开发提供一个良好的资源。为了充分了解Bacilysin以便更系统地研究其功能,本文对Bacilysin的研究进展进行了较全面的概括和介绍。
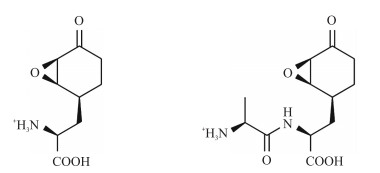
1 Bacilysin的发现及化学结构式的鉴定1946年,Abraham等[8]从英国牛津地区土壤中分离的枯草芽胞杆菌菌株中发现一种抗菌物质,它能够引起液体培养基中金黄色葡萄球菌细胞的快速溶解,并能够在固体琼脂平板上产生明显的抑菌圈,他们把该抗菌物质命名为溶杆菌素(Bacilysin)。之后,Foster等[9]和Newton[10]分离获得Bacilysin的纯品,通过元素分析和理化性质推测其分子式为C12H18N2O5,相对分子质量为271;电化学滴定试验表明,Bacilysin为一种包含1个自由的α-氨基基团和1个自由的羧基基团的短肽。然而,Bacilysin的具体结构式未被确定,关于羰基基团的信息也很少。直到1970年,Walker等[11]通过Bacilysin的三甲基硅烷基衍生物和三氯乙酰甲酯衍生物的质谱分析证实Bacilysin相对分子质量为271,核磁共振表明其含有1个与C4相连的环氧环己酮,从而确定了其具体结构式(图 1)。溶杆菌素是由N末端L-丙氨酸残基和C末端L-不典型的氨基酸(后被命名为抗荚膜菌素,相对分子质量为200)残基组成的二肽。
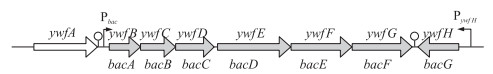
2 Bacilysin的生物合成 2.1 Bacilysin基因簇的鉴定二肽类小分子化合物作为芽胞杆菌属的一种重要次级代谢产物,其生物合成途径具有一定的代表性。Hilton等[12]利用转座子Tn917构建了模式菌株枯草芽胞杆菌(B.subtilis 168)的随机突变体文库,筛选获得了不产生Bacilysin的突变体并鉴定出转座子插入在基因ctrA和sacA区域之间,但具体插入基因未知。Steinborn等[13]发现在基因ywfA(位于基因ctrA和sacA区域之间)中插入1个新霉素抗性片段不影响Bacilysin的合成,推测ywfA不负责Bacilysin的合成;同时利用定点突变技术证明了ywfB—ywfH,并重命名为bacA—bacG(图 2),是芽胞杆菌中Bacilysin合成所必要的,但没有对各基因的具体功能进行鉴定。
关于Bacilysin的合成原料和前体,Roscoe等[14]报道添加14C标记的莽草酸后,在Bacilysin中可以检测到14C,根据它的结构推测莽草酸途径的中间产物预苯酸可能是其合成前体。Hilton等[12]发现莽草酸合成途径中aro基因被突变后,均不能产生Bacilysin;而当预苯酸脱氢酶基因tyrA和脱水酶基因pheA被破坏后仍能产生正常水平的Bacilysin,进一步证明预苯酸是Bacilysin的合成前体。
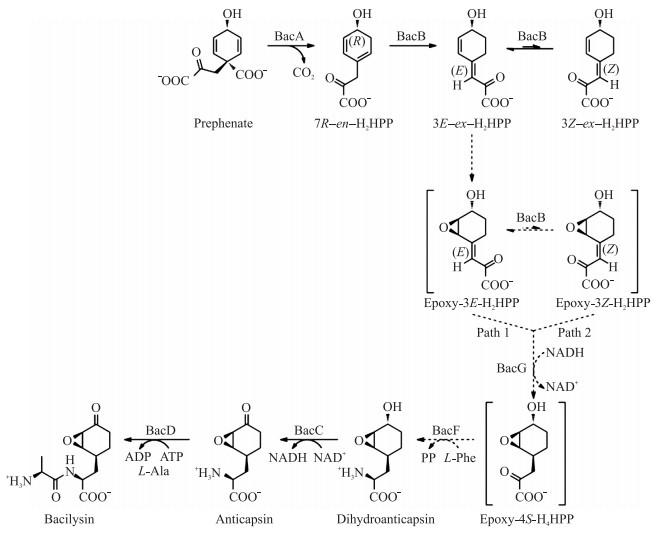
2.2 Bacilysin基因簇的各基因功能分析bac基因簇(bacA—bacG)是1个大小为6 000 bp左右的基因簇。在鸟苷五磷酸pppGpp的诱导下,bac基因簇便会启动转录表达的过程,通过连续的bacA—bacG共7个基因的分工合作合成Bacilysin。Steinborn等[13]将假定蛋白BacA—BacE进行同源性比对分析,发现BacA属于预苯酸脱羧酶,BacB属于异构酶/鸟苷酰基转移酶,BacC属于氧化还原酶/还原酶家族,BacD属于氨基甲酰-磷酸合酶L链和ATP结合域相连的D-丙氨酸-D-丙氨酸连接酶,BacE属于大环内脂输出蛋白/透性酶。2012和2013年,Parker等[15-16]对BacA—BacG蛋白分别进行了表达和纯化,利用HPLC、质谱、核磁共振等技术手段,结合生物信息学分析,明确了从预苯酸到Bacilysin的合成途径和bacA—bacG各基因在途径中的具体功能。
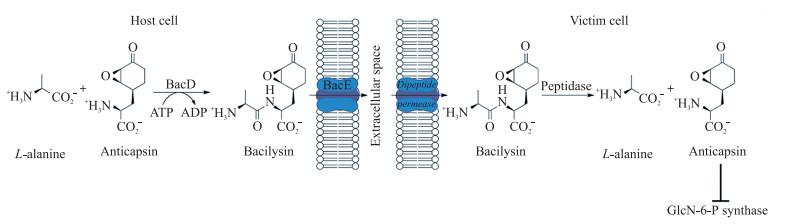
首先,在BacA烯烃异构酶作用下将合成原料预苯酸脱羧,但不发生芳香化,同时将1,4-双键转化成7R-en-H2HPP中内环的1,3-双键。其次,在BacB的催化下进行烯丙基的异构,将双键连接到产物3E-ex-H2HPP的2位酮基上。在这一过程中,包括2个反应:1)将7R-en-H2HPP催化产生3E几何异构体,然后将一部分3E-烯烃构型转化成3Z-构型并在这2种构型中达到平衡;2)在BacG催化下给环外双键加氢,形成Epoxy-4S-H4HPP,在BacF(转氨酶)的催化下,将L-苯丙氨酸作为氨基供体氨化2-酮基,形成二氢抗荚膜菌素(Dihydroanticapsin)。BacC作为1个NAD+依赖型的氧化酶,由于其具有S型选择性,因此能将Dihydroanticapsin中的羟基氧化成酮基,形成抗荚膜菌素(Anticapsin)。BacD是合成途径中最后发挥作用的酶,本质上它是1个羧肽酶,消耗ATP将L-丙氨酸通过羧化作用形成肽键而生成Bacilysin(图 3)。当Bacilysin合成以后紧接着在BacE二肽透性酶的作用下从胞内转运到胞外(图 4)。
Bacilysin结构虽然简单,但是对真菌和细菌都具有强烈的抑菌活性。Bacilysin的最初发现就是由于其能够引起金黄色葡萄球菌的溶解。Kenig等[17]发现Bacilysin还能够抑制表皮葡萄球菌、四联球菌、棒状杆菌、巨大芽胞杆菌、八联球菌、伤寒沙门氏菌、变形杆菌等病原细菌和白色假丝酵母等真菌的生长和繁殖。Chen等[18]利用薄层层析和生物自显影技术证明Bacilysin能抑制欧文氏菌的生长,对梨火疫病有很好的防治效果。Wu等[19-20]研究表明Bacilysin不仅能够有效防治铜绿微囊藻、水华束丝藻、念珠藻和鱼腥藻等有害蓝藻类,还能够显著减弱水稻黄单胞菌的毒力,降低水稻白叶枯病和细菌性条斑病的发病程度。
对于Bacilysin抗菌作用机制的研究,Kenig等[17, 21]在1976年就指出,当Bacilysin和葡萄糖胺共同添加到金黄色葡萄球菌培养基中,细胞没有被破坏溶解;葡萄糖胺能够使Bacilysin和Anticapsin的抗菌活性减弱或丧失[17];6-磷酸葡萄糖胺的产量随Bacilysin和Anticapsin浓度的增加逐渐减少[21]。因此提出假设:Bacilysin在芽胞杆菌体内合成后通过特定的转运蛋白将其运输至胞外;之后被邻近的真菌或细菌受体细胞通过二肽透性酶转入到自身细胞内部;胞内肽酶将Bacilysin水解成L-丙氨酸和Anticapsin,Anticapsin作为谷氨酰胺的结构类似物,会竞争性抑制以谷氨酰胺为唯一底物的6-磷酸葡萄糖胺合成酶的氨基转移酶活性,从而直接导致6-磷酸葡萄糖胺的合成受阻。在真菌和细菌中,6-磷酸葡萄糖胺主要被用于一些大分子的构建,如真菌几丁质、甘露糖蛋白和细菌肽聚糖,它对细胞壁的形成是必需的。因此,Bacilysin通过Anticapsin使微生物细胞壁的结构受损,细胞内含物外流,细胞溶解死亡(图 4)[22]。然而,到目前为止没有直接的证据证明Bacilysin被水解生成Anticapsin,同时也未得到Antacapsin与6-磷酸葡萄糖胺合成酶之间复合物的晶体结构。因此该假设仍需后续更为详细的试验数据加以验证。
3.2 Bacilysin调控产胞功能研究Özcengiz等[23]报道脂肽化合物类抗菌物质如表面活性素(surfactin)的突变体通常在芽胞形成或萌发过程中有缺陷。因此推测抗菌物质Bacilysin可能在芽胞形成、萌发和生长过程中也发挥着重要的作用。Hilton等[24]发现枯草芽胞杆菌Bacilysin突变体与野生型相比,对热、化学物质和溶菌酶更敏感且芽胞数量下降,而当外源添加Bacilysin后产胞效率恢复,证实了上述的推测。但目前对于Bacilysin调控产胞功能的具体机制还有待进一步研究。
4 Bacilysin生物合成的调控 4.1 培养基成分的改变对Bacilysin产量的影响通过改善芽胞杆菌培养基成分可以提高Bacilysin的合成产量。Özcengiz等[25]证实氮源是Bacilysin合成过程中最主要的调节因子,天冬氨酸比谷氨酸更好,而无机铵盐效果最差。Inaoka等[26]研究表明在培养基中添加钪可以在转录水平上提高Bacilysin基因簇的表达量,并且也使抑菌活性得到增强。
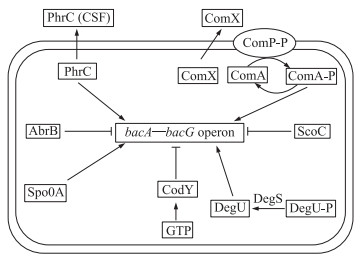
4.2 Bacilysin生物合成的基因调控Yazgan等[27]报道群集感应(Quorum sensing)系统可通过PhrC(CSF)和ComA的共同作用影响Bacilysin的生物合成;敲除phrC和comA基因后,芽胞杆菌不能合成Bacilysin。Belitsky等[28]和Karatas等[29]进一步证实bacA—bacF基因簇的激活和表达由ComA、Spo0A、AbrB和CodY这4种蛋白共同调控,它们可以同时绑定到bac基因簇的启动子上。ComA和Spo0A为正向调控子,AbrB和CodY为负向调控子。另外,Inaoka等[26]报道过渡状态调控子ScoC也能够与bacA—bacF启动子结合从而负向调控Bacilysin合成。
反应调节子DegU和其同源激酶DegS为芽胞杆菌中重要的双组分调控系统,能够正向调控芽胞杆菌bac基因簇的转录水平[30]。
鸟苷四磷酸(ppGpp)在bac基因簇转录过程中发挥关键性的作用,其含量依赖于细胞内GTP水平,而GTP水平却受到CodY蛋白介导的抑制系统调控。在野生型中,降低GTP含量或敲除codY基因,都能够提高bac基因簇的表达水平[31](图 5)。
5 问题与展望Bacilysin是一种高效的抗菌活性物质,但是到目前为止还没有大规模应用的报道。限制其应用的因素主要有以下几个方面:1)天然菌株的产量较低,不能满足产业化的需要。芽胞杆菌的Bacilysin产量0.448 g · L-1左右[32]。2)目前的研究主要集中在单纯利用发酵优化手段来提高Bacilysin的产量,提高幅度仍然有限。3)缺乏高效的分离提取工艺。目前Bacilysin的提取方法是利用凝胶过滤层析或者离子交换技术,操作繁琐且回收率低。
传统的多肽类抗菌物质大多含有线状、环状或分枝状等复杂结构,它们可以靶向细胞内或细胞膜上的位点,干扰生理代谢途径[33]。而Bacilysin与传统的抗菌药物相比,结构简单,仅含有2个氨基酸且靶标位点为细胞壁合成关键酶,具有选择性和广谱性的生物活性,也不会产生抗药性菌株。因此,Bacilysin在农业和医药领域具有广阔的应用前景。为实现Bacilysin的广泛应用,研究重点将围绕利用基因工程技术对芽胞杆菌菌株进行修饰,如bac基因簇的原始启动子替换成强启动子,负向调控基因的缺失等来提高Bacilysin的生产效率;通过药物设计手段对Bacilysin的N端或C端进行改造,提高其抗菌活性和稳定性;改进Bacilysin的分离提取工艺,降低成本及有机溶剂的污染,满足产业化生产需求;探究Bacilysin对其他有益微生物的毒害作用和在土壤、环境中的归趋问题。这些方面的深入研究可以为其开发成新型生物农药提供科学依据,为植物病害的生物防治提供新思路和新手段。
| [1] | Sonenshein A L. Control of sporulation initiation in Bacillus subtilis[J]. Current Opinion in Microbiology, 2000, 3(6): 561-566. DOI: 10.1016/S1369-5274(00)00141-7 |
| [2] | Moszer I, Jones L M, Moreira S, et al. SubtiList:the reference database for the Bacillus subtilis genome[J]. Nucleic Acids Research, 2002, 30(1): 62-65. DOI: 10.1093/nar/30.1.62 |
| [3] | Finking R, Marahiel M A. Biosynthesis of nonribosomal peptides[J]. Annual Review of Microbiology, 2004, 58: 453-488. DOI: 10.1146/annurev.micro.58.030603.123615 |
| [4] | Özcengiz G, Ö Aǧ ülür İ. Biochemistry, genetics and regulation of bacilysin biosynthesis and its significance more than an antibiotic[J]. New Biotechnology, 2015, 32(6): 612-619. DOI: 10.1016/j.nbt.2015.01.006 |
| [5] | Rogers H J, Lomakina N, Abraham E P. Observations on the structure of bacilysin[J]. Biochemical Journal, 1965, 97(2): 579-586. DOI: 10.1042/bj0970579 |
| [6] | Loeffler W, Tschen J S M, Vanittanakom N, et al. Antifungal effects of bacilysin and fengymycin from Bacillus subtilis F-29-3 A comparison with activities of other Bacillus antibiotics[J]. Journal of Phytopathology, 1986, 115(3): 204-213. DOI: 10.1111/j.1439-0434.1986.tb00878.x |
| [7] | Perry D, Abraham E P. Transport and metabolism of bacilysin and other peptides by suspensions of Staphylococcus aureus[J]. Journal of General Microbiology, 1979, 115(1): 213-221. DOI: 10.1099/00221287-115-1-213 |
| [8] | Abraham E P, Callow D, Gilliver K. Adaptation of Staphylococcus aureus to growth in the presence of certain antibiotics[J]. Nature, 1946, 158(4023): 818-821. DOI: 10.1038/158818a0 |
| [9] | Foster J W, Woodruff H B. Bacillin, a new antibiotic substance from a soil isolate of Bacillus subtilis[J]. Journal of Bacteriology, 1946, 51(3): 363-369. |
| [10] | Newton G G F. Antibiotics from a strain of Bacillus subitilis, bacilipin A and B and bacilysin[J]. British Journal of Experimental Pathology, 1949, 304(4): 306-319. |
| [11] | Walker J E, Abraham E P. The structure of bacilysin and other products of Bacillus subtilis[J]. Biochemical Journal, 1970, 118(4): 563-570. DOI: 10.1042/bj1180563 |
| [12] | Hilton M D, Alaeddinoglu N G, Demain A L. Synthesis of bacilysin by Bacillus subtilis branches from prephenate of the aromatic amino acid pathway[J]. Journal of Bacteriology, 1988, 170(1): 482-484. DOI: 10.1128/jb.170.1.482-484.1988 |
| [13] | Steinborn G, Hajirezaei M R, Hofemeister J. bac genes for recombinant bacilysin and anticapsin production in Bacillus host strains[J]. Archives of Microbiology, 2005, 183(2): 71-79. DOI: 10.1007/s00203-004-0743-8 |
| [14] | Roscoe J, Abraham E P. Experiments relating to the biosynthesis of bacilysin[J]. Biochemical Journal, 1966, 99(3): 793-800. DOI: 10.1042/bj0990793 |
| [15] | Parker J B, Walsh C T. Stereochemical outcome at four stereogenic centers during conversion of prephenate to tetrahydrotyrosine by BacABGF in the bacilysin pathway[J]. Biochemistry, 2012, 51(28): 5622-5632. DOI: 10.1021/bi3006362 |
| [16] | Parker J B, Walsh C T. Action and timing of BacC and BacD in the late stages of biosynthesis of the dipeptide antibiotic bacilysin[J]. Biochemistry, 2013, 52(5): 889-901. DOI: 10.1021/bi3016229 |
| [17] | Kenig M, Abraham E P. Antimicrobial activities and antagonists of bacilysin and anticapsin[J]. Journal of General Microbiology, 1976, 94: 37-45. DOI: 10.1099/00221287-94-1-37 |
| [18] | Chen X H, Scholz R, Borriss M, et al. Difficidin and bacilysin produced by plant-associated Bacillus amyloliquefaciens are efficient in controlling fire blight disease[J]. Journal of Biotechnology, 2009, 140: 38-44. DOI: 10.1016/j.jbiotec.2008.10.015 |
| [19] | Wu L M, Wu H J, Chen L N, et al. Bacilysin from Bacillus amyloliquefaciens FZB42 has specific bactericidal activity against harmful algal bloom species[J]. Applied and Environmental Microbiology, 2014, 80(24): 7512-7520. DOI: 10.1128/AEM.02605-14 |
| [20] | Wu L M, Wu H J, Chen L N, et al. Difficidin and bacilysin from Bacillus amyloliquefaciens FZB42 have antibacterial activity against Xanthomonas oryzae rice pathogens[J]. Scientific Reports, 2015, 13(5): 12975. |
| [21] | Kenig M, Vandamme E, Abraham E P. The mode of action of bacilysin and anticapsin and biochemical properties of bacilysin-resistant mutants[J]. Journal of General Microbiology, 1976, 94(1): 46-54. DOI: 10.1099/00221287-94-1-46 |
| [22] | Wojciechowski M, Milewski S, Mazerski J, et al. Glucosamine-6-phosphate synthase, a novel target for antifungal agents:molecular modelling studies in drug design[J]. Acta Biochimica Polonica-English Edition, 2005, 52(3): 647-653. |
| [23] | Özcengiz G, Alaeddinoglu N G. Bacilysin production by Bacillus subtilis:effects of bacilysin, pH and temperature[J]. Folia Microbiologica, 1991, 36(6): 522-526. DOI: 10.1007/BF02884030 |
| [24] | Hilton M D, Alaeddinoglu N G, Demain A L. Bacillus subtilis mutant deficient in the ability to produce the dipeptide antibiotic bacilysin:isolation and mapping of mutation[J]. Journal of Bacteriology, 1988, 170: 1018-1020. DOI: 10.1128/jb.170.2.1018-1020.1988 |
| [25] | Özcengiz G, Alaeddinoglu N G, Demain A L. Regulation of biosynthesis of bacilysin by Bacillus subtilis[J]. Journal of Industrial Microbiology, 1990, 6(2): 91-100. DOI: 10.1007/BF01576428 |
| [26] | Inaoka T, Takahashi K, Ohnishi-Kameyama M, et al. Guanine nucleotides guanosine 5'-diphosphate 3'-diphosphate and GTP co-operatively regulate the production of an antibiotic bacilysin in Bacillus subtilis[J]. Journal of Biological Chemistry, 2003, 278(4): 2169-2176. DOI: 10.1074/jbc.M208722200 |
| [27] | Yazgan A, Ozcengiz G, Marahiel M A. Tn10 insertional mutations of Bacillus subtilis that block the biosynthesis of bacilysin[J]. Biochimica et Biophysica Acta, 2001, 1518(1/2): 87-94. |
| [28] | Belitsky B R, Sonenshein A L. Genetic and biochemical analysis of CodY-binding sites in Bacillus subtilis[J]. Journal of Bacteriology, 2008, 190(4): 1224-1236. DOI: 10.1128/JB.01780-07 |
| [29] | Karatas A Y, Cetin S, Özcengiz G. The effects of insertional mutations in comQ, comP, srfA, spo0H, spo0A and abrB genes on bacilysin biosynthesis in Bacillus subtilis[J]. Biochimica et Biophysica Acta, 2003, 1626(1/2/3): 51-56. |
| [30] | Mariappan A, Makarewicz O, Chen X H, et al. Two-component response regulator DegU controls the expression of bacilysin in plant-growth-promoting bacterium Bacillus amyloliquefaciens FZB42[J]. Journal of Molecular Microbiology and Biotechnology, 2012, 22(2): 114-125. DOI: 10.1159/000338804 |
| [31] | Köroǧlu T E, Öǧülür İ, Mutlu S, et al. Global regulatory systems operating in bacilysin biosynthesis in Bacillus subtilis[J]. Journal of Molecular Microbiology and Biotechnology, 2011, 20(3): 144-155. DOI: 10.1159/000328639 |
| [32] | Walker J E, Abraham E P. Isolation of bacilysin and a new amino acid from culture filtrates of Bacillus subtilis[J]. Biochemical Journal, 1970, 118(4): 557-561. DOI: 10.1042/bj1180557 |
| [33] | Scholz R, Vater J, Budiharjo A, et al. Amylocyclicin, a novel circular bacteriocin produced by Bacillus amyloliquefaciens FZB42[J]. Journal of Bacteriology, 2014, 196(10): 1842-1852. DOI: 10.1128/JB.01474-14 |