文章信息
- 刘艺, 朱小品, 刘喜, 田云录, 刘世家, 王云龙, 张文伟, 江玲, 王益华, 万建民
- LIU Yi, ZHU Xiaopin, LIU Xi, TIAN Yunlu, LIU Shijia, WANG Yunlong, ZHANG Wenwei, JIANG Ling, WANG Yihua, WAN Jianmin
- 水稻胚乳粉质突变体flo9的表型分析和基因定位
- Phenotyping and gene-mapping of a floury endosperm mutant flo9 in rice
- 南京农业大学学报, 2018, 41(4): 616-624
- Journal of Nanjing Agricultural University, 2018, 41(4): 616-624.
- http://dx.doi.org/10.7685/jnau.201801028
-
文章历史
- 收稿日期: 2018-01-19
2. 中国农业科学院作物科学研究所, 北京 100081
2. Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China
水稻是发展中国家最主要的粮食作物, 全球约有50%的人口以稻米为主食[1]。随着人民生活水平的不断提高, 稻米的品质改良被提到一个更加重要的高度。淀粉是水稻的主要组成部分, 在含水量为14%的精米中, 淀粉占76.7%~78.4%[2]。根据链长结构和链的连接方式差异, 可将淀粉分为直链淀粉和支链淀粉两类, 两者的比例、空间构造及相互关系是影响稻米品质的主要因素[3]。因此研究淀粉的生物合成和结构对稻米的品质改良具有重要意义。
淀粉的生物合成是将前体物质ADP葡萄糖(ADP glucose, ADPG)通过一系列酶促反应转变成淀粉, 并稳定储存在胚乳造粉体中的过程[4-5]。在研究淀粉生物合成分子机制的过程中, 人们寻找或创造了大量胚乳表现异常的突变体, 其表型表现为糯性(waxy)[6]、暗色(dull)[7]、糖质(sugary)[8]、皱缩(shrunken)[9]和粉质(floury)[10]等。在水稻中, 人们已经发现了flo1—flo8等一系列胚乳粉质突变体:其中, FLO1—FLO3[11-13]编码功能未知的蛋白或者重复序列, 可能直接或间接参与淀粉的合成; FLO4编码磷酸丙酮酸双激酶, 可能通过调节胚乳的碳氮代谢影响淀粉合成[14]; FLO5编码SSⅢa, 参与支链淀粉的合成[15]; FLO6编码了一个在水稻中广泛表达的包含CBM48结构域的蛋白, 具有淀粉结合能力[16]; FLO7编码一个功能未知的蛋白, 作为一个特异的调节因子, 调控淀粉的合成和胚乳外围造粉体的发育[17]; FLO8编码UDPG焦磷酸化酶1(Ugp1), 影响水稻胚乳淀粉的合成和结构[18]。以上突变体的发现和基因的克隆鉴定, 为阐述淀粉合成及调控机制奠定了基础, 但是仍需要挖掘更多的相关基因, 进一步解析淀粉合成调控的分子机制。
本研究从辐射诱变的‘N22’突变体库中筛选获得1份粉质突变体flo9, 其胚乳表现粉质皱缩, 对该突变体种子的形态学特征、胚活力、胚乳淀粉结构和多项理化性质进行了详细的描述和分析, 同时配制杂交组合, 对该突变基因进行精细定位, 并检测了基因的突变对淀粉合成相关基因转录水平的影响。本研究结果为FLO9的进一步克隆与功能研究奠定了基础。
1 材料与方法 1.1 试验材料flo9是由籼稻品种‘N22’经60Co辐射诱变后, 经多代回交、自交筛选得到的稳定遗传的粉质突变体。由于纯合致死, 选择杂合单株与粳稻品种‘滇粳优1号’配制杂交组合并自交获得F2群体, 利用SSR和Indel标记进行基因定位。所有材料种植于南京农业大学牌楼实验基地和土桥实验基地。
1.2 种子活力测定取成熟种子20粒, 30 ℃恒温浸种24 h后, 吸干种子表面水分, 将种子放入15 mL试管中, 加入8 mL 5 g·L-1 TTC染液, 30 ℃恒温染色2 h后, 用蒸馏水冲洗3次, 观察染色情况。胚部呈红色则种子具有活力, 胚部无色则种子不具有活力, 胚部呈浅红色则种子活力较弱。
1.3 胚乳横切面扫描电镜观察将成熟种子脱壳, 用锋利的刀片从种子中间横切, 样品送至南京农业大学生命科学学院电镜实验中心, 由该中心的专业人员协助完成样品的制备, 用日立S-3000N型扫描电子显微镜观察并拍照。
1.4 半薄切片观察方法参照文献[16]。具体步骤如下:取花后18 d的胚乳, 从胚乳中部切出1 mm左右的薄片, 置于戊二醛固定液中, 4 ℃避光固定12 h后, 用PBS漂洗3次; 在4 ℃分别用30%和50%(体积分数)乙醇脱水, 每次15 min; 在-20 ℃用70%乙醇脱水2次, 每次30 min; 在-20 ℃用70%乙醇和100% LR white树脂(体积比1:2)渗透2 h, 100% LR white树脂渗透2 h, 100% LR white树脂过夜渗透; 次日, 用100% LR white树脂包埋样品, 60 ℃聚合48 h。对聚合后的胶囊修块, 用Leica RM2265全自动半薄切片机切成1 μm的切片, 并用10 g·L-1的I2-KI染色, 在Zeiss Ax10显微镜下观察拍照。
1.5 成熟种子理化性质测定将成熟种子去壳后磨成糙米粉, 过150 μm筛, 50 ℃烘干至恒质量后备用。总淀粉含量采用Megazyme starch assay kit(K-TSTA)测定。直链淀粉含量按照《米质测定方法:NY 147—1988》测定。脂肪含量采用丹麦福斯公司的全自动脂肪测定仪测定。尿素膨胀方法参照文献[19]。RVA黏度特性采用瑞典波通公司的RVA快速黏度分析仪测定。
1.6 FLO9基因定位配制flo9杂合单株(FlO9flo9)和‘滇粳优1号’的杂交组合, 挑选其F2群体中粉质不透明的种子, 浸种12 h, 采用CTAB法提取种子基因组DNA。标记选用本实验室均匀分布于12条染色体的SSR和Indel标记, 并用Primer Premier 5.0软件和Gramene(http://www.gramene.org/)开发新标记, 引物合成由金斯瑞生物科技有限公司完成, 所有定位所用引物见表 1。PCR反应体系10 μL:1 μL 10×Buffer(含Mg2+), 0.1 μL Taq(0.5 U·μL-1)DNA聚合酶, 0.3 μL dNTPs(1 mmol·L-1), 1 μL DNA模板(约15 ng), 2 μmol·L-1上、下游引物各1 μL, 6.6 μL ddH2O。PCR程序:94 ℃ 5 min; 95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 40 s, 33个循环; 72 ℃ 5 min, 4 ℃ 10 min。PCR产物采用1.125 g·L-1的聚丙烯酰胺凝胶电泳, 银染显色。
| 标记Markers | 引物(5′→3′)Primers |
| XY9-26 | TCCGACGAGCAAATCAGTCA/GGTTATTCCGCGCGTTTCT |
| XY9-27 | AGTAGGAGAATGTGCATTGTGC/CATGTCCATTCCTTGTCCGG |
| XY9-40 | TCCAAAATGTCACCAGACTACC/AGAGGCTGCTTTGTCGGCTT |
| XY9-20 | GGTGGTTTGGTGGTGAAGTC/GGCACCATGCTCAAAGAGAC |
| XY9-8 | AAAAGAAAGGCAGAGCAACT/ACAGCAGCAGCCATGAGA |
| XY9-28 | AGTGCAAACTGCCATGTATGT/TTTGGAACCCTGTAGGATTGTG |
| I9-6 | GCATGTATCGTGGACATGGA/TCCTTGATCAACACCGTCAA |
取花后18 d的胚乳, 用RNApure Plant Kit(DNAseⅠ)提取水稻胚乳RNA, 用PrimeScriptⅡ RTase将RNA反转为cDNA, 采用7500实时荧光定量PCR系统检测和分析淀粉和脂质合成相关基因的表达水平。反应体系20 μL:8 μL cDNA模板(约80 ng), 10 μmol·L-1上、下游引物各1 μL, 10 μL SYBR Premix Ex Taq Ⅱ。每个样品3次重复。PCR反应程序:95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 40个循环; 并添加熔解曲线以确定PCR产物的特异性。以水稻Ubiquitin基因作为内参, 采用2-ΔΔCT[4]方法处理试验数据。试验所用引物见前人研究[20]和表 2。
| 基因Genes | 引物(5′→3′) Primers |
| ACC1 | CATGCCAAAGACCTCGAGAA/CAGCCCAGAGAAACGAATCA |
| ACC2 | TGCATATTGCTTTGTTGGGTGCTG/TCTGTCAGCATCATCACCACTTTC |
| ACP5 | ACCATCAACAGGGTCAAGACTGC/GGCAGTGAATGCTTGACTTGGAG |
| FAT9 | TCATGTAACAAGGCCTGAGC/GAGGGAACAACATGGCGTAT |
| KSA11 | GTTGCCAAGAAGGTCCTCAA/ATGTGCCAGTCAGTCAACTC |
| 注:ACC:乙酰辅酶A羧化酶Acetyl CoA carboxylase; ACP:酰基载体蛋白Acyl carrier protein; FAT:脂酰-酰基载体蛋白硫酯酶Fatty thioesterase; KSA:酮脂酰-ACP合酶Ketoacyl-acyl carrier protein synthase. | |
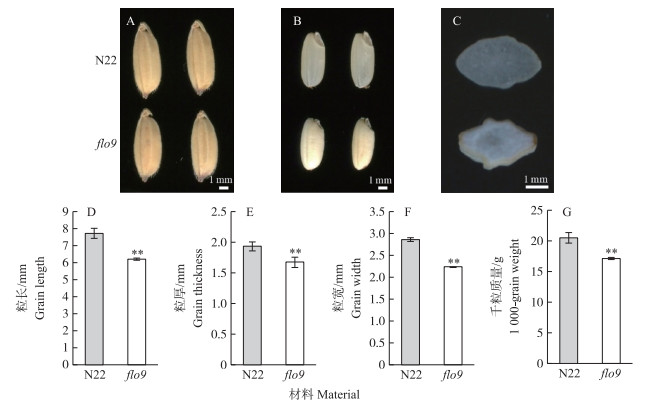
由图 1可见:flo9突变体成熟种子轻微皱缩, 胚乳外围呈粉质、不透明状, 胚乳中心透明, 而野生型成熟种子胚乳呈现透明状(图 1-A、B、C)。与野生型相比, flo9突变体种子的粒长、粒宽和粒厚分别显著减少18.6%、22.3%和14.0%(图 1-D、E、F), 千粒质量也显著下降(图 1-G)。表明flo9突变体的种子发育受到影响。
|
图 1 ‘N22’和flo9突变体种子表型分析 Figure 1 Phenotypic analysis of'N22'and flo9 mutant seeds **P < 0.01. The same as follows. |
flo9突变体经过多代回交、自交, 不能获得纯合的突变体植株, 猜测突变体纯合致死。将野生型和突变体去壳的种子在30 ℃恒温发芽3 d, 结果(图 2)发现野生型能正常发芽成苗, 而突变体不能萌发(图 2-A、D)。为了进一步验证该结果, 将野生型和突变体去壳种子进行TTC染色, 检测种子活力, 发现野生型的胚能染成红色, 而突变体的胚部不能着色(图 2-B、E)。为了进一步观察胚部的发育情况, 将野生型和突变体去壳种子在30 ℃恒温吸胀12 h, 在体视镜下观察到野生型的胚部出现萌动迹象, 而flo9突变体的胚干瘪坏死, 无萌动迹象(图 2-C、F)。说明flo9突变体种子胚部存在严重缺陷, 导致种子活力丧失, 表现为纯合致死。

|
图 2 种子活力分析 Figure 2 Analysis of seed viability A、D. 30 ℃恒温发芽3 d后的种子; B、E. TTC染色的种子; C、F.种子30 ℃恒温吸胀12 h的胚。 A, D. Seeds germinated at 30 ℃ for three days; B, E. Seeds stained with TTC; C, F. The embryos imbibed at 30 ℃ for 12 h. |
扫描电镜观察野生型和flo9突变体成熟种子胚乳的横断面, 结果(图 3)显示野生型胚乳横断面的淀粉颗粒呈不规则的多面体状, 且排列紧密, 胚乳中心和外围淀粉颗粒结构无明显差异(图 3-A、B、C), 而flo9突变体胚乳边缘的淀粉颗粒呈圆形, 并且排列疏松多孔隙(图 3-F), 胚乳中心淀粉颗粒则排列紧密, 与野生型相似(图 3-E)。

|
图 3 ‘N22’和flo9突变体成熟种子扫描电镜观察 Figure 3 Scanning electron microscopy observation of mature seeds of'N22'and flo9 mutant A、D.胚乳横断面; B、E.胚乳横断面中心区域(白色框); C、F.胚乳横断面边缘区域(黑色框)。 A, D. Endosperm cross-sections; B, E. The central area of endosperm cross-sections(white squares); C, F. The outer area of endosperm cross-sections(black squares). |
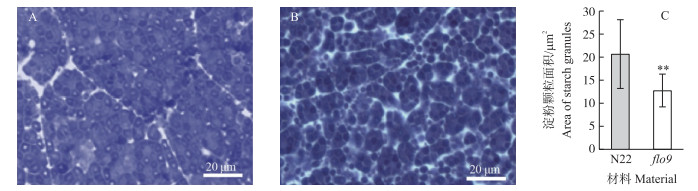
花后18 d的野生型胚乳边缘造粉体之间相互挤压, 排列紧密, 几乎没有空隙(图 4-A)。而突变体胚乳边缘造粉体多呈圆形或者椭圆形, 造粉体之间排列疏松, 可以观察到大量空隙(图 4-B)。突变体淀粉颗粒截面积显著小于野生型(图 4-C)。
|
图 4 ‘N22’(A)和flo9突变体(B)成熟种子半薄切片观察及淀粉颗粒面积(C) Figure 4 Semi-thin section observation of'N22'(A)and flo9 mutant(B)mature seeds and area of starch granules(C) |
flo9突变体总淀粉含量与野生型无显著差异(图 5-A), 但直链淀粉含量和脂质含量显著下降(图 5-B、C)。米粉在尿素膨胀体积变化上, flo9突变体始终低于野生型, 当尿素浓度为5 mol·L-1时, 野生型的米粉发生明显膨胀, 而flo9突变体无明显膨胀(图 5-D、E)。黏度分析发现, 突变体淀粉消减值、回复值和崩解值下降(图 5-F和表 3)。说明基因的突变改变了flo9突变体成熟种子的多项理化性质。

|
图 5 ‘N22’和flo9突变体种子理化性质分析 Figure 5 Analysis of physicochemical properties of'N22'and flo9 mutant seeds A、B、C.总淀粉、直链淀粉和脂质含量; D、E.淀粉在不同浓度尿素中的膨胀体积; F.成熟种子淀粉快速黏度分析。 A, B, C. The contents of total starch, amylose, and lipid; D, E. Swelling volume changes of starch in different concentration urea; F. Rapid viscosity analysis of mature seeds starch. |
| 材料 Material |
最高黏度 PKV |
热浆黏度 HPV |
崩解值 BDV |
冷胶黏度 CPV |
回复值 CSV |
消减值 SBV |
起浆温度/℃ PaT |
峰值时间/min PeT |
| N22 | 224.8 | 161.7 | 63.1 | 371.9 | 210.3 | 147.2 | 75.9 | 5.9 |
| flo9 | 118.7 | 103.7 | 15.0 | 230.8 | 127.1 | 112.1 | 75.9 | 6.0 |
| Note:PKV:Peak viscosity; HPV:Hot paste viscosity; BDV:Breakdown; CPV:Cool paste viscosity; CSV:Consistence; SBV:Setback; PaT:Pasting temperature; PeT:Peak time. | ||||||||
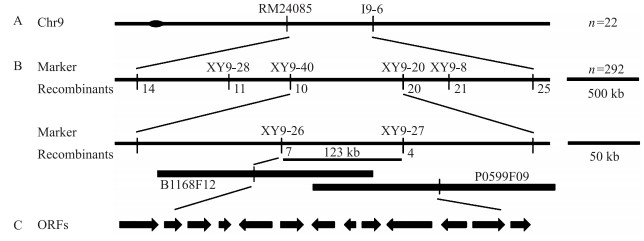
在flo9杂合单株(FlO9flo9)和‘滇粳优1号’的F2代群体中挑选粉质极端个体进行基因的精细定位。利用22个极端个体, 将目标突变基因连锁在第9染色体长臂RM24085和I9-6两标记之间(图 6-A); 利用292个极端个体将目标基因定位在XY9-26和XY9-27两标记之间, 物理距离123 kb(图 6-B)。利用Gramene网站对该区间内的基因进行了预测, 发现该区间内共有13个ORF(图 6-C), 其中7个ORF具有明确的功能注释, 1个编码转座子蛋白, 2个ORF编码保守的假定蛋白, 剩余3个ORF编码功能未知的表达蛋白(表 4)。
|
图 6 FLO9的精细定位 Figure 6 Fine mapping of FLO9 A. FLO9与标记RM24085和I9-6连锁; B.利用292个粉质极端个体将FLO9定位在123 kb区间内; C.定位区间内有13个候选基因。 A. FLO9 was linked with RM24085 and I9-6;B. FLO9 was located in a 123 kb region based on 292 individuals; C. Thirteen candidate genes in the mapping region. |
| 位点Locus | 功能描述Description of function |
| LOC_Os09g20340 | 含DUF630/DUF632结构域的蛋白DUF630/DUF632 domains containing protein |
| LOC_Os09g20350 | DRE结合因子2 DRE binding factor 2 |
| LOC_Os09g20360 | 表达蛋白Expressed protein |
| LOC_Os09g20370 | 表达蛋白Expressed protein |
| LOC_Os09g20380 | 转座子蛋白Transposon protein |
| LOC_Os09g20390 | 海藻糖-6-磷酸磷酸酶Trehalose-6-phosphate phosphatase |
| LOC_Os09g20400 | 保守假定蛋白Conserved hypothetical protein |
| LOC_Os09g20410 | 表达蛋白Expressed protein |
| LOC_Os09g20420 | 保守假定蛋白Conserved hypothetical protein |
| LOC_Os09g20430 | 电子转运蛋白SCO1/SenC家族蛋白Electron transport protein SCO1/SenC family protein |
| LOC_Os09g20440 | 琥珀酸脱氢酶和延胡索酸还原酶铁硫蛋白Succinate dehydrogenase and fumarate reductase iron-sulfur protein |
| LOC_Os09g20450 | 推定转座子蛋白, CACTA, En/Spm亚类Transposon protein, putative, CACTA, En/Spm sub-class |
| LOC_Os09g20460 | 含VQ结构域的蛋白VQ domain containing protein |
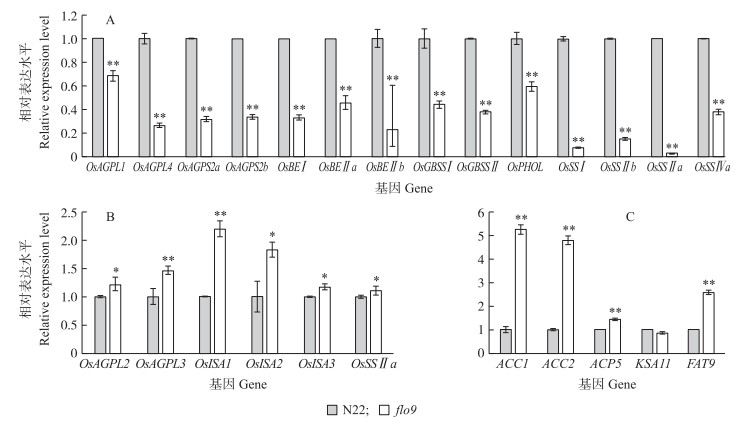
用花后18 d种子提取总RNA, 对淀粉合成相关基因进行转录水平分析, 发现大量与淀粉合成相关的基因显著下调, 其中OsAGPL4、OsAGPS2a、OsSSⅠ、OsSSⅡb、OsSSⅢa、OsBEⅠ、和OsBEⅡb分别下调73%、68%、92%、85%、97%、67%和77%(图 7-A)。部分淀粉合成相关的基因上调(图 7-B)。由于flo9突变体中脂质含量显著下降, 检测了部分参与脂质合成相关基因的表达情况, 发现除KSA11外, flo9突变体中参与脂质合成的主要基因均显著上调(图 7-C)。
|
图 7 花后18 d‘N22’和flo9突变体种子淀粉(A、B)和脂质(C)合成相关基因表达水平 Figure 7 Expression level of starch(A, B)and lipid(C)synthesis genes of'N22'and flo9 mutant seeds at 18 day after flowering *P < 0.05, **P < 0.01. |
本研究表明, flo9突变体的成熟种子粒长、粒宽、粒厚和千粒质量都显著降低, 同时种子活力丧失, 表现出纯合致死的表型。flo9突变体淀粉颗粒的发育和淀粉的合成受阻, 直链淀粉和支链淀粉的比例和空间构造发生改变。flo9突变体中脂质含量显著降低, 但主要参与脂质合成的部分基因的转录水平显著上调, 暗示flo9中脂质合成相关基因的表达可能受到反馈调节。利用314个粉质极端个体, 将目标基因定位在Chr9上123 kb内, 该区间有13个ORF。对每个ORF进行分析发现, ORF1/LOC_Os09g20340编码具有DUF630/DUF632结构域的未知功能的蛋白。ORF2/LOC_Os09g20350编码脱水反应元件结合蛋白(DRE), 在植物反应和适应非生物胁迫方面扮演着重要的角色[21]。ORF6/LOC_Os09g20390编码海藻糖-6-磷酸磷酸酶, 参与海藻糖-6-磷酸的代谢[22]。ORF10/LOC_Os09g20430编码线粒体蛋白细胞色素C氧化酶1(SCO1), 该蛋白对呼吸链中复合物Ⅳ的正确组装至关重要, 在拟南芥中影响植物的胚胎建成[23-24]。ORF11/LOC_Os09g20440编码琥珀酸脱氢酶和富马酸还原酶铁硫蛋白。ORF12/LOC_Os09g20450编码CACTA家族转座子[25]。ORF13/LOC_Os09g20460编码植物特异性蛋白VQ(缬氨酸和谷氨酰胺基序)蛋白, 参与植物对生物和非生物胁迫的应答[26], 近年来发现VQ蛋白通过调控胚乳发育以调控拟南芥的种子大小[27-28]。在水稻中, 以上候选基因均未有参与淀粉、脂质合成或者种子发育的报道, 所以FLO9是一个影响造粉体发育, 调控淀粉与脂质合成并控制种子发育的新基因。该结果为候选基因的预测和克隆提供了较好的理论基础。
在对玉米、水稻和拟南芥等植物淀粉合成和种子发育的研究中, 发现控制种子发育和淀粉合成的相关基因在单子叶植物和双子叶植物中存在较高的保守性。近年来, 在水稻中已经报道了8个粉质突变体(flo1—flo8), 粉质皱缩是水稻胚乳突变体的常见类型, 但是同时具有胚胎致死表型的突变体却鲜有报道。玉米中报道的相对较多, 多为影响线粒体功能的基因。如EMP7编码线粒体特异性的E型PPR蛋白, 参与RNA的编辑, 在emp7突变体中, 线粒体功能受损, 导致呼吸速率显著下降, 突变体籽粒表现出粉质皱缩且胚胎致死的表型[29]。DEK35编码线粒体P型PPR蛋白, 该基因的突变降低了nad4的拼接效率, 从而影响线粒体复合物Ⅰ的组装和NADH脱氢酶活性, 突变体籽粒同样表现为粉质皱缩且胚胎致死的表型[30]。我们的研究表明flo9突变体成熟种子粉质皱缩并伴随胚胎致死的表型, 猜测FLO9可能与线粒体的功能相关, 但还有赖于进一步开展基因的精细定位和测序工作, 以确定目标基因。本研究为FLO9目标基因的确定和后续的功能研究打下了基础。
| [1] |
谢华安, 张建福, 王乌齐, 等. 超级稻育种实践和前景[J].
分子植物育种, 2006, 4(3): 4-10.
Xie H A, Zhang J F, Wang W Q, et al. Practice and prospect on breeding of super-hybridization rice in China[J]. Molecular Plant Breeding, 2006, 4(3): 4-10. (in Chinese with English abstract) |
| [2] |
黄发松, 孙宗修, 胡培松, 等. 食用稻米品质形成研究的现状与展望[J].
中国水稻科学, 1998, 12(3): 172-176.
Huang F S, Sun Z X, Hu P S, et al. Present situations and prospects for the research on rice grain quality forming[J]. Chinese Journal of Rice Science, 1998, 12(3): 172-176. (in Chinese with English abstract) |
| [3] | Juliano B O. Rice grain quality:problems and challenges[J]. Cereal Foods World, 1990, 35(2): 245-253. |
| [4] |
彭城. 水稻籽粒中调控淀粉合成的关键基因FLO6的图位克隆和功能分析[D]. 南京: 南京农业大学, 2013.
Peng C. Map-based cloning and functional analysis of a key gene flo6 involved in regulation of starch synthesis in rice endosperm[D]. Nanjing: Nanjing Agricultural University, 2013(in Chinese with English abstract). |
| [5] | Pérez S, Bertoft E. The molecular structures of starch components and their contribution to the architecture of starch granules:a comprehensive review[J]. Starch/Stärke, 2010, 62: 389-420. DOI: 10.1002/star.201000013 |
| [6] | Sato H, Suzuki Y, Sakai M, et al. Molecular characterization of Wx-mq, a novel mutant gene for low-amylose content in endosperm of rice[J]. Breeding Science, 2002, 52: 131-135. DOI: 10.1270/jsbbs.52.131 |
| [7] | Zeng D, Yan M, Wang Y, et al. Du1, encoding a novel Prp1 protein, regulates starch biosynthesis through affecting the splicing of Wxb pre-mRNAs in rice(Oryza sativa L. )[J]. Plant Molecular Biology, 2007, 65(4): 501-509. |
| [8] | James M G, Robertson D S, Myers A M. Characterization of the maize gene sugary1, a determinant of starch composition in kernels[J]. Plant Cell, 1995, 7(4): 417-429. DOI: 10.1105/tpc.7.4.417 |
| [9] | Kawagoe Y, Kubo A, Satoh H, et al. Roles of isoamylase and ADP-glucose pyrophosphorylase in starch granule synthesis in rice endosperm[J]. Plant Journal, 2005, 42(2): 164-174. DOI: 10.1111/tpj.2005.42.issue-2 |
| [10] | Lending C R, Larkins B A. Effect of the floury-2 locus on protein body formation during maize endosperm development[J]. Protoplasma, 1992, 171: 123-133. DOI: 10.1007/BF01403727 |
| [11] | Satoh H, Omura T. New endosperm mutations induced by chemical mutagens in rice(Oryza sativa L.)[J]. Japanese Journal of Breeding, 1981, 31(3): 447-457. |
| [12] | She K C, Kusano H, Koizumi K, et al. A novel factor FLOURY ENDOSPERM2 is involved in regulation of rice grain size and starch quality[J]. Plant Cell, 2010, 22(10): 3280-3294. DOI: 10.1105/tpc.109.070821 |
| [13] | Hamada S, Suzuki K, Suzuki Y. Development of a new selection method and quality improvement of sugary-1 rice mutants[J]. Breeding Science, 2014, 63(5): 461-467. DOI: 10.1270/jsbbs.63.461 |
| [14] | Kang H G, Park S, Matsuoka M, et al. White-core endosperm floury endosperm-4 in rice is generated by knockout mutations in the C4-type pyruvate orthophosphate dikinase gene(OsPPDKB)[J]. Plant Journal, 2005, 42(6): 901-911. DOI: 10.1111/tpj.2005.42.issue-6 |
| [15] | Ryoo N, Yu C, Park C S, et al. Knockout of a starch synthase gene OsSS Ⅲa/Flo5 causes white-core floury endosperm in rice(Oryza sativa L.)[J]. Plant Cell Reports, 2007, 26(7): 1083-1095. DOI: 10.1007/s00299-007-0309-8 |
| [16] | Peng C, Wang Y, Liu F, et al. FLOURY ENDOSPERM6 encodes a CBM48 domain-containing protein involved in compound granule formation and starch synthesis in rice endosperm[J]. Plant Journal, 2014, 77(6): 917-930. DOI: 10.1111/tpj.2014.77.issue-6 |
| [17] | Zhang L, Ren Y L, Lu B Y, et al. FLOURY ENDOSPERM7 encodes a regulator of starch synthesis and amyloplast development essential for peripheral endosperm development in rice[J]. Journal of Experimental Botany, 2016, 67(3): 633-647. DOI: 10.1093/jxb/erv469 |
| [18] | Long W H, Dong B N, Wang Y H, et al. FLOURY ENDOSPERM8, encoding the UDP-glucose pyrophosphorylase 1, affects the synthesis and structure of starch in rice endosperm[J]. Journal of Plant Biology, 2017, 60(5): 513-522. DOI: 10.1007/s12374-017-0066-3 |
| [19] |
潘鹏屹, 朱建平, 王云龙, 等. 水稻粉质胚乳突变体ws的表型分析及基因克隆[J].
中国水稻科学, 2016, 30(5): 447-457.
Pan P Y, Zhu J P, Wang Y L, et al. Phenotyping and gene cloning of a floury endosperm mutant ws in rice[J]. Chinese Journal of Rice Science, 2016, 30(5): 447-457. (in Chinese with English abstract) |
| [20] | Ohdan T, Francisco P B Jr, Sawada T, et al. Expression profiling of genes involved in starch synthesis in sink and source organs of rice[J]. Journal of Experimental Botany, 2005, 56(422): 3229-3244. DOI: 10.1093/jxb/eri292 |
| [21] | Ke Y G, Yang Z J, Yu S W, et al. Characterization of OsDREB6 responsive to osmotic and cold stresses in rice[J]. Journal of Plant Biology, 2014, 57(3): 150-161. DOI: 10.1007/s12374-013-0480-0 |
| [22] | Kretzschmar T, Pelayo M A, Trijatmiko K R, et al. A trehalose-6-phosphate phosphatase enhances anaerobic germination tolerance in rice[J]. Nature Plants, 2015, 1: 1-5. |
| [23] | Fujimoto M, Yamada A, Kurosawa J, et al. Pleiotropic role of the Sco1/SenC family copper chaperone in the physiology of Streptomyces[J]. Microbial Biotechnology, 2012, 5(4): 477-488. DOI: 10.1111/mbt.2012.5.issue-4 |
| [24] | Steinebrunner I, Landschreiber M, Krause-Buchholz U, et al. HCC1, the Arabidopsis homologue of the yeast mitochondrial copper chaperone SCO1, is essential for embryonic development[J]. Journal of Experimental Botany, 2011, 62(1): 319-330. DOI: 10.1093/jxb/erq269 |
| [25] | Yang Q, Li Z, Li W, et al. CACTA-like transposable element in ZmCCT attenuated photoperiod sensitivity and accelerated the postdomestication spread of maize[J]. Proc Natl Acad Sci USA, 2013, 110(42): 16969-16974. DOI: 10.1073/pnas.1310949110 |
| [26] |
张高原, 张一卉, 李景娟, 等. 植物VQ蛋白研究进展[J].
农业生物技术学报, 2017, 25(1): 142-149.
Zhang G Y, Zhang Y H, Li J J, et al. Research progress of plant VQ protein[J]. Journal of Agricultural Biotechnology, 2017, 25(1): 142-149. (in Chinese with English abstract) |
| [27] | Wang A H, Garcia D, Zhang H Y, et al. The VQ motif protein IKU1 regulates endosperm growth and seed size in Arabidopsis[J]. Plant Journal, 2010, 63(4): 670-679. DOI: 10.1111/j.1365-313X.2010.04271.x |
| [28] | Song W B, Zhao H M, Zhang X B, et al. Genome-wide identification of VQ motif-containing proteins and their expression profiles under abiotic stresses in maize[J]. Frontiers in Plant Science, 2016, 6: 1-13. |
| [29] | Sun F, Wang X, Bonnard G, et al. Empty pericarp7 encodes a mitochondrial E-subgroup pentatricopeptide repeat protein that is required for ccmFN editing, mitochondrial function and seed development in maize[J]. Plant Journal, 2015, 84(2): 283-295. DOI: 10.1111/tpj.12993 |
| [30] | Chen X, Feng F, Qi W, et al. Dek35 encodes a PPR protein that affects cis-splicing of mitochondrial nad4 intron 1 and seed development in maize[J]. Molecular Plant, 2017, 10(3): 427-441. DOI: 10.1016/j.molp.2016.08.008 |




