文章信息
- 曾振灵, 王勉之, 孙永学
- ZENG Zhenling, WANG Mianzhi, SUN Yongxue
- 环境中噬菌体携带耐药基因传播的研究进展
- Research advances of bacteriophages in contributing antimicrobial resistance gene in the environment
- 南京农业大学学报, 2018, 41(4): 598-604
- Journal of Nanjing Agricultural University, 2018, 41(4): 598-604.
- http://dx.doi.org/10.7685/jnau.201712006
-
文章历史
- 收稿日期: 2017-12-06
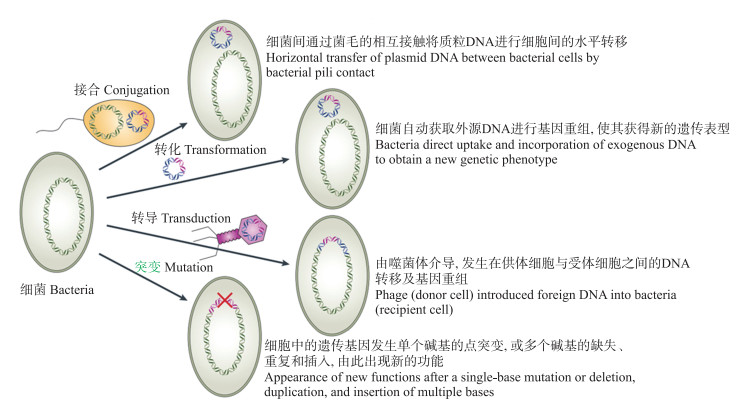
抗生素(含合成抗菌药)超量使用导致其在环境中不断残留, 新型耐药基因(antimicrobial resistant genes, ARG)[1]及耐药细菌(antimicrobial resistant bacterial, ARB)[2]严重威胁抗生素治疗感染性疾病的效果。同时, 细菌群体易通过遗传突变(genetic mutation, GM)或通过ARG水平转移(horizontal gene transfer, HGT)将基因向子代遗传或远距离传播给其他菌株, 造成了耐药性的传播, 从而增加了感染性疾病的治疗难度。相比于细菌自身突变获得的耐药性, 耐药基因通过移动遗传元件(mobile genetic element, MGE)进行的水平转移则是耐药性广泛传播中最为关键的一环, 此过程一般由插入序列、转座子、整合子、质粒或噬菌体等介导[3-4]。其中, 由噬菌体介导的基因转移是细菌基因DNA重组和转移的方式之一(图 1)[5], 也是最容易被忽视的一种。
噬菌体(bacteriophages或phages)作为细菌病毒, 是地球上最丰富和最多样化的生物体, 其数量是细菌的10~100倍[6-7]。噬菌体分为13个科[8], 其中长尾噬菌体科(Siphoviridae)、肌尾噬菌体科(Myoviridae)和足状噬菌体科(Podoviridae)3个科共15个属是最常见的噬菌体种类[8]。噬菌体可以通过转导作用与其宿主细菌进行遗传物质的交换, 以利于繁殖(短期)或促进其进化(长期)。曾有研究表明, 噬菌体的遗传交换不仅可以在紧密相关的细菌物种间发生, 也可以发生在远缘的物种间, 甚至高度保守的16S rRNA基因序列中也发现过噬菌体的基因[9]。因此, 理论上环境中大量存在的噬菌体不仅可以通过转导作用与细菌基因进行重组并携带相关基因在环境中传播, 同时在自然环境中可能协助并促进ARG的水平传播。虽然与人类相关的病原菌噬菌体基因组很少或几乎不携带ARG[10], 但是非人源性的环境样品噬菌体基因组(例如动物粪便、污水废水、淡水和海洋环境)却包含大量的ARG[11]。这些结果表明, 噬菌体基因组携带了大量的ARG, 并且在传播抗生素耐药性过程中发挥了重要作用, 因此不应低估因噬菌体转导作用而导致的ARG的水平转移作用。
本文将从荧光定量PCR(qPCR)方法和全基因组测序方法两方面综述全球不同环境样本中噬菌体基因组携带ARG的情况以及其多样性和丰度的研究进展, 并讨论其潜在危害性, 希望引起更多的关注。
1 基于qPCR探究噬菌体携带ARG在环境中的传播最近的研究结果表明, 噬菌体在ARG的出现和传播中可能发挥比以前预期更多的重要作用, 在不同来源的介质中噬菌体携带ARG的情况见表 1[12-22]。在这些研究中, Colomer-Lluch等[12]使用实时荧光定量PCR(qPCR)检测到城市污水和河水样品噬菌体DNA中2种编码β-内酰胺酶基因(blaTEM和blaCTX-M)和1个编码青霉素结合蛋白的基因(mecA)的存在; Marti等[15]对不同医院污水排出口样品中噬菌体DNA中的ARG进行定量, 结果显示存在高水平表达的β-内酰胺酶基因(blaTEM、blaCTX-M和blaSHV), 以及对氟喹诺酮类药物敏感性降低的耐药基因(qnrA、qnrB和qnrS)。同时, Shousha等[19]对鸡肉样品进行研究, 证明了噬菌体DNA中的一些耐药基因(blaTEM和blaCTX-M)可以转移到易感的大肠杆菌菌株中, 并使其变得对氨苄西林具有抗性。
| 采样地点 Site |
样品类型 Types |
耐药基因的绝对丰度/g-1或mL-1 Abundance of ARG |
检测方法 Methods |
参考文献 Reference |
| 屠宰场和养殖场, 西班牙 Slaughterhouses and farms, Spain |
动物粪便及污水 Fecal and wastewater |
blaTEM(103~104)、blaCTX-M(102~103)mecA(101~103) | qPCR | [12] |
| 略夫雷加特河, 西班牙 Llobregat river, Spain |
城市污水 Raw urban wasterwater |
blaTEM(104), blaCTX-M-9、mecA(102) | qPCR | [13] |
| 西班牙巴塞罗那 Barcelona, Spain |
城市污水 Raw urban wastewater |
blaTEM、sul1(105.5~104.4), blaCTX-M、qnrA、qnrB、qnrS(103) | qPCR | [14] |
| 西班牙加泰罗尼亚区 Catalonia, Spain |
医院污水 Hospitals wastewater |
blaSHV、qnrS(104), blaTEM、qnrA、qnrB、blaCTX-M(-) | qPCR | [15] |
| 西班牙圣保罗医院 Sant Pau Hospital, Spain |
人类粪便 Human fecal samples |
blaTEM(105)、blaCTX-M-1(103)、qnrA(103~105), mecA、rmA、qnrS(-) | qPCR | [16] |
| 西班牙巴塞罗那 Barcelona, Spain |
城市污水 Raw urban wastewater |
qnrA、qnrS、blaTEM、blaCTX-M | qPCR | [17] |
| 加拿大农业食品研究农场 Agri-Food farm, Canada |
粪便-土壤模型 Fecal-soil samples |
strA、strB、blaOXA-20(-), aadA(101~102)、sul1(102) | 构建模型(基于qPCR) Modelling(based on qPCR) |
[18] |
| 奥地利维也纳鸡肉市场 Viena markets, Austria |
包装, 冷藏(4 ℃)的鸡肉 Chicken meat |
tet(A)、aphA1、blaTEM-1、floR(+)、tet(B)、tet(C)、blaCMY、blaIMP、blaOXA、blaPSE、catI(-) | qPCR | [19] |
| 西班牙巴塞罗那大都会区 Barcelona Metropolitan area, Spain |
污水处理池的出水口水样 Raw urban wastewater |
blaTEM、blaCTX-M、sul1 | 构建模型(基于qPCR) Modelling(based on qPCR) |
[20] |
| 略夫雷加特河, 地中海区域 Llobregat river, Barcelona |
水样和河床底泥 Water and sediment |
blaTEM、sul1 blaCTX-M、qnrA、qnrS、mecA(+) | qPCR | [21] |
| 泰尔河和医院 Ter river and hospital |
废水样 Wastewater |
blaTEM、blaNDM、blaKPC、tetW、ermB、qnrS、vanA(+) | qPCR | [22] |
| 注:“+”表示检测阳性, “-”表示检测阴性。‘+’and‘-’mean positive and negative test result, respectively. | ||||
除了直接调查研究环境样品中噬菌体携带ARG的特性外, 相关学者同样深入研究了不同因素对噬菌体携带及传播耐药基因的影响。Colomer-Lluch等[17]研究诱导因子对粪便污水中噬菌体携带耐药基因qnrA和qnrS丰度的影响, 观察到城市污水样品处理螯合剂, 如EDTA和柠檬酸钠均能显著增加ARG的丰度, 而丝裂霉素C和环丙沙星并没有显著影响噬菌体中ARG的丰度。Calero-Caceres等[20]研究pH、氯离子和紫外线等对噬菌体携带并传播耐药基因的影响, 发现噬菌体中耐药基因在不同理化条件下, 比在细菌中的保存时间更长, 表明了环境中噬菌体携带耐药基因可能会对特定ARG有保护作用。
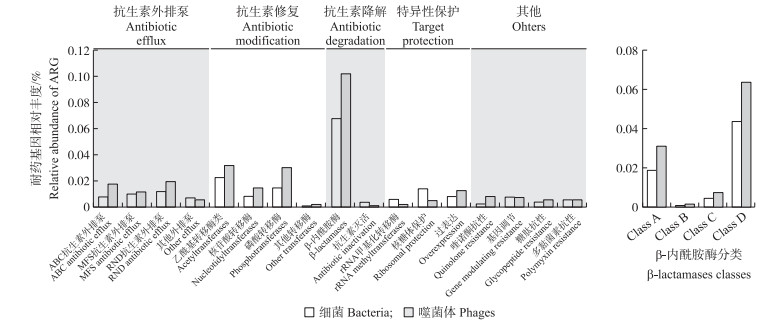
综上所述, 近几年的研究发现, 用传统方法检测到噬菌体DNA中携带了高水平的β-内酰胺酶基因(主要集中在A和D型[23]的blaTEM、blaCTX-M和blaSHV)和氟喹诺酮类耐药基因(qnrA、qnrB和qnrS), 以及零星的其他耐药基因如对磺胺类耐药的sul1、对大环内酯耐药的ermB及耐万古霉素的vanA和耐四环素类的tetW。但是, 其他耐药基因是否被携带及携带的环境偏好性如何仍需要进一步的研究; 另外, 也需要进一步明确噬菌体携带耐药基因的环境背景, 为以后定向的机制研究和遏制噬菌体携带耐药基因的传播打下基础。
上述的研究结果不仅证明了噬菌体具有携带ARG在环境中扩散的能力, 也暗示人为的输入可能促进ARG的传播(如螯合剂的使用可以提高ARG的丰度), 这应引起公众的关注。可喜的是, 随着科技的进步, 第二、三代测序技术的成熟, 如今可以结合宏基因组的方法, 更深层地研究耐药基因在噬菌体基因组的排布规律、耐药基因和细菌群落及噬菌体群落的关系, 为探究其传播机制助力。
2 基于宏基因组探究噬菌体携带ARG在环境中的传播近两年来, 随着测序手段的进步, 第二代和第三代测序已陆续推出, 宏基因组DNA的测序已经从过去的鸟枪(shotgun)法的效率低、测序深度不够向更优的方法过渡。而测序又提供了显著优于传统定量的方法, 因为它允许更好地了解补测体的自然结构和功能, 同时使用序列和基于功能的探究方法揭示细菌及噬菌体基因组中存在的ARG或ARG样基因。因此, 通过分析整合NCBI、MG-RAST等数据库的海量测序数据, 一方面可以了解噬菌体携带相关基因(如耐药基因和致病基因)的情况; 另一方面, 由于噬菌体在疾病治疗方面的巨大潜力[7], 对从环境中分离得到的噬菌体进行宏基因组测序, 不仅可以明确治疗用噬菌体的安全性[24], 还可以通过对基因序列的分析寻找耐药基因的携带情况。例如刘小东等[25]从医院污水中分离了一株能裂解弗氏柠檬酸杆菌的噬菌体, 为治疗和控制弗氏柠檬酸杆菌提供物质基础, 而对大量的不同来源的噬菌体基因组进行组合分析可以窥探其携带耐药基因的信息。
基于此, 通过分析已经公布的噬菌体宏基因组去挖掘耐药基因成为可能。Subirats等[26]对医院污水样品进行噬菌体及细菌基因组测序, 基于抗生素作用机制分类, 用基因组方法分析噬菌体中ARG的丰度, 发现β-内酰胺酶基因丰度最高, 特别是A和D型(图 2)。同时对噬菌体群落及细菌群落构建生态网络关系, 为后续ARG的定位提供了技术支持, 这些结果间接证明了qPCR结果的可靠性。Lekunberri等[27]从NCBI数据库下载不同来源环境样品数据(表 2), 挖掘噬菌体中ARG及其丰度信息, 发现在这些序列中, 由于细菌DNA的污染, 细菌基因组中ARG的存在会导致噬菌体中ARG的丰度可能被高估。因此, 在所有组装的噬菌体基因组中Lekunbervi等[27]检测了16S rRNA基因的含量, 高于0.5%的基因组被排除, 结果表明:选取的所有人粪便、淡水和海水噬菌体基因组均符合条件(16S rRNA基因含量小于0.02%); 虽然有些猪粪便和污水中的病毒基因组具有相对高水平的潜在细菌污染(16S rRNA基因含量0.02%~0.50%), 但是也包括在分析中; 另外有2个猪粪便病毒基因组因为它们的高水平细菌基因特异性污染(16S rRNA基因含量大于1%)而被排除。
| 噬菌体基因组样品名称 Sample name of phage genome |
样品类型 Types |
样品来源 Origin |
测序后片段数 Number of sequencing reads |
| Barcelona RS | 未经处理污水Raw urban wastewater | 巴塞罗那Barcelona | 1 043 225 |
| Addis ababa RS | 未经处理污水Raw urban wastewater | 亚的斯亚贝巴Addis ababa | 640 054 |
| Pittsburgh RS | 未经处理污水Raw urban wastewater | 美国匹兹堡Pittsburgh(USA) | 662 246 |
| Sewage virome | 未经处理污水Raw urban wastewater | 美国密歇根Michigan(USA) | 31 915 |
| Sow C28ap | 猪粪Swine fecal | 德国Germany | 64 227 |
| Sow P28ap | 猪粪Swine fecal | 德国Germany | 55 502 |
| Sow C14pp | 猪粪Swine fecal | 德国Germany | 55 851 |
| Sow P14pp | 猪粪Swine fecal | 德国Germany | 38 955 |
| Piglet C12 | 猪粪Swine fecal | 德国Germany | 39 670 |
| Piglet P12 | 猪粪Swine fecal | 德国Germany | 26 115 |
| SARVir063005 | 海水Seawater | 马尾藻海Sargasso Sea | 412 745 |
| ArcticVir2002 | 海水Seawater | 北冰洋The Arctic Ocean | 376 909 |
| BBCVir96to04 | 海水Seawater | 哥伦比亚湾Columbia Bay | 435 584 |
| GOMVir94to01 | 海水Seawater | 墨西哥湾Gulf of Mexico | 302 449 |
| ElBerbera | 淡水Freshwater | 毛里塔尼亚Mauritani | 75 921 |
| Hamdoun | 淡水Freshwater | 毛里塔尼亚Mauritani | 39 404 |
| IIij | 淡水Freshwater | 毛里塔尼亚Mauritani | 62 059 |
| Molomhar | 淡水Freshwater | 毛里塔尼亚Mauritani | 75 709 |
| Montrose | 淡水Freshwater | 美国密歇根Michigan(USA) | 1 854 527 |
| 57th street | 淡水Freshwater | 美国密歇根Michigan(USA) | 2 202 471 |
| Montrose | 淡水Freshwater | 美国密歇根Michigan(USA) | 1 547 157 |
| Wilmette | 淡水Freshwater | 美国密歇根Michigan(USA) | 1 220 239 |
| Twins_ virome_pool1 | 人粪便Human fecal | 美国人American | 286 146 |
| Twins_ virome_pool2 | 人粪便Human fecal | 美国人American | 245 471 |
| Twins_ virome_pool3 | 人粪便Human fecal | 美国人American | 359 698 |
| Twins_ virome_pool4 | 人粪便Human fecal | 美国人American | 208 551 |
| hmp_Phage_GE66EV001 | 人粪便Human fecal | 美国人American | 30 873 |
| hmp_Phage_GE66EV002 | 人粪便Human fecal | 美国人American | 132 569 |
| hmp_Phage_GE66EV003 | 人粪便Human fecal | 美国人American | 61 104 |
| hmp_Phage_GE66EV004 | 人粪便Human fecal | 美国人American | 148 781 |
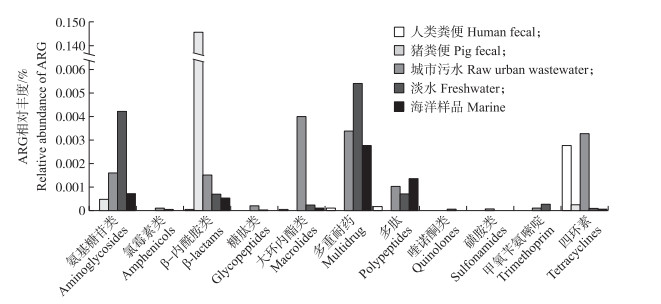
排除细菌污染后的结果显示, 人粪便病毒中ARG含量平均为0.003%, 在污水中为0.02%, 但猪粪中噬菌体基因组显示了相对高丰度的ARG(0.04%~0.45%)。对ARG多样性的统计结果表明, 水体及底泥样品噬菌体基因组有最高的ARG多样性, 猪粪中主要为编码β-内酰胺酶、氨基糖苷类和四环素类的耐药基因, 而人类粪便噬菌体基因组中ARG的丰度和多样性均较低, 仅编码四环素核糖体保护蛋白基因的丰度较高(图 3)。这个结果也恰好证实了最近的一项研究结论, 即人相关噬菌体基因组很少携带ARG[10]。从这些宏基因组分析结果来看, 水源性的噬菌体基因组中ARG的高丰度和多样性可能表明水体中噬菌体的多样性比我们想象的复杂得多, 其对ARG的传播可能起到了主要作用; 同时, 不同来源样品中的噬菌体基因组ARG主要以β-内酰胺酶抗性为主, 结果与qPCR检测结果一致, 表明结果的可靠性; 但不同来源环境噬菌体基因组携带喹诺酮类耐药基因几乎没有发现, 与qPCR检测结果不一致, 仍需要以后进行持续研究。
总而言之, 这些结果证明了来自不同环境的噬菌体基因组可作为ARG的另一重要存储库, 并且能够携带ARG进行水平传播这个令人信服的结论。因此, 针对噬菌体在自然界中协助ARG的获取和传播的作用不应该被低估。此外, 绝大多数的细菌和噬菌体在自然环境中的相互作用可以促进基因转移, 一些研究显示, 转导频率可能比我们以前认为的大几个数量级[28]。这些使噬菌体成为潜在的促进抗生素耐药性传播的载体, 因此在今后解决全球抗生素耐药性危机中应该重视和研究噬菌体在采集和动员ARG传播的机制。
3 结论与展望自然环境是一个巨大的耐药基因和耐药生物体的储库, 越来越多的证据也表明人类肠道微生物中ARG很可能来自环境介质中的耐药细菌[29]。qPCR方法证明了噬菌体可以作为载体传播ARG并在自然界中广泛分布; 而通过高通量测序产生的基因组数据则揭示了不同环境样品噬菌体基因组中耐药基因种类的多样性, 这些结果表明耐药基因不仅通过质粒而且还会通过噬菌体在这些生物之间发生了水平转移。因此, 针对噬菌体携带耐药基因转导传播的机制和控制策略应从以下几方面进一步深入研究。
第一, 明确畜禽养殖环境中不同耐药基因通过噬菌体传播的生态遗传背景。畜禽养殖圈舍及其周边环境(如池塘、菜地、处理池和周边河流)共同组成了微小核心体, 畜禽粪便排出后, 首先流向周边环境, 然后向更远区域扩散。而明确动物源环境中噬菌体基因组耐药基因及亚型的基因环境、生态分布和携带特性, 获得耐药基因通过噬菌体传播的生态遗传背景图谱是进一步研究其分子传播机制最重要的一环[30]。
第二, 选择最重要、流行最广的耐药基因优先进行深入的分子传播机制研究, 探究控制其传播的方法。虽然噬菌体可能作为耐药基因储存库并传播耐药基因, 但是其转导传播能力依然明显弱于细菌间的直接传播[27]。因此, 选择最优先等级耐药基因进行深入的分子传播机制研究, 可以更精准有效地为后期临床用药及控制策略服务, 同时丰富耐药性水平传播的理论。例如碳青霉烯类抗生素耐药基因(blaNDM和blaKPC)在噬菌体中的检出, 提示噬菌体在转导传播该基因的过程中作出了重要“贡献”, 而碳青霉烯类抗生素作为人医临床抗革兰阴性菌最有效的药物之一, 随着噬菌体基因组中碳青霉烯类抗生素耐药基因的检出, 由于噬菌体转导作用的介入将大大增强这些耐药基因的传播能力, 可能严重威胁该药的临床作用。因此, 针对碳青霉烯类抗生素耐药基因的噬菌体转导机制研究应该作为最优先等级尽快开展相关试验。
第三, 对接噬菌体治疗。研究噬菌体携带耐药基因的转导传播机制, 必然涉及到噬菌体相关的分离、培养及鉴定工作, 以此为依托可以快速开展噬菌体治疗相关的研究工作。噬菌体作为潜在抗生素佐剂[31-32], 有能力在将来服务于临床, 而噬菌体转导机制的研究又可以避免噬菌体治疗时的耐药基因再传播的可能, 一举两得。
总之, 噬菌体作为自然环境中最重要的生物体之一, 无论是从转导传播耐药基因的“负面影响”谈之, 还是潜在的作为抗生素佐剂治疗的“正面影响”谈之, 我们都应该对其深入研究早做准备。
| [1] | Liu Y Y, Wang Y, Walsh T R, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China:a microbiological and molecular biological study[J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168. DOI: 10.1016/S1473-3099(15)00424-7 |
| [2] | Walsh T R, Weeks J, Livermore D M, et al. Dissemination of NDM-1 positive bacteria in the New Delhi environment and its implications for human health:an environmental point prevalence study[J]. The Lancet Infectious Diseases, 2011, 11(5): 355-362. DOI: 10.1016/S1473-3099(11)70059-7 |
| [3] |
杨凤霞, 毛大庆, 罗义, 等. 环境中抗生素抗性基因的水平传播扩散[J].
应用生态学报, 2013, 24(10): 2993-3002.
Yang F X, Mao D Q, Luo Y, et al. Horizontal transfer of antibiotic resistance genes in the environment[J]. Chinese Journal of Applied Ecology, 2013, 24(10): 2993-3002. (in Chinese with English abstract) |
| [4] |
于帅, 李锦, 毛大庆, 等. 抗生素抗性基因在废(污)水处理系统的来源、传播扩散、归趋以及污染控制研究进展[J].
环境化学, 2013, 32(11): 2060-2071.
Yu S, Li J, Mao D Q, et al. Sources, dissemination, fate and pollution control of antibiotic resistance genes in wastewater(sewage)treatment system[J]. Environmental Chemistry, 2013, 32(11): 2060-2071. (in Chinese with English abstract) |
| [5] | Andersson D I, Hughes D. Antibiotic resistance and its cost:is it possible to reverse resistance?[J]. Nat Rev Microbiol, 2010, 8(4): 260-271. DOI: 10.1038/nrmicro2319 |
| [6] | Weinbauer M G. Ecology of prokaryotic viruses[J]. FEMS Microbiol Rev, 2004, 28(2): 127-181. DOI: 10.1016/j.femsre.2003.08.001 |
| [7] |
王盛, 童贻刚. 噬菌体治疗研究进展[J].
微生物学通报, 2009, 36(7): 1019-1024.
Wang S, Tong Y G. Recent advance in bacteriophage therapy[J]. Microbiology China, 2009, 36(7): 1019-1024. (in Chinese with English abstract) |
| [8] |
冯烨, 刘军, 孙洋, 等. 噬菌体最新分类与命名[J].
中国兽医学报, 2013, 33(13): 1954-1958.
Feng Y, Liu J, Sun Y, et al. An introduction to current classification and nomenclature of bacterial viruses[J]. Chinese Journal of Veterinary Science, 2013, 33(13): 1954-1958. (in Chinese with English abstract) |
| [9] | Del Casale A, Flanagan P V, Larkin M J, et al. Analysis of transduction in wastewater bacterial populations by targeting the phage-derived 16S rRNA gene sequences[J]. FEMS Microbiol Ecol, 2011, 76: 100-108. DOI: 10.1111/fem.2011.76.issue-1 |
| [10] | Enault F, Briet A, Bouteille L, et al. Phages rarely encode antibiotic resistance genes:a cautionary tale for virome analyses[J]. ISME J, 2017, 11(1): 237-247. DOI: 10.1038/ismej.2016.90 |
| [11] | Muniesa M, Colomer-Lluch M, Jofre J. Potential impact of environmental bacteriophages in spreading antibiotic resistance genes[J]. Future Microbiol, 2013, 8: 739-751. DOI: 10.2217/fmb.13.32 |
| [12] | Colomer-Lluch M, Imamovic L, Jofre J, et al. Bacteriophages carrying antibiotic resistance genes in fecal waste from cattle, pigs, and poultry[J]. Antimicrob Agents Chemother, 2011, 55(10): 4908-4911. DOI: 10.1128/AAC.00535-11 |
| [13] | Colomer-Lluch M, Jofre J, Muniesa M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples[J]. PLoS One, 2011, 6(3): e17549. DOI: 10.1371/journal.pone.0017549 |
| [14] | Calero-Caceres W, Melgarejo A, Colomer-Lluch M, et al. Sludge as a potential important source of antibiotic resistance genes in both the bacterial and bacteriophage fractions[J]. Environ Sci Technol, 2014, 48(13): 7602-7611. DOI: 10.1021/es501851s |
| [15] | Marti E, Variatza E, Balcazar J L. Bacteriophages as a reservoir of extended-spectrum beta-lactamase and fluoroquinolone resistance genes in the environment[J]. Clin Microbiol Infect, 2014, 20(7): 456-459. DOI: 10.1111/1469-0691.12446 |
| [16] | Quir S P, Colomer-Lluch M, Martnez C A, et al. Antibiotic resistance genes in the bacteriophage DNA fraction of human fecal samples[J]. Antimicrob Agents Chemother, 2014, 58: 606-609. DOI: 10.1128/AAC.01684-13 |
| [17] | Colomer-Lluch M, Jofre J, Muniesa M. Quinolone resistance genes(qnrA and qnrS)in bacteriophage particles from wastewater samples and the effect of inducing agents on packaged antibiotic resistance genes[J]. J Antimicrob Chemother, 2014, 69(5): 1265-1274. DOI: 10.1093/jac/dkt528 |
| [18] | Ross J, Topp E. Abundance of antibiotic resistance genes in bacteriophage following soil fertilization with dairy manure or municipal biosolids, and evidence for potential transduction[J]. Appl Environ Microbiol, 2015, 81(22): 7905-7913. DOI: 10.1128/AEM.02363-15 |
| [19] | Shousha A, Awaiwanont, Sofka D, et al. Bacteriophages isolated from chicken meat and the horizontal transfer of antimicrobial resistance genes[J]. Appl Environ Microbiol, 2015, 81(14): 4600-4606. DOI: 10.1128/AEM.00872-15 |
| [20] | Calero-Caceres W, Muniesa M. Persistence of naturally occurring antibiotic resistance genes in the bacteria and bacteriophage fractions of wastewater[J]. Water Res, 2016, 95: 11-18. DOI: 10.1016/j.watres.2016.03.006 |
| [21] | Calero-Caceres W, Mendez J, Martin-Diaz J, et al. The occurrence of antibiotic resistance genes in a Mediterranean river and their persistence in the riverbed sediment[J]. Environ Pollut, 2017, 223: 384-394. DOI: 10.1016/j.envpol.2017.01.035 |
| [22] | Lekunberri I, Villagrasa M, Balczar J L, et al. Contribution of bacteriophage and plasmid DNA to the mobilization of antibiotic resistance genes in a river receiving treated wastewater discharges[J]. Science of the Total Environment, 2017, 601/602: 206-209. DOI: 10.1016/j.scitotenv.2017.05.174 |
| [23] |
蔡文辉, 张文莉, 付英梅, 等. 内酰胺酶抑制剂研究进展[J].
中国抗生素杂志, 2013, 38(11): 805-809.
Cai W H, Zhang W L, Fu Y M, et al. Advances in β-lactamase inhibitors[J]. Chinese Journal of Antibiotics, 2013, 38(11): 805-809. DOI: 10.3969/j.issn.1001-8689.2013.11.002 (in Chinese with English abstract) |
| [24] |
崔泽林, 郭晓奎, 汤荣, 等. 基于基因组学的肺炎克雷伯菌噬菌体安全性评估[J].
基因组学与应用生物学, 2017, 36(30): 1114-1122.
Cui Z L, Guo X K, Tang R, et al. Safety assessment of Klebsiella pneumoniae phages based on genomics[J]. Genomics and Applied Biology, 2017, 36(30): 1114-1122. (in Chinese with English abstract) |
| [25] |
刘小东, 王伟, 黄勇, 等. 一株弗氏柠檬酸杆菌噬菌体IME_CF2的分离、测序及全基因组分析[J].
安徽医科大学学报, 2016, 51(3): 315-319.
Liu X D, Wang W, Huang Y, et al. Isolation, sequencing and complete genome sequence analysis of bacteriophage IME-CF2 infecting Citrobacter freundii[J]. Acta Universitatis Medicinalis Anhui, 2016, 51(3): 315-319. (in Chinese with English abstract) |
| [26] | Subirats J, Sanchez-melsio A, Borrego C M, et al. Metagenomic analysis reveals that bacteriophages are reservoirs of antibiotic resistance genes[J]. Int J Antimicrob Agents, 2016, 48(2): 163-167. DOI: 10.1016/j.ijantimicag.2016.04.028 |
| [27] | Lekunberri I, Subirats J, Borrego C M, et al. Exploring the contribution of bacteriophages to antibiotic resistance[J]. Environ Pollut, 2017, 220: 981-984. DOI: 10.1016/j.envpol.2016.11.059 |
| [28] | Muniesa M, Imamovic L, Jofre J. Bacteriophages and genetic mobilization in sewage and faecally polluted environments[J]. Microb Biotechnol, 2011, 4(6): 725-734. DOI: 10.1111/mbt.2011.4.issue-6 |
| [29] | Wright G D. Antibiotic resistance in the environment:a link to the clinic?[J]. Curr Opin Microbiol, 2010, 13(5): 589-594. DOI: 10.1016/j.mib.2010.08.005 |
| [30] |
窦春玲, 郭雪萍, 尹大强. 污水处理厂抗生素抗性基因分布和去除研究进展[J].
环境化学, 2013, 32(10): 1886-1893.
Dou C L, Guo X P, Yin D Q. Review on distribution and removal of antibiotic resistance genes(ARG)in wastewater treatment plants(WWTPs)[J]. Environmental Chemistry, 2013, 32(10): 1886-1893. (in Chinese with English abstract) |
| [31] | Carol P. Phage renaissance:new hope against antibiotic resistance[J]. Environ Health Perspect, 2013, 121: A49-A53. |
| [32] | Kingwell K. Bacteriophage therapies re-enter clinical trials[J]. Nat Rev Drug Discov, 2015, 14: 515-516. DOI: 10.1038/nrd4695 |