文章信息
- 周济, Tardieu Francois, Pridmore Tony, Doonan John, Reynolds Daniel, Hall Neil, Griffiths Simon, 程涛, 朱艳, 王秀娥, 姜东, 丁艳锋
- ZHOU Ji, Tardieu Francois, Pridmore Tony, Doonan John, Reynolds Daniel, Hall Neil, Griffiths Simon, CHENG Tao, ZHU Yan, WANG Xiu'e, JIANG Dong, DING Yanfeng
- 植物表型组学:发展、现状与挑战
- Plant phenomics:history, present status and challenges
- 南京农业大学学报, 2018, 41(4): 580-588
- Journal of Nanjing Agricultural University, 2018, 41(4): 580-588.
- http://dx.doi.org/10.7685/jnau.201805100
-
文章历史
- 收稿日期: 2018-05-31
2. Earlham Institute, Norwich Research Park, Norwich, NR4 7UZ, UK;
3. University of East Anglia, Norwich Research Park, Norwich, NR4 7TJ, UK;
4. INRA, University of Montpellier, LEPSE, 2 Place Viala, E34060 Montpellier, France;
5. University of Nottingham, Nottingham, NG7 2RD, UK;
6. Aberystwyth University, IBERS, Aberystwyth, SY23 3DA, UK;
7. John Innes Centre, Norwich Research Park, Norwich, NR4 7UH, UK
2. Earlham Institute, Norwich Research Park, Norwich, NR4 7UZ, UK;
3. University of East Anglia, Norwich Research Park, Norwich, NR4 7TJ, UK;
4. INRA University of Montpellier, LEPSE, 2 Place Viala, E 34060 Montpellier, France;
5. University of Nottingham, Nottingham, NG7 2RD, UK;
6. Aberystwyth University, IBERS, Aberystwyth, SY23 3DA, UK;
7. John Innes Centre, Norwich Research Park, Norwich, NR4 7UH, UK
在生命科学领域中, 表型(phenotype)一般指生物个体或群体, 在特定条件下(如各类环境和生长阶段等)所表现出的可观察的形态特征。生物学上的表型概念最早由丹麦遗传学家Wilhelm Johannsen在1911年提出, 他认为生物体的表型是基因型(genotype, 即生物体遗传构成)和环境因素(environmental factors)复杂交互的结果:基因型是表型得以表达的内因, 而环境是各类形态特征得以显现的外部条件[1]。20世纪90年代, 随着基因、基因表达产物及各类遗传关联分析研究的开展, 美国凯斯西储大学流行病学和生物统计学专家Nicholas Schork率先在疾病研究(disease research)中提出了与基因组学相对应的表型组学(phenomics)概念[2]。自此, 针对人、动物和植物单一或系列表型(phenome)的研究开始受到广泛关注[3], 并逐渐发展成生物学中的一个重要分支[4-5]。
植物表型研究始于20世纪末, 其核心是获取高质量、可重复的性状数据, 进而量化分析基因型和环境互作效应(genotype-by-environment interactions, GxE)及其对产量、质量、抗逆等相关的主要性状的影响[6-7]。相对于单一性状, 植物表型组能为植物研究提供全面的科学证据[8]。特别是伴随着基因组学的快速发展, 表型组学的理论基础和研究方法在过去10年间得到了极大的完善[9]。2011年, 澳大利亚植物表型研究所(Australian Plant Phenomics Facility, CSIRO)植物学家Robert Furbank在总结分析当时主流表型技术的基础上, 提出了著名的表型研究瓶颈问题, 包括多环境中对产量相关性状的多季重复测量、遥感技术在田间监测中的误差和关键表型的高通量分析等[10]。为解决这些问题, 世界上很多顶尖科研团队和商业机构开发了一系列高通量、高精度表型工具, 涵盖了环境传感(environmental sensor)、非侵入式成像(non-invasive imaging)、反射光谱(reflectance spectroscopy)、机器人技术(robotics)、机器视觉(computer vision)和高通量细胞表型筛选(high-content screening)等技术领域[10-12]。
上述技术方法显著促进了植物表型的研究进程。2012年, 西班牙巴塞罗那大学的作物学家Jose Luis Araus比较作物基因组选择和高通量表型组技术的研究应用后指出, 表型获取技术是整个作物育种中比较薄弱的环节, 所以需重点提高该技术的精度、通量, 降低价格成本[13]。美国亚利桑那州立大学Maricopa研究中心作物生理学家Jeffrey White和墨西哥国际玉米小麦改良中心(CIMMYT)小麦生理学家Matthew Reynolds分别总结了玉米和小麦育种中所使用的田间表型采集技术和表型分析方法(phenotypic analysis)[14-15], 为此后的田间表型研究建立了初步框架。这一时期的室内表型技术大都由商业机构推动, 其中最著名的当属德国LemnaTec公司的高通量室内植物表型筛分系统(Scanalyzer HTS)。该系统已被广泛应用于监测可控环境中的植物形态特征, 如通过比较拟南芥(Arabidopsis thaliana)表型差异进行突变体筛选[16]; 通过量化植株形态特征分析大麦对干旱反应的性状[17]; 在不同栽培条件下对玉米多个生长表型参数和产量表现进行分析等[18]。其他比较著名的室内表型平台还有捷克PSI公司(Photon Systems Instruments)的PlantScreenTM系统, 主要用于拟南芥和豌豆(Pisum sativum L.)等植物叶片叶绿素荧光成像和测量等[19-20]。
表型组学的发展与20世纪90年代的基因分型技术(genotyping)不断优化的发展轨迹十分相似。表型组学是跨学科的研究, 其理论核心、研究方法和标准规范的建立和完善需要由生物学家、计算机科学家、统计学家和软硬件工程师等共同推进[21-22]。因此, 康奈尔大学的植物分子遗传育种学家Susan McCouch于2013年提出了下一代表型研究(next-generation phenotyping)的概念[23]。她认为表型组研究应与高分辨率连锁图谱(high-resolution linkage mapping)、全基因组关联分析(genome-wide association studies)和基因组选择模型(genomic selection models)等技术紧密结合:通过建立强大的表型分型系统精确表征影响不同物种的细胞、器官和组织在不同发育阶段、不同年份、不同环境中表型差异的遗传学基础。此后, 多个致力于植物基础研究的团队也相继提出了系统化表型组学的研究策略, 即不单要从细胞到群体层面获取各类相关联的表型性状(traits), 更重要的是表型组研究要为揭示性状调控的分子机制(mechanism)和阐明基因功能(gene functions)提供大数据和决策支持[24-25]。
表型组理论的发展为表型组学技术的快速发展奠定了基础。近年来, 由于越来越多的植物和性状参数需要被快速和准确测量, 很多世界顶尖科研机构都把研究重心转移到诸如试验设计、定量分析和结论阐释等实际问题的解决等方面[18]。国际植物表型协会(International Plant Phenotyping Network, IPPN)主席、德国尤利希研究中心的植物学家Ulrich Schurr针对通量、扩展性和准确性等问题详细探讨了室内、外表型技术在作物遗传育种研究中的发展和应用现状[12]; 法国里昂大学的David Rousseau和英国诺丁汉大学的Tony Pridmore从计算机视觉角度介绍了植物成像、图像分析以及它们在定性表型分析中的应用价值[26]; 德国尤利希表型研究中心的Hanno Scharr基于高通量图像分析技术讨论了定量表型分析的设计方式和分析方法[27]; 德国莱布尼兹植物遗传遗传学与作物研究所(IPK)的分子遗传学家Thomas Altmann从生物试验设计角度出发介绍了室内、外表型技术在生物试验中的应用场景[18]。
2017年, 基于世界最新的表型研究成果, 法国植物表型协会主席、法国国家农业研究院(INRA)作物生理生态学家Francois Tardieu和诺丁汉大学植物学家Malcolm Bennett共同提出了多层次表型组(multi-scale phenomics)研究的构想[28]。他们指出表型组研究领域正在进入一个全新的发展阶段:如何把室内、外表型研究中产生的巨量图像和传感器数据转化为有意义的生物学知识将成为下一个表型组学研究的瓶颈。特别是针对与等位基因变异(allelic variants)和环境控制(environment control)紧密相关的动态性状, 只有革命性的数据处理和动态建模才能解决这一技术难题。图 1总结了当今国际植物表型界对表型组学研究进程的共识:为提高现代作物育种的选择速度、提高全球气候变化影响下作物的适应力和产量稳定性, 需要在完善大规模、高通量表型数据采集环节的自动化和遥感技术的基础上, 发展和利用全新的统计方法设计大数据生物试验[29]; 使用复杂的数据管理手段对表型数据集进行注释(data annotation)、标准化(data calibration)和存储(data storage)[30]; 基于本体论(ontology)进行数据的优化整合[31]; 引入最新的机器学习(machine learning)和深度学习(deep learning)等人工智能(artificial intelligence)方法对多维表型组数据集(multi-dimensional phenotypic datasets)进行分析[32]; 进而萃取可靠的性状特征信息(feature extraction of phenotypic information), 最终挖掘出有意义的生物学知识, 并解决实际的科学问题(knowledge mining)。
|
图 1 植物表型组学研究策略 Figure 1 The strategy of plant phenomics research |
现代多层次表型采集技术可以获取多维植物表型数据集, 而数据解析方法则致力于从数据集提取性状, 以获得高可信度、可重复的生物学结论[33]。根据不同的应用载体平台, 表型采集技术大致可分为手持、人载、车载、田间实时监控、大型室内外自动化平台、航空机载以及不同级别的卫星成像平台等。如图 2所示:由英国诺维奇科学研究院和南京农业大学植物表型组学研究中心周济实验室领导开发的多层次作物表型平台可以从空中、近地面以及田间等不同空间尺度进行作物表型分析, 实现了不同作物的研究目标。其中包括:通过装载三维激光成像仪(PlantEyeTM)的Phenospex田间表型平台[34]可以动态比较小麦近等基因系(near isogenic lines)的发育特征, 基于机器学习算法的SeedGerm系统可以大规模筛查种子萌发和出芽表型[35], 基于移动设备(如智能手机)拍摄的作物图像序列的开源软件Leaf-GP可以自动分析多个生长表型指标[36], 此外, 还有基于物联网技术(IoT)的分布式实时作物监测平台CropQuant[37]、农用田间表型机器人CropQuant-Robot及高通量航空图像分析平台AirSurf等。

|
图 2 英国诺维奇科学研究院使用的多层次作物表型平台 Figure 2 Multi-scale crop phenotyping platforms at Norwich Research Park, UK |
当然, 室内表型设施也是多层次表型研究中的重要组成部分。例如, 华中农业大学和华中科技大学共同开发的高通量室内水稻表型设施(high-throughput rice phenotyping facility, HRPF)[38], 德国IPK和浙江大学生命科学学院共同研发的基于LemnaTec系统的高通量植物生长和干旱反应分析系统[39]。本文将着重介绍讨论各类田间作物表型技术。
2.1 基于航空影像的表型技术(aerial plant phenotyping)基于航空影像的表型技术一般指在卫星、飞艇、直升机、固定翼微小型飞机和无人机(unmanned aerial vehicle, UAV)等飞行设备上装载近远程光学遥感成像仪来进行大规模田间表型采集。因为航空表型技术可以在短时间内对多个田间实验地块进行取样, 所以这类技术通常被应用于大规模作物育种和栽培试验。卫星影像[40]和软式飞艇[41]被用于研究不同作物品种在田间的生长规律和遗传差异; 改装后的载人直升机被应用于测量数以千计的田间小区的冠层温度和倒伏情况[42]; 各类无人机和固定翼微小型飞机被用于获取不同试验点的冠层结构变化信息等[43]。
航空表型技术在过去的5年中发展迅速, 其主要原因是由于飞行器和遥感硬件的成本下降、技术可靠性提高以及近远程传感设备性能的提升[33]。由于商业和开源图像处理软件的进步, 研究人员可以结合全球定位系统(global positioning system, GPS)和无人机影像信息来创建精确的正射影像图(orthomosaic map)[41], 用来计算分析植物覆盖率和光合作用[44]。近几年, 一些半专业的无人机(如大疆的DJI系列)已可以搭载价格相对低廉的高清可见光(red-green-blue, RGB)相机和轻型激光雷达(LiDAR), 通过重建高密度的三维点云(point clouds)获得植物高度和生长表型数据[45]; 专业无人机和固定翼飞机搭载价格不菲的多光谱(multispectral)、高光谱(hyperspectral)和热成像(thermal imaging)设备也被逐渐应用到对重要作物冠层性状(如氮素营养状况和冠层温度等)的高精度采样中[46]。
航空影像技术的高通量、大规模及相对经济的实验成本等特性使这项技术被大量应用于大规模田间表型数据的采集。但是, 这项技术需要在相对稳定的天气条件下进行操作:风力不能太强(风速通常要低于35 km·h-1), 不能在大雨、浓雾、云层较厚(特别是高空表型技术, 如卫星)等情况下运行。此外, 因为光谱设备对光照度要求很高, 不同时间段获取的图像信息可能会出现较大的偏差。这些因素影响了该技术在应用中的数据质量、使用频率及试验数据的连续性和可靠性。值得指出的是, 由于各国航空法和人员培训政策上的不断变化[41], 该技术在今后作物研究中的大规模应用存在一定的不确定性。此外, 海量航空影像数据的产生显著增加了科研人员在性状萃取和后续量化分析上的工作量和工作难度[28]。因此, 在应用推广这类表型技术时应特别注意并优先研发切实可行的后期数据解析方案。
2.2 田间移动和固定表型平台(ground-based phenotyping platforms)田间移动表型平台通常指将成像组件加装到现有农用车辆上后所构建的车载表型采集系统。例如, 在拖拉机上装载传感器(如彩色摄像机和激光距离扫描仪)后可以把拖拉机改装为移动式车载表型平台[47]; 单轮或多轮人力推车也可以被设计为田间表型装置来测量株高较低的农作物[48]; 在农用车辆上加装吊杆可以移动大型密闭舱, 加装人工光源来测量田间植株或小区的多光谱数据[49]; 通过车载自动化机械臂可以控制热红外相机和低成本的LiDAR相机来获取田间表型数据[50]。
使用上述车载表型平台的最大问题是传感器校准和数据预处理。由于不同传感器组(sensor group)在不同试验点和环境条件下的差异很大, 校准传感器组来保证多区域数据的可靠性和可比性至今仍是一项艰巨的任务。如果需要在整个生长季节中连续获取不同监测点的作物图像和传感器数据, 获取的表型数据上必须添加试验元数据(如时间、空间坐标和各类试验参数等)。车载表型设备不但要加装复杂的控制系统, 还要由训练有素的专业人员来操作。因此, 车载表型技术代价相对高昂, 实际应用中也有较多的限制[51]。
机器人技术和智能机器人的研发可以在一定程度上改善这一技术问题[52]。例如, 专为野外表型分析而设计的全自动机器人通过使用多个GPS系统互作而获得厘米级的精确定位, 加装模块化传感器的机器人可以完成一些基于图像的表型采集任务[53]。植入智能图像分析系统的简易机器人已被用来分析葡萄育种中的浆果外形和颜色的性状鉴定[48]。这类移动式表型机器人逐渐成为田间移动表型技术发展的新方向。但是无论是车载还是机器人系统, 共同的问题是难以在短时间内完成高频率、多地点的表型采集。例如, 捕获开花期中瞬息即逝的花药从花中挤压出性状(anther extrusion), 至今没有合适的智能技术手段来实现从发现性状、移动至观察点、选择合适设备到采集表型数据的监测过程。
为了解决车载和机器人系统的技术缺陷, 近几年作物研究中使用了配备有价格高昂的近红外三维激光扫描仪、多光谱或高光谱等传感仪的大型轨道式田间表型监测平台。其中比较著名的系统有苏黎世联邦理工学院(ETH Zürich)的Field Phenotyping Platform(FIP)[34]、Phenospex公司的Field Scanner[54]和LimnaTec公司的Field Scanalyzer平台[55]。它们通过程控系统对固定数量的植株或小区进行长时间、多次数(一般每天8~12次)的扫描监测。因为大型轨道平台可以带动沉重的高精度监测仪, 这类技术又被称作深度植物表型技术(deep plant phenotyping)[56]。近年来, 英国的洛桑实验站(Rothamsted Research)、诺维奇科研院的约翰英纳斯中心(John Innes Centre)和厄尔汉姆研究中心(Earlham Institute)、苏黎世联邦理工学院、美国的Maricopa研究中心、南京农业大学植物表型组学研究中心等都建设了这类表型平台, 为育种项目中研究作物生长和环境互作等提供大规模的田间表型数据。由于这类系统需要定制、价格昂贵(至少需要千万元的硬件和基建投资), 同时需要建立一支团队在安装、调试、运行、保养和后期分析上提供技术支持, 因此这类平台一般很难在多地点的大型育种和栽培项目中使用。而且, 田间轨道式表型平台至今仍有多个技术难关未能攻克, 比如多个激光仪在风、雨条件下的点云采集、三维校准和高质量植物的精确重建等。特别需要指出的是, 由于大型轨道表型平台通常依靠专门的商业软件来完成硬件调控、数据管理和性状分析, 且商业公司对自身知识产权的保护(intellectural property), 因此限制了科研人员根据各自科学问题对平台和分析软件的二次开发和技术推广。
2.3 田间手持和分布式表型设备(handheld and distributed phenotyping devices)小型手持设备在提高田间表型数据采集灵活性的基础上大大降低了采集硬件的成本。使用数码相机和三维重建软件可以通过完全开源(open source)的运动恢复结构技术(structure from motion, SfM)和多视点立体视觉技术(multi-view stereo, MVS)对数十幅二维图像序列进行植物形态的三维重建[57]; 使用立体相机装置(stereo imaging rig)可以对三维冠层和叶片空间结构进行分析[58]; 通过安装在手持杆上的笔记本电脑可以调控红外线温度等传感器计算归一化植被指数(normalized difference vegetation index, NDVI)[50]; 在小型手持设备中集成发光二极管(LED)和可见光及红外传感器等可以测量叶面透光率与光合效率效能等关键参数[59]。
手持表型装置的缺陷是人工和软件成本相对偏高。为了获取高质量、可重复的表型数据集, 该技术需要通过手动方式来选择合适的采集区域, 然后使用分析软件标准化数据来提高数据的可靠性。虽然手持表型设备通常简单易用, 它仍需要具有一定生物和技术背景的人员进行操作。因此, 人工测量的规模和通量一般较小, 而且操作者必须遵循标准化的流程来完成采集任务。
针对现代高频率、多地点、标准化的表型采集需求, 通过集成低成本传感器建立的分布式全生长季田间表型监测平台的重要性开始显现。比如, 基于“开放硬件”设计(open hardware design)思想的经济型、模块化的电子仪, 通过制造独立运行的田间小型工作站来完成小区的作物生长及微环境(micro-environment)的测量[60]; 通过通用分组无线业务(general packet radio service, GPRS)或者无线通讯(radio transmission)的传输模式, 可以完成多地点的数据互联和校验[61]。在集成多个低成本传感器的基础上, 日本东京大学开发了开放式田间服务器(open field server, OpenFS), 并通过网络云服务来实现对植物在不同环境下的长期观察[62]。
这类技术还包括由英国诺维奇科学研究院开发的基于IoT技术的分布式作物表型监测平台CropQuant[37]。通过单板电脑(如树莓派电脑、Raspberry Pi Computers和英特尔的Galileo系列)来调控多个低成本环境传感器, 对田间气候和土壤信息(如土壤含水量和温度)进行连续测量。使用二次开发的成像软件可以动态控制对田间作物的连续拍摄(如根据不同光照条件自动调整成像模式), 同时在田间可以使用基于Python的开源图像分析库[63]来完成初步的表型分析, 从而实现对作物全生育期关键性状的高通量、高频率表型分析。由于微型单板机在外接无线适配器后可建立可扩展的网状网络(expandable mesh network), 因此多地点的植物生长表型和气候数据均可通过多工作站的物联网互联完成校准。最后, 性状和环境数据可以输出至嵌入式神经元网络(embedded neural network)进行机器学习, 以此来分析和预测作物生长和环境互作效应。多年的作物田间数据亦可用于作物生长模型的建立, 预测不同作物品种或不同基因型之间的生长差异, 提供作物精确管理和农业生产中的智能化决策。值得指出的是, 田间分布式表型技术面临的主要问题是如何完成大规模田间工作站的生产、安装、运行、维护及后期大数据的综合分析。
2.4 表型组数据解析(phenotypic analysis)无论是固定式(如田间分布式和固定式大型表型平台)还是移动式(航空、田间移动、手持式表型设备)的表型系统, 如何在特定的时间点或时间段内获取研究人员感兴趣的性状是最重要的表型采集任务。然而, 如果不能把各类表型采集技术获取的大规模数据(以数万亿字节计, terabytes data)转化为研究人员可以理解的信息(以数千字节计, kilobytes data), 那无论采用什么样的技术平台都是没有实际意义的。因此, 数据解析在表型组学研究中的重要性不言而喻。
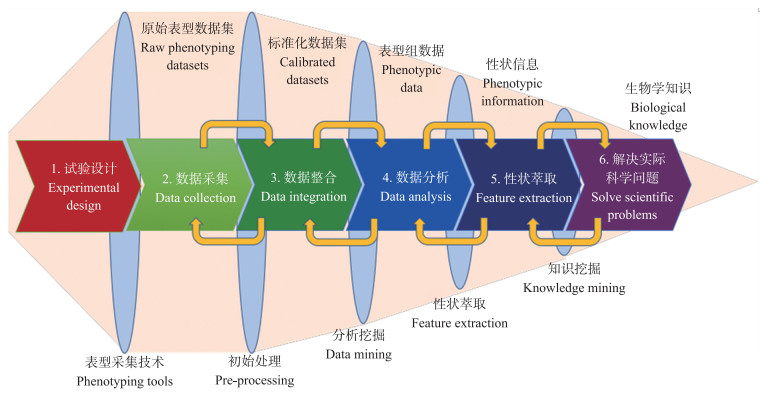
表型组数据包括可视图像数据、传感器数据及试验元数据等多种数据集, 因此数据解析实际包含从最初数据采集到最终细化分析的整个过程。图 3展示了田间表型研究各阶段的数据解析环节, 可分为数据采集、数据整合和数据分析3部分。在数据采集环节中, 需要考虑各试验点和各种试验方法间的数据预处理, 包括基本数据删选、数据校准和多试验的数据交叉验证(cross reference); 在数据整合环节中, 需要考虑对包含表型图像、环境传感、试验设计和各种元数据的多维数据集的整合, 以此分类管理采集的多维表型组数据集; 而在分析环节, 需重点考虑分析结果的可视化表现(data visualisation)、各类性状在不同试验条件下的对比、GxE动态互作分析、生长预测建模以及模型与实际性状之间的差异及原因分析等。

|
图 3 田间表型采集、表型数据整合及表型组数据解析过程 Figure 3 The analytic procedure in plant phenomics research, including multi-scale field phenotyping, multi-dimensional data integration, and phenotypic analysis |
随着各类计算机视觉算法、图像处理和机器学习分类方法(classification methods)在表型数据解析中的应用[64-67], 许多商业或科研团队研发了各类自动分析管线软件(analysis pipeline software)对植物大小、形状、生长动态等重要性状进行提取。由商业机构研发的软件包一般针对特定的硬件系统或分析任务而设计, 因此很难被广泛应用。通过使用开源软件库如OpenCV[63]、SciKit-Image[68]、Scikit-Learn[69]和TensorFlow[70]等设计开发的自动化表型分析流程, 有望解决表型组学研究中的数据解析瓶颈。研发这类分析流程需要开发者或开发团队具备数学、算法设计、软硬件开发、生命科学等多方面的专业背景, 因此需要多个科研团队之间的学科交叉和长期合作。从长远发展考虑, 我国的科研体系也需要针对性培养可引领世界的跨学科综合性人才, 为将来在育种、栽培、功能和调控机制等方面的科研工作提供技术和人才的储备。
3 植物表型组学的展望表型组学是突破未来作物学研究和应用的关键研究领域, 通过表型分析来描述关键性状可以为育种、栽培和农业实践提供基于大数据的决策支持。但是, 没有针对性的全表型标记在植物研究领域中是没有太多实际意义的。即使只针对某一个物种, 全表型测量会产生海量、无重点的数据集。在今后相当长的时间内, 数据解析方法很难从这类数据集中高效地总结共性规律, 并提取有生物学意义的信息。对于希望在植物研究中使用表型组学的团队而言, 这样的科研方式也是比较低效的。因此, 表型组研究需要明确的目标是, 在快速筛选关键性状集的基础上, 为针对性解决科学问题提供决策数据, 体现研究的实际应用价值。需重点指出的是, 表型组学研究不等同于表型仪器的购置。各类表型设备只是完成了数据采集工作, 而真正对生物学有意义的表型组信息需要由跨学科团队的紧密合作来完成。无论是利用F1/F2等分离群体开展植物数量性状位点(quantitative trait locus)分析, 还是利用近等基因系等开展基因克隆研究, 选择表型设备和分析手段应明确目的, 即为更好解决具体的生物学问题服务。因此, 认为只要购买了先进表型仪器就能完成整个性状测量、数据分析和生物知识构建等, 这是对表型组学研究的误解。
表型组学的巨大潜力还体现在其与其他组学研究(omics research, 如基因组学、表观组学、转录组学、蛋白组学、代谢组学和离子组学等)知识的综合分析[71], 量化分析特定表型的遗传规律, 对作物细胞、器官、群体不同层次的监控, 作物在不同发育阶段的动态性状的获取, 并与其他组学分析结果融合, 可对重要生命过程进行多方位的解释。中国幅员辽阔, 各地区实际情况差别较大, 有必要针对不同生态环境中的作物表型组和其他组学数据进行整合和交叉验证, 揭示农业植物生物学规律, 切实支撑中国各类作物的生理学、发育学、遗传学、育种、栽培以及农业大田生产等研究, 提升中国作物遗传育种、栽培管理和农业生产服务能力。
| [1] | Johannsen W. The genotype conception of heredity[J]. Int J Epidemiol, 2014, 43(4): 989-1000. DOI: 10.1093/ije/dyu063 |
| [2] | Schork N J. Genetics of complex disease:approaches, problems, and solutions[J]. Am J Respir Crit Care Med, 1997, 156: S103-S109. DOI: 10.1164/ajrccm.156.4.12-tac-5 |
| [3] | Siebner H R, Callicott J H, Sommer T, et al. From the genome to the phenome and back:linking genes with human brain function and structure using genetically informed neuroimaging[J]. Neuroscience, 2009, 164(1): 1-6. DOI: 10.1016/j.neuroscience.2009.09.009 |
| [4] | Bilder R M, Sabb F W, Cannon T D, et al. Phenomics:the systematic study of phenotypes on a genome-wide scale[J]. Neuroscience, 2009, 164(1): 30-42. DOI: 10.1016/j.neuroscience.2009.01.027 |
| [5] | Houle D, Govindaraju D R, Omholt S. Phenomics:the next challenge[J]. Nat Rev Genet, 2010, 11(12): 855-866. DOI: 10.1038/nrg2897 |
| [6] | Tester M, Langridge P. Breeding technologies to increase crop production in a changing world[J]. Science, 2010, 327(5967): 818-822. DOI: 10.1126/science.1183700 |
| [7] | Ribaut J M, de Vicente M C, Delannay X. Molecular breeding in developing countries:challenges and perspectives[J]. Curr Opin Plant Biol, 2010, 13(2): 213-218. |
| [8] | Finkel E. With 'Phenomics' plant scientists hope to shift breeding into overdrive[J]. Science, 2009, 325(5939): 380-381. DOI: 10.1126/science.325_380 |
| [9] | Bevan M W, Uauy C, Wulff B B H, et al. Genomic innovation for crop improvement[J]. Nature, 2017, 543(7645): 346-354. DOI: 10.1038/nature22011 |
| [10] | Furbank R T, Tester M, Berry S, et al. Phenomics:technologies to relieve the phenotyping bottleneck[J]. Trends Plant Sci, 2011, 16(12): 635-644. DOI: 10.1016/j.tplants.2011.09.005 |
| [11] | McCoy J P. High-content screening:getting more from less[J]. Nat Methods, 2011, 8(5): 390-391. DOI: 10.1038/nmeth.1599 |
| [12] | Fiorani F, Schurr U. Future scenarios for plant phenotyping[J]. Annu Rev Plant Biol, 2013, 64: 267-291. DOI: 10.1146/annurev-arplant-050312-120137 |
| [13] | Cabrera-Bosquet L, Crossa J, von Zitzewitz J, et al. High-throughput phenotyping and genomic selection:the frontiers of crop breeding converge[J]. J Integr Plant Biol, 2012, 54(5): 312-320. DOI: 10.1111/jipb.2012.54.issue-5 |
| [14] | White J W, Andrade-Sanchez P, Gore M A, et al. Field-based phenomics for plant genetics research[J]. Fild Crop Res, 2012, 133: 101-112. DOI: 10.1016/j.fcr.2012.04.003 |
| [15] | Pask A, Pietragalla J, Mullan D, et al. Physiological Breeding Ⅱ:A Field Guide to Wheat Phenotyping[M]. Mexico: CIMMYT, 2012. |
| [16] | Arvidsson S, Pérez-Rodríguez P, Mueller-Roeber B. A growth phenotyping pipeline for Arabidopsis thaliana integrating image analysis and rosette area modeling for robust quantification of genotype effects[J]. New Phytol, 2011, 191(3): 895-907. DOI: 10.1111/nph.2011.191.issue-3 |
| [17] | Chen D, Neumann K, Friedel S, et al. Dissecting the phenotypic components of crop plant growth and drought responses based on high-throughput image analysis[J]. Plant Cell, 2014, 26(12): 4636-4655. DOI: 10.1105/tpc.114.129601 |
| [18] | Junker A, Muraya M M, Weigelt-Fischer K, et al. Optimizing experimental procedures for quantitative evaluation of crop plant performance in high throughput phenotyping systems[J]. Front Plant Sci, 2014, 5: 770. |
| [19] | Bourdais G, Burdiak P, Gauthier A, et al. Large-scale phenomics identifies primary and fine-tuning roles for crks in responses related to oxidative stress[J]. PLoS Genet, 2015, 11: 1-36. |
| [20] | Humplík J F, Lazár D, Fürst T, et al. Automated integrative high-throughput phenotyping of plant shoots:a case study of the cold-tolerance of pea(Pisum sativum L.)[J]. Plant Methods, 2015, 11: 1-11. DOI: 10.1186/s13007-015-0043-0 |
| [21] | Goff S A, Vaughn M, McKay S, et al. The iPlant collaborative:cyberinfrastructure for plant biology[J]. Front Plant Sci, 2011, 2: 34. |
| [22] | McCouch S R, Baute G J, Bradeen J, et al. Feeding the future[J]. Nature, 2013, 499(7456): 23-24. DOI: 10.1038/499023a |
| [23] | Cobb J N, Declerck G, Greenberg A, et al. Next-generation phenotyping:requirements and strategies for enhancing our understanding of genotype-phenotype relationships and its relevance to crop improvement[J]. Theor Appl Genet, 2013, 126(4): 867-887. DOI: 10.1007/s00122-013-2066-0 |
| [24] | Cardona A, Tomancak P. Current challenges in open-source bioimage informatics[J]. Nat Methods, 2012, 9: 661-665. DOI: 10.1038/nmeth.2082 |
| [25] | Fritsche-Neto R, Borém A. Phenomics:How Next-Generation Phenotyping is Revolutionizing Plant Breeding[M]. Basel: Springer International Publishing Switzerland, 2015. |
| [26] | Rousseau D, Dee H, Pridmore T. Imaging methods for phenotyping of plant traits[M]//Kumar J. Phenomics in Crop Plants: Trends, Options and Limitations. New Delhi: Springer India, 2015: 61-74. |
| [27] | Scharr H, Dee H, French A P, et al. Special issue on computer vision and image analysis in plant phenotyping[J]. Mach Vis Appl, 2016, 27: 607-609. DOI: 10.1007/s00138-016-0787-1 |
| [28] | Tardieu F, Cabrera-Bosquet L, Pridmore T, et al. Plant phenomics, from sensors to knowledge[J]. Curr Biol, 2017, 27: R770-R783. DOI: 10.1016/j.cub.2017.05.055 |
| [29] | Rahaman M M, Chen D, Gillani Z, et al. Advanced phenotyping and phenotype data analysis for the study of plant growth and development[J]. Front Plant Sci, 2015, 6: 619. |
| [30] | Shrestha R, Matteis L, Skofic M, et al. Bridging the phenotypic and genetic data useful for integrated breeding through a data annotation using the crop ontology developed by the crop communities of practice[J]. Front Physiol, 2012, 3: 326. |
| [31] | Li Y F, Kennedy G, Ngoran F, et al. An ontology-centric architecture for extensible scientific data management systems[J]. Futur Gener Comput Syst, 2013, 29: 641-653. DOI: 10.1016/j.future.2011.06.007 |
| [32] | Libbrecht M W, Noble W S. Machine learning applications in genetics and genomics[J]. Nat Rev Genet, 2015, 16(6): 321-332. DOI: 10.1038/nrg3920 |
| [33] | Tardieu F, Simonneau T, Muller B. The physiological basis of drought tolerance in crop plants:a scenario-dependent probabilistic approach[J]. Annu Rev Plant Biol, 2018, 69: 733-759. DOI: 10.1146/annurev-arplant-042817-040218 |
| [34] | Kirchgessner N, Liebisch F, Yu K, et al. The ETH field phenotyping platform FIP:a cable-suspended multi-sensor system[J]. Funct Plant Biol, 2017, 44: 154-168. DOI: 10.1071/FP16165 |
| [35] | Benjamins R, Lanfermeijer F, Westland B, et al. Automated germination analysis[C]//Syngenta ISSS Monterey, 2017. |
| [36] | Zhou J, Applegate C, Alonso A D, et al. Leaf-GP:an open and automated software application for measuring growth phenotypes for Arabidopsis and wheat[J]. Plant Methods, 2017, 13: 1-30. DOI: 10.1186/s13007-016-0152-4 |
| [37] | Zhou J, Reynolds D, Corn T L, et al. CropQuant:an automated and scalable field phenotyping platform for crop monitoring and trait measurements to facilitate breeding and digital agriculture[J]. bioRxiv, 2017. DOI: 10.1101/161547 |
| [38] | Yang W, Guo Z, Huang C, et al. Combining high-throughput phenotyping and genome-wide association studies to reveal natural genetic variation in rice[J]. Nat Commun, 2014, 5: 5087. DOI: 10.1038/ncomms6087 |
| [39] | Chen D, Neumann K, Friedel S, et al. Dissecting the phenotypic components of crop plant growth and drought responses based on high-throughput image analysis[J]. Plant Cell, 2014, 26(12): 4636-4655. DOI: 10.1105/tpc.114.129601 |
| [40] | Zaman-Allah M, Vergara O, Araus J L, et al. Unmanned aerial platform-based multi-spectral imaging for field phenotyping of maize[J]. Plant Methods, 2015, 11: 35. DOI: 10.1186/s13007-015-0078-2 |
| [41] | Sankaran S, Khot L R, Espinoza C Z, et al. Low-altitude, high-resolution aerial imaging systems for row and field crop phenotyping:a review[J]. Eur J Agron, 2015, 70: 112-123. DOI: 10.1016/j.eja.2015.07.004 |
| [42] | Chapman S C, Merz T, Chan A J, et al. Pheno-Copter:a low-altitude, autonomous remote-sensing robotic helicopter for high-throughput field-based phenotyping[J]. Agronomy, 2014, 4(2): 279-301. DOI: 10.3390/agronomy4020279 |
| [43] | Jonckheere I, Fleck S, Nackaerts K, et al. Review of methods for in situ leaf area index determination:part Ⅰ. Theories, sensors and hemispherical photography[J]. Agric For Meteorol, 2004, 121(1): 19-35. |
| [44] | Gracia-Romero A, Kefauver S C, Vergara-Diaz O, et al. Comparative performance of ground versus aerially assessed RGB and multispectral indices for early-growth evaluation of maize performance under phosphorus fertilization[J]. Front Plant Sci, 2017, 8: 2004. DOI: 10.3389/fpls.2017.02004 |
| [45] | Watanabe K, Guo W, Arai K, et al. High-throughput phenotyping of sorghum plant height using an unmanned aerial vehicle and its application to genomic prediction modeling[J]. Front Plant Sci, 2017, 8: 1-11. |
| [46] | Verger A, Vigneau N, Cheeron C, et al. Green area index from an unmanned aerial system over wheat and rapeseed crops[J]. Remote Sens Environ, 2014, 152: 654-664. DOI: 10.1016/j.rse.2014.06.006 |
| [47] | Stark B, Zhao T, Chen Y. An analysis of the effect of the bidirectional reflectance distribution function on remote sensing imagery accuracy from Small Unmanned Aircraft Systems[C]//International Conference on Unmanned Aircraft Systems(ICUAS), Arlingto, USA, 2016. |
| [48] | Cendrero-Mateo M P, Muller O, Albrecht H, et al. Field Phenotyping:Challenges and Opportunities[M]. Terrestrial Ecosystem Research Infrastructures, Boca Raton: CRC Press, 2017, 53-80. |
| [49] | Svensgaard J, Roitsch T, Christensen S. Development of a mobile multispectral imaging platform for precise field phenotyping[J]. Agronomy, 2014, 4(3): 322-336. DOI: 10.3390/agronomy4030322 |
| [50] | Crain J L, Wei Y, Barker J, et al. Development and deployment of a portable field phenotyping platform[J]. Crop Sci, 2016, 56(3): 965-975. DOI: 10.2135/cropsci2015.05.0290 |
| [51] | Reina G, Milella A, Rouveure R, et al. Ambient awareness for agricultural robotic vehicles[J]. Biosyst Eng, 2016, 146: 114-132. DOI: 10.1016/j.biosystemseng.2015.12.010 |
| [52] | Nielsen S H, Jensen K, Jorgensen Q J, et al. A low cost, modular robotics tool carrier for precision agriculture research[C]//11th International Conference on Precision Agriculture, Indiana, USA, 2012. |
| [53] | Grimstad L, From P. The thorvald Ⅱ agricultural robotic system[J]. Robotics, 2017, 6(4): 24. DOI: 10.3390/robotics6040024 |
| [54] | Vadez V, Kholová J, Hummel G, et al. LeasyScan:a novel concept combining 3D imaging and lysimetry for high-throughput phenotyping of traits controlling plant water budget[J]. J Exp Bot, 2015, 66(18): 5581-5593. DOI: 10.1093/jxb/erv251 |
| [55] | Virlet N, Sabermanesh P, Sadeghitehran K, et al. Field Scanalyzer:an automated robotic field phenotyping platform for detailed crop monitoring[J]. Funct Plant Biol, 2017, 44: 143-153. DOI: 10.1071/FP16163 |
| [56] | Hawkesford M J, Lorence A. Plant phenotyping:increasing throughput and precision at multiple scales[J]. Funct Plant Biol, 2017, 44: 5-7. |
| [57] | Rose J C, Paulus S, Kuhlmann H. Accuracy analysis of a multi-view stereo approach for phenotyping of tomato plants at the organ level[J]. Sensors, 2015, 15: 9651-9665. DOI: 10.3390/s150509651 |
| [58] | Müller-Linow M, Pinto-Espinosa F, Scharr H, et al. The leaf angle distribution of natural plant populations:assessing the canopy with a novel software tool[J]. Plant Methods, 2015, 11: 11. DOI: 10.1186/s13007-015-0052-z |
| [59] | Kuhlgert S, Austic G, Zegarac R, et al. MultispeQ Beta:a tool for large-scale plant phenotyping connected to the open PhotosynQ network[J]. R Soc Open Sci, 2016, 3(10): 160592. DOI: 10.1098/rsos.160592 |
| [60] | Millet E, Welcker C, Kruijer W, et al. Genome-wide analysis of yield in Europe:allelic effects as functions of drought and heat scenarios[J]. Plant Physiol, 2016, 172(2): 749-764. |
| [61] | Villarrubia G, de Paz J F, de Iglesia D H L, et al. Combining multi-agent systems and wireless sensor networks for monitoring crop irrigation[J]. Sensors, 2017, 17(8): 1775. DOI: 10.3390/s17081775 |
| [62] | Hirafuji M, Yoichi H. Creating high-performance/low-cost ambient sensor cloud system using OpenFS(open field server)for high-throughput phenotyping[C]//SICE Annual conferencer, Tokyo, Japan, 2011. |
| [63] | Rosebrock A. Practical Python and OpenCV[M]. New York: Pyimageseach, 2015. |
| [64] | Tsaftaris S A, Minervini M, Scharr H. Machine learning for plant phenotyping needs image processing[J]. Trends in Plant Science, 2016, 21(12): 989-991. DOI: 10.1016/j.tplants.2016.10.002 |
| [65] | Pound M P, Atkinson J A, Townsend A J, et al. Deep machine learning provides state-of-the-art performance in image-based plant phenotyping[J]. Gigascience, 2017, 6(10): 1-10. |
| [66] | Eliceiri K, Berthold M, Goldberg I, et al. Biological imaging software tools[J]. Nat Methods, 2012, 9(7): 697-710. DOI: 10.1038/nmeth.2084 |
| [67] | King A. Technology:the future of agriculture[J]. Nature, 2017, 544(7651): S21-S23. DOI: 10.1038/544S21a |
| [68] | van der Walt S, Schönberger J L, Nunez-Iglesias J, et al. Scikit-image:image processing in python[J]. PeerJ, 2014, 2: e453. DOI: 10.7717/peerj.453 |
| [69] | Pedregosa F, Varoquaux G, Gramfort A, et al. Scikit-learn:machine learning in python[J]. J Mach Learn Res, 2011, 12: 2825-2830. |
| [70] | Rosebrock A. Deep Learning for Computer Vision with Python[M]. New York: Pyimageseach, 2017. |
| [71] | Lin E, Lane H Y. Machine learning and systems genomics approaches for multi-omics data[J]. Biomark Res, 2017, 5: 2. DOI: 10.1186/s40364-017-0082-y |




