文章信息
- 林达, 洪萍, 李国亮, 曹罡
- LIN Da, HONG Ping, LI Guoliang, CAO Gang
- DLO Hi-C染色体构象捕获技术
- Digestion-ligation-only Hi-C(DLO Hi-C)for chromosome conformation capture
- 南京农业大学学报, 2018, 41(4): 577-579
- Journal of Nanjing Agricultural University, 2018, 41(4): 577-579.
- http://dx.doi.org/10.7685/jnau.201806031
-
文章历史
- 收稿日期: 2018-06-21
2. 华中农业大学作物遗传改良国家重点实验室, 湖北 武汉 430070
2. National Key Laboratory of Crop Genetic Improvement, Huazhong Agricultural University, Wuhan 430070, China
自1993年人类基因组计划实施到今天, 基因组学的发展经历了2个重要阶段。第1阶段是以人类基因组计划为代表, 该计划采用经典的Sanger测序技术对人类基因组进行了测序, 定义了人类基因组的线性序列, 同时极大地促进了测序技术的发展, 开启了基因组学时代; 第2阶段以“人类基因组DNA元件百科全书计划”为代表, 该计划延续了人类基因组计划, 对人类基因组线性序列进行了解读, 发现了大量的基因转录调控元件, 并对这些转录调控元件进行了系统的注释[1]。
很多基因组的转录调控元件, 如增强子、沉默子等, 都是离散地分布在全基因组中, 这些调控元件与目的基因往往距离很远, 调控机制很难得到较好的解释, 三维基因组学就是为了解决这个难以回答的问题而孕育出来的一门学科[2]。三维基因组学主要想解释的问题是:基因组中的染色体是如何进行折叠的, 基因组上的远距离调控元件是如何发生相互作用的。对基因组三维结构的研究也预示着基因组学第3次发展浪潮的到来[3]。
Hi-C技术[4]是研究基因组三维结构的主要工具之一, 它是基于经典的染色体构象捕获技术(chromosome conformation capture, 3C)发展而来的。经典3C技术是先用甲醛固定细胞中的染色质, 然后进行内切酶酶切后DNA再连接, 这样就能将空间接近的序列片段聚拢在一起, 之后通过测序分析这些连接处的特征, 获得1张详细的染色质互作图谱。但3C技术聚焦于2个特定位点之间的互作, 最终结果是基于观测特异性扩增片段的有无, 因此仅能“一对一”地研究基因组中某一特定DNA片段在染色质中与另一特定DNA片段的相互作用。
随着高通量测序技术和生物信息学的发展, 人们希望寻求一种方法能够克服3C技术通量低的缺陷, 在全基因组的范围内分析染色体互作位点。2009年马萨诸塞大学的Job Dekker研究团队首次提出Hi-C的概念。它以整个细胞核为研究对象, 运用分子标记与新一代测序(NGS)技术, 研究整个染色质中DNA在空间位置上的关系, 通过对染色质内全部DNA的相互作用模式进行捕获, 来获得高分辨率的染色质三维结构信息。
Hi-C主要包括以下步骤:6碱基或4碱基内切酶酶切固定后的染色质; 内切酶切口补平、生物素标记; 邻近DNA片段末端连接; 未连接的DNA片段末端去生物素; DNA纯化以及超声波打断; 链霉亲和素磁珠特异性分离纯化DNA; DNA片段末端修补以及测序接头连接; 高通量测序分析。
自Hi-C技术提出以来, 已经有了很多的应用[5-6], 但是其自身存在较多缺陷, 其中包括:试验成本高, 需要生物素标记以及链霉亲和素磁珠选择吸附; 需要很大的测序量, 测序成本高, 不是一般实验室所能承受的; 噪音大, 链霉亲和素磁珠会吸附很多杂质DNA片段, 导致测序结果有用数据过低; 试验过程繁琐, 周期长, 成功率低, 往往要经过几次试验才能得到可用的试验结果; 不能对文库进行质量检测, 只有测序结果出来后才能对文库的质量进行分析, 试验风险性很大; 分析难度大, 要从海量的数据中去除噪音, 对生物信息学分析造成很大的难度。
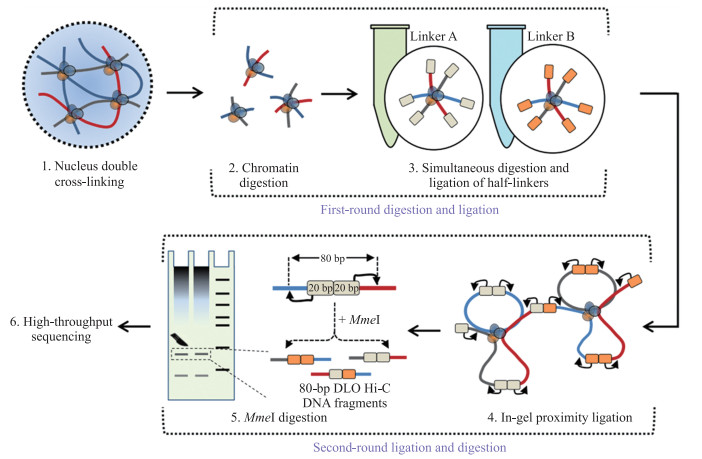
为了解决这些问题, 我们开发了只基于酶切酶连的DLO Hi-C(digestion-ligation-only Hi-C)技术(图 1)[7], 该方法采用同时酶切酶连(simultaneous digestion and ligation)[8]的方式, 用限制性内切酶以及DNA连接酶将1个带有MmeI酶切位点的20 bp接头(linker)连接在了染色体内切酶切口的黏性末端上, 再通过离心的方法去除多余的接头, 然后对连有接头的染色体片段进行T4 DNA酶邻近酶连、蛋白酶K消化、酚仿抽提, 得到纯化后的DLO Hi-C总DNA(total DNA of DLO Hi-C), 再用MmeI内切酶将DLO Hi-C总DNA进行酶切消化, 并用非变性Page胶回收释放出80 bp左右的DLO Hi-C片段(DLO Hi-C DNA fragment), 连上Illumina高通量测序接头, PCR扩增, 最终得到DLO Hi-C文库。
DLO Hi-C只需要简单的酶切酶连就能获取质量远高于传统Hi-C的数据。DLO Hi-C与传统Hi-C方法相比, 主要优势在于:使用EGS(ethylene glycol bis(csuccinimidyl succinate))和甲醛水溶液对细胞进行双交联固定, 可以捕获DNA-蛋白弱互作, 同时避免了后期试验过程中解交联的发生; 不需要生物素标记, 很大程度上节约了成本; 试验周期短, 只需要进行简单的酶切酶连步骤, 文库只需要2 d即可构建完成; 数据噪音更小, 用Page胶通过片段大小选择回收DLO Hi-C片段, 测序所得数据基本上是有效数据, 并且离心去除多余的linker的同时, 也去除了未结合蛋白的基因组碎片, 降低了小片段自环化产生的噪音; 不通过测序即可对文库随机连接噪音进行检测, 将连有linker A和linker B的2份样品混合, 再进行T4 DNA连接酶邻近酶连, 能够根据linker A和linker B的连接情况来判断文库质量; 构建的DNA-linker-DNA长度为80 bp, 可以减少测序量; 数据分析简单, 根据linker的序列信息, 降低了噪音去除、数据提取以及后期分析的难度。
我们采用in situ DLO Hi-C文库构建方法, 大约20 h就可以构建1个DLO Hi-C文库, 而传统的Hi-C文库构建, 一般需要36 h。不仅如此, 因为我们取消了生物素标记以及链霉亲和素吸附的步骤, 大大降低了试验成本, 整个试验的花费只需要100美元左右, 试验成本远低于传统Hi-C。由于DLO Hi-C的插入片段仅有80 bp, 只需要SE100测序平台即可对互作DNA进行测序, 大大降低了测序成本。
我们将DLO Hi-C应用于K562细胞系, 与来自同一细胞系的已有Hi-C数据进行比较, 结果显示:采用DLO Hi-C方法, 利用6.32×108条测序原始序列(raw reads), 找到了12 780个染色体Loop; 而利用经典的in situ Hi-C方法, 利用1.37×109条测序原始序列, 只找到了6 057个染色体Loop。
我们将DLO Hi-C应用于白血病单核THP-1细胞系, 成功地检测到了THP-1细胞系已知的染色体易位。对于新检测到的染色体易位, 我们进行了HCR(hybridization chain reaction)验证, 证明我们检测到的染色体易位是真实的。
DLO Hi-C文库构建过程简单、成本较低、成功率高, 不需要复杂的DNA超声仪器设备, 任何实验室都可以进行试验, 将在一定程度上推动三维基因组学技术的普及, 后期在癌细胞染色体变异检测、宏基因组组装、新物种基因组组装以及基因组调控元件互作分析中有着广泛的应用前景。
该成果在《Nature Genetics》以“Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture”为题发表。
| [1] | Maher B. ENCODE:the human encyclopaedia[J]. Nature, 2012, 489(7414): 46-48. DOI: 10.1038/489046a |
| [2] | Dekker J, Belmont A S, Guttman M, et al. The 4D nucleome project[J]. Nature, 2017, 549(7671): 219-226. DOI: 10.1038/nature23884 |
| [3] |
李国亮, 阮一骏, 谷瑞升, 等. 起航三维基因组学研究[J].
科学通报, 2014, 59(13): 1165-1172.
Li G L, Ruan Y J, Gu R S, et al. Emergence of 3D genomics[J]. Chinese Science Bulletin, 2014, 59(13): 1165-1172. (in Chinese with English abstract) |
| [4] | Lieberman-Aiden E, Dekker J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome[J]. Science, 2009, 326(5905): 289. |
| [5] | Dixon J R, Jung I, Selvaraj S, et al. Chromatin architecture reorganization during stem cell differentiation[J]. Nature, 2015, 518(7539): 331-336. DOI: 10.1038/nature14222 |
| [6] | Ke Y, Yu Y, Chen X, et al. 3D chromatin structures of mature gametes and structural reprogramming during mammalian embryogenesis[J]. Cell, 2017, 170(2): 367-381. DOI: 10.1016/j.cell.2017.06.029 |
| [7] | Lin D, Hong P, Zhang S, et al. Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture[J]. Nature Genetics, 2018, 50(5): 754-763. DOI: 10.1038/s41588-018-0111-2 |
| [8] | Cost G J. Enzymatic ligation assisted by nucleases:simultaneous ligation and digestion promote the ordered assembly of DNA[J]. Nature Protocols, 2007, 2(9): 2198-2202. DOI: 10.1038/nprot.2007.325 |