文章信息
- 向亚男, 黄蕊蕊, 顾婷婷, 甘立军
- XIANG Yanan, HUANG Ruirui, GU Tingting, GAN Lijun
- 基于RNA-Seq的拟南芥不定芽再生过程的基因表达谱分析
- Analysis of RNA-Seq-based expression profiles during adventitious shoot regeneration in Arabidopsis thaliana
- 南京农业大学学报, 2018, 41(2): 308-320
- Journal of Nanjing Agricultural University, 2018, 41(2): 308-320.
- http://dx.doi.org/10.7685/jnau.201703026
-
文章历史
- 收稿日期: 2017-03-21
2. 南京农业大学园艺学院, 江苏 南京 210095
2. College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China
植物不定芽的再生是一个多级发育过程, 其中包括体细胞对于植物激素信号的感知与传递、启动细胞分裂增殖、获得具有器官再生能力的去分化以及形成器官的再分化过程[1-3]。1986年, Feldmann等[4]建立了拟南芥不定芽再生的高效培养体系。首先将拟南芥外植体预培养在富含生长素的愈伤组织诱导培养基(callus induction medium, CIM)上, 再将外植体移至富含细胞分裂素的芽诱导培养基(shoot induction medium, SIM)[5-6]。不定芽再生被广泛运用于植物快速繁殖和作物遗传改良等生物技术过程中[1, 7]。因此, 深入研究两步培养法中不定芽发生的机制具有重要的理论和应用价值。
拟南芥过量表达细胞分裂素的hoc突变体在不含植物激素的培养基上能由根外植体发育形成完整植株[8], 而过量表达细胞分裂素负调控因子A型ARR则会抑制不定芽的形成[9]。对生长素应答启动子DR5的研究发现, 在诱导愈伤组织阶段生长素响应被显著促进[10]。不定芽再生过程早期生长素相关基因如AUX/IAA在CIM预培养时表达量上调[11]。细胞分裂素调节基因在SIM上培养时表达量上调[12], SIM培养基上芽的形成会促进芽顶端分生组织细胞分裂素响应信号的再分布[13]。此外, 生长素和细胞分裂素信号途径还会调节芽感应能力, 诱导其他基因的表达。CUC2(CUP-SHAPED COTYLEDON2)基因的表达在增殖的愈伤组织中明显上调[14]。而一些与分生组织形成相关的基因比如WUS(WUSCHEL)、CLV3(CLAVATA3)等基因则不在愈伤组织中表达[13]。还有研究表明在芽出现之前许多编码信号和转录元件的基因显著上调, 在这些阶段有大量基因调节活动在发生改变[15], 因此深入探讨在愈伤组织形成及离体芽再生阶段相关基因的表达及其与植物激素的关系, 对于研究不定芽再生的分子机制具有重要意义。
Xu等[16]利用基因芯片技术对愈伤组织形成阶段的根外植体和地上部外植体进行了动态转录组分析, 发现过量表达转录因子HB52和CRF3的外植体在不添加生长素的培养基上也能产生愈伤组织。Che等[11]利用基因芯片技术研究了拟南芥3种再生途径的基因表达情况, 发现了一系列可以作为不同发育过程分子标签的基因。这些研究在一定程度上揭示了部分发育过程的基因调控机制, 但所使用基因探针数目有限, 只能检测到有限基因的改变。我们采用了RNA-Seq技术对两步培养法中愈伤组织形成阶段及不定芽再生阶段基因的表达情况进行了测序、比较和分析, 发现两个阶段众多差异表达的基因及转录物功能单位, 并对显著富集的生长素与细胞分裂素合成代谢途径相关基因进行了分析, 为揭示不定芽发生的分子和激素调控机制提供理论依据。
1 材料与方法 1.1 试验材料植物材料拟南芥为Columbia生态型(Col-0)。1/2 MS培养基为自配。RNA提取试剂盒、反转录试剂盒等均购自TaKaRa公司。
1.2 愈伤组织生成及不定芽再生培养拟南芥种子经春化、消毒后直线播撒在含1/2 MS培养基的培养皿上, 置于22 ℃、16 h光照/8 h黑暗的条件下培养。培养7 d后, 随机切取根尖5 mm的根段, 将切取的根段外植体放在富含生长素的愈伤组织诱导培养基(CIM, 含2.2 μmol·L-1 2, 4-氯化苯氧乙酸, 0.2 μmol·L-1激动素的B5培养基)上培养4 d后, 再将外植体转移至芽诱导培养基(SIM, 含5.0 μmol·L-1异戊烯基腺嘌呤, 0.9 μmol·L-1 3-吲哚乙酸的B5培养基)上继续培养, 培养温度为21 ℃, 光照24 h。
1.3 总RNA的提取分别取CIM培养基上培养0 d(C0), CIM培养4 d(C4)和SIM培养3 d(S3)的拟南芥根外植体提取总RNA, 每个样本点进行3个生物学重复。为了保证提取RNA的质量, 每个时间点选取150个根外植体。采用RNeasy Plant Mini Kit(Tiagen, China)提取总RNA。利用DNase Ⅰ(Qiagen, UK)对样品中DNA进行消化处理。所有RNA样本用10 g·L-1琼脂糖凝胶电泳检查RNA样本的完整性, 然后用紫外分光光度计测量RNA浓度, 分析D260/D280和D260/D230值, 检验RNA样品的浓度和纯度。
1.4 建立cDNA文库和Illumina测序通过带有Oligo(dT)的磁珠富集mRNA, 富集的mRNA利用Oligo(dT)引物和反转录酶合成cDNA的第1、2条链。经限制性内切酶NlaⅢ剪切后, 对所得的cDNA片段进行纯化, 末端修复及3′末端加‘A’碱基, 并将DNA片段两端连接上特定的测序接头。电泳回收105 bp大小的DNA片段, PCR扩增富集测序样本。以上具体试验操作步骤均按照Illumina Gene Expression Sample Prep Kit说明书进行。随后将已加入接头的DNA片段绑定在流通池(flow cell)上进行反应, 扩增不同片段。在下一步反应中, 4种荧光标记的染料采用边合成边测序(sequencing by synthesis)的原理, 使用Illumina HiSeqTM 2000 system对文库进行测序。
1.5 数据分析拟南芥参考基因序列来自Illumina iGenomes网站(http://support.illumina.com/sequencing/sequencing_software/igenome.html;Arabidopsis_thaliana_Ensembl_TAIR10.tar.gz), 使用TopHat 2.0.6(http://ccb.jhu.edu/software/tophat/index.shtml)将得到的有效读段(clean reads)分别比对到拟南芥参考基因序列(TAIR10)。利用Bowtie 2(http://bowtie bio.sourceforge.net/index.shtml)将所得的clean reads对应到拼接好的转录物上, 比较不同样品间基因的表达差异, 使用FPKM法来计算基因表达量。通过比较不同时间点的转录水平(FPKM)来计算差异倍数(fold change, FC)。将错误发现率(false discovery rate, FDR)≤0.05, 且差异倍数不低于2倍(即log2[FC]的绝对值≥1)的基因确定为差异表达基因, 并比较了与细胞分裂素、生长素以及芽顶端分生组织相关基因的差异表达情况。通过DAVID(https://david.ncifcrf.gov/home.jsp)工具对转录物序列进行GO(Gene Ontology)功能注释和GO功能分类统计。在GO功能注释及分类统计时, 将P≤0.05的GO定义为显著富集。
2 结果与分析 2.1 RNA-Seq测序和评估本研究采用两步培养体系来再生不定芽, 首先在富含生长素的CIM上对外植体进行预培养诱导其伤口形成愈伤组织, 然后转至富含细胞分裂素的SIM上诱导不定芽的形成(图 1-A)[17]。拟南芥在CIM上预培养4 d(C4)后, 在根外植体端点部分出现膨大, 逐渐形成愈伤组织。再将其移至SIM培养基, 培养3 d(S3)外植体膨大更明显, 培养至6 d(S6)后根外植体上可见绿点, 继续培养绿点颜色逐渐加深。在SIM上培养至20 d(S20)明显可见不定芽的形成(图 1-B)。分别提取C0、C4和S3时间点的总RNA, 构建cDNA文库, 进行RNA-Seq试验。
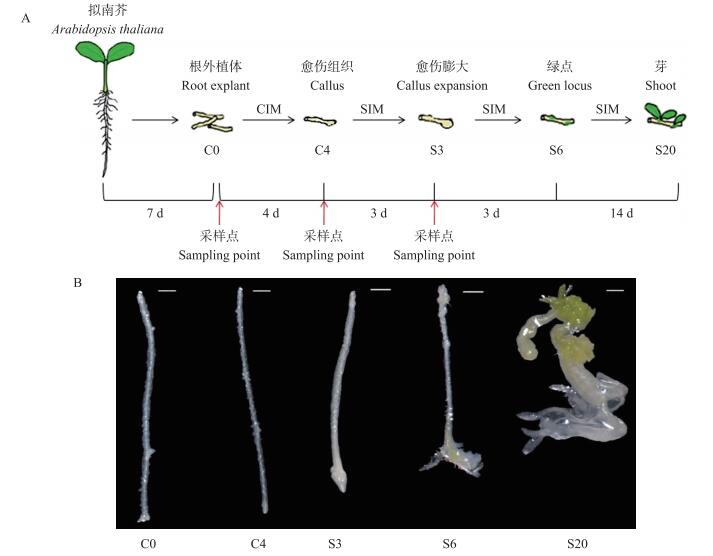
|
图 1 拟南芥芽再生培养体系
Figure 1 The in vitro shoot regeneration system with root explants in Arabidopsis thaliana
A.拟南芥不定芽再生培养体系, 红色箭头所指为提取RNA的3个时间点; C0、C4、S3、S6、S20分别代表CIM培养0 d、CIM培养4 d、SIM培养3 d、SIM培养6 d、SIM培养20 d。下同。 B.5个不同时期拟南芥根外植体的生长状态, 标尺=500 μm。 A.The in vitro shoot regeneration system using root as explants in Arabidopsis thaliana, red arrows show times during development when RNA samples were taken; C0, C4, S3, S6 and S20 presented 0 d on CIM, 4 d CIM, 3 d SIM, 6 d SIM, 20 d SIM, respectively. The same as follows. B.The pictures of Arabidopsis thaliana root explants in different stages of de novo shoot regeneration, bar=500 μm. |
采取C0、C4和S3三个时间点样本进行RNA-Seq分析, 将3个时间点样本分别与拟南芥参考基因进行比对, 统计所得有效读段数及其有效读段的比例(表 1)。
| 指标Index | C0-Ⅰ | C0-Ⅱ | C0-Ⅲ | C4-Ⅰ | C4-Ⅱ | C4-Ⅲ | S3-Ⅰ | S3-Ⅱ | S3-Ⅲ |
| 匹配读段数/106 Total number of mapped reads | 34.6 | 34.6 | 36.0 | 36.0 | 37.9 | 36.0 | 37.0 | 36.1 | 35.4 |
| 匹配读段所占比例/% Mapped reads percentage | 81.7 | 84.8 | 85.3 | 86.3 | 85.5 | 85.1 | 85.6 | 85.4 | 84.8 |
| Read-1*/106 | 21.2 | 20.4 | 21.1 | 20.9 | 22.1 | 21.1 | 21.6 | 21.1 | 20.9 |
| Read-2*/106 | 21.2 | 20.4 | 21.1 | 20.9 | 22.1 | 21.1 | 21.6 | 21.1 | 20.9 |
| 未匹配读段数/106 Total number of unmapped reads | 7.7 | 6.2 | 6.2 | 5.7 | 6.4 | 6.3 | 6.2 | 6.1 | 6.3 |
| 注: Ⅰ、Ⅱ和Ⅲ代表不同取样时间点的3个生物学重复; Read-1*和Read-2*分别表示正义链和反义链的有效读段(clean reads)数。 Note: Ⅰ, Ⅱ and Ⅲ presented 3 biological repeats of different sampling points; Read-1* and Read-2* showed forward and reverse primer derived reads, respectively. | |||||||||
为了检测在愈伤组织形成阶段基因的差异表达情况, 我们将C4时间点的基因表达量和C0时间点的进行了比对。FDR≤0.05且差异倍数不低于2倍(|log2FC(C4/C0)|≥1)的基因确定为差异表达基因。依据这些标准, 我们共检测到4 332个差异表达基因, 有1 399个基因上调, 2 933个基因下调。表 2为C4与C0(愈伤组织形成阶段)比较前50个差异基因。最显著上调的基因AT3G59580编码转录因子Nin-like家族蛋白。另有编码转录因子TCP家族蛋白的基因AT3G18550、LBD19基因AT2G45410和WRKY 63基因AT1G66600显著上调。另外, 一些与胚胎形成相关的基因如晚期胚胎富集蛋白基因(late embryogenesis abundant protein genes, LEA):AT4G21020、AT2G03740、AT2G03850也明显上调。编码与生长素相关的GH3.12(AT5G13320)、GH3.3(AT2G23170)及编码细胞分裂素氧化酶2(cytokinin oxidase 2, CKX2, AT2G19500)的基因也发生了变化。
| 基因ID Gene ID |
差异倍数 log2FC(C4/C0) |
P值 P-value |
错误发现率 FDR |
功能注释 Descriptions |
| 表达量增加的基因 Up-regulated genes | ||||
| AT3G59580 | 10.960 600 | 1.12E-141 | 3.63E-140 | 转录因子 NLP9 Transcription factor NLP9 |
| AT3G45760 | 10.493 980 | 2.43E-64 | 2.63E-63 | 核苷酸转移酶家族蛋白 Nucleotidyltransferase family protein |
| AT1G33960 | 10.167 820 | 3.17E-71 | 3.82E-70 | 核苷三磷酸水解酶 AIG1 Nucleoside triphosphate hydrolases AIG1 |
| AT5G58390 | 9.407 965 | 7.10E-120 | 1.78E-118 | 过氧化物酶 67 Peroxidase 67 |
| AT3G13630 | 9.350 780 | 9.41E-71 | 1.12E-69 | 未知蛋白 Unknown protein |
| AT5G52730 | 9.234 108 | 1.08E-45 | 7.93E-45 | 铜运输蛋白 Copper transport protein |
| AT1G74140 | 9.121 313 | 3.13E-51 | 2.61E-50 | 膜内丝氨酸蛋白酶家族蛋白 Intramembrane serine protease family protein |
| AT3G60420 | 9.011 520 | 1.33E-137 | 4.06E-136 | 磷酸甘油酸变位酶家族蛋白 Phosphoglycerate mutase family protein |
| AT5G24210 | 8.997 218 | 3.08E-40 | 1.99E-39 | α/β水解酶超家族蛋白 Alpha/beta-Hydrolases superfamily protein |
| AT1G56710 | 8.900 146 | 1.11E-104 | 2.23E-103 | 果胶裂解酶类似物家族蛋白 Pectin lyase-like family protein |
| AT5G59810 | 8.823 784 | 1.69E-33 | 9.25E-33 | 亚麻酶家族蛋白 SBT5.4 Subtilase family protein SBT5.4 |
| AT3G28510 | 8.771 441 | 2.28E-124 | 5.98E-123 | 含有P环的核苷三磷酸水解酶超家族蛋白 P-loop containing nucleoside triphosphate hydrolases superfamily protein |
| AT2G26400 | 8.652 720 | 2.31E-184 | 1.20E-182 | 顺式还原酮加双氧酶 3 Acireductone dioxygenase 3 |
| AT5G24200 | 8.607 502 | 1.95E-78 | 2.65E-77 | α/β水解酶超家族蛋白 Alpha/beta-Hydrolases superfamily protein |
| AT5G56810 | 8.589 908 | 1.93E-30 | 9.63E-30 | 含有F-box结构域的蛋白 F-box domains-containing protein |
| AT4G31870 | 8.231 147 | 1.83E-26 | 8.11E-26 | 谷胱甘肽过氧化物酶 7 Glutathione peroxidase 7 |
| AT4G21020 | 8.200 845 | 2.23E-58 | 2.18E-57 | 晚期胚胎富集蛋白 Late embryogenesis abundant protein(LEA)family protein |
| AT5G13320 | 8.177 742 | 1.62E-157 | 6.25E-156 | 生长素响应家族蛋白GH3.12 Auxin-responsive GH3 family protein GH3.12 |
| AT2G19500 | 8.126 221 | 4.74E-23 | 1.88E-22 | 细胞分裂素脱氢酶2 Cytokinin dehydrogenase 2 |
| AT4G31380 | 8.014 105 | 2.28E-168 | 1.00E-166 | FPF1类似蛋白 FPF1-like protein 1 |
| AT5G24160 | 7.987 202 | 1.41E-17 | 4.65E-17 | 鲨烯环氧酶6 Squalene epoxidase 6 SQE6 |
| AT2G03740 | 7.985 489 | 1.71E-22 | 6.64E-22 | 含有LEA结构域的蛋白 LEA domain-containing protein |
| AT2G03850 | 7.984 408 | 8.52E-246 | 8.10E-244 | 晚期胚胎富集蛋白 LEA family protein |
| AT2G23170 | 7.966 611 | 0 | 0 | 生长素响应家族蛋白GH3.3 Auxin-responsive GH3 family protein GH3.3 |
| AT5G59390 | 7.916 237 | 4.03E-81 | 5.69E-80 | 含有XH/XS结构域的蛋白 XH/XS domain-containing protein |
| AT2G18140 | 7.868 948 | 9.43E-113 | 2.14E-111 | 过氧化物酶14 Peroxidase 14 |
| AT3G18550 | 7.853 049 | 1.16E-20 | 4.23E-20 | 转录因子TCP18 Transcription factor TCP18 |
| AT5G09970 | 7.833 744 | 3.43E-69 | 4.01E-68 | 细胞色素CYP78A7 Cytochrome CYP78A7 |
| AT5G59330 | 7.779 557 | 1.60E-21 | 6.03E-21 | 脂质转移蛋白 Lipid-transfer protein |
| AT2G05380 | 7.755 803 | 7.09E-155 | 2.66E-153 | 富含甘氨酸蛋白S3 Glycine-rich protein S3 |
| AT4G11890 | 7.743 940 | 1.46E-15 | 4.46E-15 | 蛋白激酶超家族蛋白ARCK1 Protein kinase superfamily protein ARCK1 |
| AT1G71150 | 7.670 446 | 1.42E-121 | 3.63E-120 | 未知蛋白 Unknown protein |
| AT2G45410 | 7.666 772 | 8.77E-155 | 3.28E-153 | 转录因子LBD19 Transcription factor LBD19 |
| AT5G16980 | 7.654 911 | 8.60E-76 | 1.12E-74 | 锌结合脱氢酶家族蛋白 Zinc-binding dehydrogenase family protein |
| AT1G66600 | 7.645 328 | 2.87E-89 | 4.65E-88 | 转录因子WRKY63 Transcription factor WRKY63 |
| AT1G22090 | 7.643 044 | 8.84E-20 | 3.14E-19 | 蛋白质EMB2204 Protein EMB2204 |
| AT1G52130 | 7.624 207 | 3.62E-38 | 2.23E-37 | 甘露糖结合凝集素超家族蛋白 Mannose-binding lectin superfamily protein |
| AT3G57260 | 7.608 860 | 6.66E-19 | 2.29E-18 | 葡聚糖酶BGL2 Glucanase BGL2 |
| AT5G52700 | 7.588 621 | 7.04E-16 | 2.18E-15 | 铜转运蛋白 Copper transport protein |
| AT1G65680 | 7.583 108 | 3.95E-30 | 1.95E-29 | 扩张蛋白B2 Expansin-B2 |
| AT2G05540 | 7.544 622 | 7.93E-142 | 2.57E-140 | 富含甘氨酸蛋白 Glycine-rich protein |
| AT4G24000 | 7.488 220 | 6.86E-18 | 2.28E-17 | 纤维素合酶类似蛋白G2 Cellulose synthase-like protein G2 |
| AT2G02320 | 7.447 018 | 3.97E-15 | 1.19E-14 | 韧皮部蛋白PP2-B7 Phloem protein PP2-B7 |
| AT1G29100 | 7.445 327 | 2.33E-17 | 7.59E-17 | 重金属转运蛋白 Heavy metal transport protein |
| AT5G22980 | 7.442 985 | 1.10E-13 | 3.12E-13 | 丝氨酸羧肽酶类似物47 Serine carboxypeptidase-like 47 |
| AT1G67770 | 7.408 316 | 1.16E-145 | 3.93E-144 | 末端EAR1类似物2 Terminal EAR1-like 2 |
| AT3G12900 | 7.404 723 | 2.10E-15 | 6.38E-15 | 亚铁依赖型加氧酶超家族蛋白 Fe(Ⅱ)-dependent oxygenase superfamily protein |
| AT1G11600 | 7.401 719 | 9.83E-293 | 1.40E-290 | 细胞色素CYP77B1 Cytochrome CYP77B1 |
| AT2G41850 | 7.339 567 | 0 | 0 | 聚半乳糖醛酸酶ADPG2 Polygalacturonase ADPG2 |
| AT5G25040 | 7.326 759 | 2.99E-41 | 1.98E-40 | 未知蛋白 Unknown protein |
| 表达量减少的基因 Down-regulated genes | ||||
| AT3G19710 | -14.741 6 | 8.85E-201 | 5.31E-199 | 蛋氨酸转氨酶BCAT4 Methionine aminotransferase BCAT4 |
| AT3G01190 | -13.907 7 | 4.31E-310 | 6.93E-308 | 过氧化物酶27 Peroxidase 27 |
| AT4G22666 | -13.640 5 | 1.35E-224 | 1.05E-222 | 脂质转移蛋白 Lipid-transfer protein |
| AT1G54970 | -13.421 1 | 2.24E-157 | 8.61E-156 | 富含脯氨酸蛋白1(PRP1)Proline-rich protein 1 |
| AT5G42180 | -13.291 5 | 0 | 0 | 过氧化物酶64 Peroxidase 64 |
| AT2G39510 | -12.923 8 | 2.77E-59 | 2.75E-58 | WAT1相关蛋白 WAT1-related protein |
| AT3G18200 | -12.893 6 | 3.43E-284 | 4.65E-282 | EamA转运类似物家族蛋白 EamA-like transporter family protein |
| AT5G57625 | -12.716 7 | 1.21E-98 | 2.26E-97 | CAP超家族蛋白 CAP superfamily protein |
| AT4G13770 | -12.617 1 | 2.31E-225 | 1.82E-223 | 细胞色素P45083A1 Cytochrome P45083A1 |
| AT2G36100 | -12.584 0 | 0 | 0 | 凯式带膜蛋白1 Casparian strip membrane protein 1 |
| AT3G01260 | -12.524 1 | 2.96E-198 | 1.74E-196 | 半乳糖变旋酶类似物超家族蛋白 Galactose mutarotase-like superfamily protein |
| AT1G31050 | -12.513 2 | 2.41E-242 | 2.25E-240 | bHLH DNA结合超家族蛋白 bHLH DNA-binding superfamily protein |
| AT3G05920 | -12.501 0 | 3.29E-233 | 2.77E-231 | 重金属转运蛋白 Heavy metal transport protein |
| AT1G14220 | -12.416 1 | 8.55E-172 | 3.90E-170 | 核糖核酸酶T2家族蛋白 Ribonuclease T2 family protein |
| AT1G19900 | -12.396 0 | 3.35E-127 | 9.10E-126 | 乙二醛氧化酶相关蛋白 Glyoxal oxidase-related protein |
| AT1G64780 | -12.393 6 | 0 | 0 | 铵基转运物1 Ammonium transporter 1 |
| AT1G12090 | -12.347 8 | 1.57E-148 | 5.45E-147 | 伸展蛋白类似蛋白Extensin-like protein |
| AT2G43880 | -12.244 8 | 1.67E-149 | 5.86E-148 | 果胶裂解酶类似物超家族蛋白 Pectin lyase-like superfamily protein |
| AT3G06460 | -12.233 0 | 2.78E-221 | 2.08E-219 | GNS1/SUR4膜蛋白家族GNS1/SUR4 membrane protein family |
| AT5G36140 | -12.206 3 | 2.14E-53 | 1.86E-52 | 细胞色素CYP716A2 Cytochrome CYP716A2 |
| AT3G55230 | -12.021 2 | 0 | 0 | 抗病响应家族蛋白 Disease resistance-responsive family protein |
| AT1G73330 | -11.963 0 | 0 | 0 | 干旱抑制蛋白4(DR4)Drought-repressed 4 |
| AT2G15370 | -11.876 6 | 2.94E-126 | 7.86E-125 | 岩藻糖转移酶5 Fucosyltransferase 5 |
| AT1G14160 | -11.805 8 | 4.32E-165 | 1.80E-163 | CASP类似蛋白1A1 CASP-like protein 1A1 |
| AT5G46900 | -11.785 4 | 3.66E-214 | 2.60E-212 | 脂质转移蛋白 Lipid-transfer protein |
| AT4G25250 | -11.731 8 | 1.32E-99 | 2.50E-98 | 果胶甲酯酶抑制物家族蛋白 Pectin methylesterase inhibitor family protein |
| AT1G65310 | -11.695 1 | 1.59E-189 | 8.52E-188 | 木葡聚糖水解酶17 Xyloglucan hydrolase 17 |
| AT5G44130 | -11.684 0 | 8.69E-38 | 5.29E-37 | 类似FASCICLIN的阿拉伯半乳聚糖蛋白13前体FASCICLIN-like arabinogalactan protein 13 precursor |
| AT4G08410 | -11.624 3 | 2.33E-120 | 5.86E-119 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT5G15150 | -11.616 2 | 3.84E-168 | 1.67E-166 | 转录因子HB-3 Transcription factor HB-3 |
| AT5G06630 | -11.585 3 | 7.84E-89 | 1.26E-87 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT3G45680 | -11.555 5 | 5.06E-193 | 2.78E-191 | 蛋白NRT1/PTR家族2.3 Protein NRT1/PTR FAMILY 2.3 |
| AT5G61420 | -11.541 6 | 1.63E-241 | 1.50E-239 | 转录因子MYB28 Transcription factor MYB28 |
| AT4G14060 | -11.498 0 | 4.71E-89 | 7.58E-88 | 多聚乙酰环化酶和脂质转运物超家族蛋白Polyketide cyclase and lipid transport superfamily protein |
| AT3G21180 | -11.492 8 | 1.47E-113 | 3.38E-112 | 钙离子运输ATP酶9 Calcium-transporting ATPase 9 |
| AT2G28780 | -11.472 3 | 1.60E-126 | 4.29E-125 | 未知蛋白 Unknown protein |
| AT3G28290 | -11.443 1 | 3.10E-90 | 5.10E-89 | 跨膜蛋白AT14A Transmembrane protein AT14A |
| AT1G47600 | -11.420 4 | 0 | 0 | 芥子酶4 Myrosinase 4 |
| AT4G13390 | -11.419 6 | 2.97E-125 | 7.84E-124 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT5G04960 | -11.419 2 | 1.69E-151 | 6.07E-150 | 果胶酯酶抑制物Pectinesterase inhibitor |
| AT4G22217 | -11.394 7 | 8.19E-144 | 2.72E-142 | 防御素类似物蛋白100 Defensin-like protein 100 |
| AT4G15290 | -11.387 7 | 2.43E-60 | 2.46E-59 | 纤维素合酶类似物蛋白B5 Cellulose synthase-like protein B5 |
| AT4G30170 | -11.358 9 | 0 | 0 | 过氧化物酶45 Peroxidase 45 |
| AT2G34910 | -11.303 8 | 5.65E-161 | 2.26E-159 | 未知蛋白 Unknown protein |
| AT1G20190 | -11.291 9 | 1.61E-208 | 1.06E-206 | 扩张蛋白A11 Expansin-A11 |
| AT3G60280 | -11.268 6 | 6.47E-90 | 1.06E-88 | 蛋白质UCC3 Protein UCC3 |
| AT4G11320 | -11.216 6 | 0 | 0 | 半胱氨酸蛋白酶Cysteine proteinase |
| AT2G37700 | -11.192 9 | 1.34E-132 | 3.90E-131 | 蛋白CER1类似物2 Protein CER1-like 2 |
| AT3G53980 | -11.176 4 | 3.17E-155 | 1.19E-153 | 脂质转移蛋白 Lipid-transfer protein |
| AT5G14330 | -11.157 5 | 3.52E-149 | 1.23E-147 | 跨膜蛋白 Transmembrane protein |
| Note:FDR:False discovery rate. The same as follows. | ||||
同时, 我们也分析了愈伤组织形成阶段下调的基因, 发现下调倍数最高的基因AT3G19710编码蛋氨酸转氨酶BCAT4。BCAT4转录物可由伤害诱导在韧皮部大量积累[18]。另几个明显下调的基因AT3G01190、AT5G42180和AT4G30170, 它们都编码第三类过氧化物酶, 这一类酶与生长素的分解代谢、创伤愈合、木质化等功能相关。编码凯氏带膜域蛋白1(casparian strip membrane domain protein 1, CASP1)的基因AT2G36100表达量也出现下降。CASPs在细胞壁的修饰方面具有重要的指导作用, 它通过与分泌型过氧化物酶的相互作用来调节木质素的沉积和凯氏带的形成[19]。另外编码转录因子bHLH DNA结合家族蛋白(AT1G31050)、HD-ZIP homeobox 3(AT5G15150)、MYB28(AT5G61420)的基因明显表达下调。
此外, 我们还利用Gene Ontology(GO)功能注释分类体系分别对2个阶段差异基因进行功能注释和分析。在GO功能分类体系中, 我们发现愈伤组织形成阶段差异表达的4 332个基因中有4 137条转录物具有具体功能定义。它们分别富集在3个功能范畴, 这3个功能可被划分为更详细的446个类别, 其中生物学过程有282个功能亚类, 细胞组分有29个功能亚类, 分子功能有135个。图 2为愈伤组织形成阶段前20个显著富集的差异基因功能条目, 从图 2可以看出:核转录因子活性、激素刺激响应、生长素刺激响应、生长素信号调节通路、激素水平调节等是显著富集的功能条目。
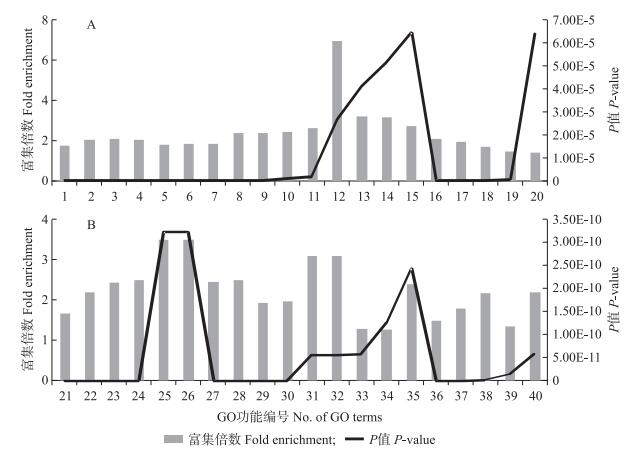
|
图 2 拟南芥愈伤组织形成阶段前20个显著富集的转录物GO功能
Figure 2 TOP 20 highly enriched GO(Gene Ontology)terms during the callus formation of A.thaliana
A.显著上调的富集转录物功能条目。The upregulated gene subsets with annotated GO terms. 1.转录调节 Regulation of transcription; 2.有机物质反应 Response to organic substance; 3.激素刺激响应 Response to hormone stimulus; 4.内源刺激响应 Response to endogenous stimulus; 5.转录 Transcription; 6.依赖DNA的转录调节 Regulation of transcription, DNA-dependent; 7.RNA代谢调节 Regulation of RNA metabolic process; 8.激素刺激细胞反应 Cellular response to hormone stimulus; 9.激素信号 Hormone-mediated signaling; 10.生长素刺激响应 Response to auxin stimulus; 11.乙烯刺激响应 Response to ethylene stimulus; 12.子叶发育 Cotyledon development; 13.生长素信号调节通路 Auxin mediated signaling pathway; 14.激素水平调节 Regulation of hormone levels; 15.乙烯调节的信号通路 Ethylene mediated signaling pathway; 16.转录因子活性 Transcription factor activity; 17.转录调节活性 Transcription regulator activity; 18.DNA结合 DNA binding; 19.固有膜 Intrinsic to membrane; 20.膜完整性 Integral to membrane. B.显著下调的富集转录物功能。The downregulated gene subsets with annotated GO terms. 21.氧化还原 Oxidation reduction; 22.次级代谢过程 Secondary metabolic process; 23.外部封装结构 External encapsulating structure organization; 24.细胞壁 Cell wall organization; 25.过氧化氢分解代谢 Hydrogen peroxide catabolic process; 26.过氧化氢细胞响应 Cellular response to hydrogen peroxide; 27.四吡咯结合 Tetrapyrrole binding; 28.亚铁血红素结合 Heme binding; 29.铁离子结合 Iron ion binding; 30.电子载体活性 Electron carrier activity; 31.氧化还原酶活性 Oxidoreductase activity; 32.过氧化物酶活性 Peroxidase activity; 33.离子结合 Ion binding; 34.阳离子结合 Cation binding; 35.氧气结合 Oxygen binding; 36.内膜系统 Endomembrane system; 37.膜外区 Extracellular region; 38.质外体 Apoplast; 39.固有膜 Intrinsic to membrane; 40.固定于膜 Anchored to membrane. |
将S3时间点的基因表达量和C4时间点的进行了比对(FDR≤0.05且|log2FC(S3/C4)|≥1)。发现在不定芽再生阶段有2 910个差异表达的基因, 其中上调的基因有2 231个, 下调的基因有679个。表 3列出了本阶段前50个差异表达的基因。其中有多个基因在愈伤组织形成阶段表达量降低, 而在不定芽再生阶段却显著增加, 如编码CAP家族蛋白的AT5G57625和编码CASP1的AT2G36100, 编码过氧化物酶64的AT5G42180和编码过氧化物酶27的AT3G01190也在不定芽再生阶段出现明显上调。在生长素信号下游起作用并影响侧根形成的转录因子PUCHI以及其共同作用元件LBD16在本阶段出现了明显的下调。
| 基因ID Gene ID |
差异倍数 log2FC(S3/C4) |
P值 P-value |
错误发现率 FDR |
功能注释 Descriptions |
| 表达量增加的基因 UP-regulated genes | ||||
| AT5G57625 | 13.561 87 | 7.84E-116 | 3.62E-114 | CAP超家族蛋白 CAP superfamily protein |
| AT3G45680 | 13.350 20 | 0 | 0 | 未知蛋白 Unknown protein |
| AT1G54970 | 12.790 29 | 2.00E-137 | 1.20E-135 | 富含脯氨酸蛋白 PRP1 Proline-rich protein 1 |
| AT4G08410 | 12.592 97 | 1.85E-142 | 1.20E-140 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT3G19270 | 12.496 03 | 4.11E-220 | 6.57E-218 | 脱落酸羟化酶Abscisic acid 8′-hydroxylase 4 |
| AT5G42180 | 12.139 10 | 3.45E-256 | 8.21E-254 | 过氧化物酶64 Peroxidase 64 |
| AT1G62540 | 12.117 13 | 4.16E-109 | 1.75E-107 | 含有黄素的单氧酶FMO GS-OX2 Flavin-containing monooxygenase GS-OX2 |
| AT5G06630 | 11.927 27 | 3.26E-94 | 1.07E-92 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT4G13390 | 11.906 70 | 7.43E-136 | 4.42E-134 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT1G29660 | 11.739 98 | 8.44E-161 | 6.95E-159 | GDSL脂肪酶GDSL lipase |
| AT3G60280 | 11.670 82 | 3.76E-101 | 1.37E-99 | 蛋白质UCC3 Protein UCC3 |
| AT3G62680 | 11.647 08 | 1.50E-124 | 7.83E-123 | 富含脯氨酸蛋白PRP 3 Proline-rich protein 3 |
| AT3G01190 | 11.611 29 | 7.95E-158 | 6.30E-156 | 过氧化物酶27 Peroxidase 27 |
| AT2G36100 | 11.606 82 | 0 | 0 | 凯氏带膜蛋白1 Casparian strip membrane protein 1 |
| AT5G06640 | 11.578 06 | 1.23E-113 | 5.52E-112 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT3G55230 | 11.457 04 | 1.78E-313 | 7.80E-311 | 抗病响应家族蛋白Disease resistance-responsive family protein |
| AT2G16005 | 11.441 89 | 2.44E-74 | 5.65E-73 | 含有MD-2相关脂质识别域蛋白MD-2-related lipid recognition domain-containing protein |
| AT4G22666 | 11.377 19 | 3.03E-120 | 1.49E-118 | 脂质转移蛋白 Lipid-transfer protein |
| AT2G20520 | 11.347 54 | 1.20E-57 | 2.02E-56 | 类似成束蛋白的阿拉伯半乳聚糖蛋白6 Fasciclin-like arabinogalactan protein 6 |
| AT5G04960 | 11.276 25 | 2.73E-147 | 1.87E-145 | 果胶酯酶抑制物46 Pectinesterase inhibitor 46 |
| AT2G24980 | 11.273 30 | 8.62E-122 | 4.32E-120 | 富含脯氨酸的伸展蛋白类似物家族蛋白Proline-rich extensin-like family protein |
| AT1G14220 | 11.252 01 | 1.94E-118 | 9.28E-117 | 核糖核酸酶T2家族蛋白Ribonuclease T2 family protein |
| AT1G27740 | 11.246 73 | 7.54E-172 | 6.97E-170 | 转录因子bHLH54 Transcription factor bHLH54 |
| AT1G30870 | 11.205 71 | 1.65E-272 | 4.59E-270 | 过氧化物酶7 Peroxidase 7 |
| AT2G34910 | 11.157 99 | 2.19E-151 | 1.60E-149 | 未知蛋白 Unknown protein |
| AT1G47600 | 11.038 63 | 7.12E-315 | 3.28E-312 | 芥子酶4 Myrosinase 4 |
| AT3G54580 | 11.014 35 | 4.36E-231 | 8.02E-229 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT3G54590 | 10.996 14 | 1.79E-165 | 1.59E-163 | 富含羟脯氨酸糖蛋白 Hydroxyproline-rich glycoprotein |
| AT5G14330 | 10.762 64 | 1.65E-138 | 1.02E-136 | 未知蛋白 Unknown protein |
| AT1G13710 | 10.561 70 | 3.02E-63 | 5.70E-62 | 细胞色素P450 78A5 Cytochrome P450 78A5 |
| AT5G61350 | 10.559 36 | 5.34E-82 | 1.43E-80 | 受体类似物蛋白激酶Receptor-like protein kinase |
| AT5G14340 | 10.553 26 | 4.60E-81 | 1.21E-79 | 转录因子MYB40 Transcription factor MYB40 |
| AT2G45180 | 10.526 65 | 1.76E-66 | 3.52E-65 | 脂质转移蛋白 Lipid-transfer protein |
| AT5G35190 | 10.516 67 | 8.25E-101 | 2.99E-99 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT5G22410 | 10.445 95 | 4.47E-130 | 2.51E-128 | 过氧化物酶60 Peroxidase 60 |
| AT4G15680 | 10.441 70 | 4.56E-95 | 1.51E-93 | 单巯基谷氧还蛋白S4 Monothiol glutaredoxin-S4 |
| AT5G03960 | 10.407 19 | 9.01E-91 | 2.79E-89 | IQ结构域12 IQ-domain 12 |
| AT2G34490 | 10.380 47 | 2.46E-186 | 2.74E-184 | 细胞色素450 710A2 Cytochrome P450 710A2 |
| AT1G62980 | 10.372 33 | 2.19E-156 | 1.69E-154 | 扩张蛋白A18 Expansin-A18 |
| AT3G05920 | 10.361 89 | 7.02E-99 | 2.47E-97 | 重金属运输蛋白 Heavy metal transport protein |
| AT4G11210 | 10.288 31 | 0 | 0 | 抗病响应家族蛋白Disease resistance-responsive family protein |
| AT3G06460 | 10.261 96 | 3.97E-96 | 1.34E-94 | GNS1/SUR4膜蛋白家族GNS1/SUR4 membrane protein family |
| AT3G05770 | 10.247 59 | 0 | 0 | 未知蛋白 Unknown protein |
| AT2G21140 | 10.228 64 | 1.75E-118 | 8.42E-117 | 富含脯氨酸蛋白2(PRP2)Proline-rich protein 2 |
| AT1G23720 | 10.225 68 | 0 | 0 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT2G44110 | 10.216 31 | 6.29E-71 | 1.37E-69 | MLO类似蛋白15 MLO-like protein 15 |
| AT3G18200 | 10.207 22 | 4.16E-95 | 1.38E-93 | EamA转运类似物家族蛋白EamA-like transporter family protein |
| AT3G28550 | 10.143 51 | 0 | 0 | 富含脯氨酸的伸展蛋白类似物家族蛋白 Proline-rich extensin-like family protein |
| AT2G05100 | 10.112 77 | 4.84E-71 | 1.06E-69 | 光合系统Ⅱ集光复合体Photosystem Ⅱ light harvesting complex |
| AT2G20750 | 10.090 03 | 2.34E-139 | 1.47E-137 | 扩张蛋白B1 Expansin-B1 |
| 表达量减少的基因 Down-regulated genes | ||||
| AT2G41850 | -13.341 20 | 0 | 0 | 聚半乳糖醛酸酶ADPG2 Polygalacturonase ADPG2 |
| AT5G22430 | -8.722 50 | 1.16E-31 | 1.02E-30 | 伸展蛋白家族蛋白 Extensin family protein |
| AT2G15590 | -8.675 76 | 1.29E-40 | 1.49E-39 | 未知功能蛋白 Protein of unknown function |
| AT1G18100 | -8.299 30 | 5.41E-58 | 9.22E-57 | PEBP家族蛋白 PEBP family protein |
| AT2G03740 | -8.000 38 | 1.12E-22 | 7.10E-22 | 含有LEA结构域蛋白 LEA domain-containing protein |
| AT4G12500 | -7.957 78 | 1.74E-08 | 5.24E-08 | pEARLI1相似脂质转运蛋白3 pEARLI1-like lipid transfer protein 3 |
| AT4G30140 | -7.865 81 | 3.42E-251 | 7.87E-249 | GDSL脂肪酶GDSL lipase |
| AT5G58390 | -7.762 65 | 2.88E-117 | 1.36E-115 | 过氧化物酶67 Peroxidase 67 |
| AT1G76610 | -7.747 30 | 4.32E-19 | 2.35E-18 | 未知功能蛋白 Protein of unknown function |
| AT1G03790 | -7.351 61 | 1.96E-28 | 1.53E-27 | 转录因子C3H家族蛋白 C3H family protein |
| AT2G18130 | -7.324 10 | 1.24E-11 | 4.58E-11 | 酸性磷酸酶11(PAP11)Purple acid phosphatase 11 |
| AT1G67770 | -7.191 10 | 1.40E-146 | 9.50E-145 | 末端EAR1类似物2 Terminal EAR1-like 2 |
| AT5G10500 | -7.119 56 | 1.53E-13 | 6.30E-13 | 未知蛋白 Unknown protein |
| AT3G48300 | -7.113 78 | 7.79E-12 | 2.90E-11 | 细胞色素P450 71A23 Cytochrome P450 71A23 |
| AT1G21970 | -7.031 63 | 5.41E-14 | 2.28E-13 | 转录因子NF-YB家族蛋白 NF-YB family protein |
| AT4G12480 | -6.962 85 | 1.32E-21 | 7.99E-21 | 脂质转运蛋白EARLI1 Lipid transfer protein EARLI 1 |
| AT1G25310 | -6.942 29 | 1.17E-11 | 4.33E-11 | 转录因子MEE8 Transcription factor MEE8 |
| AT1G04150 | -6.918 41 | 3.29E-94 | 1.08E-92 | 磷酸核糖基转移酶家族蛋白 Phosphoribosyltransferase family protein |
| AT2G46990 | -6.639 95 | 1.50E-207 | 2.03E-205 | 生长素响应蛋白IAA20 Auxin-responsive protein IAA20 |
| AT1G62421 | -6.629 93 | 1.52E-10 | 5.26E-10 | 未知蛋白 Unknown protein |
| AT3G02670 | -6.623 97 | 6.72E-39 | 7.37E-38 | 富含甘氨酸蛋白 Glycine-rich protein |
| AT2G45410 | -6.604 74 | 1.54E-143 | 1.01E-141 | 转录因子LBD19 Transcription factor LBD 19 |
| AT1G21990 | -6.588 18 | 4.51E-84 | 1.26E-82 | F-box蛋白 F-box protein |
| AT1G56360 | -6.564 52 | 1.48E-29 | 1.20E-28 | 酸性磷酸酶6(PAP6)Purple acid phosphatase 6 |
| AT1G68320 | -6.553 62 | 2.47E-114 | 1.12E-112 | 转录因子MYB62 Transcription factor MYB62 |
| AT2G41225 | -6.552 37 | 1.02E-09 | 3.35E-09 | 未知蛋白 Unknown protein |
| AT1G29600 | -6.545 06 | 3.84E-18 | 1.99E-17 | 转录因子C3H家族蛋白 C3H family protein |
| AT4G18190 | -6.508 33 | 7.18E-10 | 2.38E-09 | 嘌呤透性酶6 Purine permease 6 |
| AT3G03670 | -6.466 89 | 2.92E-09 | 9.32E-09 | 过氧化物酶28 Peroxidase 28 |
| AT4G37850 | -6.449 27 | 1.96E-57 | 3.29E-56 | 转录因子bHLH25 Transcription factor bHLH25 |
| AT2G03850 | -6.320 60 | 1.95E-231 | 3.61E-229 | 晚期胚胎富集蛋白 LEA family protein |
| AT5G47440 | -6.264 57 | 5.60E-245 | 1.22E-242 | 未知功能植物蛋白 Plant protein of unknown function |
| AT5G18560 | -6.261 66 | 0 | 0 | 转录因子PUCHI Transcription factor PUCHI |
| AT4G03950 | -6.210 38 | 8.56E-05 | 0.0001953 | 磷酸盐转运物类似蛋白1 Phosphate-translocator-like protein 1 |
| AT1G48325 | -6.197 16 | 6.03E-22 | 3.71E-21 | 未知蛋白 Unknown protein |
| AT4G09940 | -6.171 98 | 2.11E-265 | 5.57E-263 | 含有P环的核苷三磷酸水解酶超家族蛋白 P-loop containing nucleoside triphosphate hydrolases superfamily protein |
| AT5G13670 | -6.079 33 | 5.66E-06 | 1.43E-05 | WAT1相关蛋白WAT1-related protein |
| AT3G27590 | -6.069 53 | 2.34E-07 | 6.53E-07 | 未知蛋白 Unknown protein |
| AT3G15534 | -6.016 10 | 1.40E-06 | 3.71E-06 | 未知蛋白 Unknown protein |
| AT3G60650 | -6.006 11 | 1.12E-208 | 1.56E-206 | 未知蛋白 Unknown protein |
| AT5G56960 | -5.944 23 | 8.95E-73 | 2.02E-71 | 转录因子bHLH041 Transcription factor bHLH041 |
| AT5G42223 | -5.906 75 | 1.04E-19 | 5.83E-19 | 防御素类似蛋白114 Defensin-like protein 114 |
| AT1G44542 | -5.902 20 | 2.22E-46 | 2.95E-45 | 环化酶家族蛋白 Cyclase family protein |
| AT2G42430 | -5.901 55 | 2.27E-316 | 1.06E-313 | 转录因子LBD16 Transcription factor LBD16 |
| AT1G61750 | -5.891 57 | 2.42E-06 | 6.29E-06 | 分泌蛋白 Secretory protein |
| AT2G14960 | -5.876 69 | 0 | 0 | 生长素响应家族蛋白GH3.1 Auxin-responsive GH3 family protein GH3.1 |
| AT4G12490 | -5.874 92 | 1.09E-14 | 4.79E-14 | pEARLI1相似脂质转运蛋白2 pEARLI1-like lipid transfer protein 2 |
| AT3G62610 | -5.850 46 | 3.27E-184 | 3.55E-182 | 转录因子MYB11 Transcription factor MYB 11 |
| AT5G02900 | -5.846 07 | 7.03E-31 | 5.98E-30 | 细胞色素P450 96A13 Cytochrome P450 96A13 |
| AT4G08300 | -5.832 71 | 3.63E-10 | 1.23E-09 | WAT1相关蛋白 WAT1-related protein |
此外, 转录因子RSL4(AT1G27740)和MYB40(AT5G14340)的基因表达量增加, 编码转录因子TZF4 (AT1G03790), NF-YB家族蛋白(AT1G21970), bHLH家族蛋白(AT1G25310、AT4G37850、AT5G56960), LBD19 (AT2G45410), MYB62 (AT1G68320), C3H家族蛋白(AT1G29600)的基因表达量降低。
在不定芽再生阶段, 比较S3与C4表达量所得2 910个差异基因中有2 804条转录物具有具体功能定义, 共得到403个功能注释, 分别包含234个生物学过程亚类、42个细胞组分亚类, 127个分子功能亚类。图 3为不定芽再生阶段前20个显著富集(P < 0.05)的功能条目。其中激素刺激响应、生长素刺激响应、乙烯刺激响应、生长素内稳态和转录因子活性等是显著下调的富集功能条目。
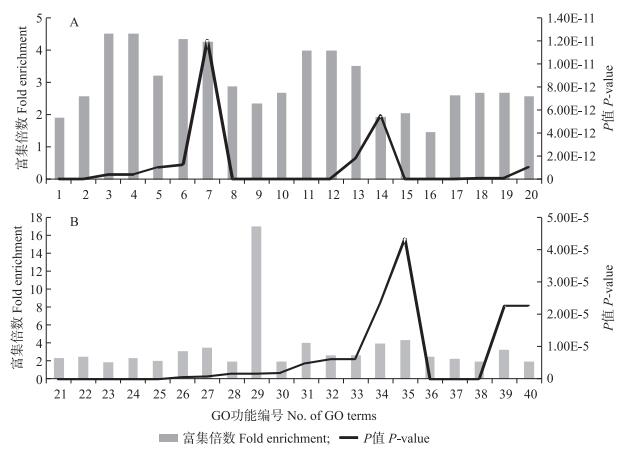
|
图 3 拟南芥不定芽再生阶段培养前20个显著富集的转录物GO功能条目
Figure 3 TOP 20 highly enriched GO terms during shoot regeneration of A.thaliana
A.显著上调的富集转录物功能条目。The upregulated gene subsets with annotated GO terms. 1.氧化还原 Oxidation reduction; 2.次级代谢 Secondary metabolic process; 3.过氧化氢分解 Hydrogen peroxide catabolic process; 4.过氧化氢细胞响应 Cellular response to hydrogen peroxide; 5.光合作用 Photosynthesis; 6.过氧化氢代谢过程 Hydrogen peroxide metabolic process; 7.糖苷代谢过程 Glycoside metabolic process; 8.四吡咯结合 Tetrapyrrole binding; 9.电子载体结合 Electron carrier activity; 10.亚铁血红素结合 Heme binding; 11.过氧化物酶活性 Peroxidase activity; 12.氧化还原酶活性 Oxidoreductase activity; 13.抗氧化活性 Antioxidant activity; 14.铁离子结合 Iron ion binding; 15.胞外区 Extracellular region; 16.内膜系统 Endomembrane system; 17.质外体 apoplast; 18.叶绿体类囊体膜 Chloroplast thylakoid membrane; 19.质体类囊体膜 Plastid thylakoid membrane; 20.类囊体膜 Thylakoid membrane. B.显著下调的富集转录物功能条目。The downregulated gene subsets with annotated GO terms. 21.有机物质响应 Response to organic substance; 22.激素刺激响应 Response to hormone stimulus; 23.转录调节 Regulation of transcription; 24.内源刺激响应 Response to endogenous stimulus; 25.转录 Rranscription; 26.生长素刺激响应 Response to auxin stimulus; 27.乙烯刺激响应 Response to ethylene stimulus; 28.依赖DNA的转录调节 Regulation of transcription, DNA-dependent; 29.生长素内稳态 Auxin homeostasis; 30.RNA代谢调节 Regulation of RNA metabolic process; 31.脂质定位 Lipid localization; 32.生长素调节信号 Hrmone-mediated signaling; 33.激素刺激的细胞响应 Cellular response to hormone stimulus; 34.脂质运输 Lipid transport; 35.生长素信号调节通路 Auxin mediated signaling pathway; 36.转录因子活性 Transcription factor activity; 37.转录调节子活性 Transcription regulator activity; 38.DNA结合 DNA binding; 39.脂质结合 Lipid binding; 40.胞外区 Extracellular region. |
通过对比生长素、细胞分裂素相关基因在2个阶段的表达量变化(图 4)。可知在愈伤组织形成阶段4个生长素合成相关家族基因YUC(YUCCA)表达量显著降低, 其中YUC5下调最明显, 但在不定芽再生阶段它们的变化并不明显; 5个生长素相关家族基因GH3(GH3.2、GH3.3、GH3.5、GH3.6和GH3.12)表达量增加, 其中GH3.3增加了约250倍, GH3.12增加了288倍, 但在不定芽再生阶段它们的表达量减少; 7个生长素响应基因表达量在愈伤组织形成阶段增加, 其中IAA19增加了21倍, IAA20增加了25倍, IAA30增加了64倍。另外, SAUR(SMALL AUXIN-UP RNAs)基因的表达达量在愈伤组织形成阶段都明显减少, 但在不定芽再生阶段表达量都明显增加。
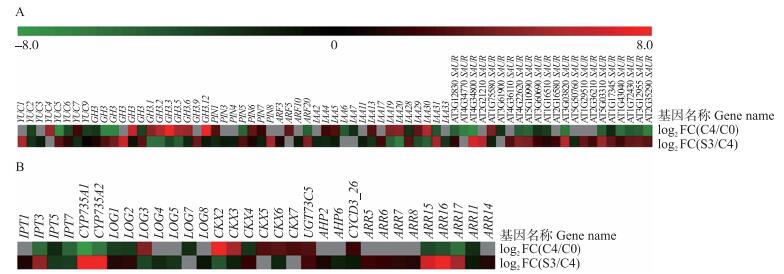
|
图 4 拟南芥愈伤组织形成阶段及不定芽再生阶段与生长素(A)和细胞分裂素(B)相关基因的表达量 Figure 4 Transcript levels of genes related to auxin(A)and cytokinin(B)during callus formation and shoot regeneration of A.thaliana |
异戊烯基转移酶(isopentenyltransferase, IPT)是细胞分裂素合成的关键酶, 在愈伤组织形成阶段IPT基因的表达量发生明显下降。另一个细胞分裂素合成的重要酶类细胞色素单氧酶P450(CYP735A1和CYP735A2)以及催化细胞分裂素从无活性向活性形式转化的LOG家族基因均出现了一定程度的下调。与此同时CKX2、CKX3、CKX5和CKX6等4个细胞分裂素氧化酶基因的表达量在愈伤组织形成阶段都出现了一定程度的上调, 其中CKX2、CKX3上调倍数最多:CKX2上调了约278倍, CKX3上调约40倍。当外植体移至SIM培养基后IPT1、IPT3、CYP735A1、CYP735A2、LOG1、LOG2的表达量出现明显增加, 特别是CYP735A1和CYP735A2, 分别上调了879和440倍。另外, CKX2、CKX3和CKX6表达量减少。A型ARR基因能被细胞分裂素快速诱导, 从图 4也可以看出A型ARR基因表达量在不定芽再生阶段均有所增加, 且ARR15和ARR16最为显著。
2.5 拟南芥芽顶端分生组织相关基因的表达芽顶端分生组织(shoot apical meristem, SAM)在植物的生长发育过程中具有重要的功能。在离体芽的再生过程中一些与SAM相关基因的表达也发生了改变(表 4)。其中:WUS(AT2G17950)的表达在不定芽再生阶段最为明显, 在愈伤组织形成阶段未检测到其表达。其次是RAP2.6, 它在不定芽再生阶段显著表达, 同时在愈伤组织形成过程中表达量显著降低。另外, CUC1、CUC2和CUC3在不定芽再生阶段均有表达, 但CUC2在2个阶段均出现表达上调, 且在愈伤组织形成阶段表达显著。ESR1(ENHANCER OF SHOOT REGENERATION 1)和PUCHI在愈伤组织形成阶段均显著表达。
| 基因名称 Gene short name |
基因ID Gene ID |
愈伤组织形成阶段 Callus formation |
不定芽再生阶段 Shoot regeneration |
|||
| 差异倍数 log2FC(C4/C0) |
错误出现率 FDR |
差异倍数 log2FC(S3/C4) |
错误出现率 FDR |
|||
| WUS | AT2G17950 | — | — | 7.188 693 | 1.84E-13 | |
| CUC1 | AT3G15170 | — | — | 2.687 777 | 4.84E-22 | |
| CUC2 | AT5G53950 | 6.003 334 | 1.75E-128 | 1.119 877 | 4.46E-27 | |
| CUC3 | AT1G76420 | — | — | 1.231 715 | 0.074 954 61 | |
| ESR1 | AT1G12980 | 5.019 877 | 0.000 542 341 | 1.956 636 | 0.000 030 8 | |
| RAP2.6 | AT1G43160 | -7.508 208 | 2.79E-67 | 5.187 398 | 7.63E-31 | |
| RAP2.6L | AT5G13330 | 2.028 171 | 9.51E-105 | 2.045 683 | 3.66E-159 | |
| BBM | AT5G17430 | 3.806 910 | 6.08E-251 | -3.687 839 | 3.87E-238 | |
| PUCHI | AT5G18560 | 5.372 501 | 0 | -6.261 660 | 0 | |
| CLE2 | AT4G18510 | -2.271 201 | 1.19E-69 | 2.428 083 | 1.83E-78 | |
| AGL24 | AT4G24540 | — | — | 5.238 352 | 2.49E-11 | |
| CLE41 | AT3G24770 | — | — | 2.306 391 | 3.42E-64 | |
| WOX4 | AT1G46480 | — | — | 2.431 537 | 7.21E-96 | |
| NAC2 | AT3G15510 | 2.808 793 | 8.01E-101 | -3.950 705 | 4.85E-146 | |
| WOX5 | AT3G11260 | 5.976 016 | 0 | -4.630 332 | 6.53E-253 | |
| AGL20 | AT2G45660 | -7.286 607 | 3.48E-27 | — | — | |
| 注:“—”未测出。“—”indicates no detection. | ||||||
愈伤组织形成和不定芽再生阶段是不定芽形成的2个关键阶段, 许多基因的表达情况在这2个阶段发生显著改变并决定了细胞去分化和再分化的命运, 因此我们对这2个阶段基因的差异表达情况进行了分析。王兴春等[20]曾利用RNA-Seq技术研究了以下胚轴为外植体的不定芽再生早期的基因表达情况, 选用的是CIM培养7 d和SIM培养2 d的材料。CIM培养4 d时外植体即可见愈伤组织的膨大, 我们利用CIM培养4 d和SIM培养3 d的根外植体材料可以更好地研究愈伤组织形成和不定芽再生阶段的基因表达情况。Che等[11]利用22 810个基因探针对拟南芥根外植体不同再生途径中的基因进行分析, 检测到不定芽形成途径中478个上调表达基因和397个下调表达基因。Xu等[16]利用22 746个基因探针对地上部外植体和根外植体在愈伤组织形成阶段的基因表达情况进行了分析, 检测到1 342个差异表达基因。由于基因芯片的局限性, 检测到的基因数目有限。我们利用RNA-Seq技术检测到愈伤组织形成阶段的4 332个差异表达基因与不定芽再生阶段的2 910个差异表达基因, 为研究不定芽再生过程的基因表达情况提供更有效的数据依据。我们还利用GO功能注释分类体系分别对2个阶段差异表达基因进行功能分析, 发现生长素和细胞分裂素的合成、代谢相关途径的差异表达基因最为丰富。
在愈伤组织形成阶段细胞分裂素分解代谢途径显著富集, CKX家族部分基因显著表达。与此同时, 我们发现细胞分裂素合成相关家族基因IPT, CYP735A1、CYP735A2以及LOG家族基因均出现了一定程度的表达下调。Takei等[21]通过RT-PCR发现用生长素处理拟南芥1 h CYP735A的表达量即明显下调。Werner等[22]发现生长素不仅影响细胞分裂素的生物合成, 而且还通过调节CKX家族基因来影响细胞分裂素的降解。因此, 我们认为这一现象与CIM中高浓度的生长素有关。在愈伤组织形成阶段, Che等[12]发现生长素响应基因AUX/IAA表达量增加, 我们对AUX/IAA家族基因进行了全面分析也发现多个IAA家族基因表达量增加。我们还发现与生长素合成相关的YUC家族基因表达量出现了一定程度的下调, 同时GH3家族部分基因表达量出现了显著增加。GH3家族基因编码吲哚乙酸氨基酸合成酶催化IAA与氨基酸的结合, 从而改变IAA的活性[23]。这些基因表达量的改变可能也与CIM培养基中高浓度生长素有关。这些变化可能是CIM培养基上高浓度生长素负反馈调节的结果, 从而维持愈伤组织形成阶段细胞分裂素和生长素的动态平衡。
Che等[11]研究发现, ARR15、ARR16在不定芽再生途径中表达量增加。我们在不定芽再生阶段也检测到它们表达量增加。王兴春等[20]检测到在不定芽再生阶段IPT5的表达量下降, 但并未发现其他IPT基因的变化。我们除检测到IPT5、IPT7的表达量减少, 还发现与细胞分裂素合成及活性有关的CYP735A1、CYP735A2和LOG家族基因在该阶段出现了明显的表达量增加, 与细胞分裂素代谢相关的CKX家族部分基因表达量减少。虽然有研究表明细胞分裂素在4 h内即可抑制IPT1、IPT3、IPT5和IPT7基因的表达[24], 但随着在SIM上培养时间的增加, 外源细胞分裂素的含量可能逐渐减少。为了维持促进不定芽形成的细胞分裂素水平, 其他一些与细胞分裂素合成相关的基因表达量开始增加。
SAM的发育与不定芽的再生关系密切, 我们发现有多个基因在愈伤组织形成阶段表达量增加, 可见在第4天虽然外植体还只是处于愈伤组织阶段, 但与芽顶端分生组织相关的多个基因已经开始表达, 外植体已经获得形成芽的能力。CUC2作为根外植体获得芽再生形成能力的标志基因[14], 在愈伤组织形成阶段表达量显著上调。在不定芽再生阶段有10个与SAM相关的基因表达上调, 4个基因表达下调。其中WUS、RAP2.6、AGL24上调幅度较大, 可以看出WUS在芽分生组织形成的基因调控过程中具有重要地位[25]。
转录因子AP2/EREBP家族基因参与多个生长发育过程, 该家族有144个成员[26], 研究表明多个AP2家族成员参与了不定芽再生过程, 如ESR1[27]。Banno等[27]认为在外植体获得器官形成能力后ESR1可以促进芽的形成, 我们在愈伤组织形成和不定芽再生阶段均检测到ESR1的表达, 且ESR1的表达在愈伤组织形成阶段更显著。Che等[11]研究发现转录因子RAP2.6L在不定芽形成过程出现了上调。我们也检测到RAP2.6L的上调表达, 但不同的是我们在愈伤组织形成阶段和不定芽再生早期阶段均检测到RAP2.6L上调表达。同时, 还发现另一个AP2转录因子家族成员RAP 2.6在愈伤组织形成阶段表达量显著减少, 而在不定芽再生阶段表达量显著增加。我们推测可能RAP2.6更特异性地参与芽再生的调控过程。在不定芽再生阶段另一个AP2转录因子PUCHI及其共同作用元件LBD16表达量明显减少。PUCHI会限制早期侧根原基的细胞增殖, 并与LBD16、LBD18共同作用来调控侧根原基的发育及侧根的形成[28]。Yadav等[29]认为器官形成早期的主要调控过程和愈伤组织形成过程是一个和侧根形成类似的过程。因此PUCHI的表达量减少可能解除了对侧根原基增殖的限制, 从一方面促进了不定芽的再生。
除了与不定芽再生有关的转录因子编码基因ESR1、PUCHI、RAP2.6、LBD16外, 我们还发现多个转录因子基因表达量发生改变, 它们属于不同转录因子家族, 如Nin-like家族、LBD家族、bHLH家族、MYB家族和C3 H家族等。拟南芥编码1 500个转录因子[15]且芽的再生是一个非常复杂的过程。因此, 在愈伤组织形成阶段和不定芽再生阶段必然还蕴含大量的转录因子调度调节以及各生理信号的交互作用有待我们进一步去发掘, 以期解释其内部的分子生物学机制。
| [1] | Deklerk G J, Amholdt-Schmitt B, Lieberei R, et al. Regeneration of roots, shoots and embryos:physiological, biochemical and molecular aspects[J]. Biologia Plantarum, 1997, 39(1): 53-66. DOI: 10.1023/A:1000304922507 |
| [2] | Sangwan R S, Harada H. Chemical regulation of callus growth, organogenesis, plant regeneration, and somatic embryogenesis in Antirrhinum majus tissue and cell cultures[J]. Journal of Experimental Botany, 1975, 26(6): 868-881. DOI: 10.1093/jxb/26.6.868 |
| [3] | Dhaliwal H S, Ramesar-Fortner N S, Yeung E C, et al. Competence, determination, and meristemoid plasticity in tobacco organogenesis in vitro[J]. Canadian Journal of Botany, 2003, 81(6): 611-621. DOI: 10.1139/b03-047 |
| [4] | Feldmann K A, Marks M D. Rapid and efficient regeneration of plants from explants of Arabidopsis thaliana[J]. Plant Science, 1986, 47(1): 63-69. |
| [5] | Sangwan R S, Bourgeois Y, Brown S, et al. Characterization of competent cells and early events of Agrobacterium-mediated genetic transformation in Arabidopsis thaliana[J]. Planta, 1992, 188(3): 439-456. |
| [6] | Valvekens D, Montagu M V, Lijsebettens M V, et al. Agrobacterium tumefaciens mediated transformation of Arabidopsis thaliana root explants using kanamycin selection[J]. Proc Natl Acad Sci USA, 1988, 85(15): 5536-5540. DOI: 10.1073/pnas.85.15.5536 |
| [7] | Sangwan R S, Sangwan-Norreel B S, Harada H, et al. In vitro techniques and plant morphogenesis:fundamental aspects and practical applications[J]. Plant Biotechnology, 1997, 14(2): 93-100. |
| [8] | Catterou M, Dubois F, Smets R, et al. hoc:an Arabidopsis mutant overproducing cytokinins and expressing high in vitro organogenic capacity[J]. The Plant Journal, 2002, 30(3): 273-287. DOI: 10.1046/j.1365-313X.2002.01286.x |
| [9] | Ren B, Liang Y, Deng Y, et al. Genome-wide comparative analysis of type-A Arabidopsis response regulator genes by overexpression studies reveals their diverse roles and regulatory mechanisms in cytokinin signaling[J]. Cell Research, 2009, 19(10): 1178-1190. DOI: 10.1038/cr.2009.88 |
| [10] | Gordon S P, Heisler M G, Reddy G V, et al. Pattern formation during de novo assembly of the Arabidopsis shoot meristem[J]. Development, 2007, 134(19): 3539-3548. DOI: 10.1242/dev.010298 |
| [11] | Che P, Lall S, Nettleton D, et al. Gene expression programs during shoot, root, and callus development in Arabidopsis tissue culture[J]. Plant Physiology, 2006, 141(2): 620-637. |
| [12] | Che P, Gingerich D J, Lall S, et al. Global and hormone-induced gene expression changes during shoot development in Arabidopsis[J]. Plant Cell, 2002, 14(11): 2771-2785. DOI: 10.1105/tpc.006668 |
| [13] | Su Y H, Chen Z J, Su Y X, et al. Pattern analysis of stem cell differentiation during in vitro Arabidopsis organogenesis[J]. Frontiers of Biology, 2010, 5(5): 464-470. DOI: 10.1007/s11515-010-0820-0 |
| [14] | Motte H, Verstraeten I, Werbrouck S, et al. CUC2 as an early marker for regeneration competence in Arabidopsis root explants[J]. Journal of Plant Physiology, 2011, 168(13): 1598-1601. DOI: 10.1016/j.jplph.2011.02.014 |
| [15] | Riechmann J L, Heard J, Martin G, et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499): 2105-2110. DOI: 10.1126/science.290.5499.2105 |
| [16] | Xu K, Liu J, Fan M, et al. A genome-wide transcriptome profiling reveals the early molecular events during callus initiation in Arabidopsis multiple organs[J]. Genomics, 2012, 100(2): 116-124. |
| [17] | Cary A J, Che P, Howell S H. Development events and shoot meristem gene expression patterns during shoot development in Arabidopsis thaliana[J]. The Plant Journal, 2002, 32(6): 867-877. DOI: 10.1046/j.1365-313X.2002.01479.x |
| [18] | Schuster J, Knill T, Reichelt M, et al. BRANCHED-CHAIN AMINOTRANSFERASE4 is part of the chain elongation pathway in the biosynthesis of methionine-derived glucosinolates in Arabidopsis[J]. The Plant Cell, 2006, 18(10): 2664-2679. DOI: 10.1105/tpc.105.039339 |
| [19] | Naseer S, Lee Y, Lapierre C, et al. Casparian strip diffusion barrier in Arabidopsis is made of a lignin polymer without suberin[J]. Proc Natl Acad Sci USA, 2012, 109(25): 10101-10106. DOI: 10.1073/pnas.1205726109 |
| [20] |
王兴春, 杨致荣, 张树伟, 等. 拟南芥不定芽发生早期的数字基因表达谱分析[J].
生物工程学报, 2013, 29(2): 189-202.
Wang X C, Yang Z R, Zhang S W, et al. Digital gene expression profiling analysis of the early adventitious shoot formation in Arabidopsis thaliana[J]. Chinese Journal of Biotechnology, 2013, 29(2): 189-202. (in Chinese with English abstract) |
| [21] | Takei K, Yamaya T, Sakakibara H. Arabidopsis CYP735A1 and CYP735A2 encode cytokinin hydroxylases that catalyze the biosynthesis of trans-zeatin[J]. Journal of Biological Chemistry, 2004, 279(40): 41866-41872. DOI: 10.1074/jbc.M406337200 |
| [22] | Werner T, Köllmer I, Bartrina I, et al. New insights into the biology of cytokinin degradation[J]. Plant Biology, 2006, 8: 371-381. DOI: 10.1055/s-2006-923928 |
| [23] | Hagen G, Kleinschmidt A, Guilfoyle T. Auxin-regulated gene expression in intact soybean hypocotyl and excised hypocotyl sections[J]. Planta, 1984, 162(2): 147-153. |
| [24] | Miyawaki K, Matsumoto-Kitano M, Kakimoto T. Expression of cytokinin biosynthetic isopentenyltransferase genes in Arabidopsis:tissue specificity and regulation by auxin, cytokinin, and nitrate[J]. The Plant Journal, 2004, 37(1): 128-138. DOI: 10.1046/j.1365-313X.2003.01945.x |
| [25] | Traas J, Monéger F. Systems biology of organ initiation at the shoot apex[J]. Plant Physiology, 2010, 152(2): 420-427. DOI: 10.1104/pp.109.150409 |
| [26] | Riechmann J L, Meyerowitz E M. The AP2/EREBP family of plant transcription factors[J]. Biological Chemistry, 1998, 379(6): 633-646. |
| [27] | Banno, H, Ikeda Y, Niu Q W, et al. Overexpression of Arabidopsis ESR1 induces initiation of shoot regeneration[J]. The Plant Cell, 2001, 13(12): 2609-2618. DOI: 10.1105/tpc.13.12.2609 |
| [28] | Kang N Y, Lee H W, Kim J. The AP2/EREBP gene PUCHI co-acts with LBD16/ASL18 and LBD18/ASL20 downstream of ARF7 and ARF19 to regulate lateral root development in Arabidopsis[J]. Plant and Cell Physiology, 2013, 54(8): 1326-1334. DOI: 10.1093/pcp/pct081 |
| [29] | Yadav S R, Bishopp A, Helariutta Y, et al. Plant development:early events in lateral root initiation[J]. Current Biology, 2010, 20(19): R843-R845. DOI: 10.1016/j.cub.2010.09.010 |




