文章信息
- 刘子文, 胡曼曼, 王致远, 张瑜竣, 谷晗, 游佳, 王益华, 江玲, 刘玲珑
- LIU Ziwen, HU Manman, WANG Zhiyuan, ZHANG Yujun, GU Han, YOU Jia, WANG Yihua, JIANG Ling, LIU Linglong
- 水稻心白突变体whc的理化性质和基因定位
- Physicochemical properties and gene mapping of white-core mutant whc in rice
- 南京农业大学学报, 2018, 41(2): 231-239
- Journal of Nanjing Agricultural University, 2018, 41(2): 231-239.
- http://dx.doi.org/10.7685/jnau.201706007
-
文章历史
- 收稿日期: 2017-06-07
水稻(Oryza sativa)是世界上重要的粮食作物之一, 而淀粉是水稻种子中主要的组成成分, 约占水稻胚乳干物质总量的80%[1-2], 因此淀粉的积累对水稻产量[3]和品质的形成具有极其重要的经济价值。淀粉的形成是一个复杂的酶促过程, 最后以直链淀粉和支链淀粉2种形式在造粉体中贮藏[4-5]。支链淀粉的结构有助于淀粉颗粒结晶结构的形成[6]。贮藏淀粉的形成伴随着胚乳细胞内造粉体和淀粉粒的增殖和发育[7-9], 需要多种酶和调控因子的协同作用[10-13]。目前, 人们对该过程的了解并不十分透彻, 因此研究水稻淀粉的生物合成具有一定的理论意义。
淀粉合成、代谢相关基因的突变多会引起淀粉含量或结构的改变。目前已报道的淀粉相关突变体主要包括粉质、皱缩、糯性、垩白等类型。其中已报道的粉质突变体有flo1、flo2、flo3、flo4、flo5、flo6、flo7等。突变体flo1定位于第5染色体, 其突变后直链淀粉含量增加, 粒型变化不明显[14]。flo2突变体籽粒变小、淀粉品质下降、直链淀粉含量降低[15]; FLO2是一个植物特异性表达的基因, 其编码的蛋白具有一个三角状四肽重复结构域, 与碱性螺旋-环-螺旋结构相互作用共同调节贮藏蛋白和淀粉合成相关基因的表达[15]。FLO3基因突变后, 其表型与flo2相似[16], 该突变体相对分子质量为16×103的球蛋白含量降低, 从而导致胚乳粉质。flo4是一个T-DNA插入突变体, 插入突变后使丙酮酸磷酸双激酶(OsPPDKB)失去功能, 导致胚乳中心部位粉质不透明[17]。flo5由于OsSSc Ⅲa基因的缺失, 导致胚乳呈现出心白的表型[18-19]。组织培养过程中获得的flo6突变体, 其N端具有与淀粉异构酶ISA1结合的功能域, 从而调控淀粉合成[20]。flo7突变体的胚乳外围呈现粉质状, 中心部分表现正常[21]; FLO7编码一个包含高度保守结构域DUF1338的未知功能蛋白。尽管目前已经克隆以上基因, 但淀粉合成还有许多未知事件。因此, 鉴定新的粉质相关突变体并克隆其基因, 进一步了解淀粉合成与代谢途径, 有助于稻米品质改良。
本研究对粳稻品种‘中花11’(ZH11)胚乳心白突变体(white core, whc)的形态学特征、理化性质、淀粉结构等性状进行分析, 将突变控制基因定位于第4染色体, 并克隆该基因, 为淀粉合成与代谢途径的研究提供了新的资源。
1 材料与方法 1.1 试验材料突变体whc是粳稻品种‘中花11’(ZH11)经60Co-γ辐照诱变得到的一个稳定遗传的胚乳心白突变体材料。将突变体whc与‘N22’配制F1杂交组合, 获得F1植株的种子(F2代), 从中挑选出表现为胚乳心白的籽粒, 用于WHC基因的遗传连锁分析。亲本和F1植株均于2014年正季种植于南京农业大学土桥实验基地。于抽穗期, 随机选取野生型和突变体各20株, 考查株高、穗长、有效分蘖数、千粒质量等农艺性状, 计算平均值和标准差。
1.2 成熟种子直链淀粉含量和蛋白质含量测定直链淀粉含量测定采用《米质测定方法:NY 83—1988》[22], 蛋白质含量测定利用FOSS公司生产的近红外快速含量分析仪, 参照孙成效等[23]方法, 每个样品重复3次, 取平均值。
1.3 成熟种子支链淀粉链长分布的测定利用DNA测序的荧光糖电泳法, 用APTS标记的麦芽七糖作为标准样, 野生型与突变体whc种子的米粉样品经处理后, 使用3130XL测序仪(Applied Biosystems, USA)电泳。电泳完毕后, 利用GeneMapper 4.0软件导出数据, 并用Microsoft Excel® 2007进行数据分析。
1.4 成熟种子横截面扫描电镜观察分别选取野生型和突变体干燥的成熟种子, 用刀背在其中部敲打使之自然断裂, 取断裂后的籽粒为样品, 以导电胶粘着于样品台上, 使横断面向上, 利用真空镀膜仪(Hitachi, Japan)对横断面镀金, 使用S-3000N(Hitachi, Japan)型扫描电镜, 于20 kV的加速电压下对米粒横截面的不同部位进行观察和拍照[24]。
1.5 米粉的尿素膨胀在1.5 mL Ep管中分别加入1 mL不同浓度的尿素溶液(浓度为0~9 mol·L-1, 用乙酸调节pH=6.0)。在相同尿素浓度的Ep管中分别加入20 mg野生型和whc的精米粉, 混匀, 25 ℃孵育24 h。室温下8 000 r·min-1离心20 min, 室温静置1 h。计算Ep管中可溶尿素部分的体积, 淀粉颗粒的膨胀体积=总体积-可溶部分体积。
1.6 突变基因whc的精细定位采用SDS法[25]提取基因组DNA。利用本实验室已有的水稻分子标记库筛选出ZH11和‘N22’之间具有多态性的SSR和InDel标记, 从中选出均匀分布于12条水稻染色体的标记, 间距在10~15 cM之间, 从F2群体中选择22个极端胚乳心白个体进行初步连锁分析。确定连锁标记后, 进一步利用299个胚乳心白个体进行验证并缩小连锁区间。在连锁标记所在染色体区段利用Primer Premier 5.0软件开发新的标记, 筛选多态性标记, 利用重要的交换单株和新选取的130个极端胚乳心白个体进行精细定位。采用dCAPS Finder 2.0软件(http://helix.wustl.edu/dcaps/dcaps.html)将包含突变位点前后各25个碱基的野生型和突变体序列输入软件开发dCAPS标记, 另一侧的引物采用Primer Premier 5.0设计。精细定位引物和dCAPS标记序列见表 1。
| 标记 Marker |
正向引物序列(5′ →3′) Forward primer sequence |
反向引物序列(5′ →3′) Revers primer sequence |
产物大小/bp Product size |
限制性内切酶 Restriction enzyme |
| d1 | GTCATTAGGTCCTTTAAGCACA | CCTCTCTACCCATCTAGATC | 180 | |
| Z-1 | CTCGGCTAGCTTGCGTTTCTTGC | CCGTTAGCGAGGTTTCCGTTCC | 131 | |
| Z-14 | CAGGACGAGATCCAAGCCAAGG | TCGTCCTCGCCACCACTACTCC | 143 | |
| I4-10 | TCCGTTGATATCGTGCCTAA | GGATGTACAGGTGGATTGAAGG | 150 | |
| I4-15 | ATGTAACCCGGCCAGAGTG | CCATTAACTGGTCGGAATCG | 160 | |
| RM127 | GTGGGATAGCTGCGTCGCGTCG | AGGCCAGGGTGTTGGCATGCTG | 210 | |
| RM255 | TGTTGCGTGTGGAGATGTG | CGAAACCGCTCAGTTCAAC | 150 | |
| RM317 | CATACTTACCAGTTCACCGCC | CTGGAGAGTGTCAGCTAGTTGA | 108 | |
| RM349 | TTGCCATTCGCGTGGAGGCG | GTCCATCATCCCTATGGTCG | 140 | |
| dCAPS | GATGACATGCAAAATATGTC | GGAACCTCTCTACCCATCTAGATC | 136 | BglⅡ |
利用实时荧光定量PCR技术进行水稻淀粉合成相关基因的表达分析。总RNA的提取采用RNA Prep Pure Plant Kit(TIANGEN), 然后使用SuperScriptⅡ反转录酶(TaKaRa)反转录得到cDNA, 采用荧光染料预混液SYBR® Premix Ex TaqTM Ⅱ(TaKaRa), 在ABI Prism 7500的定量RT-PCR仪上进行qRT-PCR反应。反应体系为1.8 μL cDNA模板, 10 μL 2×SYBR® Premix Ex TaqTM Ⅱ, 10 μmol·L-1 PCR正反向引物各0.8 μL, ddH2O补足至20 μL。反应程序采用两步法:第一步95 ℃预变性30 s, 95 ℃ 5 s, 60 ℃ 30 s, 循环数为40;第二步95 ℃ 15 s, 60 ℃ 1 min, 95 ℃ 15 s。淀粉合成相关基因的扩增引物序列来自Ohdan等[26]的研究报道, 用2-ΔΔCT法对实时定量PCR的结果进行分析[27], 以水稻Actin基因作为内参。
2 结果与分析 2.1 野生型与突变体whc的表型分析相比于野生型, 成熟的突变体whc的种子胚乳表现为中心粉质不透明(图 1-A, B)。在主要籽粒性状方面(图 1-C, D, E), 突变体whc的千粒质量明显减少, 下降了约14.5%;而粒宽、有效分蘖数则有不同程度的增加; 此外, 突变体whc的株高、穗长等农艺性状与野生型相比没有明显差异(表 2)。

|
图 1 野生型ZH11与突变体whc的表型比较 Figure 1 Phenotypic comparison of the wild type ZH11(WT)and whc A.糙米表型, B.胚乳横截面, 标尺均为1 mm; C.粒长; D.粒宽; E.千粒质量。**P < 0.01。下同。 A.Appearance of brown rice, B.Cross sections of endosperm, bar=1 mm; C.Grain length; D.Grain width; E.1 000-grain weight. **P < 0.01. The same as follows. |
| 农艺性状Agronomic traits | ZH11 | whc |
| 株高/cm Plant height | 122.25±3.92 | 102.00±10.69 |
| 穗长/cm Panicle length | 24.57±2.13 | 21.85±2.58 |
| 每株有效分蘖数No.of effective panicles per plant | 10.85±3.41 | 13.57±8.72* |
| 剑叶长/cm Flag leaf length | 29.11±5.36 | 28.56±5.39 |
| 剑叶宽/cm Flag leaf width | 1.67±0.11 | 1.44±0.21 |
| 倒二叶长/cm Penultimate leaf length | 44.03±5.71 | 43.19±8.43 |
| 倒二叶宽/cm Penultimate leaf width | 1.28±0.19 | 1.10±0.13 |
| 注: *表示野生型与突变体在0.05水平差异显著。*means significant difference between the wild type and whc mutant at 0.05 level. | ||
对成熟种子稻米品质分析结果显示, 相对于野生型, 突变体whc总蛋白质含量变化不显著(图 2-A), 但直链淀粉含量显著降低, 减少了约24.7%(图 2-B)。由此可见, WHC基因的突变主要影响种子中直链淀粉的积累。
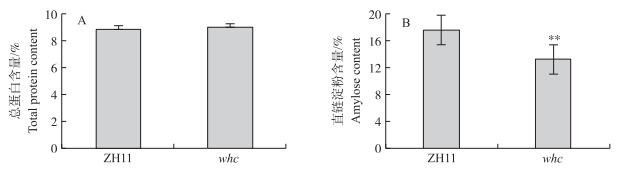
|
图 2 野生型ZH11与whc的总蛋白和直链淀粉含量 Figure 2 Total protein and amylose contents of wild type ZH11 and whc |
野生型和突变体whc成熟种子横断面的扫描电镜结果显示:野生型种子淀粉颗粒呈多面体结构, 形状规则, 排列紧密充实(图 3-A, B, C); 而突变体whc种子心白部位淀粉粒呈球形或不规则形状, 大小不一, 淀粉粒之间距离较大, 排列疏松(图 3-D, E, F)。因此在光线通过时会发生散射, 导致whc突变体籽粒外观呈不透明表型。表明WHC基因的突变导致种子胚乳中淀粉颗粒结构的异常。

|
图 3 野生型和突变体whc的扫描电镜观察 Figure 3 Observation of scanning electron micrographs of wild type and whc mutant A~C.野生型胚乳的扫描电镜观察; D~F.whc胚乳的扫描电镜观察。B和E分别是对应A和D红色框部分的扩大图, C和F分别是对应B和E中红色框部分的扩大图。 A-C.Scanning electron micrographs of wild type; D-F.Scanning electron micrographs of whc mutant. Panels B and E are the enlargement corresponding to red boxes in panels A and D respectively, and panels C and F are the enlargement corresponding to red boxes in panels B and E respectively. |
利用DNA测序的荧光糖电泳法, 对野生型和突变体whc种子的支链淀粉链长分布进行测定, 结果显示:与野生型相比, 突变体聚合度(degree of polymerization, DP)为8~9的短链及13~14的中短链比例有所减少, 其余聚合度的链长比例均有所增加(图 4-A, B)。因此, WHC基因的突变会引起支链淀粉结构的改变。
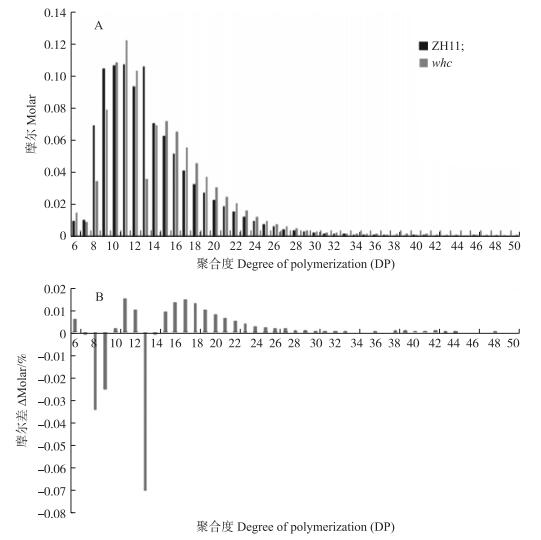
|
图 4 野生型和突变体whc的支链淀粉链长分布 Figure 4 Chain length distribution profiles of amylopectin in whc and wild type endosperm A.whc和野生型的聚合度; B.whc与ZH11聚合度差值。 A.Degree of polymerization of whc and its wild type; B.Difference of degree of polymerization between whc and ZH11. |
通过测定样品的膨胀体积来衡量淀粉粒的溶解度。从图 5可见:在低浓度尿素溶液(0~5 mol·L-1)中, 突变体和野生型的膨胀体积几乎没有差别。在6 mol·L-1时, 两者的溶解量出现差异; 随着尿素浓度的继续增加, 野生型和突变体的膨胀体积间差异随之增大。尿素膨胀结果表明, whc突变体中支链淀粉结构发生改变。

|
图 5 野生型和突变体whc籽粒中淀粉的尿素溶解性比较 Figure 5 Comparison of gelatinization properties in wild type and mutant whc starch granules A.野生型与突变体米粉在不同浓度尿素溶液中的膨胀, 尿素溶度为0~9 mol·L-1。五角星表示该浓度下突变体与野生型存在差异。B.不同尿素浓度下野生型和突变体米粉的膨胀体积比较(n=3)。*P < 0.05, **P < 0.01。下同。 A.Gelatinization properties of wild type and whc starch powder in urea solutions(from 0-9 mol·L-1). The star indicates significant difference between wild type and whc at the concentration. B The swelling volume of wild type and whc starch powder in urea solutions at various concentrations(n=3).*P < 0.05, **P < 0.01. The same as follows. |
收获whc与‘N22’所配制的F1植株的种子, 脱壳后观察到部分种子表现为类似whc的胚乳心白表型。从F2代种子中挑选出22粒突变体表型的种子, 进行连锁分析, 仅发现一处连锁位点, 初步将WHC定位在第4染色体长臂上的I4-10与I4-15分子标记之间(图 6-A)。继续扩大定位群体, 利用筛选出的多态性标记, 进一步缩小定位区间(图 6-B)。最终利用1 130个极端个体, 将WHC定位在RM349与d1分子标记之间的158 kb内(图 6-C)。

|
图 6 基因WHC的精细定位 Figure 6 Fine mapping of WHC A.基因WHC与标记I4-10和I4-15连锁; B.利用299个隐性分离单株将WHC定位到RM127与I4-15两个引物之间; C.利用1 130个隐性分离单株将WHC定位到158 kb的区间内。 A.WHC was linked with markers I4-10 and I4-15;B.WHC was located between markers RM127 and I4-15 based on 299 segregated recessive plants; C.WHC was located in a 158 kb region based on 1 130 segregated recessive plants. |
通过NCBI和Gramene(http://www.gramene.org/)网站预测, 该区间内有20个基因, 7个为假想蛋白, 其余注释为3-酮脂酰载体蛋白合酶、2OG-Fe(Ⅱ)加氧酶、环核苷酸门控通道蛋白、异戊烯转移酶、SAM同源结构域包含蛋白、富脯氨酸类蛋白、环氧水解酶、外被体ε亚基、氯通道电压门控家族蛋白、C2结构域包含蛋白、三角状四肽类螺旋结构域包含蛋白、VQ结构域包含蛋白和细胞循环控制蛋白。对该区间内的一些基因进行测序, 发现whc中Os04g0645100基因编码区与野生型存在一处非同义突变。Os04g0645100大小为13 kb, 共有23个外显子和22个内含子, 包含一个植物特异的高度保守的TPR(三角状四肽重复)基序。该基因开放阅读框(ORF)的第14个外显子上第214个碱基发生了由A到T的突变(图 7-A, B), 导致其编码的精氨酸突变为终止密码子, 蛋白翻译提前终止。

|
图 7 突变体基因WHC的候选基因分析 Figure 7 Analysis of candidate gene for WHC A. WHC基因的结构与突变的位置; B. WHC基因发生了A到T(红色字体)的单碱基突变; C. dCAPS标记的验证。BglⅡ可以酶切野生型ZH11, 但无法酶切whc与极端隐性单株(1~9泳道)。 A. Structure of WHC gene and mutation site; B. A single base mutation of A to T(in red)in WHC; C. Validation of dCAPS marker. ZH11 could be cut by BglⅡ, but whc and extreme recessive plants couldn′t(lane 1 to 9). |
通过查阅文献, 发现该基因是一个已经报道的粉质胚乳基因FLO2[15]。为了进一步验证该测序结果, 我们根据突变位点设计了dCAPS标记(含有BglⅡ酶切位点), 对F2后代进行共分离验证(图 7-C)。酶切结果显示, 野生型可以被BglⅡ酶酶切, 而突变体亲本及纯合隐性单株均无法被酶切, 因此该位点在F2后代中与WHC共分离。由此确定FLO2基因即为WHC的目标基因。
2.7 突变体whc种子中淀粉合成相关基因的表达分析突变体whc的成熟种子中存在明显的心白表型, 猜测其淀粉合成可能受到一定程度的影响。利用定量RT-PCR方法对突变体中与淀粉合成相关基因的表达进行分析。结果表明:突变体whc中编码ADP-葡萄糖焦磷酸化酶大小亚基的OsAGPS2b(编码小亚基2Ba)、OsAGPL1(编码大亚基Ⅰ)以及OsAGPL2(编码大亚基Ⅱ)的基因表达水平极显著下调; 编码淀粉分支酶ⅡA的OsBE Ⅱa基因表达水平显著下调(图 8), 这与上述结果(图 3-B)中突变体的支链淀粉长链的聚合度比例增加相符。由此可知, 突变体whc中ADP-葡萄糖焦磷酸化酶大小亚基及其淀粉分支酶基因的表达异常, 进而可能影响其淀粉的合成。此外, 与野生型相比, 突变体中编码异淀粉酶Ⅰ(OsISA1)、酶Ⅱ(OsISA2)和酶Ⅲ(OsISA3), 腺苷二磷酸葡萄糖焦磷酸化酶(AGPase)小亚基Ⅰ(OsAGPS1)和大亚基Ⅲ(OsAGPL3), 淀粉合酶Ⅱa(OsSS Ⅱa)以及颗粒结合型淀粉合酶Ⅱ(OsGBSS Ⅱ)均明显下调; 而ADP-葡萄糖焦磷酸化酶小亚基Ⅱa(OsAGPS2a)、大亚基Ⅳ(OsAGPSL4), 淀粉合酶Ⅰ(OsSS1)、Ⅱb(OsSS Ⅱb)、Ⅱc(OsSS Ⅱc)、Ⅲb(OsSS Ⅲb)、Ⅳa(OsSS Ⅳa)、Ⅳb(OsSS Ⅳb), 颗粒结合型淀粉合酶Ⅰ(OsGBSS1)和淀粉分支酶Ⅰ(OsBE1)的表达均没有显著变化。
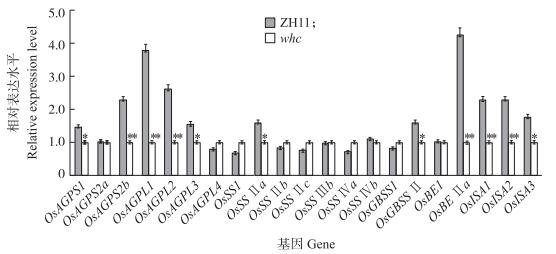
|
图 8 与淀粉合成相关基因的定量PCR分析 Figure 8 Real-time PCR analysis of starch synthesis-related genes |
粉质胚乳突变体是一类常见的胚乳突变体。当胚乳中蛋白质体和淀粉体充实不足, 相互间存在的空气会形成一种光学特性, 从而表现出粉质不透明状[28]。种子的发育过程, 是淀粉贮藏和蛋白快速、高水平积累的过程, 该发育进程的调控机制目前仍有很多未知因素[29]。
本研究中, 我们通过筛选60Co-γ诱变粳稻品种‘中花11’的突变体库, 获得一个稳定遗传的胚乳中心粉质(心白)突变体whc。whc突变体的粒长无明显变化, 千粒质量降低14.5%, 粒宽增加。与野生型相比, whc的直链淀粉含量降低24.7%, 总蛋白质含量无明显差异, 支链淀粉链长分布组成改变; whc突变体的心白部分淀粉颗粒大小不一、排列疏松, 这可能是导致其种子心白表型的直接原因。
利用图位克隆的方法, 我们发现whc突变体中一个已报道的具有TPR基序的FLO2基因发生了单碱基的替换, 导致蛋白翻译提前终止。FLO2在发育中的种子和叶中特异高表达, flo2突变降低了胚乳中储存淀粉和储存蛋白的含量; 此外, 突变体flo2中粒长、粒宽、粒厚、千粒质量都低于野生型[15]。由于whc为FLO2的一个新的等位变异, 我们推测由于突变位点不同, 从而导致理化性质和农艺性状有所不同。Wu等[30]报道3个新的flo2等位突变体, 由于环境和遗传背景的差异, 导致产生与野生型不同的外观、直链淀粉含量、支链淀粉链长结构等理化性质, 本研究的结果与之相一致。
whc突变体胚乳中储存淀粉的结构与野生型有很大的差异。一些涉及淀粉合成的相关基因表达量显著降低。AGPase活性强烈影响谷物淀粉的产量, 其突变往往导致淀粉无法正常形成[11], 而whc突变体中编码AGPase大小亚基的基因表达均下调。ISA1具有淀粉脱支酶的活性, 对于形成精密的支链淀粉结构至关重要[7], whc突变体中ISA1表达下调可能影响了支链淀粉的结构。BEⅡa是一类淀粉分支酶, 倾向于转移葡聚糖中短链(DP 6-15)[31], whc突变体中BE Ⅱa基因表达量显著下调, 这可能是造成突变体中支链淀粉链长成簇分布变化的原因。因此, whc胚乳中淀粉颗粒异常与这些基因的表达下调直接或间接相关。
WHC具有一个由34个串联重复氨基酸组成的TPR基序, 该基序含有螺旋-转角-螺旋结构(bHLH), 介导蛋白-蛋白之间的相互作用[32-33]。我们猜测whc在转录复合体中通过与bHLH的相互作用, 影响水稻胚乳的发育, 从而直接或间接调节贮藏淀粉和蛋白的积累。
目前关于粉质胚乳突变体虽有一些研究报道, 但粉质胚乳的形成机制和调控的分子机制仍不是十分清楚。本研究发现的WHC基因作为FLO2的一个新的等位基因, 为进一步阐明水稻淀粉形成的分子机制奠定了基础; 同时该突变体具有与报道的flo2不同的理化性质, 可对稻米品质的进一步改良提供创新种质。
农业部长江中下游粳稻生物学与遗传育种重点实验室和江苏省现代作物生产协同创新中心对本研究给予支持, 谨致谢意。
| [1] |
方鹏飞, 李三峰, 焦桂爱, 等. 水稻粉质胚乳突变体flo7的理化性质及基因定位[J].
中国水稻科学, 2014, 28(5): 447-457.
Fang P F, Li S F, Jiao G A, et al. Physicochemical property analysis and gene mapping of a floury endosperm mutant flo7 in rice[J]. Chinese Journal of Rice Science, 2014, 28(5): 447-457. (in Chinese with English abstract) |
| [2] | Giroux M J, Boyer C, Feix G, et al. Coordinated transcriptional regulation of storage product genes in the maize endosperm[J]. Plant Physiol, 1994, 106(2): 713-722. DOI: 10.1104/pp.106.2.713 |
| [3] | Shewry P R, Napier J A, Tatham A S. Seed storage proteins:structures and biosynthesis[J]. Plant Cell, 1995, 7(7): 945-956. DOI: 10.1105/tpc.7.7.945 |
| [4] | Nakamura Y. Towards a better understanding of the metabolic system for amylopectin biosynthesis in plants:rice endosperm as a model tissue[J]. Plant Cell Physiol, 2002, 43(7): 718-725. DOI: 10.1093/pcp/pcf091 |
| [5] | James M G, Denyer K, Myers A M. Starch synthesis in the cereal endosperm[J]. Curr Opin Plant Biol, 2003, 6(3): 215-222. DOI: 10.1016/S1369-5266(03)00042-6 |
| [6] | Buléon A, Colonna P, Planchot V, et al. Starch granules:structure and biosynthesis[J]. Int J Biol Macromol, 1998, 23(2): 85-112. DOI: 10.1016/S0141-8130(98)00040-3 |
| [7] | Kubo A, Fujita N, Harada K, et al. The starch-debranching enzymes isoamylase and pullulanase are both involved in amylopectin biosynthesis in rice endosperm[J]. Plant Physiol, 1999, 121(2): 399-409. DOI: 10.1104/pp.121.2.399 |
| [8] | Nishi A, Nakamura Y, Tanaka N, et al. Biochemical and genetic analysis of the effects of amylose-extender mutation in rice endosperm[J]. Plant Physiol, 2001, 127(2): 459-472. DOI: 10.1104/pp.010127 |
| [9] | Fujita N, Kubo A, Suh D S, et al. Antisense inhibition of isoamylase alters the structure of amylopectin and the physicochemical properties of starch in rice endosperm[J]. Plant Cell Physiol, 2003, 44(6): 607-618. DOI: 10.1093/pcp/pcg079 |
| [10] | Hirose T, Terao T. A comprehensive expression analysis of the starch synthase gene family in rice(Oryza sativa L.)[J]. Planta, 2004, 220(1): 9-16. DOI: 10.1007/s00425-004-1314-6 |
| [11] | Kawagoe Y, Kubo A, Satoh H, et al. Roles of isoamylase and ADP-glucose pyrophosphorylase in starch granule synthesis in rice endosperm[J]. Plant J, 2005, 42(2): 164-174. DOI: 10.1111/tpj.2005.42.issue-2 |
| [12] | Woo M O, Ham T H, Ji H S, et al. Inactivation of the UGPase1 gene causes genic male sterility and endosperm chalkiness in rice(Oryza sativa L.)[J]. Plant J, 2008, 54(2): 190-204. DOI: 10.1111/j.1365-313X.2008.03405.x |
| [13] | Kawasaki T, Mizuno K, Baba T, et al. Molecular analysis of the gene encoding a rice starch branching enzyme[J]. Mol Gen Genet, 1993, 237(1/2): 10-16. |
| [14] | Satoh H, Omura T. New endosperm mutations induced by chemical mutagens in rice(Oryza sativa L.)[J]. Jpn J Breeding, 1981, 31(3): 316-326. DOI: 10.1270/jsbbs1951.31.316 |
| [15] | She K C, Kusano H, Koizumi K, et al. A novel factor FLOURY ENDOSPERM2 is involved in regulation of rice grain size and starch quality[J]. Plant Cell, 2010, 22(10): 3280-3294. DOI: 10.1105/tpc.109.070821 |
| [16] | Nishio T, Iida S. Mutants having a low content of 16-kDa allergenic protein in rice(Oryza sativa L.)[J]. Theor Appl Genet, 1993, 86(2): 317-321. |
| [17] | Kang H G, Park S, Matsuoka M, et al. White-core endosperm floury endosperm-4 in rice is generated by knockout mutations in the C4-type pyruvate orthophosphate dikinase gene(OsPPDKB)[J]. Plant J, 2005, 42(6): 901-911. DOI: 10.1111/tpj.2005.42.issue-6 |
| [18] | Ryoo N, Yu C, Park C S, et al. Knockout of a starch synthase gene OsSSⅢa/Flo5 causes white-core floury endosperm in rice(Oryza sativa L.)[J]. Plant Cell Rep, 2007, 26(7): 1083-1095. DOI: 10.1007/s00299-007-0309-8 |
| [19] | Fujita N, Yoshida M, Kondo T, et al. Characterization of SSⅢa-deficient mutants of rice:the function of SSⅢa and pleiotropic effects by SSⅢa deficiency in the rice endosperm[J]. Plant Physiol, 2007, 144(4): 2009-2023. DOI: 10.1104/pp.107.102533 |
| [20] | Peng C, Wang Y H, Liu F, et al. FLOURY ENDOSPERM6 encodes a CBM48 domain-containing protein involved in compound granule formation and starch synthesis in rice endosperm[J]. Plant J, 2014, 77(6): 917-930. DOI: 10.1111/tpj.2014.77.issue-6 |
| [21] | Zhang L, Ren Y L, Lu B Y, et al. FLOURY ENDOSPERM7 encodes a regulator of starch synthesis and amyloplast development essential for peripheral endosperm development in rice[J]. J Exp Bot, 2016, 67(3): 633-647. DOI: 10.1093/jxb/erv469 |
| [22] |
中华人民共和国农业部发布. 米质测定方法: NY 147-1988[S]. 北京: 中国标准出版社, 2002.
The Ministry of Agriculture of the People's Republic of China. Determination of quality of rice: NY 147-1988[S]. Beijing: China Standard Press, 2002(in Chinese). |
| [23] |
孙成效, 段彬伍, 谢黎虹, 等. 利用近红外透射光谱技术同步测定糙米的多项品质指标初报[J].
中国水稻科学, 2006, 20(4): 451-454.
Sun C X, Duan B W, Xie L H, et al. Determination of several quality characters of brown rice by near infrared transmission spectroscopy[J]. Chinese Journal of Rice Science, 2006, 20(4): 451-454. (in Chinese with English abstract) |
| [24] |
康海岐, 常红叶. 杂交水稻主要亲本材料的垩白性状及其胚乳结构电镜扫描[J].
中国农学通报, 2007, 23(4): 180-185.
Kang H Q, Chang H Y. Study on chalkiness characters and endosperm structures of the main parents' kernel of hybrid rice[J]. Chinese Agricultural Science Bulletin, 2007, 23(4): 180-185. (in Chinese with English abstract) |
| [25] | Wu K S, Tanksley S D. Abundance, polymorphism and genetic mapping of microsatellites in rice[J]. Mol Gen Genet, 1993, 241(1): 225-235. |
| [26] | Ohdan T, Francisco P B Jr, Sawada T, et al. Expression profiling of genes involved in starch synthesis in sink and source organs of rice[J]. J Exp Bot, 2005, 56(422): 3229-3244. DOI: 10.1093/jxb/eri292 |
| [27] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI: 10.1006/meth.2001.1262 |
| [28] |
金田蕴, 李辉, 郭涛, 等. 水稻稳定高垩白率突变体的获得与理化特性分析[J].
作物学报, 2010, 36(1): 121-132.
Jing T Y, Li H, Guo T, et al. Analysis of physiological and biochemical characteristics of six mutants with stable high percentage of chalkiness in rice grains[J]. Acta Agronomica Sinica, 2010, 36(1): 121-132. (in Chinese with English abstract) |
| [29] | Sabelli P A, Larkins B A. The development of endosperm in grasses[J]. Plant Physiol, 2009, 149(1): 14-26. DOI: 10.1104/pp.108.129437 |
| [30] | Wu Y P, Pu C H, Lin H Y, et al. Three novel alleles of FLOURY ENDOSPERM2(FLO2)confer dull grains with low amylose content in rice[J]. Plant Sci, 2015, 233: 44-52. DOI: 10.1016/j.plantsci.2014.12.011 |
| [31] | Nakamura Y, Utsumi Y, Sawada T, et al. Characterization of the reactions of starch branching enzymes from rice endosperm[J]. Plant Cell Physiol, 2010, 51(5): 776-794. DOI: 10.1093/pcp/pcq035 |
| [32] | D'Andrea L D, Regan L. TPR proteins:the versatile helix[J]. Trends Biochem Sci, 2003, 28(12): 655-662. DOI: 10.1016/j.tibs.2003.10.007 |
| [33] | Chadli A, Bruinsma E S, Stensgard B, et al. Analysis of Hsp90 cochaperone interactions reveals a novel mechanism for TPR protein recognition[J]. Biochemistry, 2008, 47(9): 2850-2857. DOI: 10.1021/bi7023332 |




