文章信息
- 李洁, 周浩, 郁建锋, 姚文, 顾志良
- LI Jie, ZHOU Hao, YU Jianfeng, YAO Wen, GU Zhiliang
- 鹅VNN1基因的克隆及组织表达分析
- Molecular cloning and tissue expression analysis of VNN1 gene in goose
- 南京农业大学学报, 2018, 41(1): 140-146
- Journal of Nanjing Agricultural University, 2018, 41(1): 140-146.
- http://dx.doi.org/10.7685/jnau.201701006
-
文章历史
- 收稿日期: 2017-01-03
2. 常熟理工学院生物与食品工程学院, 江苏 常熟 215500
2. College of Biological Science and Food Technology, Changshu Institute of Technology, Changshu 215500, China
泛酰巯基乙胺酶(pantetheinase)是一类泛酰巯基乙胺水解酶, 它能水解泛酰巯基乙胺形成泛酸和半胱胺[1]。泛酰巯基乙胺酶存在于多种哺乳动物组织中, 马、牛、猪、大鼠和奶牛的肝脏、肾、心脏和肌肉组织中均有发现, 鸽子和鸡的肝脏提取物中也有极高的酶活性[2]。Vanin是其中一种泛酰巯基乙胺酶, Vanin家族的不同基因编码不同的泛酰巯基乙胺酶亚型[3]。Vanin基因的亚型包括VNN1、VVN2和VNN3, 小鼠有两种Vanin基因:VNN1和VNN3, 位于10号染色体的10A2B1;人有3种Vanin基因:VNN1、VVN2和VNN3, 位于6号染色体的q22-24[4-5]。在鸡cDNA文库中发现了位于3号染色体的VNN1基因[6]。VNN1是Vanin家族中研究最多的一个亚型, 其编码一种在血管周围胸腺基质细胞表达的具有糖基磷脂酰肌醇(glycosylphosphatidylinositol, GPI)锚定蛋白结构的膜分子蛋白(VNN1), 参与调控胸腺归巢中的细胞黏附, 相对分子质量为70 000[7]。
VNN1蛋白不仅对底物具有催化作用, 还在肝脏脂类代谢中发挥重要作用。小鼠和人类肝脏中的VNN1主要在肝小叶中心区的细胞表达, 肝脏产生的VNN1能释放到血清中, 且肝脏和血清中的VNN1均具有泛酰巯基乙胺酶活性[8]。人服用非诺贝特或禁食能显著提高血浆VNN1的酶活性[9]。而对小鼠, 诱导剂PPARα能诱发肝脏VNN1的表达, 口服甘油三酯或非诺贝特, 导致小鼠肝脏VNN1的表达量大幅度升高[10], 提示VNN1可能参与脂类代谢。VNN1基因在db/db小鼠和高脂日粮诱导的肥胖小鼠肝脏中表达, 这些小鼠都出现严重的肝脏脂肪变性, 产生胰岛素抵抗, 并激活肝脏的糖异生途径; 而在特异性敲除VNN1基因的小鼠中, 肝脏脂肪变性得到改善; 另外在慢性炎症的db/db小鼠中, 肝脏VNN1酶活性升高[11]。对VNN1缺陷小鼠进行基因芯片分析, 发现了一系列差异表达基因富集在脂肪酸生物合成、脂肪酸代谢、甘油酯类代谢、类固醇生物合成等途径中, 说明VNN1在脂质代谢中发挥了很重要的作用[12]。
目前有关VNN1基因的研究主要集中在人、小鼠等方面, 关于禽类VNN1的研究很少。鹅肉是一种健康的禽类肉食品, 质嫩鲜美, 蛋白质含量高, 脂肪含量低, 不饱和脂肪酸含量高, 且多为人体必需的不饱和脂肪酸, 其脂肪营养价值优于鸡、鸭等家禽。目前尚未有鹅VNN1基因的相关报道。本试验将以太湖鹅为材料, 通过RT-PCR和快速扩增cDNA末端(rapid amplification cDNA ends, RACE)方法获得鹅VNN1基因全长cDNA, 并对所得序列进行生物信息学分析, 运用实时荧光定量PCR (qPCR)检测鹅VNN1基因在不同组织中mRNA水平的表达规律, 为进一步研究该基因在鹅脂类代谢中的作用奠定基础。
1 材料与方法 1.1 试验材料从常熟市江南畜禽食品公司选取3只6周龄健康太湖鹅, 采集胸肌、腿肌、肝脏、心脏、肺、脾、肾、肌胃、腺胃、小肠、脂肪和脑12种组织样本经液氮速冻后放置于-80 ℃冰箱中备用。
1.2 RNA的提取用Trizol试剂(Invitrogen, USA)提取鹅胸肌、腿肌、肝脏、心脏、肺、脾、肾、肌胃、腺胃、小肠、脂肪和脑12种组织的总RNA, NanoDrop2000 (Thermo Scientific, USA)测定其浓度并用甲醛变性琼脂糖凝胶电泳检验RNA质量, 置于-80 ℃保存备用。
1.3 引物设计根据红原鸡(Gallus gallus)的VNN1基因序列(GenBank ID:NM_001039288.2)设计鹅VNN1基因部分片段引物, 结合鸡VNN1基因的序列设计5′-RACE和3′-RACE的特异性引物。同时根据克隆得到的鹅VNN1基因cDNA序列设计实时荧光定量qPCR引物, 选用GAPDH作为内参。引物均由生工生物工程(上海)有限公司合成(表 1)。
| 引物(对)名称 Primer (paris)name | 引物(对)序列(5′→3′) Primer (paris)sequences | 扩增片段长度/bp Amplified fragment size | 用途 Purpose |
| ggaVNN1 qF1/R5 | GACTCTGAAGGGAAACTGGT/ATACCTCAAATTCCCCAGGG | 880 | 克隆鹅VNN1中间片段 |
| gooseVNN1R1 | AAGGGTGTCTCGAAGGTGAC | 541 | 5′RACE |
| 5′RACE outer primer | CATGGCTACATGCTGACAGCCTA | 5′RACE | |
| gooseVNN1R2 | TACTTGTGGTAACGAGCCACCA | 5′RACE | |
| 5′RACE inner primer | CGCGGATCCACAGCCTACTGATGATCAGTCGATG | 5′RACE | |
| GooseVNN1spF1 | GACTGCGCAGACCAAGTTTG | 571 | 3′RACE |
| 3′RACE outer primer | TACCGTCGTTCCACTAGTGATTT | 3′RACE | |
| gooseVNN1spF2 | CGTCTTTCCAGAGGTCTTGT | 3′RACE | |
| 3′RACE inner primer | CGCGGATCCTCCACTAGTGATTTCACTATAGG | 3′RACE | |
| gooseVNN1 qF1/Rq | GACTCTGAAGGGAAACTGGT/AACGCCAAACTTCCCAAAGG | 129 | qPCR |
| GAPDH qF/qR | TCTTCACCACCATGGAGAAG/CAGGACGCATTGCTGACAAT | 154 | qPCR |
以鹅肝脏组织总RNA为模板, 通过RT-PCR获得cDNA, 然后以ggaVNN1 qF1/R5为引物对扩增鹅VNN1部分cDNA片段。反应体系:10×PCR Buffer 2.5 μL, 10 mmol·L-1 dNTPs 2 μL, 10 μmol·L-1的引物对ggaVNN1 qF1/R5各1 μL, rTaq (5 U·μL-1)0.2 μL, 1 μL cDNA, ddH2O 17.3 μL。PCR扩增条件:94 ℃ 3 min; 94 ℃ 30 s, 58 ℃ 30 s, 72 ℃ 1 min, 共32个循环; 72 ℃ 10 min; 4 ℃保温。PCR产物在10 g·L-1琼脂糖凝胶中电泳, 回收PCR产物片段后送生工生物工程(上海)有限公司测序。
1.5 鹅VNN1基因5′端和3′端的序列扩增及克隆与测序5′端序列和3′端序列的扩增分别按照5′-Full RACE Kit和3′-Full RACE Kit (TaKaRa公司)的说明书步骤, 进行2轮巢式PCR反应, 反应产物用10 g·L-1的琼脂糖凝胶电泳鉴定, 用DNA回收试剂盒(天根公司)回收目的片段, 回收产物与pMD19-T载体(TaKaRa公司)连接后转化至大肠杆菌DH5α感受态细胞, 经过蓝白斑筛选后挑选阳性克隆进行菌液PCR检测, 提取阳性菌的质粒测序。
1.6 鹅VNN1基因的组织表达分析将太湖鹅的胸肌、腿肌、心脏、肝脏、脾、肾、肌胃、腺胃、小肠、脂肪和脑共12种组织的RNA稀释成100 ng·μL-1, 每组3个个体, 分别取5 μL RNA进行反转录(RT)反应, 得到的cDNA产物采用实时荧光定量PCR (qPCR)检测VNN1和甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase, GAPDH)基因的表达。反应体系(20 μL):SYBR Premix Ex Taq Ⅱ (2×)10 μL, ddH2O 6.8 μL, ROX Reference DyeⅡ (50×)0.4 μL, 10 μmol·L-1的上游和下游引物各0.4 μL, cDNA 2 μL。qPCR反应条件:95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 40个循环。
1.7 生物信息学分析用DNAMAN 7.0软件分析克隆得到鹅cDNA序列, 同时推导出氨基酸序列并进行物种间序列的同源性比对; 用MEGA 7.0软件构建系统进化树; 用ExPASy生物信息门户中的ProtParam和ProtScale工具分别分析蛋白质的理化性质和亲水性, 用SWISS-MODEL预测蛋白质三级结构。
1.8 数据收集和分析PCR结果采用2-ΔΔCT法分别计算每个样品VNN1和GAPDH基因表达的相对值, 用Excel 2016绘制VNN1在不同组织中mRNA表达量的柱状图, 采用Origin 7.0软件进行差异显著性分析。
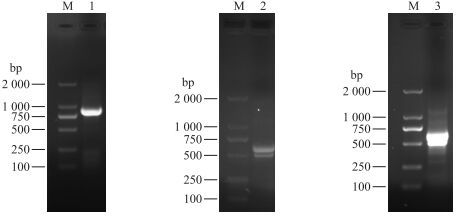
2 结果与分析 2.1 鹅VNN1基因全长cDNA克隆及序列分析以太湖鹅肝脏总RNA为模板, 反转录得到cDNA, 用引物对ggaVNN1 qF1/R5扩增得到一中间片段, 经克隆测序后确定该序列长度为880 bp (图 1-A), 与NCBI上其他物种序列对比, 证明所得序列为VNN1基因中间片段序列。根据得到的鹅VNN1基因中间片段序列设计5′-RACE特异性引物gooseVNN1R1和gooseVNN1R2, 分别与5′RACE outer primer和inner primer配对, 进行2轮巢式PCR扩增及克隆测序后得到长度为541 bp的序列(图 1-B)。根据已获得的鹅VNN1基因中间片段序列设计3′RACE特异性引物gooseVNN1spF1和gooseVNN1spF2, 分别与3′RACE outer primer和inner primer配对, 进行2轮巢式PCR扩增及克隆测序后得到长度为571 bp的序列(图 1-C)。用DNAMAN 7.0软件将上述3个片段拼接得到长度为1 924 bp的鹅VNN1全长cDNA序列(GenBank登录号:KY399733), 包含35 bp的5′UTR、417 bp的3′UTR及1 476 bp的CDS序列, 起始密码子为ATG, 终止密码子为TAA, 共编码491个氨基酸(图 2)。
|
图 1 鹅VNN1基因cDNA扩增 Figure 1 The amplification products of goose VNN1 cDNA M:DL2000分子量标准; 1.鹅中间片段扩增产物; 2. 5′RACE扩增产物; 3. 3′RACE扩增产物。 M:DL2000 marker; 1. The amplification products of goose middle fragment; 2. The 5′RACE amplification product; 3. The 3′RACE amplification product. |

|
图 2 鹅VNN1基因核苷酸序列及推导的氨基酸序列 Figure 2 Nucleotide and amino acid sequence of VNN1 gene from goose |
用ExPASy的ProtParam工具分析鹅VNN1基因编码蛋白质的理化性质, 该蛋白由7 646个原子组成, 分子式为C2487H3780N632O723S24, 相对分子质量为54 870.61, 理论等电点为5.23;氨基酸组成方面, 常见的20种氨基酸中缬氨酸(Val)含量最高, 为8.8%, 色氨酸(Trp)含量最低, 为1.4%, 带负电荷氨基酸总数(Asp+Glu)为56个, 带正电荷氨基酸总数(Arg+Lys)为40个; 不稳定指数为43.95, 脂肪系数为81.41, 总平均亲水性为-0.081。
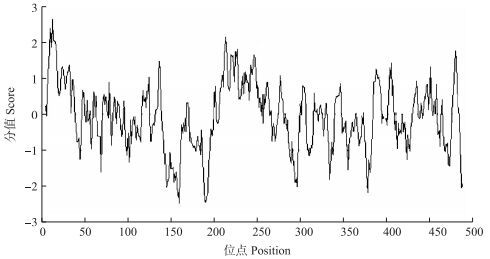
用ExPASy的ProtScale工具以Hphob./Kyte & Doolittle为标度做亲疏水性分析, 发现该蛋白质的第14位氨基酸分值最大, 为2.622, 表明该位点具有很强的疏水性; 而在第160位的分值最小, 为-2.467, 表明该位点具有很强的亲水性(图 3)。
|
图 3 鹅VNN1蛋白的疏水性和亲水性分布图 Figure 3 The hydrophobic distribution of goose VNN1 protein |
用SWISS-MODEL同源建模并在线预测鹅和鸭VNN1蛋白的三级结构, 鹅与鸭属于近缘物种, 在两者的三级结构中均发现有高度相似的折叠和螺旋(图 4)。

|
图 4 预测的鹅(A)和绿头鸭(B)VNN1蛋白三级结构 Figure 4 The prediction of three-dimensional structure of VNN1 protein in goose (A) and mallard (B) |
运用DNAMAN 7.0软件将鹅VNN1基因CDS序列和其他物种序列进行同源性比对, 发现鹅VNN1基因CDS区与绿头鸭(Anas platyrhynchos, XM_005009567)核苷酸同源性最高, 为95.66%, 与红原鸡(Gallus gallus, XM_015284109)的核苷酸同源性为89.86%, 而与热带爪蟾(Xenopus tropicalis, XM_004914589)的核苷酸同源性最低, 为60.86%;将鹅VNN1氨基酸序列和其他物种的氨基酸序列比对, 发现其与绿头鸭的氨基酸同源性最高, 为95.11%, 与红原鸡的氨基酸同源性为89.23%, 而鸿雁(Anser cygnoides domesticus, XM_013175489)虽然比鹅少127个氨基酸, 但其同源性也较高, 为73.73%, 与其他物种的氨基酸同源性大多在62%~65%区间, 与热带爪蟾的氨基酸同源性最低, 为60.45% (表 2)。
| 物种 Species | GenBank登录号 GenBank accession No. | 核苷酸同源性/% Nucleotide homology | 氨基酸同源性/% Amino acid homology | 编码氨基酸数 No. of encoded amino acid |
| 绿头鸭Anas platyrhynchos | XM_005009567 | 95.66 | 95.11 | 490 |
| 鸿雁Anser cygnoides domesticus | XM_013175489 | 73.98 | 73.73 | 364 |
| 人Homo sapiens | NM_004666 | 63.27 | 62.81 | 513 |
| 红原鸡Gallus gallus | XM_015284109 | 89.86 | 89.23 | 491 |
| 小鼠Mus musculus | NM_011704 | 63.67 | 62.36 | 512 |
| 大鼠Rattus norvegicus | NM_001025623 | 62.81 | 62.74 | 512 |
| 猕猴Macaca mulatta | XM_001102153 | 63.11 | 64.35 | 513 |
| 黑猩猩Pan troglodytes | XM_001169280 | 63.46 | 63.39 | 513 |
| 牛Bos taurus | NM_001024556 | 65.12 | 66.02 | 510 |
| 水牛Bubalus bubalis | XM_006059301 | 63.53 | 65.13 | 514 |
| 山羊Capra hircus | XM_005684767 | 65.33 | 65.95 | 506 |
| 绵羊Ovis aries | XM_004011338 | 65.14 | 65.95 | 506 |
| 野猪Sus scrofa | NM_214133 | 62.24 | 63.01 | 513 |
| 热带爪蟾Xenopus tropicalis | XM_004914589 | 60.86 | 60.45 | 488 |
利用MEGA 7.0软件分析鹅VNN1氨基酸序列与绿头鸭、鸿雁、人等物种的氨基酸序列的进化关系, 并以距离矩阵邻接法(neighbor-joining method, NJ)构建分子进化树, 通过自举检验获得系统分支的置信度, 重复次数1 000次。由图 5可知:VNN1基因在系统进化中, 哺乳动物形成一个很大的分支; 鹅与鸿雁聚成一支, 且自展值为100, 说明鹅与鸿雁亲缘关系最近, 鹅与鸿雁所在的一支又与绿头鸭、红原鸡聚成一支。

|
图 5 基于NJ法构建的VNN1氨基酸序列系统进化树 Figure 5 Phylogenetic tree reconstructed based on VNN1 amino acid sequence with neighbor-joining method |
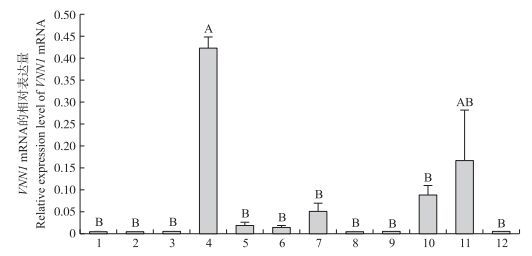
由图 6的实时荧光定量结果可知:VNN1 mRNA在太湖鹅肝脏、脂肪、小肠、肾、脾、肺和胸肌中均有表达, 其中肝脏的表达量极显著高于小肠、肾、脾和肺(P < 0.01), 肝脏VNN1与脂肪VNN1的mRNA表达量没有显著差异, 在胸肌中表达量极低。
|
图 6 太湖鹅VNN1基因在不同组织中的表达量(内参基因:GAPDH; n=3) Figure 6 The expression of Taihu goose VNN1 gene in various tissues 1.胸肌Chest muscle; 2.腿肌Thigh muscle; 3.心脏Heart; 4.肝脏Liver; 5.脾Spleen; 6.肺Lung; 7.肾Kidney; 8.肌胃Muscular stomach; 9.腺胃Glandular stomach; 10.小肠Intestine; 11.脂肪Adipose tissue; 12.脑Brain. |
本研究通过RT-qPCR、RACE等技术成功克隆了鹅VNN1基因(GenBank登录号:KY399733)全长cDNA序列, 利用DNAMAN软件拼接得到完整的长度为1 924 bp的序列, 包括1 476 bp的开放阅读框, 共编码491个氨基酸。
利用DNAMAN 7.0软件对鹅VNN1基因与绿头鸭、红原鸡、人、牛等物种的VNN1基因及推导的氨基酸序列进行同源性分析, 发现鹅的碱基序列和氨基酸序列均与绿头鸭的同源性最高, 表明鹅与绿头鸭的亲缘性较近; 鸿雁比鹅少了127个氨基酸, 但是鹅与鸿雁的氨基酸序列同源性仍有73.73%, 从MEGA 7.0构建的进化树可以看出, 鹅与鸿雁聚成一支, 且自展值为100, 说明鹅与鸿雁亲缘关系较近, 鹅与鸿雁所在的一支又与绿头鸭聚成一支。这与虞德兵等[13]分析鸭科进化关系的结果一致, 说明VNN1基因在不同物种间的系统发育与亲缘关系一致。Maras等[14]从猪肾脏中分离出泛酰巯基乙胺酶, 其部分氨基酸序列与人VNN1基因和小鼠VNN1基因的相似性分别为83.2%和79.7%, 表明它与人VNN1和小鼠VNN1基因有很高的同源性。Caldwell等[6]发现鸡VNN1基因与其他物种的VNN1基因同源性很高, 说明VNN1氨基酸序列在长期进化过程中高度保守。
本试验发现该VNN1基因在肝脏、脂肪、小肠、肾、脾、肺和胸肌中均有表达, 其中在肝脏中的表达量极显著高于除了脂肪以外的其他10种组织。与鹅的表达相似, 鸽子和鸡的肝脏提取物中也有极高的泛酰巯基乙胺酶活性[2]; Li等[15]在4周龄鸡中检测VNN1基因的表达情况, 也发现VNN1基因在肝脏中表达量最高; 但是有报道指出VNN1蛋白在小鼠肾脏的表达量比在肝脏中高[16-17], 说明VNN1基因表达模式存在物种依赖性。脂类代谢是重要的生物过程之一, 近年的不少研究均发现VNN1在脂类代谢中发挥重要作用。Motomura等[18]研究发现小鼠饲喂高脂日粮1 d, 其肝脏VNN1 mRNA水平上调; 在体外培养的HuH-7细胞中, 只要加入0.01 mmol·L-1的油酸就能显著上调VNN1 mRNA的表达。另有研究证明对小鼠禁食会促进肝脏甘油三酯和血浆游离脂肪酸的积累, 而将小鼠VNN1基因敲除会加剧肝脏甘油三酯的积累。使用基因芯片进行检测分析, 发现VNN1的缺失与一系列参与肝脏脂肪变性的基因相关[19]。禽类的肝脏是脂类代谢的主要场所, 鹅VNN1基因在肝脏的高表达说明其可能在脂类代谢中发挥重要作用。
| [1] | Duprè S, Graziani M T, Rosei M A, et al. The enzymatic breakdown of pantethine to pantothenic acid and cystamine[J]. Eur J Biochem, 1970, 16(3): 571-578. DOI: 10.1111/ejb.1970.16.issue-3 |
| [2] | Wittwer C T, Schweitzer C, Pearson J, et al. Enzymes for liberation of pantothenic acid in blood:use of plasma pantetheinase[J]. Am J Clin Nutr, 1989, 50(5): 1072-1078. DOI: 10.1093/ajcn/50.5.1072 |
| [3] | Martin F, Malergue F, Pitari G, et al. Vanin genes are clustered (human 6q22-24 and mouse 10A2B1) and encode isoforms of pantetheinase ectoenzymes[J]. Immunogenetics, 2001, 53(4): 296-306. DOI: 10.1007/s002510100327 |
| [4] | Galland F, Malergue F, Bazin H, et al. Two human genes related to murine vanin-1 are located on the long arm of human chromosome 6[J]. Genomics, 1998, 53(2): 203-213. DOI: 10.1006/geno.1998.5481 |
| [5] | Granjeaud S, Naquet P, Galland F. An ESTs description of the new Vanin gene family conserved from fly to human[J]. Immunogenetics, 1999, 49(11/12): 964-972. |
| [6] | Caldwell R B, Kierzek A M, Arakawa H, et al. Full-length cDNAs from chicken bursal lymphocytes to facilitate gene function analysis[J]. Genome Biol, 2005, 6(1): R6. |
| [7] | Aurrand-Lions M, Galland F, Bazin H, et al. Vanin-1, a novel GPI-linked perivascular molecule involved in thymus homing[J]. Immunity, 1996, 5(5): 391-405. DOI: 10.1016/S1074-7613(00)80496-3 |
| [8] | Rommelaere S, Millet V, Gensollen T, et al. PPARalpha regulates the production of serum Vanin-1 by liver[J]. FEBS Lett, 2013, 587(22): 3742-3748. DOI: 10.1016/j.febslet.2013.09.046 |
| [9] | van Diepen J A, Jansen P A, Ballak D B, et al. PPAR-alpha dependent regulation of vanin-1 mediates hepatic lipid metabolism[J]. J Hepatol, 2014, 61(2): 366-372. DOI: 10.1016/j.jhep.2014.04.013 |
| [10] | Rakhshandehroo M. Peroxisome proliferator-activated receptor alpha target genes[EB/OL].[2010-8-9]. https://www.hindawi.com/journals/ppar/2010/612089. |
| [11] | Chen S, Zhang W, Tang C, et al. Vanin-1 is a key activator for hepatic gluconeogenesis[J]. Diabetes, 2014, 63(6): 2073-2085. |
| [12] | Rommelaere S, Millet V, Manh T P V, et al. Sox17 regulates liver lipid metabolism and adaptation to fasting[J]. PLoS ONE, 2014, 9(8). |
| [13] |
虞德兵, 陆应林, 徐昊翔, 等. 基于线粒体COⅠ基因序列分析家鸭系统发育关系[J].
南京农业大学学报, 2011, 34(6): 109-114.
Yu D B, Lu Y L, Xu H X, et al. Analysis of phylogenetic relationship based on mitochondrial COⅠ gene sequence in domestic duck[J]. Journal of Nanjing Agricultural University, 2011, 34(6): 109-114. DOI: 10.7685/j.issn.1000-2030.2011.06.020 (in Chinese with English abstract) |
| [14] | Maras B, Barra D, Duprè S, et al. Is pantetheinase the actual identity of mouse and human vanin-1 proteins[J]. FEBS Lett, 1999, 461(3): 149-152. DOI: 10.1016/S0014-5793(99)01439-8 |
| [15] | Li Y, Wang X, Yu J, et al. MiR-122 targets the vanin1 gene to regulate its expression in chicken[J]. Poult Sci, 2016, 95(5): 1145-1150. DOI: 10.3382/ps/pew039 |
| [16] | Pitari G, Malergue F, Martin F, et al. Pantetheinase activity of membrane-bound Vanin-1:lack of free cysteamine in tissues of Vanin-1 deficient mice[J]. FEBS Lett, 2000, 483(2/3): 149-154. |
| [17] | Ruan B H, Cole D C, Wu P, et al. A fluorescent assay suitable for inhibitor screening and vanin tissue quantification[J]. Anal Biochem, 2010, 399(2): 284-292. DOI: 10.1016/j.ab.2009.12.010 |
| [18] | Motomura W, Yoshizaki T, Takahashi N, et al. Analysis of vanin-1 upregulation and lipid accumulation in hepatocytes in response to a high-fat diet and free fatty acids[J]. J Clin Biochem Nutr, 2012, 51(3): 163-169. DOI: 10.3164/jcbn.12-06 |
| [19] | Leone T C, Weinheimer C J, Kelly D P. A critical role for the peroxisome proliferator-activated receptor alpha (PPARalpha) in the cellular fasting response:the PPARalpha-null mouse as a model of fatty acid oxidation disorders[J]. Proc Natl Acad Sci USA, 1999, 96(13): 7473-7478. DOI: 10.1073/pnas.96.13.7473 |




