文章信息
- 李庆玲, 沈丹宇, 于佳, 赵媛媛, 朱也, 窦道龙
- LI Qingling, SHEN Danyu, YU Jia, ZHAO Yuanyuan, ZHU Ye, DOU Daolong
- 基于环介导等温扩增技术快速检测瓜果腐霉菌
- Rapid detection of Pythium aphanidermatum by loop-mediated isothermal amplification
- 南京农业大学学报, 2018, 41(1): 79-87
- Journal of Nanjing Agricultural University, 2018, 41(1): 79-87.
- http://dx.doi.org/10.7685/jnau.201703025
-
文章历史
- 收稿日期: 2017-03-21
腐霉菌(Pythium)隶属于卵菌门, 是一类重要的植物病原菌。目前已经鉴定的腐霉菌有120余种[1], 并且它们大多数是腐生性植物病原菌, 主要通过土壤传播并具有广泛的宿主范围。它们通常侵染幼苗的根和种子, 并引起苗期前后的猝倒、种子腐烂、根腐和果实腐烂等症状[2]。腐霉菌能引起很多重要作物的根腐病, 包括小麦、玉米、辣椒、黄瓜、番茄等[3]。大多数腐霉菌可以产生游动孢子, 并伴随灌溉水或雨水转移到新的宿主上[4]。另外, 腐霉菌偏好高温、高湿环境, 更容易在温室培养的作物上大面积发生病害, 造成严重的经济损失[5]。
瓜果腐霉菌(Pythium aphanidermatum)能引起多种农作物的猝倒病和瓜果腐烂病, 寄主范围很广, 可以侵染玉米引起茎腐[6], 侵染黄瓜、番茄等引起猝倒病、果腐, 在苗期发病尤为严重, 常造成大量死苗[7-8]。病菌侵入果实种皮后, 在运输、贮存的过程中, 遇到合适的温度、湿度条件时, 可以通过伤口深入果实内部, 最终导致果实腐烂溃败, 失去产品价值[8-9]。瓜果腐霉菌在高温和高湿的条件下, 对禾本科坪草同样具有很强的致病力, 可造成坪草大面积病害甚至死亡[1]。
由于腐霉菌引起的病害症状与其他植物病害相似性高, 尤其病害初期症状不明显, 使得相关病害的诊断较为困难[9]。病害的诊断主要依赖病原菌的分离与鉴定, 但是受到该属病原菌分离培养难, 腐生种类多, 不同种菌丝形态与分生孢子相似性极高等因素的制约[10], 传统鉴定方法需要专业丰富的病原菌识别经验, 且耗时长, 不利于大规模样品的分析鉴定。近年来, 已经开发了基于聚合酶链式反应(PCR)的一系列分子检测方法, 其中常规PCR和实时定量PCR测定是2种广泛使用的检测技术, 并且已经应用于超过20种致病性腐霉菌的特异性诊断[11]。例如, Cullen等[12]以ITS区域作为靶标序列, 设计终极腐霉菌的特异性引物, 并使用常规PCR测定法检测终极腐霉菌。然而, 上述PCR方法需要操作经验丰富的实验员和相对昂贵的仪器设备如PCR仪、荧光定量PCR仪[13]。因此, 这些方法并不适用于大田操作的基层工作者和一些资源有限的实验室。
环介导等温扩增(loop-mediated isothermal amplification, LAMP)是一种新开发的分子检测技术, 通过具有链置换活性的BstDNA聚合酶在等温条件下扩增, 扩增效率在1 h内可达到109拷贝数。LAMP方法使用4~6条引物分别识别靶标DNA相应的区域, 具有高特异性、快速和简单扩增靶基因的优势[14]。在反应体系中添加显色染料, 例如SYBR Green、羟基萘酚蓝(HNB)或钙黄绿素, 检测结果可直接通过肉眼观察判定[15]。这些优点使得LAMP方法与其他诊断方法相比, 在病原菌的快速检测中更具吸引力。迄今为止, 该技术已成功应用于真菌、细菌、卵菌和病毒的检测[16-20]。目前包括腐霉菌在内的大部分病原菌的LAMP检测方法都是以ITS序列作为靶标, 如Fukuta等[19]依据群结腐霉菌(Pythium myriotylum)的ITS序列设计LAMP特异性引物, 并成功将群结腐霉菌与其他39种腐霉菌和11种疫霉菌区分开; Takahashi等[20]以ITS序列作为检测靶标, 建立了能特异性检测Pythium helicoides的LAMP检测方法。
目前, 腐霉菌的LAMP检测技术主要以ITS序列作为靶标, 但ITS序列在不同物种间很保守, 尤其在相近物种间序列差异更小, 因此特异性不高是其缺陷。本研究以检测靶标基因为切入点, 通过比较基因组学方法, 在瓜果腐霉菌基因组上选择有序列特异性的基因作为检测靶标, 建立了基于颜色判定的LAMP检测方法, 并对该LAMP方法的特异性、灵敏度和人工模拟接种试验进行了系统分析。
1 材料与方法 1.1 供试菌株供试菌株的寄主、来源和数量见表 1。腐霉和疫霉菌株的培养采用10%(质量分数)V8固体培养基[21], 其他真菌在马铃薯葡萄糖固体培养基(PDA)上培养[22]。从新鲜的菌落边缘切取1 cm × 1 cm菌丝块, 置于20 mL 10%的V8(腐霉、疫霉)或PDA(真菌)培养液中, 在25 ℃黑暗环境中静置培养3 d后收集菌丝, 随后放在-70 ℃保存, 为提取菌株基因组DNA做准备。
| 种名Species | 寄主Host | 来源Source | 菌株数量No.of isolates |
| 瓜果腐霉菌Pythium aphanidermatum | 番茄Solanum lycopersicum | 山东Shandong Province | 2 |
| 终极腐霉菌Pythium ultimum | 草莓Fragaria ananassa | 山东Shandong Province | 4 |
| 刺腐霉菌Pythium spinosum | 土壤Soil | 江苏Jiangsu Province | 2 |
| 旋柄腐霉菌Pythium helicoides | 大豆Glycine max | 江苏Jiangsu Province | 2 |
| 畸雌腐霉菌Pythium irregulare | 胡萝卜Daucus carota | 江苏Jiangsu Province | 1 |
| 强雄腐霉菌Pythium arrhenomanes | 小麦Triticum aestivum | 河南Henan Province | 1 |
| 齿孢腐霉菌Pythium hydnosporum | 土壤Soil | 江苏Jiangsu Province | 1 |
| 群结腐霉菌Pythium myriotylum | 西瓜Citrullus lanatus | 江苏Jiangsu Province | 1 |
| 宽雄腐霉菌Pythium dissotocum | 松树Pinus linn | 江苏Jiangsu Province | 1 |
| 链状腐霉菌Pythium catenulatum | 苹果Malus pumila | 江苏Jiangsu Province | 1 |
| 大豆疫霉菌Phytophthora sojae | 大豆Glycine max | 江苏Jiangsu Province | 1 |
| 辣椒疫霉菌Phytophthora capsici | 甜辣椒Capsicum annuum | 江苏Jiangsu Province | 1 |
| 烟草疫霉菌Phytophthora parasitica | 烟草Nicotiana benthamiana | 江苏Jiangsu Province | 1 |
| 灰霉菌Botrytis cinerea | 葡萄Vitis vinifera | 江苏Jiangsu Province | 1 |
| 梨腐烂菌Valsa pyri | 梨Pyrus sinkiangensis | 江苏Jiangsu Province | 1 |
| 棉花黄萎菌Verticillium dahliae | 棉花Gossypium hirsutum | 江苏Jiangsu Province | 1 |
| 立枯丝核菌Rhizoctonia solani | 棉花Gossypium hirsutum | 江苏Jiangsu Province | 1 |
| 禾谷镰刀菌Fusarium graminearum | 小麦Triticum aestivum | 河南Henan Province | 1 |
甜菜碱(Betaine)、MgSO4、羟基萘酚蓝(HNB)购自Sigma公司; Bst DNA聚合酶购自New England Biolabs公司; 5×PsBuffer、10 mmol·L-1 dNTPs、5 mmol·L-1 dNTPs购自TaKaRa公司; Loopamp反应管购自日本荣研化学株式会社; 移液管购自Eppendorf公司; 电热恒温水浴锅(DK-8D)购自上海森信实验仪器有限公司; PCR扩增仪购自TaKaRa公司。
1.3 靶标基因筛选与引物设计从腐霉菌基因组数据库网站(http://pythium.plantbiology.msu.edu)下载了已公布的6种腐霉菌的基因组序列, 通过比较基因组的方法, 将瓜果腐霉菌的所有基因序列用BLASTn(E-value < 1E-10)比对其他5种腐霉菌的基因组, 找到瓜果腐霉菌特异的基因作为靶标基因。利用在线工具PrimerExplore V4设计瓜果腐霉菌靶标基因的LAMP特异性引物。
1.4 LAMP反应体系及条件的优化LAMP反应体系包括:2.5 μL 10×ThermoPol Buffer, 4 mmol·L-1 MgSO4, 1.4 mmol·L-1 dNTPs, 内引物FIP和BIP各1.6 μmol·L-1, 外引物F3和B3各0.2 μmol·L-1, 环引物LB 0.8 μmol·L-1, 0.8 mol·L-1甜菜碱, 0.1% Triton X-100, 20 mmol·L-1 Tris-HCl(pH8.8), 10 mmol·L-1 KCl, 10 mmol·L-1(NH4)2SO4, 8 U·μL-1 Bst DNA polymerase, 180 μmol·L-1羟基萘酚蓝(HNB), 2 μL模板DNA, 灭菌水补至25 μL。LAMP反应程序为:水浴锅64 ℃反应扩增60 min, 然后观察扩增产物的颜色变化, 如果颜色由紫色变为天蓝色, 表示待检物中存在瓜果腐霉菌, 如果颜色没有变化仍是紫色, 表示待检物中不存在瓜果腐霉菌。LAMP反应结束后, 反应产物经20 g·L-1的琼脂糖凝胶电泳再次确认, 如果出现典型梯形条带即为阳性结果, 反之为阴性结果。
为了确定最适合的反应体系, 对反应体系中的2个关键参数温度和MgSO4浓度进行了调整优化。根据Bst DNA聚合酶反应温度设置了60、62、64、66、68 ℃共5个温度梯度, 并设置了2、4、6、8、10、12 mmol·L-1共6个浓度梯度的MgSO4进行LAMP扩增反应, 随后通过凝胶电泳评估扩增效果, 从而确定最适合的反应条件。
1.5 LAMP特异性检测采用CTAB法分别提取了24种菌株的基因组DNA, 并进行LAMP扩增, 重复3次。反应结束后通过肉眼观察反应物的颜色变化和进行20 g·L-1的琼脂糖凝胶电泳2种方法评估LAMP反应的特异性。
1.6 LAMP灵敏度检测瓜果腐霉菌的基因组DNA模板经浓度测定后, 进行10倍系列稀释, 使得质量浓度梯度依次为100 ng·μL-1、10 ng·μL-1、1 ng·μL-1、100 pg·μL-1、10 pg·μL-1、1 pg·μL-1、100 fg·μL-1、10 fg·μL-1。分别取2.0 μL作为LAMP反应模板, 进行3次重复。反应结束后通过肉眼观察反应物的颜色变化和进行20 g·L-1的琼脂糖凝胶电泳2种方法鉴定是否发生了LAMP扩增, 确定LAMP方法的灵敏度。同时基于该靶标基因也进行了普通PCR和Real-time PCR反应的灵敏度检测, 并与LAMP方法的灵敏度进行了比较。
1.7 人工接种植株的发病组织检测为了验证LAMP检测方法在实际应用中的可行性, 通过人工接种植株的方法进行试验。选取4~6周长势良好的番茄和黄瓜叶片, 将瓜果腐霉菌丝块在以上2种植物叶片的背面上接种, 24 h后去掉菌丝块并收集发病组织提取基因组DNA, 作为LAMP检测反应的模板。此外, 为了评估人工接种植株后不同时间点的检测效果, 本研究将瓜果腐霉菌丝块接种在黄瓜叶片上, 在接种后0、6、12、18、24 h观察叶片的发病情况, 并分别将菌丝块去除后提取植物侵染部位的总DNA, 利用LAMP方法进行检测。
2 结果与分析 2.1 靶标基因的筛选和LAMP特异性引物本研究利用比较基因组学的方法, 在瓜果腐霉基因组上鉴定序列特异的基因作为检测靶标。首先将瓜果腐霉菌基因组上的12 312个基因的核酸序列通过BLAST的方法比对其他5种已测序腐霉菌的所有基因序列, 得到了1 000多个瓜果腐霉基因在其他腐霉菌中有同源序列, 但序列差异较大, 于是将这些基因作为候选的检测靶标。我们从中分析并找到1个编码Trypsin蛋白酶的基因, 该基因全长786 bp, 与其他腐霉菌的同源序列差异较大(图 1), 核酸水平的序列相似度不到50%, 将这个基因作为本研究的靶标基因。随后, 基于这个基因的特异区域设计了LAMP检测的特异性引物(图 1, 表 2)。

|
图 1 靶标基因在不同腐霉菌中的多序列比对和LAMP引物设计 Figure 1 Nucleotide sequence alignment of the target gene among different Pythium species and the design of LAMP primers LAMP引物用横线标记。 LAMP primers are represented by lines. |
| 引物名称Primer name | 序列Sequence(5′→ 3′) |
| F3(Forward outer) | GCCTGCTTGAGGTCAACG |
| B3(Backward outer) | CCTCACCACAGCCATCAC |
| FIP(Forward inner)(F1c+F2) | CGCACACAGCATTGTGTCCGTGGTCGTGACCAACGAGGA |
| BIP(Backward inner)(B1c+B2) | CGAAGGTGACTCTGGCGGTCCAGCTCACGACGCCAATC |
| LF(Loop fordward primer) | TGGTTGTCATTTTGGCCTTACAC |
| LB(Loop backward primer) | CACTGGTGGTCAAGAAGGCTGAC |
为了获得最佳的反应结果, 本研究对反应温度和MgSO4浓度这两个关键的参数进行了优化。由于Bst DNA聚合酶的最适温度范围为60~65 ℃, 因此设置了60、62、64、66、68 ℃共5个温度梯度。如图 2-A所示:在设置的5个温度条件下, 都能正常得到扩增产物, 但64 ℃条件下的扩增产物电泳条带亮度略高于其他温度, 说明该温度条件下的扩增效果最好。MgSO4浓度设置为2、4、6、8、10、12 mmol·L-1 6个梯度, 结果如图 2-B所示:MgSO4浓度为2 mmol·L-1和12 mmol·L-1时无法进行扩增, 浓度为4、6、8、10 mmol·L-1时扩增效果差异不大, 但浓度为6 mmol·L-1时扩增效果略优于其他浓度。基于以上结果, 本研究将LAMP反应体系中的温度设为64 ℃, MgSO4浓度设为6 mmol·L-1, 用于后续研究。

|
图 2 反应体系中温度(A)和MgSO4浓度(B)的优化 Figure 2 Optimization of temperature(A)and MgSO4 concentration(B) M:DNA marker. |
利用设计的特异性引物对参试的2个不同地理来源瓜果腐霉(2个菌株分别采集于山东省泰安市和济南市)和17种不同卵菌近缘种及5种常见病原真菌进行了LAMP检测。基于LAMP反应结束后的HNB显色变化(图 3-A), 2个瓜果腐霉菌株的反应管均呈现天蓝色, 为阳性结果, 而其他腐霉菌、疫霉菌和真菌反应管均为紫色, 为阴性结果。同时, LAMP反应产物经20 g·L-1琼脂糖凝胶电泳验证, 结果如图 3-B所示, 只有2个瓜果腐霉菌株的扩增产物出现了典型梯形条带即为阳性结果, 而其他腐霉菌、疫霉菌和真菌都没有出现梯形条带, 即为阴性结果。以上结果表明, 该LAMP方法能特异性地检测不同地理来源的瓜果腐霉菌, 而且具有很好的种特异性。

|
图 3 LAMP反应的特异性试验 Figure 3 Specificity of LAMP detection A. HNB可视化显色检测; B.20 g·L-1琼脂糖凝胶电泳检测。M.DNA marker; 1、2.瓜果腐霉菌; 3~6.终极腐霉菌; 7、8.刺腐霉菌; 9、10.旋柄腐霉菌; 11.畸雌腐霉菌; 12.强雄腐霉菌; 13.齿孢腐霉菌; 14.群结腐霉菌; 15.宽雄腐霉菌; 16.链状腐霉菌; 17.大豆疫霉菌; 18.辣椒疫霉菌; 19.烟草疫霉菌; 20.灰霉菌菌; 21.梨腐烂菌; 22.棉花黄萎菌; 23.立枯丝核菌; 24.禾谷镰刀菌。 A.Detection of LAMP products using HNB as a visual indicator; B.Detection of LAMP products by agarose gel electrophoresis. M.DNA marker; Tubes and lanes 1-24:1, 2.Pythium aphanidermatum; 3-6.Pythium ultimum; 7, 8.Pythium spinosum; 9, 10.Pythium helicoides; 11.Pythium irregulare; 12.Pythium arrhenomanes; 13.Pythium hydnosporum; 14.Pythium myriotylum; 15.Pythium dissotocum; 16.Pythium catenulatum; 17.Phytophthora sojae; 18.Phytophthora capsici; 19.Phytophthora parasitica; 20.Botrytis cinerea; 21.Valsa pyri; 22.Verticillium dahliae; 23.Rhizoctonia solani; 24.Fusarium graminearum. |
将10倍梯度稀释的瓜果腐霉基因组DNA进行LAMP、普通PCR和Real-time PCR反应, 以此来比较这3种方法的灵敏度。LAMP扩增产物的凝胶电泳图显示, 瓜果腐霉菌的DNA浓度为100 pg·μL-1时呈阳性结果, 而10 pg·μL-1时呈现阴性结果(图 4-A), 表明LAMP反应检测灵敏度为100 pg·μL-1。普通PCR引物的灵敏度检测结果显示, PCR反应的检测灵敏度为1 ng·μL-1(图 4-B)。基于该基因进行的Real-time PCR检测结果显示, 基因组DNA浓度为10 pg·μL-1时不能特异性产生扩增曲线(图 4-C, D), 说明Real-time PCR方法的最低检测灵敏度为100 pg·μL-1。以上结果表明LAMP检测方法的灵敏度与Real-time PCR反应灵敏度一样, 是普通PCR反应灵敏度的100倍。
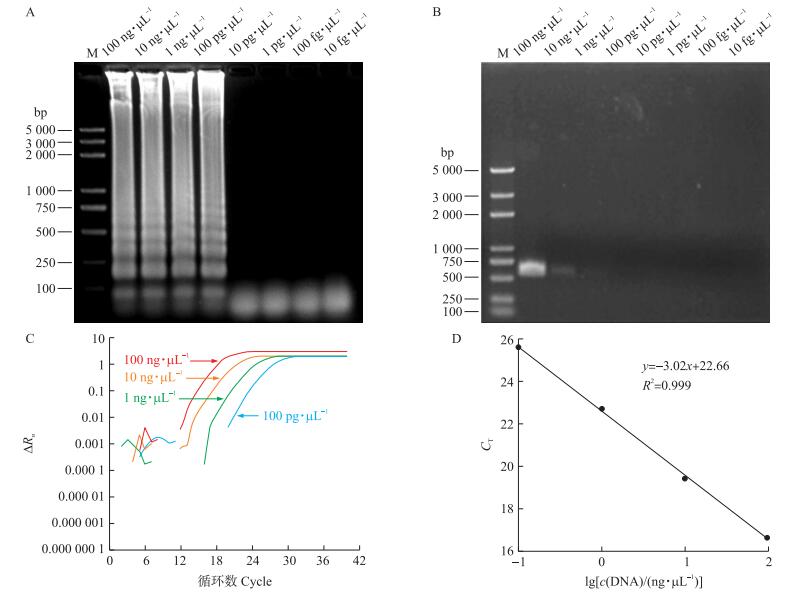
|
图 4 LAMP与普通PCR、Real-time PCR检测方法的灵敏度比较 Figure 4 Comparison of sensitivity among LAMP, conventional PCR and Real-time PCR methods A.LAMP方法的琼脂糖凝胶电泳检测; B.PCR方法的琼脂糖凝胶电泳检测; C.Real-time PCR扩增曲线; D.Real-time PCR的标准曲线及直线方程。 A.Detection of LAMP products by agarose gel electrophoresis; B.Detection of PCR products by agarose gel electrophoresis; C.Amplication plot for Real-time PCR; D.Standard curve obtained by plotting the lg of a known DNA concentration. |
将瓜果腐霉菌分别接种番茄和黄瓜叶片24 h后, 均能看到明显的水质状病斑(图 5-A), 表明瓜果腐霉菌已成功侵染这2种植物叶片。随后分别提取发病组织基因组DNA, 作为LAMP检测反应的DNA模板, 图 5-B显示以上2种作物的发病组织中都能用LAMP方法检测到瓜果腐霉菌。为了进一步评估人工接种植株后不同时间点的检测效果, 将瓜果腐霉菌丝块接种在黄瓜叶片上, 在接种后0、6、12、18、24 h观察叶片的发病情况, 发现接种6 h时能看到非常轻微的病斑(病斑直径小于1 mm), 在接种12 h时能看到轻微的水质状病斑, 病斑直径为1~2 mm, 在接种18 h和24 h时病斑更为明显。随后对以上接种不同时间点的植物发病组织提取DNA, 分别进行了LAMP检测。结果如图 5-C所示:接种0 h时没有检测到瓜果腐霉菌, 而在接种6、12、18和24 h时都检测到了瓜果腐霉菌, 说明在瓜果腐霉菌的侵染早期就能检测到该菌。以上结果表明该LAMP方法在人为模拟田间发病组织的条件下, 能够排除寄主植物DNA等的干扰, 准确地检测出目标菌。
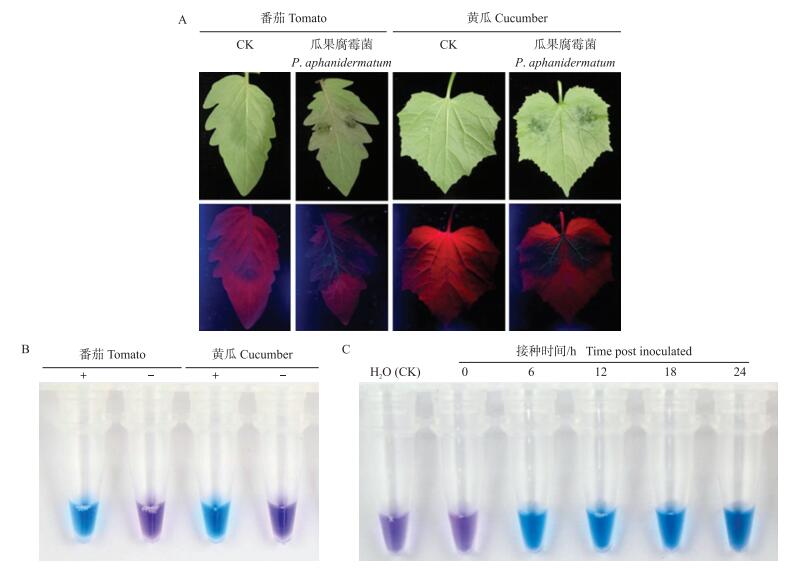
|
图 5 LAMP方法检测人工接种发病组织 Figure 5 Detection of inoculated plant tissues by LAMP assay A.接种瓜果腐霉菌24 h后的植株发病症状, CK表示未接种瓜果腐霉菌的植物; B.可视化显色检测, “+”表示在发病叶片中能检测到瓜果腐霉菌, “-”表示在未接种的健康叶片中没有检测到瓜果腐霉菌; C.接种后不同时间的检测结果。 A.Disease symptom after inoculation of P.aphanidermatum for 24 h, CK represents plant leaf without inoculation of P.aphanidermatum; B.Detection of LAMP products using HNB as a visual indicator, "+"represents positive results while"-"represents negative results; C.Detection results of different time points after inoculation. |
瓜果腐霉菌能造成瓜果腐烂病和猝倒病, 在苗期发病尤为严重, 对农作物生产造成了严重损失。因此, 对瓜果腐霉菌的早期快速、准确检测显得尤为重要。本文应用环介导等温扩增技术(LAMP)检测瓜果腐霉菌。与传统的PCR检测方法相比, LAMP方法具有显著的优点:1)LAMP在恒温条件下就可以进行扩增反应的特性, 大大降低了检测成本(普通的水浴锅即可进行); 2)反应过程耗时短, 只需1 h左右; 3)特异性高, 必须在4~6条内外引物与靶标序列完全匹配的情况下才能进行LAMP反应; 4)结果观测简便, 在扩增前加入染料HNB, 反应结束后直接通过肉眼观察颜色的变化就能判定阳性和阴性结果。基于以上几个优点, LAMP检测技术已经被成功运用于植物病原菌的检测中[23-25]。
特异性是分子检测的一个基本要求, 目前大部分真菌和卵菌的分子检测均是以ITS序列作为靶标开展的[26-28]。ITS序列在真菌和卵菌中普遍存在, 而且在基因组中的拷贝数很多, 因此基于ITS序列设计的分子检测具有微量、灵敏度高等优点。随着高通量测序的发展, 越来越多的病原菌完成了全基因组测序, 为分子检测的靶标筛选提供了数据支撑。本文利用比较基因组学方法, 将瓜果腐霉菌的所有基因比对其他5种完成测序的腐霉菌, 筛选到了对瓜果腐霉菌有序列特异性的基因作为靶标序列, 并基于其中一个编码Trypsin蛋白酶的靶标基因, 成功建立了能特异性检测出瓜果腐霉菌的LAMP方法。本研究为今后开展腐霉菌和其他病原菌的分子检测提供了新的思路:不局限于保守的ITS序列, 而是利用越来越丰富的基因组序列, 通过生物信息学分析手段, 找到对目标菌特异的序列作为检测靶标, 提高病原菌检测的效率。
灵敏度是分子检测的另一个重要指标。本研究建立的瓜果腐霉菌的LAMP方法最低检测灵敏度为100 pg·μL-1, 而之前已报道的几种其他腐霉菌在以ITS序列为靶标建立的LAMP方法的最低检测灵敏度为100 fg·μL-1[19-20], 说明本研究的检测灵敏度还不够高, 推测与使用的靶标序列有关。ITS序列在基因组上的拷贝数多, 因此其灵敏度高, 但特异性相对较差; 而本研究使用的靶标基因在基因组上只有1个拷贝, 因此灵敏度相对低, 但特异性较好。后期计划继续优化LAMP反应体系或使用不同的靶标基因, 以此来提高灵敏度。
本研究建立的LAMP检测方法具有操作简单、快速准确、结果直观的特点, 可以为瓜果腐霉菌所致病害的早期预警提供可靠的技术支持, 也适用于在生产中的推广应用。
| [1] | Martin F N, Loper J E. Soilborne plant diseases caused by Pythium spp.:ecology, epidemiology, and prospects for biological control[J]. Critical Reviews in Plant Sciences, 1999, 18(2): 111-181. DOI: 10.1016/S0735-2689(99)00389-5 |
| [2] | Larkin R P, English J T, Mihail J D. Effects of infection by Pythium spp. on root system morphology of alfalfa seedlings[J]. Phytopathology, 1995, 85(4): 430-435. DOI: 10.1094/Phyto-85-430 |
| [3] | Hendrix F F Jr, Campbell W A. Pythiums as plant pathogens[J]. Annual Review of Phytopathology, 1973, 11(1): 77-98. DOI: 10.1146/annurev.py.11.090173.000453 |
| [4] | Postma J, Geraats B P J, Pastoor R, et al. Characterization of the microbial community involved in the suppression of Pythium aphanidermatum in cucumber grown on rockwool[J]. Phytopathology, 2005, 95(7): 808-818. DOI: 10.1094/PHYTO-95-0808 |
| [5] | Kageyama K, Suzuki M, Priyatmojo A, et al. Characterization and identification of asexual strains of Pythium associated with root rot of rose in Japan[J]. Journal of Phytopathology, 2003, 151(9): 485-491. DOI: 10.1046/j.1439-0434.2003.00757.x |
| [6] |
王晓鸣, 吴全安, 刘晓娟, 等. 寄生玉米的6种腐霉及其致病性研究[J].
植物病理学报, 1994(4): 343-346.
Wang X M, Wu Q A, Liu X J, et al. Identification and pathogenicity of Pythium spp. isolated from maize[J]. Acta Phytopathologica Sinica, 1994(4): 343-346. (in Chinese with English abstract) |
| [7] |
郭世保, 陈俊华, 史洪中. 黄瓜苗期主要病害的识别与防治[J].
现代农村科技, 2015(1): 27.
Guo S B, Chen J H, Shi H Z. Identification and control of major diseases of cucumber seedling[J]. Modern Agricultural Science and Technology, 2015(1): 27. (in Chinese with English abstract) |
| [8] |
潘金菊, 于婷, 尚玉珂, 等. 生防菌BMP-11对瓜果腐霉病菌的抑制活性及其鉴定[J].
植物保护学报, 2008, 35(4): 311-316.
Pan J J, Yu T, Shang Y K, et al. Antifungal activity of antagonistic strain BMP-11 against Pythium aphanidermatum and its identification[J]. Acta Phytophylacica Sinica, 2008, 35(4): 311-316. (in Chinese with English abstract) |
| [9] |
张旭, 尚楠, 张宝, 等. 抗瓜果腐霉芽孢杆菌优良菌株的筛选及生物学特性[J].
食品科学, 2012, 33(5): 138-143.
Zhang X, Shang N, Zhang B, et al. Screening and biological characteristics of Bacillus sp. with high anti-fungal activity against Pythium aphanidermatum[J]. Food Science, 2012, 33(5): 138-143. (in Chinese with English abstract) |
| [10] | Schroeder K L, Okubara P A, Tambong J T, et al. Identification and quantification of pathogenic Pythium spp. from soils in eastern Washington using real-time polymerase chain reaction[J]. Phytopathology, 2006, 96(6): 637-647. DOI: 10.1094/PHYTO-96-0637 |
| [11] | Botton S A, Pereira D I B, Costa M M, et al. Identification of Pythium insidiosum by nested PCR in cutaneous lesions of Brazilian horses and rabbits[J]. Current Microbiology, 2011, 62(4): 1225-1229. DOI: 10.1007/s00284-010-9781-4 |
| [12] | Cullen D W, Toth I K, Boonham N, et al. Development and validation of conventional and quantitative polymerase chain reaction assays for the detection of storage rot potato pathogens, Phytophthora erythroseptica, Pythium ultimum and Phoma foveata[J]. Journal of Phytopathology, 2007, 155(5): 309-315. DOI: 10.1111/jph.2007.155.issue-5 |
| [13] | Morisset D, Stebih D, Cankar K, et al. Alternative DNA amplification methods to PCR and their application in GMO detection:a review[J]. European Food Research and Technology, 2008, 227(5): 1287-1297. DOI: 10.1007/s00217-008-0850-x |
| [14] | Notomi T, Okayama H, Masubuchi H, et al. Loop-mediated isothermal amplification of DNA[J]. Nucleic Acids Research, 2000, 28(12): e63. DOI: 10.1093/nar/28.12.e63 |
| [15] | Goto M, Honda E, Ogura A, et al. Colorimetric detection of loop-mediated isothermal amplification reaction by using hydroxy naphthol blue[J]. Biotechniques, 2009, 46(3): 167-172. DOI: 10.2144/000113072 |
| [16] | Fukuta S, Kato S, Yoshida K, et al. Detection of tomato yellow leaf curl virus by loop-mediated isothermal amplification reaction[J]. Journal of Virological Methods, 2003, 112: 35-40. DOI: 10.1016/S0166-0934(03)00187-3 |
| [17] | Dai T T, Lu C C, Lu J, et al. Development of a loop-mediated isothermal amplification assay for detection of Phytophthora sojae[J]. FEMS Microbiology Letters, 2012, 334(1): 27-34. DOI: 10.1111/fml.2012.334.issue-1 |
| [18] | Rigano L A, Marano M R, Castagnaro A P, et al. Rapid and sensitive detection of Citrus Bacterial Canker by loop-mediated isothermal amplification combined with simple visual evaluation methods[J]. BMC Microbiology, 2010, 10(1): 176-184. DOI: 10.1186/1471-2180-10-176 |
| [19] | Fukuta S, Takahashi R, Kuroyanagi S, et al. Development of loop-mediated isothermal amplification assay for the detection of Pythium myriotylum[J]. Letters in Applied Microbiology, 2014, 59(1): 49-57. DOI: 10.1111/lam.2014.59.issue-1 |
| [20] | Takahashi R, Fukuta S, Kuroyanagi S, et al. Development and application of a loop-mediated isothermal amplification assay for rapid detection of Pythium helicoides[J]. FEMS Microbiology Letters, 2014, 355(1): 28-35. DOI: 10.1111/fml.2014.355.issue-1 |
| [21] |
郑小波.
疫霉菌及其研究技术[M]. 北京: 中国农业出版社, 1997, 81-82.
Zheng X B. Phytophthora and Its Research Technology[M]. Beijing: China Agriculture Press, 1997, 81-82. (in Chinese with English abstract) |
| [22] | Fukuta S, Takahashi R, Kuroyanagi S, et al. Detection of Pythium aphanidermatum in tomato using loop-mediated isothermal amplification(LAMP) with species-specific primers[J]. European Journal of Plant Pathology, 2013, 136(4): 689-701. DOI: 10.1007/s10658-013-0198-3 |
| [23] |
戴婷婷, 陆辰晨, 沈浩, 等. 基于环介导等温扩增技术检测橡树疫霉菌[J].
南京农业大学学报, 2013, 36(3): 23-28.
Dai T T, Lu C C, Shen H, et al. Rapid diagnostic methods for Phytophthora ramorum using LAMP[J]. Journal of Nanjing Agricultural University, 2013, 36(3): 23-28. DOI: 10.7685/j.issn.1000-2030.2013.03.004 (in Chinese with English abstract) |
| [24] |
沈浩, 戴婷婷, 吴翠萍, 等. 基于环介导等温扩增技术检测大豆北方茎溃疡病菌[J].
南京农业大学学报, 2015, 38(2): 255-260.
Shen H, Dai T T, Wu C P, et al. The tef1α-LAMP method for rapid detection of Diaporthe phaseolorum var. caulivora[J]. Journal of Nanjing Agricultural University, 2015, 38(2): 255-260. DOI: 10.7685/j.issn.1000-2030.2015.02.012 (in Chinese with English abstract) |
| [25] |
戴婷婷, 陆辰晨, 郑小波. 环介导等温扩增技术在病原物检测上的应用研究进展[J].
南京农业大学学报, 2015, 38(5): 695-703.
Dai T T, Lu C C, Zheng X B. Application research progress of Loop-mediated isothermal amplification in the pathogenic microorganism[J]. Journal of Nanjing Agricultural University, 2015, 38(5): 695-703. DOI: 10.7685/j.issn.1000-2030.2015.05.001 (in Chinese with English abstract) |
| [26] | Belkhiri A, Buchko J, Klassen G R. The 5S ribosomal RNA gene in Pythium species:two different genomic locations[J]. Molecular Biology and Evolution, 1992, 9(6): 1089-1102. |
| [27] | Buchko J, Klassen G R. Detection of length heterogeneity in the ribosomal DNA of Pythium ultimum by PCR amplification of the intergenic region[J]. Current Genetics, 1990, 18(3): 203-205. DOI: 10.1007/BF00318381 |
| [28] | Wang P H, Wang Y T, White J G. Species-specific PCR primers for Pythium developed from ribosomal ITS1 region[J]. Letters in Applied Microbiology, 2003, 37(2): 127-132. DOI: 10.1046/j.1472-765X.2003.01353.x |




