文章信息
- 许乐峰, 许昕阳, 张欢, 赵志刚, 郑天慧, 赵婕妤, 曾召琼, 刘喜, 陈赛华, 万建民
- XU Lefeng, XU Xinyang, ZHANG Huan, ZHAO Zhigang, ZHENG Tianhui, ZHAO Jieyu, ZENG Zhaoqiong, LIU Xi, CHEN Saihua, WAN Jianmin
- 水稻晚开花突变体lft1的鉴定与基因克隆
- Identification and map-based gene cloning of a late flowering mutant lft1 in rice(Oryza sativa L.)
- 南京农业大学学报, 2018, 41(1): 38-48
- Journal of Nanjing Agricultural University, 2018, 41(1): 38-48.
- http://dx.doi.org/10.7685/jnau.201705034
-
文章历史
- 收稿日期: 2017-05-19
2. 中国农业科学院作物研究所, 北京 100081
2. Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
水稻开花期性状在遗传上受众多数量性状位点调控, 且受外界光照和温度等因素共同影响。国内外学者利用不同的群体定位了大量的水稻开花期QTL位点。据Gramene网站(http://www.gramene.org/qtl/index.html)不完全统计显示, 利用不同的群体已经定位水稻开花期QTL达上千个, 分布于12条染色体上, 而至今克隆的基因却为数不多。抽穗期性状属于典型的数量性状, 目前对于抽穗期的遗传研究主要从自然变异入手, 即利用抽穗期存在差异的自然亲本构建F2分离群体, 重组自交系(recombinant inbred lines, RILs)群体、DH(double haploid)群体、近等基因系(near-isogenics lines, NILs)、染色体片段置换系(chromosome segment substitution lines, CSSLs)等鉴定调控抽穗期的QTL位点, 并在构建高世代回交材料的基础上对QTL位点进行定位和克隆[1]。也有研究利用Nipponbare/Kasalath F2群体首先定位了5个QTL位点, Hd1~Hd5; 随后在回交自交群体BC1F5中又检测到Hd7、Hd8和Hd11位点, 在高世代回交自交群体中又检测到Hd6、Hd10等其他QTL位点[2-6]。在此基础上利用高世代回交群体开展基因的精细定位和克隆, 其中第1个被克隆的主效QTL为Hd1, 它与拟南芥CONSTANS(CO)基因同源。不同于CO长日照下促进开花的功能, Hd1在长日照条件下延迟开花, 但在短日照条件下促进开花, 具有双重功能[7]。Kojima等[8]克隆了水稻的Hd3a基因, Hd3a与拟南芥成花素基因FT同源, 同样也促进开花。在距离Hd3a约11.5 kb的位置上存在Hd3a的同源基因RFT1, 同样被认为是开花信号, 能在短日照下促进生长时期的转变, 当Hd3a与RFT1同时缺失时, 水稻出现无法开花的现象, 说明两者对于水稻营养生长向生殖生长转化不可或缺[9]。Hayama等[10]用荧光差异显示的方法, 获得与拟南芥中GIGANTEA高度同源的基因, 命名为OsGI, OsGI呈节律表达并调控Hd1, Hd1通过调控Hd3a和RFT1的表达从而调控开花期, 这一调控通路在水稻和拟南芥等不同物种中都相对比较保守[11-12]。
一些水稻中特异的调控因子被发现。Doi等[13]用图位克隆法克隆了水稻开花期QTL-Ehd1。Ehd1通过在短日下诱导Hd3a的表达, 促进水稻开花, 而长日下, Ehd1的表达受到抑制[14]。Xue等[15]克隆了Ghd7基因, 该基因同时控制开花期、株高、产量, 显示一因多效。Ehd1和Ghd7被认为是水稻中特有的调控开花的转录因子, 它们通过调节下游基因的表达来影响开花期。同样, 众多转录因子通过调控Ehd1的转录调控开花期, 例如, OsMADS50和OsMADS51基因被认为作用于Ehd1的上游, 促进Ehd1过量表达, 使作物提前开花[16-17]。DTH8基因通过抑制Ehd1和Hd3a的表达延迟开花[18]。Ehd2编码C2H2锌指蛋白类转录因子, Ehd4编码CCCH锌指蛋白类转录因子, 两者均通过促进Ehd1的表达来促进开花[19-20]。然而, DTH2作为CONSTANS类蛋白, 可独立于Hd1和Ehd1通路, 直接促进Hd3a和RFT1的表达来促进开花[21]。除了转录水平的调控以外, 一些转录后水平的调控基因也被报道:Hd6编码CK2α亚基, 推测其可通过磷酸化Hd2来调控开花期[22]; Hd16编码酪蛋白激酶Ⅰ, 它可能通过磷酸化Ghd7来延迟开花[23]。除磷酸化修饰以外, 甲基化水平的调控对于开花的调控也有被报道:一类编码含有SET功能结构域蛋白的基因, 例如拟南芥中的SDG4、SDG8、SDG25和水稻中的SDG708、SDG725等, 它们可以通过影响H3K36的甲基化水平, 从而影响某些开花基因的转录, 造成生育期的延迟[24-28]。Sun等[29]发现一个T-DNA突变体lvp1(long vegetative phase 1), 利用图位克隆的方法, 克隆到一个包含SET结构域的基因, 命名为SDG724(SET domian group protein 724)。SDG724编码了一个调节H3K36甲基化水平的甲基转移酶。荧光定量检测及ChIP试验证明SDG724可通过调节OsMADS50和RFT1的H3K36me2/3水平, 影响下游开花基因的转录, 进而影响水稻的生育期。
虽然已克隆了一些调控水稻开花的基因, 但要深入了解水稻的开花作用机制, 还需要进一步丰富水稻开花期基因调控网络。本研究鉴定了一个晚开花突变体lft1, 考查了其农艺性状, 对突变性状进行了遗传分析、T-DNA插入位点的共分离分析、基因的初定位、突变位点的验证及相关基因表达水平的检测, 为进一步探讨该基因的功能及应用潜力, 丰富和完善开花期的分子作用机制奠定基础。
1 材料与方法 1.1 试验材料本研究所用材料来源于中国农业科学院路铁钢老师提供的‘日本晴’的T-DNA插入突变体库, 因其具有晚开花的表型, 命名为lft1(late flowering time1)[30]。利用该材料与野生型‘日本晴’组配了lft1/日本晴F2遗传分析群体; 与‘9311’组配了lft1/9311 F2定位群体; 此外, 将野生型‘日本晴’与‘9311’杂交, 获得对照日本晴/9311 F2群体。所有材料均种植在南京农业大学土桥水稻育种实验基地。
1.2 主要农艺性状调查突变体lft1和野生型‘日本晴’均种植在南京长日照条件下, 播种日期5月18日, 小区种植, 田间管理同大田生产。开花期的调查以每株主穗抽出1 cm作为该株始穗的标准, 以播种到始穗的时间(d)作为开花期。对F2分离群体进行分单株编号, 取叶片样本, 并调查每个单株的开花期。开花后40 d左右亲本混合收种, 置于50 ℃烘箱72 h, 利用万深SC-G自动种子考种分析及千粒重仪, 采集突变植株和对照亲本的粒长、粒宽、千粒质量等数据。长日照处理在南京田间自然长日照条件下进行, 平均日照时间大于13 h; 短日照处理与长日照处理同步遮光的条件下进行, 控制日照时间为10 h。
1.3 T-DNA旁邻序列的克隆与T-DNA插入的鉴定T-DNA旁邻序列的克隆在基于PCR的基因组walking方法的基础上稍作改进[31-32]。首先选择合适的平末端酶切位点对基因组DNA进行完全酶切, 然后将引物ADAR1(100 μmol·L-1)和ADAR2(100 μmol·L-1)加热80 ℃变性处理10 min后退火形成DNA接头, 将制备好的DNA接头和1 μg基因组DNA酶切产物同时加入到T4连接酶反应体系中, 16 ℃连接过夜。最后, 以连接产物为模板, 利用接头设计的引物序列APR1和PLB1引物进行首轮扩增, 扩增产物稀释后, 再用APR2和PLB2进行巢式PCR扩增, 扩增产物连接到T载体中进行测序(表 1)。基于旁邻序列设计L1和L2引物, 分别与通用R引物组合进行PCR扩增, 用于鉴定含有T-DNA插入、非T-DNA插入和杂合3种不同的基因型。基于潮霉素抗性基因设计的Hpt900L/R引物对, 以验证T-DNA的有无。
| 引物名称 Primer name |
序列(5′→3′) Primer sequence |
| ADARl | CTAATACGAGTCACTATAGCGCTCGAGCGGCCGCCGGGGAGGT |
| ADAR2 | ACCTCCCC |
| APR1 | GGATCCTAATACGAGTCACTATAGCGC |
| APR2 | CTATAGCGCTCGAGCGGC |
| PLB1 | CAAGCCTTGAATCGTCCATACTG |
| PLB2 | GTCTTGATGAGACCTGCTGC |
| L1 | GCCTCTCTAACCATCTGTGG |
| L2 | GAATCCAGTAAGTAGGCTCG |
| R | GCTCCTCGCATTCTGGTATC |
| Hpt900-L | AAGTTCGACAGCGTCTCCGAC |
| Hpt900-R | TCTACACAGCCATCGGTCCAG |
已知在水稻全基因组上分布的SSR标记有2 740个(http://www.gramene.org), 本研究首先从已报道的SSR标记中, 筛选均匀分布在12条染色体上的在‘日本晴’和‘9311’之间存在多态性的SSR标记, 用作基因的初步连锁分析。利用GRAMENE和RAP-DB数据库中的‘日本晴’和‘9311’序列信息, 在初步定位的区段内进一步寻找序列差异, 开发相应分子标记, 用于基因的精细定位(表 2)。
| 名称Name | 正向引物序列(5′→3′) Forward primer sequence | 反向引物序列(5′→3′) Reverse primer sequence |
| RM8219 | AACCATTGTTGAGCAAATTC | GATAAGCAGGGATTGGAAAG |
| RM5799 | ATCGAACCATCCAGGATGAC | TTGCACAAGAGGCAACACTC |
| RM444 | GCTCCACCTGCTTAAGCATC | TGAAGACCATGTTCTGCAGG |
| RM219 | CGTCGGATGATGTAAAGCCT | CATATCGGCATTCGCCTG |
| C9-085 | GCGTTGGGTTTCCTCAAATA | GTATAGGCCCAGAATCTAAAGTTG |
| C9-099 | CCCTCCGATCATCTGCTAAG | ATGAACCCTAACCTTCCTTGA |
| RM3912 | TGTGTGTGCCCGATCTAC | CCTCTCGATGAGCATTCC |
| RM105 | AGGTGGTGAGGGTGAACTTG | TGGGATTAGAGCTTTGGTGG |
| RM6839 | GAACAGAGGAGGAGATCGAGAGG | CTTCTTGGGAGATGCAGAAATGG |
| sA | AGAAACGGAGCTACCAACCAG | TGAGAGGATTCATTCCCACAAC |
| sB | GTTCAGGGTCTCAGATGCG | GATATGGTGTAGCCTCTTTTCTG |
| sD | GGAAGTTGATTTCCCTTTCTTTC | CATGGAAACTTCAGGCTATGG |
| sE | ATTGTTGGCTGGGTGAGTTTAC | TAGCCAGAAATGCTAATGTTGC |
| S34 | AACCTAGAGTGTTTGAGTCTGCTC | TCCTACCATAACTGGATTCATAGTT |
| SJ9-32 | CTTTATCATTCACAGCGTCC | TGTTGAGTTGGCAGATTACC |
采集新鲜的水稻叶片直接提取DNA或保存于-20 ℃冰箱备用, 采用改进的SDS法抽提水稻基因组DNA。将叶片磨成粉末状, 加入600 μL SDS提取液, 摇匀, 65 ℃温浴30 min; 加入100 μL KAC溶液, 摇匀后冰浴30 min; 加入600 μL氯仿抽提液, 1 200 r·min-1离心30 min; 取上清液, 加入-20 ℃预冷的800 μL(2倍体积上清液)无水乙醇, 摇匀后置于冰上30 min; 离心后沉淀用70%乙醇洗涤2次, 晾干后溶于TE缓冲液, -20 ℃保存。
1.6 PCR反应及电泳检测采用10 μL反应体系:DNA模板2 μL, 前、后引物(2 μmol·L-1)各1 μL, 10×PCR Buffer 1 μL, dNTP(10 mmol·L-1)0.2 μL, Taq酶0.2 μL, ddH2O 4.6 μL。PCR反应程序:94 ℃预变性5 min; 95 ℃变性30 s, 55~60 ℃退火30 s, 72 ℃延伸40 s, 34个循环; 72 ℃延伸10 min后10 ℃保存。PCR产物采用琼脂糖凝胶电泳, EB染色后检测, 或采用聚丙烯酰胺凝胶电泳检测, 用1 g·L-1AgNO3溶液进行银染显色。
1.7 实时荧光定量PCR检测基因的表达量野生型‘日本晴’和突变体lft1的幼苗首先同步生长20 d左右, 然后移至人工光照培养箱中进行长、短日照处理。培养箱温度30 ℃, 其中短日照处理条件是光照/黑暗时间为10 h/14 h, 长日照处理条件是光照/黑暗时间为14 h/10 h。经过10 d的光照处理后, 每3 h取样1次, 每次混取3~5株苗的倒数第2片叶的顶端1/3样品, 用于提取RNA或-80 ℃保存备用。采用TIANGEN公司的RNAprep Pure Plant Kit(Cat.#DP432)提取RNA, 参照TaKaRa公司的PrimeScript RT Reagent Kit with gDNA Eraser(Perfect Real Time, Cat.#047A)反转录合成cDNA。荧光定量PCR反应体系为20 μL, 包含2 μL cDNA模板、2 μL引物、10 μL SYBR Green荧光染料和6 μL RNase-free H2O, 在Bio-Rad荧光定量PCR仪上进行扩增, 每个反应3个生物学重复。利用CFXManager软件进行数据的收集和整理。检测开花期相关基因的定量引物序列见表 3。
| 基因Gene | 正向引物序列(5′→3′) Forward primer sequence | 反向引物序列(5′→3′) Reverse primer sequence |
| SDG724 | AAAATGAAGAGGCAGGGTG | CTCCCTCAACAGTCCATTTCTG |
| RFT1 | ACCCTAACCTTAGGGAGTATCTACAC | GCCTGCATGCATATACAGCTAGGCAG |
| Hd3a | AGCCCAAGTGACCCTAACCT | GTTGTAGAGCTCGGCGAAGT |
| Ghd7 | GCTTGAACCCAAACACGG | CTCATCTCGGCATAGGCTT |
| Hd1 | TCAGCAACAGCATATCTTTCTCATCA | TCTGGAATTTGGCATATCTATCACC |
| DTH8 | CAGGAGTGCGTGTCGGAGTT | GGTCGTCGCCGTTGATGGT |
| OsMADS51 | GTTTGCTCTGCTCCTACTC | ACTCCTCCTCCAGCATTGAA |
| OsMADS50 | ATGCAATGACACCAAACCATC | GGTAGTGGAGTCTGCCGATC |
| Actin | GTACAGTGTCTGGATTGGAGGAT | GGGTCCGAAGAATTAGAAGCA |
南京自然长日照条件下, 野生型‘日本晴’的平均开花期为78 d, 而lft1则推迟了20 d(表 4, 图 1)。当采用人工遮光处理后, 两者的开花期接近, 这表明lft1中的突变基因对于光周期具有较强的敏感性, 长日照条件下作用明显。
| 材料 Materials |
开花期/d Flowering time |
粒长/mm Grain length |
粒宽/mm Grain width |
千粒质量/g 1 000-grain weight |
| 日本晴 | 78.2±1.3 | 7.256±0.359 | 3.126±0.178 | 23.942±0.107 |
| lft1 | 98.4±0.9** | 7.696±0.243** | 3.144±0.247 | 26.726±0.053** |
| Note:**P < 0.01. | ||||

|
图 1 野生型(WT)‘日本晴’和突变体lft1的表型特征 Figure 1 Phenotype of the wide type(WT)'Nipponbare'and lft1 mutant A.南京自然长日照下播种后112 d植株形态(左, 日本晴; 右, lft1); B.籽粒形态(左, 日本晴; 右, lft1); C.粒宽比较(上, 日本晴; 下, lft1); D.粒长比较(左, 日本晴; 右, lft1)。 A.The plant morphology of the 112-old-day wide type and lft1 mutant under Nanjing natural field conditions(Left, Nipponbare; Right, lft1); B.Grain morphology(Left, Nipponbare; Right, lft1); C.Comparison of grain width(Left, Nipponbare; Right, lft1); D.Comparison of grain length(Left, Nipponbare; Right, lft1). |
除此之外, lft1较‘日本晴’籽粒形态上也有了明显的变化, 粒长均显著增加, 千粒质量也明显增加(表 4, 图 1)。这表明lft1中的突变基因不仅能够延迟开花期, 同时也表现出一因多效性。有意思的是, 在lft1的颖壳上连续多年均稳定观察到褐色的斑点沉积, 这说明在lft1可能还存在氧化胁迫反应。
2.2 遗传分析与T-DNA共分离分析以lft1为母本, ‘日本晴’为父本, 构建了lft1/日本晴的F2分离群体, 并在南京自然长日照下对群体的开花期进行了调查。结果发现, 群体的开花主要集中在2个时间段, 8月10日至8月25日和9月15日至9月17日, 呈现明显的双峰分布, 且早开花与晚开花的分布基本符合3 : 1分离规律, 这说明晚开花的表型由1对隐性核基因控制。
为验证开花早晚的分离与T-DNA插入的关系, 我们首先克隆了T-DNA旁邻序列。经测序, 发现T-DNA插入点位于第3染色体上基因Os03g0107500与Os03g0107600之间, 且T-DNA为反向插入。针对插入位点, 我们设计了2对引物L2/R与L1/R, 其中前者可以在‘日本晴’中扩增385 bp的片段, 而在lft1中无法有效扩增; 后者能够与T-DNA的左边界匹配, 在lft1中扩增322 bp的片段, 而在‘日本晴’中无法扩增。利用L1/R、L2/R和潮霉素抗性标记基因上特异性的引物Hpt900, 对分离群体中开花早晚个体进行了鉴定, 结果显示:无论是早开花还是晚开花的个体, 均呈现有或无T-DNA插入(N)、T-DNA纯合(T)和T-DNA插入位点杂合(H)3种基因型(表 5), 说明T-DNA的插入与开花期早晚表型之间不存在共分离, 即lft1中的晚开花表型并不是由于第3染色体的T-DNA插入引起的。
| 引物和基因型 Primer and genotype |
早开花单株 Early flowering individuals |
晚开花单株 Late flowering individuals |
||||||||||||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | |||
| Primer | L1/R | + | + | + | + | + | + | + | - | + | + | - | - | - | + | + | + | + | - | + | + | |
| L2/R | + | - | + | + | + | - | - | + | - | - | + | + | + | - | - | + | + | + | + | + | ||
| Hpt900 | R | R | R | R | R | R | R | S | R | R | S | S | S | R | R | R | R | S | R | R | ||
| Genotype | H | T | H | H | H | T | T | N | T | T | N | N | N | T | T | H | H | N | H | H | ||
| HD | 81 | 77 | 82 | 80 | 76 | 83 | 82 | 84 | 81 | 85 | 85 | 76 | 91 | 83 | 113 | 111 | 112 | 113 | 113 | 113 | ||
| 注: “+”表示有相应扩增产物, “-”表示无相应扩增产物; “R”或“S”分别表示Hpt900有扩增产物或无扩增产物, 且叶片潮霉素抗性鉴定阳性或阴性; “T”“H”和“N”分别表示T-DNA插入位点纯合、杂合或没有插入。HD表示花期, 从播种至始穗的时间(d)。 Note: “+”indicates products amplified; “-”indicates none amplified products; “R”or“S”indicates positive and negative results of Hpt900 PCR amplification, respectively, and leaf hygromycin resistance identification; “T”“H”and“N”respectively indicates homozygous, heterozygous or none of T-DNA insertion. HD means flowering time, which was presented by days from sowing to heading. |
||||||||||||||||||||||
我们组配了lft1与‘9311’的分离群体用于基因的定位。为排除籼粳交超亲迟熟的影响, 分析突变基因的效应, 我们将野生型‘日本晴’与‘9311’进行杂交, 构建了对照日本晴/9311 F2群体。通过对2个群体同步进行开花期调查后发现, 2个品种开花期均呈现正态分布, 这说明在‘日本晴’与‘9311’之间存在多个影响开花期的位点。有意思的是, 我们将lft1/9311 F2群体与对照日本晴/9311 F2群体比较后发现, 两者开花期分布范围基本一致, 但在lft1/9311 F2群体中产生了极端晚开花的个体(图 2)。这一结果表明lft1/9311 F2群体中的极端表型极有可能是由于lft1中的基因突变而引起的。
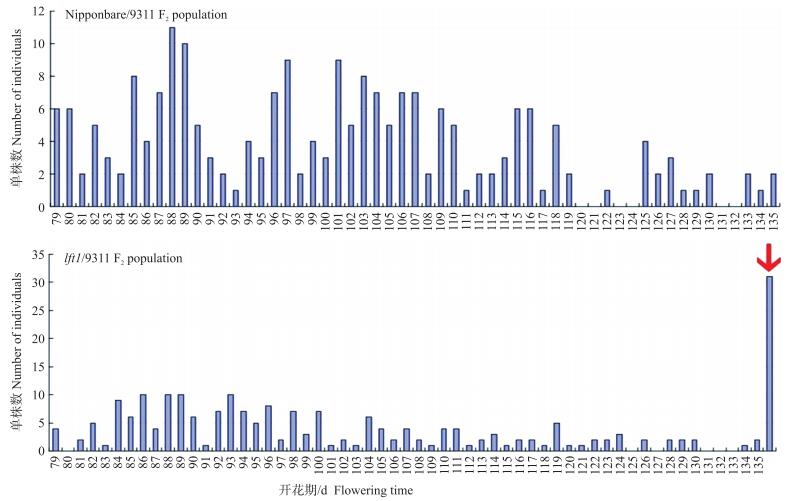
|
图 2 日本晴/9311 F2群体与lft1/9311 F2群体的开花期分布 Figure 2 Distribution of flowering time in Nipponbare/9311 F2 population and lft1/9311 F2 population 箭头所指为开花期大于135 d的极端个体。 Arrow refers to extremely late heading individuals whose flowering time is more than 135 days. |
从lft1/9311 F2群体中获得了一定数目的极端晚开花个体。从中选取10个单株, 采用121对具有多态性的分子标记进行基因的全染色体组的连锁分析。结果表明:在第9染色体长臂上的SSR标记RM8219和RM6839与目标性状存在紧密连锁(图 3-A)。在RM8219附近加密分子标记, 对58个晚开花个体进一步进行基因型鉴定后, 我们将初定位区间限定在SSR标记RM219和RM3912之间, 物理距离为2.94 Mb。随后, 在定位区间内进一步开发了在亲本间有多态的dCAPS标记C9-085和C9-099, 用于鉴定交换单株, 将lft1突变基因定位于RM219和C9-099之间约2.01 Mb的区域内(图 3-B)。
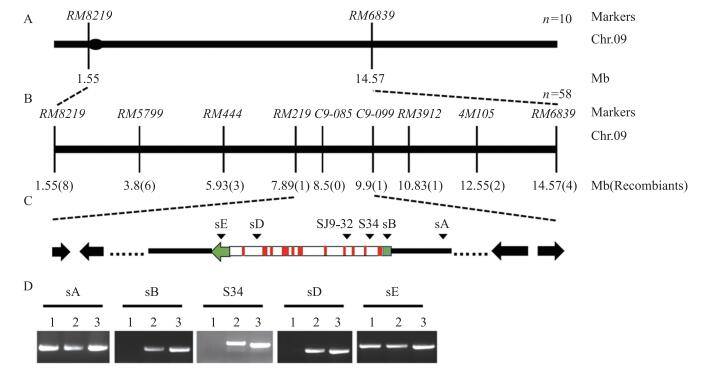
|
图 3 基因的初定位和候选基因鉴定 Figure 3 Gene initial mapping and candidate gene identification A.利用F2群体中的10株极晚开花单株进行初连锁, 将目标基因限定在第9染色体SSR标记RM8219和RM6839之间, 区间大小为2.94 Mb; B.在初定位区间内加密分子标记, 最终将目标基因定位在标记RM219和C9-099之间, 区间大小为2.01 Mb; C.标记SJ9-32位于LOC_Os09g13740基因上, 在lft1中无法有效扩增。在LOC_Os09g13740基因上设计标签标记sA、sB、S34、sD和sE; D.序列标签标记在lft1(1)、‘日本晴’(2)和‘9311’(3)中扩增电泳结果。 A.Initial linkage mapping using 10 extremely late heading individuals, the responsible gene was restricted on the long arm of chromosome 9 within a 2.94 Mb region between the SSR markers RM8219 and RM6839;B.Based on the newly developed markers, the gene locus was further mapped within a 2.01 Mb region between the markers RM219 and C9-099;C.Marker SJ9-32, locating on LOC_Os09g13740 gene, can′t be amplified in lft1 mutant, sequence tags, sA, sB, S34, sD and sE was designed; D.Electrophoresis results of amplicons in lft1(1), 'Nipponbare'(2) and'9311'(3)by sequence tags sA, sB, S34, sD and sE. |
采用标记SJ9-32进行基因型鉴定时发现, 该标记在‘日本晴’和‘9311’之间可有效实现扩增且存在多态性, 但突变体lft1和所有的极端晚开花个体都无法有效扩增, 推测在该标记位点存在片段缺失。为证实这个想法, 我们分别在该标记的上游和下游设计标记(图 3-C)进行验证。结果发现:上游分子标记sA和下游标记sE在lft1、‘日本晴’和‘9311’中均可以有效扩增, 而sB、s34和sD在‘日本晴’和‘9311’中均可以扩增到相应的DNA片段, 在lft1中则不能(图 3-D), 所有极端个体的鉴定结果与lft1一致。这些结果说明在sA和sE之间存在DNA片段的缺失, 而这一缺失片段正好包含LOC_Os09g13740基因。LOC_Os09g13740基因(即SDG724)编码一个调控组蛋白H3赖氨酸K36位残基(H3K36)甲基化的酶。据报道该基因能够介导OsMADS50和RFT1发生二甲基化、三甲基化修饰(H3K36me2/3), 进而促进开花[29]。为了进一步证实lft1晚开花表型与LOC_Os09g13740基因缺失相关, 我们将lft1和Sun等[29]报道的lvp1突变体进行了等位性测验(两者背景都是‘日本晴’), 结果F1植株开花表型与突变体一致, 证实lft1是由于SDG724基因的缺失而呈现延迟开花的表型。
2.5 开花期相关基因的表达分析SDG724编码的是一种组蛋白甲基化转移酶, 可以甲基化修饰组蛋白H3赖氨酸K36位残基(H3K36), 并且可以完成一甲基化、二甲基化、三甲基化修饰(H3K36me1/2/3)。通常认为H3K36的甲基化修饰可以介导基因的转录, 所以我们检测比较了lft1和野生型之间开花期相关基因的转录水平。结果表明:在人工长日照条件下, 突变体lft1体内的SDG724、OsMADS51、RFT1转录水平相比于野生型出现显著下调, 而其他基因如Ghd7、Hd1、DTH8、OsMADS50、Hd3a则无明显差异(图 4-A)。但在人工短日照条件下, 突变体lft1内只有SDG724表达水平相比于野生型有显著下调, 而其他基因如Ghd7、Hd1、DTH8、OsMADS50、Hd3a、OsMADS51无明显差异(图 4-B)。因此, 我们认为突变体lft1长日照条件下开花期延迟是由于其体内的OsMADS51和RFT1表达水平降低造成的。

|
图 4 长日照(A)和短日照(B)条件下野生型和突变体lft1开花期相关基因的相对表达量 Figure 4 The relative expression level of flowering time genes between wild-type and lft1 mutant under long-day(A)and short-day(B)conditions 长日照条件下光照/黑暗时间为14 h/10 h, 短日照条件下光照/黑暗时间为10 h/14 h。 Long-day condition is 14 h for light, 10 h for dark, and short-day condition is 10 h for light, 14 h for dark. |
开花期是水稻重要的农艺性状之一, 它决定了水稻生育期的长短, 决定了不同品种的地区适应性, 因而研究开花期对育种实践具有重要意义。目前, 认为调控水稻开花的通路主要有2条:一条是在拟南芥和水稻中比较保守的Hd1通路; 另一条是仅在水稻中发现的Ehd1通路。Ehd1编码一类B-type响应元件, 短日照条件下可以独立调控下游的FT类基因的表达而促进开花[33]。最新的很多文献报道, 不同基因通过调控Ehd1的表达来影响生育期, 其中Hd1、Ghd7、DTH8等通过抑制Ehd1的表达而抑制开花, 而Ehd2、Ehd4、OsMADS51、OsMADS50等则通过促进Ehd1的表达而促进开花。Ehd1可以说是整个开花期网络调控的重要节点[16-20, 34-35]。然而, 本研究中克隆的SDG724基因是通过影响甲基化修饰水平来调控开花期基因的表达, 不依赖于Hd1和Ehd1这2条已知的通路, 并且它通过影响OsMADS50和RFT1来影响开花, 丰富了对水稻开花期调控机制的认识。
开花期属于复杂的数量性状, 前期对于开花期的基因克隆多数是通过QTL的克隆实现的。为了排除遗传背景对QTL效应的影响, 对于基因的克隆往往需要制备高世代回交的材料, 如染色体片段置换系和近等基因系等, 这就使得开花期研究工作费时费力。本研究尝试采用突变体的方式来克隆开花期基因, 首先对T-DNA插入突变体, 可通过旁邻序列的克隆来获取插入位点, 分析候选基因。但是由于本研究中T-DNA的插入位点与开花期延迟表型并不关联, 因此, 我们利用定位群体展开研究。开花期表型的复杂性决定了其克隆的难度。为了能够快速锁定基因的位置, 我们将突变体与野生型同时与特定的材料进行配组, 然后分别调查对照群体与定位群体的开花期表型分布, 从而观察突变基因在群体中的效应。本研究中, lft1/9311 F2群体相比日本晴/9311 F2群体出现了明显晚开花的个体, 这是由于lft1的基因突变造成的, 因此我们采用这些极端表型的个体进行基因的初定位。在定位过程中未发现性状与基因型不符合的现象, 说明利用对照群体筛选个体的方法是可行的。但这个方法也取决于基因的效应, 我们对其他一些突变体进行类似研究时发现, 一些效应不显著的突变体在组配群体后效应并不显著, 这极有可能是受到其他开花期基因影响所导致的, 由此可见, 对于开花期基因利用突变体来进行定位仍存在一定的难度。
在水稻的遗传学研究过程中, 非常多的等位基因被鉴定出来, 等位基因的研究对进一步了解基因的功能有着重要的作用。本研究所用突变体lft1与Sun等[29]报道的突变体lvp1的表型十分相似, 经验证是SDG724基因的一个等位突变体。Lvp1与野生型在海南三亚环境下存在一定的开花期延迟, 但差异从北京长日照条件下的延迟66 d缩短到10 d左右, 这说明SDG724基因对于光周期的响应是敏感的。本研究中, 在南京自然长日照条件下lft1突变体较野生型延迟开花20 d左右, 而在同样环境下进行同步短日照遮光处理后没有观察到明显差异, 这同样说明SDG724基因对于光周期的响应是敏感的。有意思的是, lvp1与野生型在自然短日照下存在10 d的差异, 而lft1与野生型在人工短日照下并没有差异。可能的解释有两种:1)等位基因的类型不同引起的。在突变体lvp1中, SDG724基因的内含子与外显子边界处AG到AC的替换使得115 bp的内含子区域被转录, 最终引起翻译的提前终止, 而本研究中SDG724基因则完全缺失。虽然两者均引起功能丧失, 但后者是基因整体丢失, 而前者仍保留部分结构域, 这种功能域截短的蛋白是否在植株体内行使功能仍不确定。2)实验环境差异引起的。本研究中, 短日照为人工遮光处理的短日照条件, 除日照长度以外, 温度等环境因素均与外界自然长日照保持一致, 能准确比较长、短日照下的性状差异。在Sun等[29]的研究中, 短日照为海南三亚的自然环境, 这与长日照条件相比, 除了日照长度以外还存在温度等其他环境差异。植物能够感受温度变化, 并通过甲基化水平的调控来影响开花的报道很多。以拟南芥为例, 低温可以诱导拟南芥开花, 而这与FLC(flowering locus C)的组蛋白甲基化水平有密切关系[36]。SDG724编码的组蛋白甲基转移酶(histone methyltransferase), 是一种可以在组蛋白的特定位点加上甲基基团的酶[37]。因此, 不排除这样的可能性, 在海南三亚环境下lvp1与野生型的开花期差异是感受低温等环境后甲基化水平的差异而引起的。鉴于这个线索, 后续可对SDG724是否通过感受温度来调节开花展开深入的研究。Sun等[29]报道的突变体lvp1中, H3K36me2/3的转录水平显著降低, 而H3K36的甲基化主要影响基因转录的延伸过程, 这种组蛋白修饰方式大量存在于转录活化基因的编码区。该基因通过调节开花基因如OsMADS50、OsMADS51、Ehd1及RFT1的转录水平来影响开花, 但是它具体通过怎样的机制改变靶基因位点上的组蛋白的甲基化水平仍有待于进一步研究。
本研究中, 晚开花突变体lft1表型由1对隐性核基因控制。自然长日照条件下, 相较于野生型, 突变体lft1开花期时间延迟20 d左右, 籽粒形态上也有了明显的变化, 粒长和千粒质量也明显增加。通过旁邻序列克隆、共分离分析、图位克隆、等位性测验和qRT-PCR等方法, 我们确定导致突变体lft1开花延迟是SDG724基因的功能缺失, OsMADS51和RFT1的转录水平相应下调造成的。
中国农业科学院作物研究所路铁钢老师提供T-DNA插入突变体材料lft1, 中国科学院遗传研究所所储成才老师、方军老师提供lvp1突变体, 农业部长江中下游粳稻生物学与遗传育种重点实验室、长江流域杂交水稻协同创新中心、江苏省现代作物生产中心也给予支持, 谨致谢意。
| [1] |
吴晶晶, 魏子尧, 杨春艳, 等. 控制水稻抽穗延迟基因OsSET34的鉴定和图位克隆[J].
南京农业大学学报, 2015, 38(5): 704-711.
Wu J J, Wei Z Y, Yang C Y, et al. Identification and map-based cloning of OsSET34, a gene controling the delay of heading date in rice(Oryza sativa L.)[J]. Journal of Nanjing Agricultural University, 2015, 38(5): 704-711. DOI: 10.7685/j.issn.1000-2030.2015.05.002 (in Chinese with English abstract) |
| [2] | Yano M, Harushima Y, Nagamura Y, et al. Identification of quantitative trait loci controlling heading date in rice using a high-density linkage map[J]. Theoretical and Applied Genetics, 1997, 95(7): 1025-1032. DOI: 10.1007/s001220050658 |
| [3] | Takeuchi Y, Lin S Y, Sasaki T, et al. Fine linkage mapping enables dissection of closely linked quantitative trait loci for seed dormancy and heading in rice[J]. Theoretical and Applied Genetics, 2003, 107(7): 1174-1180. DOI: 10.1007/s00122-003-1364-3 |
| [4] | Yamamoto T, Lin H, Sasaki T, et al. Identification of heading date quantitative trait locus Hd6 and characterization of its epistatic interactions with Hd2 in rice using advanced backcross progeny[J]. Genetics, 2000, 154(2): 885-891. |
| [5] | Lin H X, Ashikari M, Yamanouchi U, et al. Identification and characterization of a quantitative trait locus, Hd9, controlling heading date in rice[J]. Breeding Science, 2002, 52(1): 35-41. DOI: 10.1270/jsbbs.52.35 |
| [6] | Lin S Y, Sasaki T, Yano M. Mapping quantitative trait loci controlling seed dormancy and heading date in rice, Oryza sativa L., using backcross inbred lines[J]. Theoretical and Applied Genetics, 1998, 96(8): 997-1003. DOI: 10.1007/s001220050831 |
| [7] | Yano M, Katayose Y, Ashikari M, et al. Hd1, a major photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering time gene CONSTANS[J]. Plant Cell, 2000, 12(12): 2473-2484. DOI: 10.1105/tpc.12.12.2473 |
| [8] | Kojima S, Takahashi Y, Kobayashi Y, et al. Hd3a, a rice ortholog of the Arabidopsis FT gene, promotes transition to flowering downstream of Hd1 under short-day conditions[J]. Plant and Cell Physiology, 2002, 43(10): 1096-1105. DOI: 10.1093/pcp/pcf156 |
| [9] | Komiya R, Ikegami A, Tamaki S, et al. Hd3a and RFT1 are essential for flowering in rice[J]. Development, 2008, 135(4): 767-774. DOI: 10.1242/dev.008631 |
| [10] | Hayama R, Izawa T, Shimamoto K. Isolation of rice genes possibly involved in the photoperiodic control of flowering by a fluorescent differential display method[J]. Plant and Cell Physiology, 2002, 43(5): 494-504. DOI: 10.1093/pcp/pcf059 |
| [11] | Park D H, Somers D E, Kim Y S, et al. Control of circadian rhythms and photoperiodic flowering by the Arabidopsis GIGANTEA gene[J]. Science, 1999, 285(5433): 1579-1582. DOI: 10.1126/science.285.5433.1579 |
| [12] | Hayama R, Yokoi S, Tamaki S, et al. Adaptation of photoperiodic control pathways produces short-day flowering in rice[J]. Nature, 2003, 422(6933): 719-722. DOI: 10.1038/nature01549 |
| [13] | Doi K, Izawa T, Fuse T, et al. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1[J]. Genes Dev, 2004, 18(8): 926-936. DOI: 10.1101/gad.1189604 |
| [14] | Itoh H, Nonoue Y, Yano M, et al. A pair of floral regulators sets critical day length for Hd3a florigen expression in rice[J]. Nature Genetics, 2010, 42(7): 635-638. DOI: 10.1038/ng.606 |
| [15] | Xue W Y, Xing Y Z, Weng X Y, et al. Natural variation in Ghd7 is an important regulator of flowering time and yield potential in rice[J]. Nature Genetics, 2008, 40(6): 761-767. DOI: 10.1038/ng.143 |
| [16] | Lee S, Kim J, Han J J, et al. Functional analyses of the flowering time gene OsMADS50, the putative SUPPRESSOR OF OVEREXPRESSION OF CO1/AGAMOUS-LIKE 20(SOC1/AGL20) ortholog in rice[J]. Plant Journal, 2004, 38(5): 754-764. DOI: 10.1111/tpj.2004.38.issue-5 |
| [17] | Kim S L, Lee S, Kim H J, et al. OsMADS51 is a short-day flowering promoter that functions upstream of Ehd1, OsMADS14, and Hd3a[J]. Plant Physiology, 2007, 145(4): 1484-1494. DOI: 10.1104/pp.107.103291 |
| [18] | Wei X J, Xu J F, Guo H N, et al. DTH8 suppresses flowering in rice, influencing plant height and yield potential simultaneously[J]. Plant Physiology, 2010, 153(4): 1747-1758. DOI: 10.1104/pp.110.156943 |
| [19] | Matsubara K, Yamanouchi U, Wang Z X, et al. Ehd2, a rice ortholog of the maize INDETERMINATE1 gene, promotes flowering by up-regulating Ehd1[J]. Plant Physiology, 2008, 148(3): 1425-1435. DOI: 10.1104/pp.108.125542 |
| [20] | Gao H, Zheng X M, Fei G, et al. Ehd4 encodes a novel and Oryza-genus-specific regulator of photoperiodic flowering in rice[J]. PLoS Genetics, 2013, 9(2): e1003281. DOI: 10.1371/journal.pgen.1003281 |
| [21] | Wu W X, Zheng X M, Lu G W, et al. Association of functional nucleotide polymorphisms at DTH2 with the northward expansion of rice cultivation in Asia[J]. Proc Natl Acad Sci USA, 2013, 110(8): 2775-2780. DOI: 10.1073/pnas.1213962110 |
| [22] | Takahashi Y, Shomura A, Sasaki T, et al. Hd6, a rice quantitative trait locus involved in photoperiod sensitivity, encodes the alpha subunit of protein kinase CK2[J]. Proc Natl Acad Sci USA, 2001, 98(14): 7922-7927. DOI: 10.1073/pnas.111136798 |
| [23] | Hori K, Ogiso-Tanaka E, Matsubara K, et al. Hd16, a gene for casein kinaseⅠis involved in the control of rice flowering time by modulating the day-length response[J]. Plant Journal, 2013, 76(1): 36-46. |
| [24] | Cartagena J A, Matsunaga S, Seki M, et al. The Arabidopsis SDG4 contributes to the regulation of pollen tube growth by methylation of histone H3 lysines 4 and 36 in mature pollen[J]. Developmental Biology, 2008, 315(2): 355-368. DOI: 10.1016/j.ydbio.2007.12.016 |
| [25] | Dong G F, Ma D P, Li J X. The histone methyltransferase SDG8 regulates shoot branching in Arabidopsis[J]. Biochemical and Biophysical Research Communications, 2008, 373(4): 659-664. DOI: 10.1016/j.bbrc.2008.06.096 |
| [26] | Berr A, Xu L, Gao J, et al. SET DOMAIN GROUP25 encodes a histone methyltransferase and is involved in FLOWERING LOCUS C activation and repression of flowering[J]. Plant Physiology, 2009, 151(3): 1476-1485. DOI: 10.1104/pp.109.143941 |
| [27] | Liu B, Wei G, Shi J L, et al. SET DOMAIN GROUP708, a histone H3 lysine 36-specific methyltransferase, controls flowering time in rice(Oryza sativa)[J]. New Phytologist, 2016, 210(2): 577-588. DOI: 10.1111/nph.13768 |
| [28] | Sui P F, Jin J, Ye S, et al. H3K36 methylation is critical for brassinosteroid-regulated plant growth and development in rice[J]. Plant Journal, 2012, 70(2): 340-347. DOI: 10.1111/tpj.2012.70.issue-2 |
| [29] | Sun C H, Fang J, Zhao T L, et al. The histone methyltransferase SDG724 mediates H3K36me2/3 deposition at MADS50 and RFT1 and promotes flowering in rice[J]. Plant Cell, 2012, 24(8): 3235-3247. DOI: 10.1105/tpc.112.101436 |
| [30] | Wan S Y, Wu J X, Zhang Z G, et al. Activation tagging, an efficient tool for functional analysis of the rice genome[J]. Plant Molecular Biology, 2009, 69(1/2): 69-80. |
| [31] | Siebert P D, Chenchik A, Kellogg D E, et al. An improved PCR method for walking in uncloned genomic DNA[J]. Nucleic Acids Research, 1995, 23(6): 1087-1088. DOI: 10.1093/nar/23.6.1087 |
| [32] | Sallaud C, Meynard D, van Boxtel J, et al. Highly efficient production and characterization of T-DNA plants for rice(Oryza sativa L.) functional genomics[J]. Theoretical and Applied Genetics, 2003, 106(8): 1396-1408. DOI: 10.1007/s00122-002-1184-x |
| [33] | Tsuji H, Taoka K, Shimamoto K. Regulation of flowering in rice:two florigen genes, a complex gene network, and natural variation[J]. Current Opinion in Plant Biology, 2011, 14(1): 45-52. DOI: 10.1016/j.pbi.2010.08.016 |
| [34] | Zhu S S, Wang J C, Cai M H, et al. The OsHAPL1-DTH8-Hd1 complex functions as the transcription regulator to repress heading date in rice[J]. Journal of Experimental Botany, 2017, 68(3): 553-568. |
| [35] | Nemoto Y, Nonoue Y, Yano M, et al. Hd1, a CONSTANS ortholog in rice, functions as an Ehd1 repressor through interaction with monocot-specific CCT-domain protein Ghd7[J]. Plant Journal, 2016, 86(3): 221-233. DOI: 10.1111/tpj.2016.86.issue-3 |
| [36] | Bastow R, Mylne J S, Lister C, et al. Vernalization requires epigenetic silencing of FLC by histone methylation[J]. Nature, 2004, 427(6970): 164-167. DOI: 10.1038/nature02269 |
| [37] |
宋博研, 朱卫国. 组蛋白甲基化修饰效应分子的研究进展[J].
遗传, 2011(4): 285-292.
Song B Y, Zhu W G. Advances in effector protein of histone methylation[J]. Hereditas, 2011(4): 285-292. (in Chinese with English abstract) |




