文章信息
- 潘子豪, 龚倩梅, 李科茫, 马慧, 吴宗福, 姚火春
- PAN Zihao, GONG Qianmei, LI Kemang, MA Hui, WU Zongfu, YAO Huochun
- 奶牛乳液中伊丽莎白菌按蚊亚种高度同源株的分离与鉴定
- Isolation and identification of a bacterium from milk of a dairy cow with mastitis sharing high homology to Elizabethkingia anophelis
- 南京农业大学学报, 2017, 40(6): 1088-1093
- Journal of Nanjing Agricultural University, 2017, 40(6): 1088-1093.
- http://dx.doi.org/10.7685/jnau.201709019
-
文章历史
- 收稿日期: 2017-09-11
伊丽莎白菌属于黄杆菌科伊丽莎白菌属(Elizabethkingia), 2005年单独划分为独立的属, 包括脑膜炎败血伊丽莎白菌(E.meningoseptica)、和平空间站伊丽莎白菌(E.miricola)、按蚊伊丽莎白菌(E.anophelis)和植物内生伊丽莎白菌(E.endophytica)[1]。该菌属为非发酵革兰阴性菌, 无鞭毛, 无芽孢和菌毛。伊丽莎白菌按蚊亚种(E.anophelis, EA)于2011年从冈比亚疟蚊肠道中分离并首次报道命名[2], 是一种新发现的机会致病菌。EA在环境中有快速形成生物被膜和广谱耐药性的特点, 已经成为重要的院内感染病原和新生儿脑膜炎重要致病菌。近5年来, 关于该菌感染人的案例报道逐渐增多[3-8]。2016年我国发生感染伊丽莎白菌并导致感染人员死亡的报道, Lau等[9]对中国香港地区伊丽莎白菌流行病学调查发现, 在21例感染伊丽莎白菌的临床病例中, 有17例是由EA感染引起的, 表现出比较典型的肺炎伴随菌血症并呈现高并发症和高死亡率的特征。本研究从患乳房炎的奶牛奶液中分离出伊丽莎白菌, 通过对该细菌的16S rRNA编码序列进行测序和生化反应分析, 发现该菌与伊丽莎白菌按蚊亚种人源分离株KT768343.1的相似度为95%, 该菌的发现具有潜在的公共卫生学意义。
1 材料与方法 1.1 材料 1.1.1 病料来源和菌株病料采集于江苏省内某牧场, 圈舍内荷斯坦奶牛发生乳房炎疫情, 其中2头奶牛发展为顽固性乳房炎症状, 治疗一段时间后病情未明显改变, 乳房呈明显的红肿, 奶液取样后发生分层现象。无菌条件下, 取病料, 分别涂布于绵羊血平板和麦康凯琼脂平板, 置37 ℃培养24 h观察菌落形态特征。挑取典型菌落进一步纯化, 取菌落经革兰染色后观察菌体形态及染色特征, 并分别接种THB液体培养基和含10%小牛血清的THB液体培养基, 37 ℃振荡培养12~16 h。使用VITEK 2革兰阴性菌生化鉴定卡(梅里埃诊断产品有限公司)对菌落进行生化鉴定。
1.1.2 试剂及仪器THB液体培养基、LB培养基购自美国BD公司; 无菌绵羊血购自江苏省疾控中心; 2×Taq反应混合物、PCR高保真Mix、DNA片段回收试剂盒购自大连宝生物(TaKaRa)工程公司; 胶回收试剂盒为Omega产品; 磁力搅拌器购自德国IKA公司; NGC层析系统、PCR仪购自美国BIO-RAD公司; Sephacry1 S-300凝胶层析柱购自美国GE公司; 21种抗生素购自南京鼎思生物技术有限公司。
1.2 试验方法 1.2.1 药物敏感性试验参照文献[3]的方法并稍作改进:将单菌落接种到4 mL THB液体培养基中, 于恒温摇床(180 r·min-1, 37 ℃)振荡培养; 将菌悬液培养至D600接近0.5或待新制的菌悬液调制为D600接近0.5, 经THB液体培养液1:1 000(体积比)稀释后, 再将稀释的菌液加到灭菌的96孔聚苯乙烯板中; 第1孔加180 μL, 第2~12孔每孔依次加100 μL, 加20 μL抗生素母液于第1孔, 混匀, 取100 μL于第2孔, 依次做倍比稀释至最后1孔。第1孔至第12孔药物质量浓度分别为256、128、64、32、16、8、4、2、1、0.5、0.25和0.125 μg·mL-1, 以不加抗生素为生长对照。密封后置37 ℃培养箱中, 培养12~24 h判断结果。
1.2.2 生长曲线的测定无菌操作, 取1~2个菌接种到4 mL THB液体培养基中, 在恒温摇床中(180 r·min-1, 37 ℃)振荡培养, 将菌液培养至D600接近0.6左右, 以1:100(菌液和THB液体培养基体积比)稀释后接至已灭菌的含50 mL THB液体培养基的三角瓶中, 并以未接菌的50 mL THB培养液作为空白对照, 于摇床中180 r·min-1、37 ℃培养。用分光光度计, 从0时开始, 每隔0.5 h测定培养液D600值, 当测得的D600为0.7~0.8时改为每隔1 h测1次。每次测定时, 以未接菌的培养液调零。以时间为横坐标, 以D600值为纵坐标, 制作曲线, 即为细菌在该培养条件下的生长曲线。
1.2.3 16S rRNA测序及同源性分析利用直接煮沸法提取细菌基因组。由南京金斯瑞生物科技有限公司合成细菌16S rRNA通用引物对:16S-27F:5′-AGAGTTTGATCCTGGCTCAG-3′和16S-1492R:5′-CGGCTACCTTGTTACGACTT-3′。采用40 μL扩增体系:上、下游引物各2 μL, 分离菌基因组DNA 2 μL, PCR Mix 15 μL, ddH2O 19 μL。反应条件:94 ℃ 5 min; 94 ℃ 1 min, 52 ℃ 30 s, 72 ℃ 1.5 min, 30个循环; 72 ℃ 10 min。预期PCR产物为1 500 bp左右, 送金斯瑞生物科技有限公司测序。将测得的基因序列在GenBank中进行核酸BLAST序列比对, 在Ezbiocloud数据库中进行鉴定分析, 综合结果分析其相似性。选取一定数量相似度较高种属标准菌株的16S rRNA序列, 界定某一进化关系较远的种属作为外根。使用ClustalX 1.8进行序列比对, 采用Mega 4.0构建进化树, 邻接法(Neighbor-joining)和自举(bootstrap)1 000次来验证进化树的可靠性。
2 结果与分析 2.1 细菌的分离鉴定从患顽固性乳房炎的荷斯坦奶牛奶液样品分离到的这1株细菌, 命名为JS20170427COW。该菌经24~36 h培养后, 在绵羊血平板长出直径1 mm左右的圆形、光滑菌落, 该菌落中央突起呈小丘样, 突起处呈明显淡黄色, 边缘透明, 四周平铺, 菌落周围有溶血现象。该菌在麦康凯琼脂平板上不生长。挑取绵羊血平板菌落革兰染色镜检, 可见革兰阴性短杆菌。液体培养基培养12~16 h, 革兰染色镜检, 可见革兰阴性细长杆菌, 且向内弯曲。分离菌株JS20170427COW对培养基的营养要求不高, 液体培养基中添加小牛血清可以促进该分离菌株的生长(图 1)。
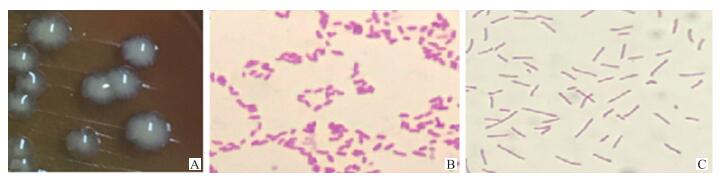
|
图 1 菌株JS20170427COW的培养特性 Figure 1 Culture characteristics of strain JS20170427COW A.培养24 h后的菌落形态; B.固体培养基培养菌镜检(×100);C.液体培养基培养菌镜检(×100)。 A. Colonies appearance after 24 hours; B.Solid culture medium microscopy(×100);C. Liquid culture microscopy(×100). |
生化鉴定结果(表 1)表明:分离菌株JS20170427COW在丙氨酸-苯丙氨酸-脯氨酸芳胺酶(APPA)、β-半乳糖苷酶(BGAL)、β-N-乙酰葡萄糖苷酶(BNAG)、谷氨酰芳胺酶(AGLTp)、脂酶(LIP)、酪氨酸芳胺酶(TyrA)、磷酸酶(PHOS)、氨基乙酰芳胺酶(GlyA)、谷氨酸-甘氨酸-精氨酸芳胺酶(GGAA)中呈阳性; 发酵α-葡萄糖(AGLU)产酸不产气, 发酵N-乙酰-β-半乳糖氨酶(NAGA)产酸且产气。在蔗糖(SAC)、D-纤维二糖(dCEL)、D-麦芽糖(dMAL)、塔格糖(dTAG)、D-甘露糖(dMNE)、葡萄糖发酵(OFF)、D-葡萄糖(dGLU)、D-甘露醇(dMAN)、古老糖(PLE)、D-海藻糖(dTRE)、D-山梨醇(dSOR)、L-阿拉伯醇(IARL)、侧金盏花醇(ADO)利用试验中都不能产酸, 表现为阴性, 其余的酶活性反应也均为阴性。
| 试验Test | 结果Result |
| APPA | + |
| H2S | - |
| BGLU | - |
| ProA | - |
| SAC | - |
| ILATk | - |
| GlyA | + |
| O129R | - |
| dCEL | - |
| BXYL | - |
| MNT | - |
| CMT | - |
| ADO | - |
| BNAG | + |
| dMAL | - |
| LIP | + |
| dTAG | - |
| AGLU | + |
| ODC | - |
| GGAA | + |
| BGAL | + |
| BAlap | - |
| 5KG | - |
| BGUR | - |
| PyrA | - |
| AGLTp | + |
| dMAN | - |
| PLE | - |
| dTRE | - |
| SUCT | - |
| LDC | - |
| IMLTa | - |
| GGT | - |
| URE | - |
| AGAL | - |
| ILATa | - |
| IARL | - |
| dGLU | - |
| dMNE | - |
| TyrA | - |
| CIT | - |
| NAGA | ⊕ |
| IHISa | - |
| ELLM | - |
| OFF | - |
| dSOR | - |
| PHOS | + |
| 注:1) “⊕”为产酸产气; “+”为阳性反应; “-”为阴性反应。“⊕”stands for production of gas and acid; “+”stands for positive, “-”stands for negative. 2) APPA:丙氨酸-苯丙氨酸-脯氨酸芳胺酶Ala-Phe-Pro-arylamidase; H2S:H2S production; BGLU:β-葡萄糖苷酶β-glucosidase; ProA:L-脯氨酸芳胺酶L-proline arylamidase; SAC:蔗糖Saccharose; ILATk:乳酸盐产碱L-lactate alkalinisation; GlyA:氨基乙酸芳胺酶Glycine arylamidase; O129R:O/129耐受O/129 resistance; dCEL:D-纤维二糖D-cellobiose; BXYL:β-木糖苷酶β-xylosidase; MNT:丙二酸盐Malonate; CMT:葡萄糖硒酸酶Courmarate; ADO:侧金盏花醇Adonitol; BNAG:β-N-乙酰葡萄糖苷酶β-N-acetyl-glucosaminidase; dMAL:D-麦芽糖D-maltose; LIP:脂酶Lipase; dTAG:塔格糖D-tagatose; AGLU:α-葡萄糖α-glucosidase; ODC:鸟氨酸脱羧酶Ornithine decarboxylase; GGAA:谷氨酸-甘氨酸-精氨酸芳胺酶Glu-Gly-Arg-arylamidase; BGAL:β-半乳糖苷酶β-galactosidase; BAlap:β-丙氨酸芳胺酶β-alanine arylamidase pNA; 5KG:5-酮-葡萄糖苷5-keto-D-gluconate; BGUR:β-葡萄糖苷酸酶β-glucoronidase; PyrA:吡咯烷基芳胺酶L-pyrrolydonyl-arylamidase; AGLTp:谷氨酰芳胺酶Glutamyl arylamidase pNA; dMAN:D-甘露醇D-mannitol; PLE:古老糖Palatinose; dTRE:D-海藻糖D-trehalose; SUCT:琥珀酸盐产碱Succinate alkalinisation; LDC:赖氨酸脱羧酶Lysine decarboxylase; IMLTa:L-苹果酸盐同化L-malate assimilation; GGT:γ-谷氨酰转移酶γ-glutamyl-transferase; URE:尿素酶Urease; AGAL:α-半乳糖苷酶α-galactosidase; ILATa:L-乳酸盐同化L-lactate assimilation; IARL:L-阿拉伯醇L-arabitol; dGLU:D-葡萄糖D-glucose; dMNE:D-甘露糖D-mannose; TyrA:酪氨酸芳胺酶Tyrosine arylamidase; CIT:柠檬酸盐Citrate; NAGA:N-乙酰-β-半乳糖氨酶β-N-acetyl-galactosa minidase; IHISa:组氨酸同化L-histidine assimilation; ELLM:埃尔曼试剂Ellman; OFF:葡萄糖发酵Fermentation/glucose; dSOR:D-山梨醇D-sorbitol; PHOS:磷酸酶Phosphatase. | |
由分离菌株生长曲线(图 2)可以看出, 接种后0~4 h该菌处于生长迟缓期, 4 h后进入对数生长期, 13.5 h开始进入平稳期, 48 h可分离的活菌数逐步减少, 细菌进入死亡期。

|
图 2 JS20170427COW的生长曲线 Figure 2 Growth curve of JS20170427COW |
以分离菌株JS20170427COW的DNA为模板, 用细菌通用引物进行16S rRNA PCR扩增, 得到约1.5 kb的DNA目的片段, 将测序分析结果在GenBank中进行BLAST比对, 发现该菌的16S rRNA与伊丽莎白按蚊亚种KT768343.1菌株的同源性最高(95%), 且处于同一进化分支(图 3)。结合细菌生理生化分析的结果, 将该菌暂命名为牛源伊丽莎白菌按蚊亚种高度同源株。
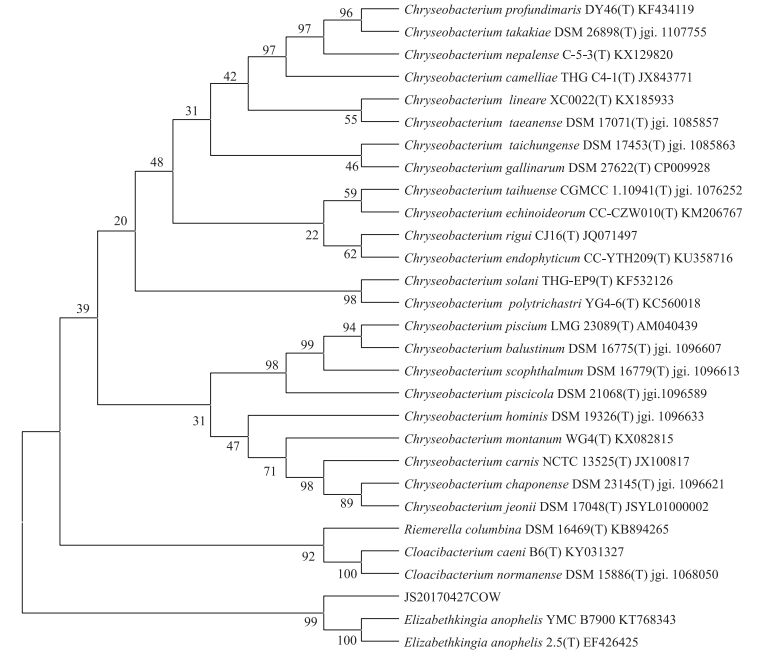
|
图 3 基于16S rRNA的Neighbour-joining进化树 Figure 3 Neighbor-joining phylogenetic tree based on 16S rRNA gene sequences Chryseobacterium金黄杆菌属; C. balustinum大比目鱼金黄杆菌; C. scophthalmum比目鱼金黄杆菌; Elizabethkingia伊丽莎白菌属; Elizabethkingia anophelis按蚊伊丽莎白; Riemerella立默氏菌属。 |
药物敏感性试验结果显示:该分离菌株JS20170427COW对利福平、红霉素、庆大霉素、氯霉素、头孢拉定、阿米卡星敏感, 对四环素类(四环素、金霉素、强力霉素)药物也均表现为敏感, 但其对杆菌肽、溶菌素、硫酸黏菌素、乳酸菌素以及多肽类药物均表现为耐药(表 2)。
| 抗生素 Antibiotic | 最小抑菌浓度/(μg·mL-1) MIC | 敏感性 Sensitivity |
| 氨苄西林Ampicillin | 128 | R |
| 青霉素G Penicillin G | 32 | I |
| 庆大霉素Gentamicin | 4 | S |
| 壮观霉素Spectinomycin | 8 | I |
| 氯霉素Chloramphenicol | 2 | S |
| 环丙沙星Ciprofloxacin | >256 | R |
| 利福平Rifampicin | <0.125 | S |
| 红霉素Erythromycin | <0.125 | S |
| 金霉素Chlortetracycline | 1 | S |
| 链霉素Streptomycin | 8 | I |
| 头孢拉定Cefradine | 4 | S |
| 阿米卡星Amikacin | 16 | S |
| 四环素Tetracycline | 4 | S |
| 杆菌肽Bacitracin | >256 | R |
| 溶菌素Lysozyme | >256 | R |
| 多黏菌素B Polymyxin B | >256 | R |
| 强力霉素Doxycycline | 4 | S |
| 卡那霉素Kanamycin | 64 | R |
| 硫酸黏菌素Colistin sulfate | >256 | R |
| 乳酸菌素Lactic acid | >256 | R |
| 注:1) MIC:Minimum inhibitory concentration; 2) S、I、R分别为敏感、中度敏感、耐药。S, I, R stand for sensitive, moderately sensitive, resistant. | ||
伊丽莎白菌属于2005年鉴定并分离, 经16S rRNA的进化树分析, 被分类为伊丽莎白菌脑膜炎亚种和伊丽莎白菌和平空间站亚种, 2011年从按蚊的肠道中分离鉴定了伊丽莎白菌按蚊亚种, 2015年从甜玉米中分离并鉴定发现了该菌属的新亚种植物内生伊丽莎白菌[10]。该菌属的所有成员均可以感染人。伊丽莎白菌是一种革兰氏阴性短杆菌, 需氧生长, 血平板上生长良好, 麦康凯平板不生长。模式生物基因序列的鉴定方法无法确定该菌属的基因型特征, 因此该菌属不同种的鉴定技术有待于进一步的完善和发展。伊丽莎白脑膜炎亚种最早从病人的血液中分离并鉴定, 被认为是一种重要的院内感染病原, 并未引起高的关注度, 而按蚊亚种(EA)因一直未被鉴定而缺乏相关的报道。关于该细菌的生化特性和分子生物学的信息缺失, 导致该病原的分离和鉴定手段空白。EA自2011年鉴定发现后不断出现其感染人的报道[4-9, 11-16], 2013年中非地区首次报道出现8岁的女性患者感染EA并诱发脑膜炎症状, 病人出现3 d的持续高热后, 从血液中分离到了EA[3]。研究人员通过该菌种16S rRNA的参考序列确认了2006年爆发的伊丽莎白菌脑膜炎亚种临床感染的病例病原为EA。新加坡、中国台湾和香港地区自2012年开始出现感染EA并导致人死亡的病例[4-5], 起初用VitekⅡ或者MALDI的全自动生化系统鉴定为伊丽莎白菌脑膜炎亚种, 后经16S rRNA鉴定为伊丽莎白菌按蚊亚种。2015年美国出现了多个感染伊丽莎白菌的病例报道。该细菌主要感染婴儿、儿童以及医院内治疗中的患者, 导致持续性发热和脑膜炎症状, 引发细菌血症并导致患者死亡, 死率24%~30%[11]。多个国家的疫病控制中心已经开展了该病原的流行病学调查并建立了数据库。2017年我国福建、山东等多名学者联合发表论文报道了两例感染EA病人出现下呼吸道感染和多器官衰竭临床症状[13]。
现有的Vitek ⅡGN、AP20E以及API20NE等全自动生化鉴定系统因缺乏该菌属的数据信息, 故不能鉴定出伊丽莎白菌以及相关的亚种, 而且伊丽莎白菌的临床分离和诊断较为困难。质谱分析技术虽能够确定伊丽莎白菌, 但需要研究者提供该菌的基因数据信息。2016年, 巴斯德研究所建立了伊丽莎白菌的数据库, 为该菌在全球的分析诊断建立有效的平台。16S rRNA的序列测定和分析如今可以实现对该细菌的基因鉴定以及种属鉴定, NCBI数据库中已经公布了部分EA的基因组序列信息, 可以用于该菌株的基因进化分析。分子指纹图谱技术是现有的能够对该菌属进行分析的重要技术, 通过多位点序列分型(MLST)和脉冲电场凝胶电泳(PFGE)等技术可以实现对不同分离株的鉴定。
EA的流行病学信息现在仍然处于空白期, 以往爆发的感染人的案例中由于当时未能确定该细菌的种型, 缺乏相关的流行病学调查的背景材料。统计已有的报道, 2004—2013年, 伊丽莎白菌感染人的案例共有21起, 16S rRNA测序结果表明其中17例为EA。Han等[17]从韩国2009—2015年医院病人的血液、呼吸道渗出液和尿液中分离出了86株伊丽莎白细菌, 51株为EA株, 比例为59%。由于该病原最早从按蚊的肠道中分离出来, 有观点认为蚊虫的叮咬可能导致EA的感染人, 但是缺乏相关的流行病学基础。2015年报道EA感染了一位33岁的女性患者并通过母源途径感染了婴儿引发了典型的细菌性脑膜炎症状。目前尚没有动物携带或者感染EA发病的报道, 本试验首次从牧场患乳房炎奶牛的乳汁中分离并鉴定出EA高度同源株, 说明该病原可能为导致奶牛乳房炎的病原, 并存在从食品中传染人的风险。EA高度同源株是否能够感染动物并引起发病还缺乏相关的流行病学调查结论。细菌的分离培养发现, JS20170427COW株在绵羊血平板上形成明显的溶血环, 液体培养基中添加血清后能够促进细菌的生长, 这些特征符合致病性菌株的特点, 其致病力还有待于进一步研究。
EA耐药性的研究是该菌株的研究热点, Han等[17]对51株EA临床株耐药谱分析研究发现, 49%的菌株对莫西沙星等新药物产生耐药性, 但是Lau等[9]和Fank等[3]发现他们的分离株对莫西沙新类药物敏感, 因此不同分离株的耐药谱差异较大, 说明EA可能存在耐药基因水平转移的特点。
目前已发表的研究结论中还未见到从乳房炎奶牛的奶液中分离出伊丽莎白菌属的报道, 因此, 本研究是首次报道从牛奶中分离到伊丽莎白菌按蚊亚种高度同源株, 将为伊丽莎白菌的流行病数据库研究提供新的资料。
| [1] | Kim K K, Kim M K, Lim J H, et al. Transfer of Chryseobacterium meningosepticum and Chryseobacterium miricola to Elizabethkingia gen. nov. as Elizabethkingia meningoseptica comb. nov. and Elizabethkingia miricola comb. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55: 1287–1293. DOI: 10.1099/ijs.0.63541-0 |
| [2] | Kampfer P, Matthews H, Glaeser S P, et al. Elizabethkingia anophelis sp. nov. isolated from the midgut of the mosquito Anopheles gambiae[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61: 2670–2675. DOI: 10.1099/ijs.0.026393-0 |
| [3] | Frank T, Gody J C, Nguyen L B, et al. First case of Elizabethkingia anophelis meningitis in the Central African Republic[J]. Lancet, 2013, 381: 1876. DOI: 10.1016/S0140-6736(13)60318-9 |
| [4] | Lau S K, Wu A K, Teng J L, et al. Evidence for Elizabethkingia anophelis transmission from mother to infant, Hong Kong[J]. Emerging Infectious Diseases, 2015, 21(2): 232–241. DOI: 10.3201/eid2102.140623 |
| [5] | Lin J N, Yang C H, Lai C H, et al. Draft genome sequence of Elizabethkingia anophelis strain EM361-97 isolated from the blood of a cancer patient[J]. Genome Announcements, 2016, 4(5): e01215–16. |
| [6] | Navon L, Clegg W J, Morgan J, et al. Notes from the field:investigation of Elizabethkingia anophelis cluster-illinois, 2014-2016[J]. Morbidity and Mortality Weekly Report, 2016, 65(48): 1380–1381. DOI: 10.15585/mmwr.mm6548a6 |
| [7] | Nicholson A C, Whitney A M, Emery B D, et al. Complete genome sequences of four strains from the 2015-2016Elizabethkingia anophelis outbreak[J]. Genome Announcements, 2016, 4(3): e00563–16. |
| [8] | Coyle A L. Elizabethkingia anophelis:exploring the outbreak of disease in the Midwest[J]. Nursing, 2017, 47(3): 61–63. DOI: 10.1097/01.NURSE.0000512887.67622.84 |
| [9] | Lau S K, Chow W N, Foo C H, et al. Elizabethkingia anophelis bacteremia is associated with clinically significant infections and high mortality[J]. Scientific Reports, 2016, 6: 26045. DOI: 10.1038/srep26045 |
| [10] | Doijad S, Ghosh H, Glaeser S, et al. Taxonomic reassessment of the genus Elizabethkingia using whole-genome sequencing:Elizabethkingia endophytica Kampfer et al. 2015 is a later subjective synonym of Elizabethkingia anophelis Kampfer et al. 2011[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(11): 4555–4559. DOI: 10.1099/ijsem.0.001390 |
| [11] | Perrin A, Larsonneur E, Nicholson A C, et al. Evolutionary dynamics and genomic features of the Elizabethkingia anophelis 2015 to 2016 Wisconsin outbreak strain[J]. Nature Communications, 2017, 8: 15483. DOI: 10.1038/ncomms15483 |
| [12] | Janda J M, Lopez D L. Mini review:new pathogen profiles:Elizabethkingia anophelis[J]. Diagnostic Microbiology and Infectious Disease, 2017, 88(2): 201–205. DOI: 10.1016/j.diagmicrobio.2017.03.007 |
| [13] | Hu S, Jiang T, Zhang X, et al. Elizabethkingia anophelis isolated from patients with multiple organ dysfunction syndrome and lower respiratory tract Infection:report of two cases and literature review[J]. Frontiers in Microbiology, 2017, 8: 382. |
| [14] | Garay J A R, Hughes G L, Koundal V, et al. Genome sequence of Elizabethkingia anophelis strain EaAs1, isolated from the Asian Malaria Mosquito Anopheles stephensi[J]. Genome Announcements, 2016, 4(2): e00084–16. |
| [15] | Breurec S, Criscuolo A, Diancourt L, et al. Genomic epidemiology and global diversity of the emerging bacterial pathogen Elizabethkingia anophelis[J]. Scientific Reports, 2016, 6: 30379. DOI: 10.1038/srep30379 |
| [16] | Teo J, Tan S Y, Liu Y, et al. Comparative genomic analysis of malaria mosquito vector-associated novel pathogen Elizabethkingia anophelis[J]. Genome Biology and Evolution, 2014, 6(5): 1158–1165. DOI: 10.1093/gbe/evu094 |
| [17] | Han M S, Kim H, Lee Y, et al. Relative prevalence and antimicrobial susceptibility of clinical isolates of Elizabethkingia species based on 16S rRNA gene sequencing[J]. Journal of Clinical Microbiology, 2017, 55(1): 274–280. DOI: 10.1128/JCM.01637-16 |




