文章信息
- 徐志辉, 黄蓉, 张钰婷, 冯元韬, 张馨玉, 仇美华
- XU Zhihui, HUANG Rong, ZHANG Yuting, FENG Yuantao, ZHANG Xinyu, QIU Meihua
- 黄瓜根际芽孢杆菌亲缘辨识特性研究
- Kin discrimination between Bacillus spp. strains from cucumber rhizosphere
- 南京农业大学学报, 2017, 40(6): 1023-1030
- Journal of Nanjing Agricultural University, 2017, 40(6): 1023-1030.
- http://dx.doi.org/10.7685/jnau.201704025
-
文章历史
- 收稿日期: 2017-04-17
2. 江苏省耕地质量与农业环境保护站, 江苏 南京 210036
2. Jiangsu Province Station of Farmland Quality and Agricultural Environment Prtection, Nanjing 210036, China
过量施用化肥、农药等化学品破坏土壤微生物区系和土壤微生物活性, 降低农田土壤养分生物转化能力, 增加作物产量对化肥供应养分的依赖性。近年来, 植物根际促生菌(plant growth-promoting rhizobacteria, PGPR)因其具有显著的促进植物生长和防控土传病害的能力以及环境友好、安全无毒的特点, 开始在农业生产上推广应用[1-3]。目前, 微生物肥料只占整个肥料产值的7%, 规模较小。限制微生物肥料产业发展的主要原因是田间应用效果的不稳定性和匮乏的土壤微生物的生态学理论研究。
自然生态系统中近亲生命体中存在合作的关系, 但这种稳定的合作关系会由于寄生或“搭便车”的伪装性种群的入侵而退化, 因此近亲种群通过分泌特有的信号物质进行交流, 而其他生物无法识别和使用, 从而保证合作系统的稳定。1964年, 英国生物学家Hamilton提出亲缘选择理论, 认为亲族之间的合作倾向与其亲近程度成正比, 即亲缘关系越近, 彼此之间的合作程度就越高[4]。合作行为有利于同源物种的基因在自然选择中的保存和进化[5]。奇异变形杆菌(Proteus mirabilis)是一种引起泌尿系统感染的革兰氏阴性病原菌, 其亲缘辨识机制已有很好的研究。奇异变形杆菌亲缘关系相近的菌株在平板上共培养时能相互融合, 而亲缘关系较远的菌株之间往往形成可见边界[6]。黏细菌(Myxobacteria)的亲缘辨识现象也有类似结果, 而有些多位点基因序列完全一致的菌株也会出现不相容现象[7], 研究者认为这种现象是独立的基因位点修饰进化的结果[8]。另外, 枯草芽孢杆菌在拟南芥根部定殖成膜过程中, 亲缘菌株在根部共存且形成混合菌株生物膜; 非亲缘菌株之间则不相容, 产生的生物膜中只有单一菌株[9]。
解淀粉芽孢杆菌SQR9是本实验室从黄瓜种植发病区的健康植株根际分离的一株根际促生菌, 在盆栽和大田试验中表现出显著的促生和生防效果[10-12]。本研究在黄瓜苗期先将菌株SQR9引入根际, 使其在根表定殖, 然后在自然土壤中培育至初花期后收集并筛选根表芽孢杆菌资源, 利用群集运动试验和16S rRNA序列特征分析研究菌群的家族分类特征, 初步探究菌株SQR9在根际对芽孢杆菌菌群活动的影响规律。
1 材料与方法 1.1 材料 1.1.1 供试菌株解淀粉芽孢杆菌(Bacillus amyloliquefaciens)SQR9及其GFP标记菌SQR9-GFP。
1.1.2 试剂卡纳霉素购自北京鼎国生物技术有限责任公司; Salkowski显色液:称取4.5 g氯化铁溶于适量水, 缓慢倒入576 mL浓硫酸, 并用ddH2O定容至1 L; 植酸钠溶液:将植酸钠、氯化钙分别溶于0.1 mol·L-1的Tris-HCl(pH7.0)中, 终浓度均为2 mmol·L-1, 测定当天混匀; 钼氨酸储备液:称取100 g (NH4)6MO7O24·4H2O溶于盛有500 mL水的烧瓶中, 加入10 mL 25%氨水, 定容至1 L; NH4VO3储备液:2.35 g分析纯NH4VO3加入盛有400 mL水的烧瓶中, 边搅动边缓慢加入20 mL稀HNO3, 定容到1 L; 终止液:在100 mL容量瓶中加入25 mL NH4VO3储备液, 再加入25 mL钼酸铵储备液, 然后加入16.25 mL浓HNO3, 最后定容至100 mL, 试验前新鲜配制; 100 mmol·L-1磷酸储备液:将适量磷酸二氢钾于105 ℃干燥2 h, 干燥器内冷却后, 准确称取1.369 g溶于蒸馏水并稀释定容至100 mL。
1.1.3 引物细菌16S通用引物, 27F:AGAGTTTGATCCTGGCTCAG; 1492R:GGTTACCTTGTTACGACTT, 由上海英骏生物技术有限公司合成。
1.1.4 供试培养基LB培养基:蛋白胨10 g, 酵母粉5 g, NaCl 3 g, ddH2O 1 L。swarming培养基:琼脂7 g·L-1, 15 mmol·L-1硫酸铵, 8 mmol·L-1七水硫酸镁, 27 mmol·L-1氯化钾, 7 mmol·L-1柠檬酸钠, 50 mmol·L-1 Tris-HCl(pH7.5), 1 μmol·L-1硫酸铁, 10 μmol·L-1硫酸锰, 0.6 mmol·L-1磷酸二氢钾, 0.86 mmol·L-1赖氨酸, 0.78 mmol·L-1色氨酸, 葡萄糖2 g·L-1。IAA诱导培养基:蛋白胨10 g, 酵母粉5 g, 氯化钠3 g, 蒸馏水1 L, 灭菌后用细菌过滤器加入3 mmol·L-1 L-色氨酸。MSgg培养基:磷酸钾缓冲液5 mmol·L-1(pH7.0), 3-吗啉丙磺酸(MOPS)溶液100 mmol·L-1(pH7.0), 氯化镁2 mmol·L-1, 氯化钙700 mmol·L-1, 氯化锰50 mmol·L-1, 氯化铁50 mmol·L-1, 氯化锌1 mmol·L-1, 硫胺2 mmol·L-1, 0.5%甘油, 0.5%谷氨酸, 色氨酸50 mg·mL-1, 苯丙氨酸50 mg·mL-1。ASW液体培养基:六水氯化镁5 g, 七水硫酸镁5 g, 氯化钾0.5 g, 氯化钠20 g, 胰蛋白胨3 g, 大豆蛋白胨1.5 g, 葡萄糖5 g, 可溶性淀粉5 g, 蒸馏水定容至1 L(pH6.8)。
1.2 试验方法 1.2.1 根际促生菌解淀粉芽孢杆菌SQR9处理后黄瓜根际芽孢杆菌的分离无菌组培黄瓜苗在1/4MS培养基中培养至长出第3片真叶, 接种SQR9-GFP菌悬液(终浓度为5×106 CFU·mL-1), 培养3 d, 移栽至自然土壤(江苏省南京市蔬菜研究所黄瓜大棚土)中, 温室培养至初花期后, 取黄瓜根, 将根部土轻轻抖落后放到装有适量无菌水的三角瓶中, 于超声仪中振荡, 将根表微生物尽可能洗脱下来, 12 000 g离心10 min, 收集菌体, 90 ℃热水中煮5 min后稀释涂布。
1.2.2 Swarming平板试验采用新配制的swarming培养基, 将LB平板上保存的解淀粉芽孢杆菌接种到3 mL swarming培养液中, 37 ℃振荡培养过夜。将10-4菌液稀释, 接种2 μL于swarming固体培养基上, 每个培养基上接种2种菌。待菌液干后, 于37 ℃培养箱正置培养2 d。观察现象并拍照。
1.2.3 细菌DNA提取、16S rRNA扩增和系统发育树的构建细菌基因组DNA采用Axygen公司生产的提取试剂盒, 具体操作参照使用手册。以提取的SQR9全基因组DNA作模板, 采用FAST Taq mix进行PCR扩增, 扩增仪为ABI(Applied BIO systems)。反应体系为25 μL, 包括:FAST Taq(2×)mix 10 μL, Forward Primer 27F(10 μmol·L-1)1 μL, Reverse Primer 1492R(10 μmol·L-1)1 μL, DNA 0.5 μL, ddH2O 10 μL。反应程序:94 ℃ 90 s; 94 ℃ 30 s, 60 ℃ 30 s, 30个循环; 72 ℃ 10 min。采用DNA回收试剂盒(Axygen)回收PCR产物后送上海英骏生物技术有限公司进行DNA测序。将菌株的16S rRNA测序结果提交至GenBank并进行多序列完全比对, 采用邻接法分析各菌株间关系, 运行1 000次自举法(bootstrap)构建系统发育。
1.2.4 生物膜测定生物膜的测定方法参照Hamon等[13]的方法稍作修改。将过夜活化的细菌接种于新鲜LB培养基中, 37 ℃、170 r·min-1培养至D600约为1.0。4 ℃离心收集菌体, 用无菌水洗涤并重悬于等体积的MSgg培养基中。生物膜定性试验采用24孔细胞培养板, 培养孔中加入2 mL MSgg培养基, 然后分别接入细菌培养物10 μL, 设置3个重复, 于37 ℃静置培养24 h后观察和监测成膜情况。
1.2.5 吲哚乙酸(IAA)测定活化待测菌株, 1 mL接种量接种于含3 mmol·L-1 L-色氨酸的100 mL LB液体培养基中, 培养条件为避光, 28 ℃、170 r·min-1振荡24 h。配置IAA溶液至质量浓度依次为0、10、20、25、40和50 mg·L-1作为标准品。另外将菌液以4 ℃、4 000 r·min-1离心15 min, 取上清液0.5 mL。将标准品以及上清液与等体积的salkowski显色液混合。室温下避光放置30 min, 测定其D530值。通过标准曲线计算相应的IAA质量浓度[14]。
1.2.6 植酸酶活性的测定接种SQR9于3 mL液体LB试管, 37 ℃、170 r·min-1振荡培养10 h, 菌液作为种子液。取1 mL种子液接种于100 mL液体ASW培养基中, 37 ℃、170 r·min-1培养发酵3 d。取200 μL发酵液4 ℃离心弃上清液待测植酸酶活性。将储备液依次稀释至0、0.2、0.4、0.8、1.2和1.6 μmol·mL-1, 使用当天配制。取100 μL各浓度磷的标准溶液和发酵液加入900 μL配制好的2 mmol·L-1植酸钠溶液中。以发酵液先加入终止液再加入植酸钠溶液作为对照。50 ℃水浴锅中静置反应30 min, 加入750 μL终止液终止酶反应, 混合液中加入1.5 mL显色液, 室温静置15 min, 采用酶标仪检测700 nm波长下的吸光值。酶活性单位(U)定义为每分钟释放1 μmol磷酸根的酶量。
1.3 数据统计与分析试验数据采用Microsoft Excel 2010统计软件进行数据统计和绘图。数据运用SPSS BASE ver.11.5软件进行方差分析, 显著性水平设定0.05水平。
2 结果与分析 2.1 初花期黄瓜根际芽孢杆菌的分离与纯化通过稀释涂布, 从初花期黄瓜根际分离到118株芽孢杆菌, 复筛首先去除带有绿色荧光标记的菌株, 然后根据菌株16S rRNA序列和菌落特征将菌株分为15组, 同时去除菌落相似且16S rRNA序列100%相同的菌株, 最后组内菌株用Biolog Gene Ⅲ进行碳源利用比较, 去除碳源利用完全相同的菌株, 并且将同一组里不同碳源利用的菌株进行拆分, 最终得到21株芽孢杆菌, 包含枯草芽孢杆菌、解淀粉芽孢杆菌、地衣芽孢杆菌、甲基营养芽孢杆菌和贝莱斯芽孢杆菌等芽孢杆菌属的种, 但主要以解淀粉芽孢杆菌为主。图 1展示了分离菌株的菌落形态, 其菌落形态差异较大。
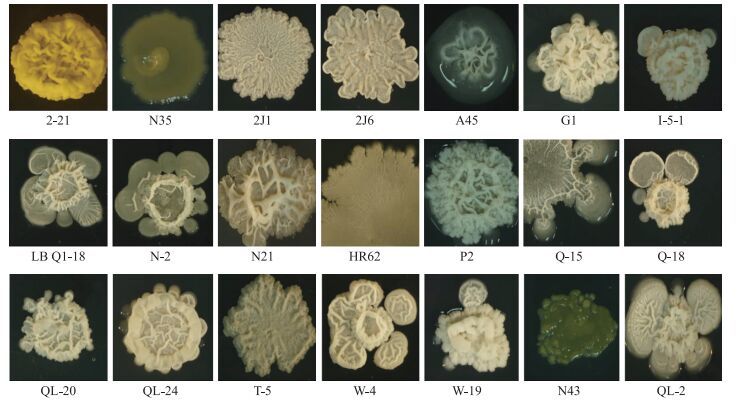
|
图 1 21株黄瓜根际芽孢杆菌的菌落形态 Figure 1 Colony morphology of 21 Bacillus strains isolated from cucumber rhizosphere |
Swarming对峙试验依据不同的试验现象分为:融合、半融合和边界3种表型。融合的情况下, 菌株与菌株之间不形成聚集的条带, 能够扩张到对方的领域中。但如果1处培养基被其中1个“菌株”占领了, 另外1个菌株也不会“强行争夺”, 二者遵循“先到先得”的原则(图 2-A)。半融合的平板现象也较多, 它们的共同点是在分界线上仍然能观察到细菌, 但存在一种“阻力”让它们不能融合, 更多的留在属于自己的领域(图 2-B)。边界指2株菌之间形成明显的分界线, 且在分界线上观察不到任何细菌。哪株菌生长快且游动能力强, 就能占领更多的区域。观察所有产生边界的平板, 发现两者之间都会留有0.5~2.0 mm的距离(图 2-C)。
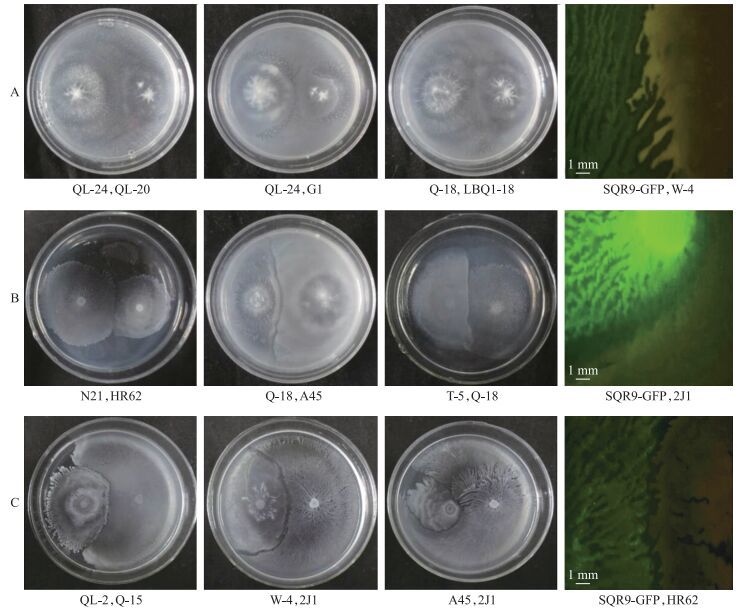
|
图 2 芽孢杆菌的3种swarming对峙表型 Figure 2 Three phenotypes of approaching Bacillus spp. swarms A.融合Merge; B.半融合Intermediate; C.边界Boundary. |
根据16S rRNA基因比对信息和群集运动表型, 将22个菌株分成A~G 7组(图 3-a), 同一群落之间出现多swarming类型说明亲缘辨识基因独立修饰进化的结果。22株菌的识别模式如图 3-b所示, 相互连接的节点代表能融合的菌株, 没有连接的是半融合与边界的情况。
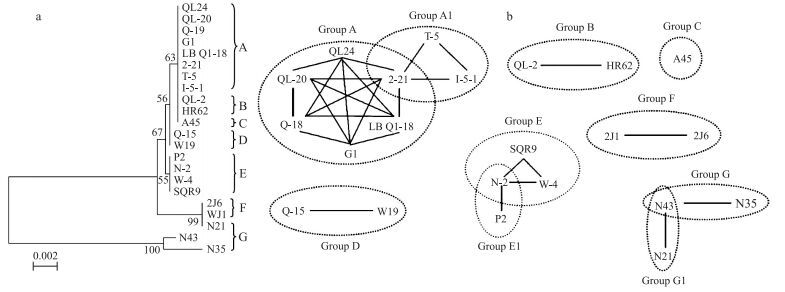
|
图 3 芽孢杆菌菌株识别的系统分类 Figure 3 Recognition groups of a sympatric Bacillus spp. a.土壤分离芽孢杆菌16S基因的系统发育重建Phylogenetic reconstruction of 16S gene from the soil isolat Bacillus; b.土壤分离芽孢杆菌的识别网络模型Recognition network model of the soil isolat Bacillus. |
将分离的22株菌株两两进行swarming对峙, 共计231组, 统计swarming表型并与16S rRNA相似度进行相关性分析。结果(图 4)表明, 从整体上看16S rRNA相似度越高, 菌株间swarming表型呈现半融合和融合的概率越大, 但也有例外。例如在融合的27对菌株中, 有25对的16S rRNA 100%相同, 而其余2对分别为0.970和0.995(表 1), 而产生边界的142对中有15对菌株保守基因相似度为100%, 说明16S rRNA并不能完全区分芽孢杆菌基因型的差异, 是由其他基因共同决定。
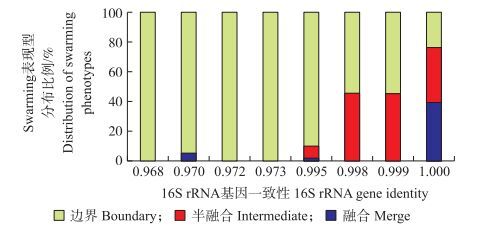
|
图 4 芽孢杆菌swarming表现型与基因型的关系分布 Figure 4 Swarming interaction phenotype in relation to phylogenetic relatedness of Bacillus spp. |
| 基因一致性 Gene identity |
类型Phenotypes | 总计 Sum |
|||||||
| 0.968 | 0.970 | 0.972 | 0.973 | 0.995 | 0.998 | 0.999 | 1.000 | ||
| 融合Merge | 1 | 1 | 25 | 27 | |||||
| 半融合Intermediate | 4 | 20 | 14 | 24 | 62 | ||||
| 边界Boundary | 3 | 19 | 11 | 6 | 47 | 24 | 17 | 15 | 142 |
| 总计Sum | 3 | 20 | 11 | 6 | 52 | 44 | 31 | 64 | 231 |
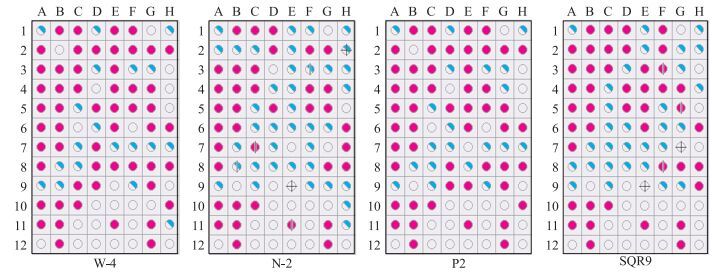
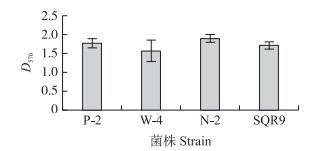
解淀粉芽孢杆菌SQR9根际成膜能力强, 施入根际后能快速定殖占领生态位。我们猜测土壤中与SQR9亲缘关系较近的菌株也有较强的生物膜形成能力。Biolog碳源利用试验表明, 菌株P2、W-4和N-2不是丢失GFP质粒的SQR9。BiologGENⅢ的碳源图谱试验(表 2, 图 5)表明, 这4株芽孢杆菌的碳源利用能力相差较大。细胞培养试验结果(图 6)表明:在实验室条件下菌株P2、W-4和N-2在24 h时生物膜形成能力接近菌株SQR9。
| 微孔板 序号 No.of microplate |
A | B | C | D | E | F | G | H |
| 1 | 阴性对照 Negative control |
蜜三糖, 棉子糖 D-raddinose |
α-D-葡糖 α-D-glucose |
D-山梨醇 D-sorbitol |
明胶 Gelatin |
果胶 Pectin |
p-羟基-苯乙酸 p-hydroxy-phenylacetic acid |
吐温40 Tween 40 |
| 2 | 糊精 Dextrin |
α-D-乳糖 α-D-lactose |
D-甘露糖 D-mannose |
D-甘露醇 D-mannitol |
氨基乙酰-L-脯氨酸 Glycyl-L-proline |
D-半乳糖醛酸 D-galacturonic acid |
丙酮酸甲酯 Methyl pyruate |
γ-氨基-丁酸 γ-amino-butryric acid |
| 3 | D-麦芽糖 D-maltose |
蜜二糖 D-melibiose |
D-果糖 D-fructose |
D-阿拉伯醇 D-arabitol |
L-丙氨酸 L-alanine |
L-半乳糖醛酸内酯 L-galactonic acid lactone |
D-乳酸甲酯 D-lactic acid methyl ester |
α-羟基-丁酸 α-hydroxy-butyric acid |
| 4 | D-海藻糖 D-trehalose |
β-甲酰-D-葡糖苷 β-methyl-D-glucoside |
D-半乳糖 D-galactose |
肌醇 Myo-inositol |
L-精氨酸 L-arginine |
D-葡糖酸 D-gluconic acid |
L-乳酸 L-lactic acid |
β-羟基-D, L丁酸 β-hydroxy-D, L butyric acid |
| 5 | D-纤维二糖 D-cellobiose |
D-水杨苷 D-salicin |
3-甲酰葡糖 3-methyl glucose |
甘油 Glycerol |
L-天冬氨酸 L-aspartic acid |
D-葡糖醛酸 D-glucuronic acid |
柠檬酸 Citric acid |
α-酮-丁酸 α-keto-butyric acid |
| 6 | 龙胆二糖 Gentiobiose |
N-乙酰-D-葡糖胺 N-acetyl-D-glucosamine |
D-果糖 D-fructose |
D-葡糖-6-磷酸 D-glucose-6-PO4 |
L-谷氨酸 L-glutamic acid |
葡糖醛酰胺 Glucuronamid |
α-酮-戊二酸 α-keto-glutaric acid |
乙酰乙酸 Acetoacetic acid |
| 7 | 蔗糖 Sucrose |
N-乙酰-β-D-甘露糖胺 N-acetyl-β-D-mannosamine |
L-果糖 L-fructose |
D-果糖-6-磷酸 D-fructose-6-PO4 |
L-组胺 L-histidine |
黏酸; 黏液酸 Mucic acid |
D-苹果酸 D-malic acid |
丙酸 Propionic acid |
| 8 | D-松二糖 D-turanose |
N-乙酰-D-半乳糖胺 N-acetyl-D-galactosamine |
L-鼠李糖 L-rhammose |
D-天冬氨酸 D-aspartic acid |
L-焦谷氨酸 L-pyroglutamic acid |
奎宁酸 Quinic acid |
L-苹果酸 L-malic acid |
乙酸 Acetic acid |
| 9 | 水苏糖 Stachyose |
N-乙酰神经氨酸 N-acetyl neuraminic acid |
肌苷 Inosine |
D-丝氨酸 D-serine |
L-丝氨酸 L-serine |
糖质酸 D-saccharic aicd |
溴-丁二酸 Bromo-succinic acid |
甲酸 Formic acid |
| 10 | 阳性对照 Positive control |
10 g·L-1 NaCl | 乳酸钠 Sodium lactate |
醋竹桃霉素 Troleandomycin |
林肯霉素, 洁霉素 Lincomycin |
万古霉素 Vancomycin |
萘啶酮酸 Nalidixic acid |
氨曲南 Aztreonam |
| 11 | pH6 SV |
40 g·L-1 NaCl | 梭链孢酸 Fusidic acid |
利福霉素 Rifamycin SV |
盐酸胍 Guanidine HCl |
四唑紫 Tetrazolium violet |
氯化锂 Lithium chloride |
丁酸钠 Sodium butyrate |
| 12 | A12 pH5 |
80 g·L-1 NaCl | D-丝氨酸 D-serine |
二甲胺四环素 Minocyline |
硫酸四癸钠 Niaproof 4 |
四唑蓝 Tetrazolium |
亚碲酸钾 Potassium tellurite |
溴酸钠 Sodium bromate |
| 注:A~H和1~12为微孔板序号, 图 5同。 Note:A-H and 1-12 are the serial number of microplate, the same as in Figure 5. |
||||||||
|
图 5 芽孢杆菌W-4、N-2、P2和SQR9的碳源利用情况 Figure 5 The carbon source utilization of Bacillus strains W-4, N-2, P2 and SQR9 |
|
图 6 筛选菌株生物膜形成能力比较 Figure 6 Comparison of biofilm formation ability of different Bacillus strains |
解淀粉芽孢杆菌SQR9在盆栽和大田试验中显示出很好的促生能力[12], 因此我们测定了其近亲芽孢杆菌的IAA合成和植酸酶分泌能力。结果表明菌株N-2产IAA能力最强, 菌株W-4、P2、SQR9产IAA能力无显著差异(图 7-A), 但菌株W-4、P2和SQR9的植酸酶分泌能力都显著高于菌株N-2(图 7-B)。

|
图 7 4株芽孢杆菌产吲哚乙酸(A)和植酸酶(B)的能力 Figure 7 IAA(A)and phytase(B)production of four Bacillus strains |
微生物肥料在促进化肥减施增效、提高农产品品质方面发挥重要作用。目前, 由于肥料菌种在复杂根际环境中存活和定殖不稳定, 导致田间效果不稳定, 从而限制生物肥料产业发展。解淀粉芽孢杆菌SQR9是本实验室从黄瓜种植发病区的健康植株根际分离的一株根际促生菌, 在盆栽和大田试验中表现出显著的促生和生防效果, 因此我们依据亲缘识别系统, 找出黄瓜根际中菌株SQR9的近亲菌株, 为进一步研究其生态学机制筛选菌种资源。
细菌群落在半固体培养基表面由接种点向周围进行的依赖鞭毛和纤毛的迁移运动称为群集运动(swarming mobility)[15]。群集运动能提高微生物的环境适应能力, 当环境中的营养耗尽时, 微生物可以游动到营养丰富的地方。土壤环境是相对封闭的, 且分散性低, 所以微生物在土壤微环境中存在种间和种内的竞争。Vos在16 cm×16 cm的土壤立方体中筛选出78株黄色黏细菌(Myxococcus xanthus), 发现该群落在较早的时期就发生了亲缘识别, 并按生态类型分类[7]。本试验从施用过菌株SQR9的黄瓜根际筛选出21株芽孢杆菌, 经过群集运动、菌落形态、Biolog碳源利用试验和16S rRNA比对分析将其分为A~G 7组。A组中的菌株大多数可以融合, 但是T-5与I-5-1只能和2-21融合, 与这组中其余菌株都产生边界或半融合, E组也是类似情况。融合没有传递性, 也就是说如果X与Y融合, Y与Z融合, 则X与Z不一定融合。如果只有一种识别基因位点, 融合现象就可以传递[16]。G组中N21、N43、N35这3株菌的基因相似性不高, 但是N43与N35、N43与N21都能融合, 这说明芽孢杆菌亲缘识别是多基因协作的结果, 只单凭16S rRNA这一个保守位点难以准确找出表现型与基因型的相关性[16-17]。另外, 亲缘识别基因位点, 比如奇异变形杆菌中的ids基因参与边界的形成, 但随着环境压力的进化也朝着多态性发展[18-21]。
植物根际促生细菌(PGPR)通过多种机制促进植物生长和增加作物产量, 如固氮、溶磷、解钾、产铁载体、产植物激素、产抗生素等, 或者通过抑制病原菌的生长等方式间接促进植物生长[14]。本研究中, 解淀粉芽孢杆菌SQR9最为亲近的3株芽孢杆菌P2、W-4和N-2都是潜在的根际促生菌, 这3株菌在生物膜形成、IAA分泌和植酸酶活性方面都接近菌株SQR9, 但它们在田间是否有促生作用还需要盆栽和大田试验验证。Stefanic等[9]研究发现相同亚家族里的枯草芽孢杆菌可以同时定殖于拟南芥根际, 而不同亚家族的枯草芽孢杆菌不能同时定殖。因此, 这3株芽孢杆菌能与菌株SQR9在黄瓜根际共存并发挥协同的植物益生功能, 将它们复配组合开发新的微生物肥料是下一步的研究重点。
| [1] | Compant S, Clément C, Sessitsch A. Plant growth-promoting bacteria in the rhizo-and endosphere of plants:their role, colonization, mechanisms involved and prospects for utilization[J]. Soil Biology and Biochemistry, 2010, 42: 669–678. DOI: 10.1016/j.soilbio.2009.11.024 |
| [2] | Compant S, Duffy B, Nowak J, et al. Use of plant growth-promoting bacteria for biocontrol of plant diseases:principles, mechanisms of action, and future prospects[J]. Applied and Environmental Microbiology, 2005, 71: 4951–4959. DOI: 10.1128/AEM.71.9.4951-4959.2005 |
| [3] |
张瑞福, 颜春荣, 张楠, 等. 微生物肥料研究及其在耕地质量提升中的应用前景[J].
中国农业科技导报, 2013, 15(5): 8–16.
Zhang R F, Yan C R, Zhang N, et al. Studies on microbial fertilizer and its application prospects in improving arable land quality[J]. Journal of Agricultural Science and Technology, 2013, 15(5): 8–16. (in Chinese with English abstract) |
| [4] | Hamilton W D. The genetical evolution of social behaviour[J]. Journal of Theoretical Biology, 1964, 7(1): 1–16. DOI: 10.1016/0022-5193(64)90038-4 |
| [5] | Gerald S W. Food sharing in vampire bats[J]. Scientific American, 1990, 6: 76–82. |
| [6] | Wenren L M, Sullivan N L, Cardarelli L, et al. Two independent pathways for self-recognition in Proteus mirabilis are linked by type VI-dependent export[J]. Microbiology, 2013, 4(4): e00374–13. |
| [7] | Vos M, Velicer G J. Social conflict in centimeter-and global-scale populations of the bacterium Myxococcus xanthus[J]. Current Biology, 2009, 19(20): 1763–1767. DOI: 10.1016/j.cub.2009.08.061 |
| [8] | West S A, Griffin A S, Gardner A. Evolutionary explanations for cooperation[J]. Current Biology, 2007, 17(16): R661–R672. DOI: 10.1016/j.cub.2007.06.004 |
| [9] | Stefanic P, Kraigher B, Anthony-Lyons N, et al. Kin discrimination between sympatric Bacillus subtilis isolates[J]. Proc Natl Acad Sci USA, 2015, 112(45): 14042–14047. DOI: 10.1073/pnas.1512671112 |
| [10] | Cao Y, Zhang Z, Ling N, et al. Bacillus subtilis SQR9 can control Fusarium wilt in cucumber by colonizing plant roots[J]. Biology and Fertility of Soils, 2011, 47: 495–506. DOI: 10.1007/s00374-011-0556-2 |
| [11] | Qiu M, Zhang R, Xue C, et al. Application of bio-organic fertilizer can control Fusarium wilt of cucumber plants by regulating microbial community of rhizosphere soil[J]. Biology and Fertility of Soils, 2012, 48: 807–816. DOI: 10.1007/s00374-012-0675-4 |
| [12] | Xu Z, Shao J, Li B, et al. Contribution of bacillomycin D in Bacillus amyloliquefaciens SQR9 to antifungal activity and biofilm formation[J]. Applied and Environmental Microbiology, 2013, 79: 808–815. DOI: 10.1128/AEM.02645-12 |
| [13] | Hamon M A, Lazazzera B A. The sporulation transcription factor Spo0A is required for biofilm development in Bacillus subtilis[J]. Molecular Microbiology, 2001, 42: 1199–1209. |
| [14] |
崔晓双, 王伟, 张如, 等. 基于根际营养竞争的植物根际促生菌的筛选及促生效应研究[J].
南京农业大学学报, 2015, 38(6): 958–966.
Cui X S, Wang W, Zhang R, et al. Screening of plant growth-promoting rhizobacteria based on rhizosphere nutrition competiveness and investigation of their promoting effect[J]. Journal of Nanjing Agricultural University, 2015, 38(6): 958–966. DOI: 10.7685/j.issn.1000-2030.2015.06.013 (in Chinese with English abstract) |
| [15] | Butler M T, Wang Q F, Harshey R M. Cell density and mobility protect swarming bacteria against antibiotics[J]. Proc Natl Acad Sci USA, 2010, 107(8): 3776–3781. DOI: 10.1073/pnas.0910934107 |
| [16] | Mitri S, Foster K R. The genotypic view of social interactions in microbial communities[J]. Annual Review of Microbiology, 2013, 47: 247–273. |
| [17] | Strassmann J E, Gilbert O M, Queller D C. Kin discrimination and cooperation in microbes[J]. Annual Review of Microbiology, 2011, 65: 349–367. DOI: 10.1146/annurev.micro.112408.134109 |
| [18] | Gibbs K A, Urbanowski M L, Greenberg E P. Genetic determinants of self identity and social recognition in bacteria[J]. Science, 2008, 321(2008): 256–259. |
| [19] | Latta R G. Gene flow, adaptive population divergence and comparative population structure across loci[J]. New Phytolist, 2004, 161(1): 51–58. |
| [20] | Le Corre V, Kremer A. Genetic variability at neutral markers, quantitative trait land trait in a subdivided population under selection[J]. Genetics, 2003, 164(3): 1205–1219. |
| [21] |
贾雄飞, 刘俊康, 徐启旺. 奇异变形杆菌周期性群集运动的研究[J].
第三军医大学学报, 2005, 27(9): 868–870.
Jia X F, Liu J K, Xu Q W. Periodic variety in swarming motility of Proteus mirabilis[J]. Acta Academiae Medicinae Militaris Tertiae, 2005, 27(9): 868–870. (in Chinese with English abstract) |




