文章信息
- 赵富林, 徐明飞, 谢青云, 单衍可, 雷静, 施志玉, 孙海凤, 刘斐
- ZHAO Fulin, XU Mingfei, XIE Qingyun, SHAN Yanke, LEI Jing, SHI Zhiyu, SUN Haifeng, LIU Fei
- 大肠杆菌表达的RNA解螺旋酶Ded1p的提纯与解螺旋机制
- Purification and mechanism of RNA helicase Ded1p expressed in Escherichia coli
- 南京农业大学学报, 2017, 40(5): 874-880
- Journal of Nanjing Agricultural University, 2017, 40(5): 874-880.
- http://dx.doi.org/10.7685/jnau.201604027
-
文章历史
- 收稿日期: 2016-04-10
DExH/D-box蛋白是RNA解螺旋酶中最大的家族, 属于超家族2(SF2) 解螺旋酶[1]。DExH/D-box蛋白参与几乎所有的RNA代谢过程, 包括转录、mRNA前体剪接、mRNA输出、核糖体生物合成、翻译起始、细胞器基因的表达, 以及RNA降解等[2-4]。此外, 最新研究表明, DExH/D-box家族蛋白还参与调控病毒的复制, 以及宿主的抗病毒反应[5], 如HIV病毒mRNA的出核需要DDX3蛋白协作[4], NS3蛋白可以促进丙型肝炎病毒的增殖[5]。DExH/D-box蛋白的这些生物学功能的发挥, 主要通过其对短双链RNA的局部扰动(解螺旋酶活性), 或对RNA上蛋白质的移除(RNP酶活性)来实现[6-7]。所以, 阐明DExH/D-box家族蛋白的双链RNA解螺旋机制, 对调控宿主基因表达和抵抗病毒入侵都具有重要意义。
体外研究表明, DExH/D-box蛋白以ATP依赖的方式催化RNA双链解螺旋[8-9]。DExH/D-box蛋白家族中的DEAD-box家族蛋白与传统的依靠在核酸链上移位来解开双链的解螺旋酶不同[10], 它是通过局部双链解开的方式进行解螺旋[11-12], 且只能解开较短的RNA-RNA或DNA-RNA双链[13]。此外, DExH/D-box蛋白解螺旋过程不需要消耗ATP[14-15], ATP水解是为了促进DEAD-box蛋白与RNA结合和蛋白的再循环[14]。对于大多数DEAD-box蛋白来说, 邻近的单链RNA会促进蛋白结合到双链区域[16], 而且对单链方向没有依赖性[17-19]。这种解螺旋机制被称为局部双链解开模式, 并且这种模式被认为是唯一适合DEAD-box蛋白在细胞内催化RNA和RNA-蛋白质复合物重组的方式[20-22]。Ded1p来自于酿酒酵母[23], 属于DExH/D-box蛋白家族中的DEAD-box家族蛋白, 它因作为染色体内编码HIS 3基因的一个重要结构域而首次获得分离, 故被命名为Ded1(defines essential domain1)[24]。Ded1p是DExH/D-box RNA解螺旋酶家族的典型代表[12]。研究表明, Ded1p主要以低聚体的形式发挥解螺旋活性[25], 但是Ded1p的解螺旋机制目前仍不明确。
单分子荧光共振能量转移(single molecule fluorescence resonant energy transfer, smFRET)技术通过检测单个分子内的荧光供体及受体间荧光能量转移效率, 来研究分子的构象变化[26]。相对于传统探测分子综合平均效应技术来说, smFRET可以对体系中的单个分子进行研究, 得到某一分子特性的分步状况, 也可以研究生物分子的动力学反应[27-28]。Syed等[10]利用smFRET技术揭示了T7解螺旋酶的分步解螺旋机制。Karunatilaka等[29]利用smFRET技术揭示了Mss116解螺旋酶介导的二型内含子折叠机制。smFRET技术近年来在探究生物学机制方面发展迅猛, 包括DNA的复制和重组, RNA的转录、折叠和催化, 蛋白的翻译、折叠和构象变化等[30-32]。
本研究以原核表达的全长Ded1p为研究对象, 以荧光标记的核酸为底物, 利用smFRET技术, 在单分子层面直观探究Ded1p对双链RNA的解螺旋机制。
1 材料与方法 1.1 试验材料Ni-NTA琼脂糖从Invitrogen(美国)购买, Capto-DEAE琼脂糖从GE(美国)购买, DNA序列从Genewiz(苏州, 中国)购买, RNA序列是用TranscriptAid T7 High Yield Transcription试剂盒(Thermo Scientific)用模板DNA转录而来, 并用非变性PAGE纯化。荧光标记的DNA和RNA从TaKaRa(大连, 中国)购买, ATP、BSA、Trolox、葡萄糖氧化酶、过氧化氢酶等都是从Sigma购买, D-葡萄糖从Amresco购买。主要RNA和DNA序列如下:
A. 41 nt RNA:5′-AAA ACA GAA CAU AAC CAA ACA AAA UAG CAC CGU AAA GAC GC-3′;
B. 16 nt RNA-6-FAM:5′-GCG UCU UUA CGG UGC U-6-FAM-3′;
C. 13 nt RNA-6-FAM:5′-GCG UCU UUA CGG U-6-FAM-3′;
D. 13 bp 3′tail RNA:5′-ACC GUA AAG ACG CAG CCA GCA UCA AUG ACA UCA GCA UC-3′;
E. Cy5-RNA-DNA-biotin:5′-Cy5-GCG UCU UUA CGG UGC UdAdGdTdTdCdAdCdG dAdAdA-biotin-3′;
F. Cy3-DNA:5′-TTT CGT(Cy3) GAA CT-3′。
1.2 Ded1p原核表达与纯化Ded1p-pET22b重组质粒源自美国Eckhard Jankowasky实验室的馈赠。将Ded1p-pET22b重组质粒转化至BL21(DE3) 大肠杆菌内, 通过菌液PCR与质粒测序, 确定质粒转化成功。然后于37 ℃培养细菌至D600达到0.6~0.8, 加入终浓度为0.5 mmol·L-1的IPTG, 于28 ℃诱导培养16 h后, 利用SDS-PAGE电泳和考马斯亮蓝染色验证目的蛋白表达情况。基于验证结果, 以同样的诱导温度和时间, 用0.5 mmol·L-1 IPTG在大量的BL21(DE3) 中诱导表达Ded1p[33]。表达完成后, 对Ded1p进行两步纯化。鉴于Ded1p上挂有6×His标签, 首先使用Ni-NTA凝胶介质进行亲和层析, 再采用Capto-DEAE凝胶阴离子交换剂进行离子交换层析, 最终得到纯净的Ded1p。
1.3 Ded1p解螺旋活性验证建立解螺旋酶体外解螺旋反应体系, 再利用非变性聚丙烯酰胺凝胶电泳(native-PAGE)验证体外表达纯化的Ded1p是否具有双链RNA解螺旋活性。本研究中涉及的所有解螺旋反应均在标准解螺旋缓冲液(helicase reaction buffer, HRB)中进行。HRB包括:40 mmol·L-1 Tris-HCl(pH8.0)、50 mmol·L-1 NaCl、0.5 mmol·L-1 MgCl2、2 mmol·L-1 DTT、1 U·μL-1RNasin、1 g·L-1 Nonidet P-40[12]。反应温度19 ℃。为了达到反应预平衡状态, 800 nmol·L-1 Ded1p先与2 nmol·L-1带有6-FAM标记的双链RNA在HRB中孵育5 min, 然后加入等浓度的MgCl2和ATP(终浓度为2 mmol·L-1)起始解螺旋反应。于19 ℃的金属浴中反应1 h后加入终止缓冲液终止反应。终止缓冲液包括:50 mmol·L-1 EDTA、10 g·L-1SDS、1 g·L-1二甲苯蓝、1 g·L-1溴酚蓝和20%甘油[12]。取反应液, 以10 V·cm-1的速度, 在1×TBE缓冲液中进行非变性聚丙烯酰胺凝胶电泳。电泳结束后, 利用激光扫描成像系统Typhoon(GE, 美国)对凝胶进行扫描成像。基于核酸底物上的6-FAM荧光标签, 可以观察到单链RNA和双链RNA的分布情况, 从而分析体外表达纯化的Ded1p是否具有解螺旋酶活性。
1.4 smFRET试验于笔者所在实验室自行搭建的、基于全内反射荧光显微镜的光学平台上进行smFRET试验。该平台主要包括:奥林巴斯IX81显微镜、PHOTOMETRICS Evolve 512电子倍增电荷耦合器(EMCCD)和50 mW的COHERENT 532 nm固态激光器。首先制备Ded1p smFRET试验的核酸底物:将D、E、F 3条核酸链按5:3:2的比例进行退火, 形成一条3′末端带有25 nt单链RNA尾巴的13 bp双链RNA底物, 再使用PEG/biotin-PEG混合液包被自制样品流动通道, 经PEG/biotin-PEG包被处理后的样品通道既可以防止蛋白的非特异性吸附, 也可以特异性的固定双链RNA[26, 34]。然后将稀释在HRB缓冲液中的RNA样品(终浓度50 pmol·L-1, Cy3-Cy5荧光对标记和生物素标记的双链RNA)加入到预处理后的样品通道中, 基于核酸分子上的生物素与通道中链霉亲和素的亲和作用, 带有荧光对标记的双链RNA固定至通道中。值得注意的是, HRB中还需添加除氧试剂和三重态淬灭剂(包括:8 g·L-1D-葡萄糖、葡萄糖氧化酶、过氧化氢酶和Trolox)以除去通道内的氧气, 延长荧光分子的寿命。最后加入等浓度的MgCl2和ATP(终浓度2 mmol·L-1)起始解螺旋反应。试验开始时开启532 nm的激光激发样品通道中的核酸样品, 通过奥林巴斯的油浸物镜(100×、1.49 N.A.)采集荧光信号。供体荧光分子(Cy3) 和受体荧光分子(Cy5) 的荧光被PHOTOMETRICS的DV2分开, 分别被投射到EMCCD的左右两边。通过Micro-Manager控制, 以100 ms每帧的速度, 采集100 s的时间轨迹。Ded1p的解螺旋反应在被ATP起始前, 由于构建的核酸底物上的Cy3和Cy5相对距离较远, 受激光激发后产生的荧光共振能量转移效率很低, 表现为低共振能量转移(FRET)值。当加入ATP后, 由于Ded1p以ATP依赖的方式打开了双链RNA, 解螺旋后的单链RNA较双链RNA更容易发生折叠和扭曲, 进而拉近了Cy3和Cy5的距离, 表现为高FRET值。本试验通过观测和分析ATP加入前后Cy3和Cy5荧光强度与FRET的变化, 来探讨Ded1p的解螺旋机制。
1.5 数据处理首先采用Image J软件对所采集的数据进行去噪声处理, 然后采用IDL软件进行trace的提取, 接着采用Matlab软件进行smFRET直方图分布的提取和单个分子时间曲线的提取, 再采用vb-FRET进行步骤拟合, 最后用Excel 2013和Matlab进行数据的统计与拟合。
2 结果与分析 2.1 Ded1p原核表达与纯化 2.1.1 Ded1p的诱导表达及验证以5 mL阳性BL21(DE3) 转化菌为基础, 37 ℃摇床培养至D600达0.6~0.8, 加入终浓度为0.5 mmol·L-1的IPTG, 于28 ℃摇床中诱导表达16 h后, 超声裂解菌体, 收集上清液, 通过SDS-PAGE验证诱导结果。如图 1所示, 诱导的菌体裂解上清液在相对分子质量70×103的位置有明显的条带, 该大小与带有6×His标签的Ded1p大小(约70.5×103)一致, 而未诱导的菌体裂解上清液在该位置则没有明显的条带, 说明Ded1p能够用0.5 mmol·L-1 IPTG诱导表达。
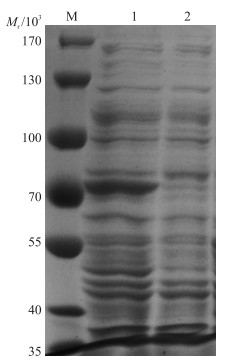
|
图 1 Ded1p诱导表达的验证 Figure 1 Expression of Ded1p M.蛋白标准品; 1. 0.5 mmol·L-1 IPTG诱导16 h; 2.无IPTG。 M. Protein marker; 1. 0.5 mmol·L-1 IPTG induced for 16 h; 2. No IPTG. |
以4 L阳性BL21(DE3) 转化菌为基础, 分装在4个1 L的锥形瓶中, 于37 ℃摇床培养至D600达0.6~0.8, 加入终浓度为0.5 mmol·L-1的IPTG, 于28 ℃摇床中诱导表达16 h后, 超声裂解菌体, 收集上清液。将上清液与平衡好的Ni-NTA凝胶在4 ℃过夜孵育后用不同浓度的咪唑洗脱目的蛋白, 最后通过SDS-PAGE电泳鉴定纯化效果。结果如图 2所示:Ni-NTA凝胶层析不足以得到1个单一的目的条带, 说明Ded1p仍含有部分杂蛋白。由于该Ded1p达不到单分子试验需要的蛋白纯度, 所以还需要进行进一步纯化。

|
图 2 Ni-NTA凝胶纯化Ded1p Figure 2 Ded1p purified by Ni-NTA agarose M.蛋白标准品; 1~20. 250 mmol·L-1咪唑洗脱液。 M. Protein maker; 1-20. Eluted by washing buffer with 250 mmol·L-1 imidazole. |
将8~14泳道代表的蛋白液合并到一起, 用结合缓冲液稀释6倍, 使NaCl浓度降低至50 mmol·L-1, 然后与平衡好的Capto-DEAE凝胶介质在4 ℃进行孵育结合, 最后进行盐离子梯度洗脱。结果如图 3可见, 在300 mmol·L-1 NaCl条件下, 能够得到条带单一的Ded1p, 该Ded1p几乎没有杂蛋白, 可用于单分子试验。

|
图 3 Capto-DEAE agarose纯化Ded1p Figure 3 Ded1p purified by Capto-DEAE agarose M.蛋白标准品; 1.结合后流出液; 2~8、9~15、16~22、23~33分别为100、150、200和300 mmol·L-1 NaCl洗脱液。 M. Protein maker; 1. Flow-through after binding; 2-8, 9-15, 16-22, 23-33 were eluted by 100, 150, 200 and 300 mmol·L-1 NaCl washing buffer, respectively. |
我们设计了2组底物进行Ded1p解螺旋酶活性验证, 一组为带有5′尾巴的16 bp dsRNA(1~5号反应), 一组为带有5′尾巴的13 bp dsRNA(6~10号反应)(图 4)。通过native-PAGE分析Ded1p是否具有双链RNA的解螺旋活性。PAGE是由丙烯酰胺和甲叉双丙烯酰胺交联形成的三维网状的结构物质, 具有分子筛效应。由于RNA所带电荷一致, native-PAGE对核酸的分离主要依赖于RNA分子的大小, 而双链RNA分子较单链RNA分子大, 所以在PAGE中移动较慢, 条带位于PAGE胶的上方; 如果双链RNA被Ded1p解开, 则2条单链RNA移动较快, 位于双链RNA的下方。为了更加明确Ded1p是否具有解螺旋活性, 我们对具有ATP和Ded1p的反应做了一次重复, 即1号和2号, 6号和7号。结果如图 4所示, 对于16 bp的双链RNA(图 4-A), 与对照组相比(4泳道), 有ATP存在时, Ded1p在1 h内几乎能够解开16 bp双链RNA(1、2泳道)。没有ATP时, Ded1p不能打开双链RNA, 并且表现出促进退火的能力, 能够将没有完全退火的单链RNA退火为双链(3泳道)。对于13 bp的双链RNA(图 4-B), 与对照组相比(9泳道), 有ATP存在时, Ded1p也能够实现双链RNA的解链(6、7泳道)。没有ATP时, Ded1p也具有退火活性, 能够将没有完全退火的单链RNA退火为双链(8泳道)。
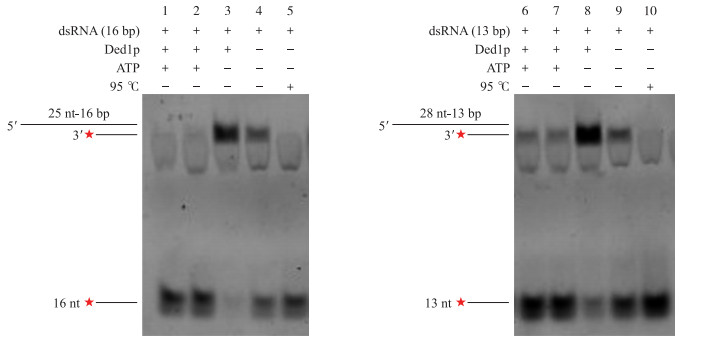
|
图 4 Ded1p解螺旋活性验证的电泳图(120 g·L-1PAGE) Figure 4 Unwinding activity of Ded1p(120 g·L-1PAGE) 1~5.以16 bp dsRNA为底物; 6~10.以13 bp dsRNA为底物。RNA以黑色线条表示, 6-FAM荧光分子以红色五角星表示。 1-5. Use 16 bp dsRNA as substrate; 6-10. Use 13 bp dsRNA as substrate. RNA was shown with black line, 6-FAM fluorescence was shown with red pentagram. |
我们设计了一条3′末端带有25 nt单链RNA尾巴的13 bp双链RNA底物, 用于Ded1p解螺旋机制的smFRET探究(图 5-A)。没有加入Ded1p和ATP时, FRET值分布在0.37附近(0.37前的小峰为荧光淬灭形成的小峰)(图 5-B), 说明此时的荧光共振能量转移效率(E)低。当加入800 nmol·L-1 Ded1p和2 mmol·L-1 ATP/Mg2+起始解螺旋反应后, FRET值分别分布在0.37和0.85附近(图 5-C)。0.37代表的是解螺旋反应起始前底物的状态, 即双链RNA表现的FRET值。0.85较0.37有显著的提高, 说明发生了高效率的能量跃迁, 认为是双链RNA的解螺旋导致, 故将高FRET值定义为底物完全解离态。
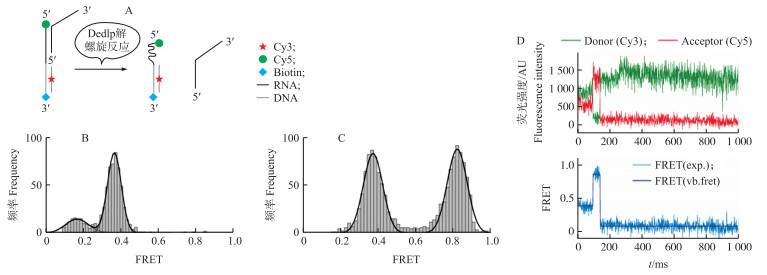
|
图 5 smFRET试验揭示Ded1p一步解开双链RNA Figure 5 smFRET experiment revealed that Ded1p unwinds dsRNA by one step A:试验设计, Ded1p起始解螺旋反应前后, 双链RNA底物构象发生变化; B:双链RNA底物的FRET分布直方图; C:起始解螺旋反应后的FRET分布直方图; D:起始反应后, 底物被532 nm激光激发时, Cy3和Cy5荧光强度的时间轨迹和FRET时间轨迹的变化。 A:Design, conformation of dsRNA substrate changes before and after Ded1p unwinds the duplex; B:The FRET histogram of dsRNA substrate; C:The FRET histogram of substrate after Ded1p initiates the unwinding reaction; D:With substrate excited by 532 nm laser, representative Cy3 and Cy5 fluorescence intensity time-trace(top panel)and FRET efficiency trajectory(bottom panel)after the reaction starts. |
进一步采集提取加入Ded1p和ATP后单个RNA分子上Cy3和Cy5荧光对的荧光强度时间轨迹与FRET时间轨迹, 结果(图 5-D)表明:Cy3的荧光强度在解螺旋反应起始后迅速降低, 而Cy5的荧光强度表现为相应的升高, 说明供体荧光分子与受体荧光分子之间产生了能量转移。对应的FRET时间轨迹也表现出同样的变化趋势, FRET由0.37跃迁至0.85。综上所述, 本试验结果表明:Ded1p以一步解螺旋的方式打开13 bp的dsRNA, 该结果在单分子层面上进一步佐证了之前的报道, 即根据生化实验结果提出的局部双链解螺旋模式[2]。
3 讨论本试验首次利用Capto-DEAE凝胶这种弱阴离子交换剂对Ded1p进行二次纯化, 得到纯度更高的Ded1p。而文献[12]报道过采用p-cellulose这种阳离子交换剂对Ded1p进行二次纯化。p-cellulose是带有磷酸基团的中等酸型阳离子交换剂, 本质是纤维素, 其活性基团位于表面, 能够分离蛋白等生物大分子。当pH值大于7时, 磷酸基团电离形成带负电的阴离子, 能够结合Ded1p上带正电的基团, 通过提高流动相中阳离子浓度, 吸附的蛋白就会被洗脱下来, 达到分离的效果, 然而p-cellulose已经停产, 这对Ded1p的提纯提出了新的挑战。我们曾尝试过多种提纯方法, 包括Q交换柱、分子筛等, 均未取得良好效果。而Capto-DEAE是带有二乙氨基乙基的弱碱型弱阴离子交换剂, 本质是琼脂糖, 能够分离大分子物质, 不会引起被分离蛋白的变性或失活, 而且其非特异性吸附很低, 交换容量比纤维素高3~4倍。当pH值稍微大于蛋白的等电点时, 蛋白带负电荷, 带正电荷的二乙氨基乙基能够将其吸附, 当提高流动相中的负离子, 被DEAE吸附的蛋白就会被洗脱下来, 达到分离纯化的效果。综合比较, 后者对Ded1p的活性保持以及分离效率都要高, 所以我们选择了Capto-DEAE进行二次纯化。
大量的晶体结构和生化试验研究表明, DExH/D-box家族的DEAD-box RNA解螺旋酶解开RNA双链的方式与传统通过移位的方式解螺旋的蛋白不同[1, 12, 14-15, 25, 35], 它是通过局部双链打开的模式进行解螺旋反应[2], 并且非共价相连但空间距离极近的一段单链DNA或RNA能够促进DEAD-box蛋白结合到双链区域, 进而发挥解螺旋酶活性[12]。DEAD-box蛋白打开双链RNA不需要水解ATP供能[14]。DEAD-box蛋白是以低聚体的形式合作发挥解螺旋作用[25]。虽然以前的研究已经揭示了很多DEAD-box蛋白的解螺旋特点, 但其解螺旋机制目前还存在很多疑点。本试验对Ded1p这个典型的DEAD-box蛋白展开研究, 从单分子水平上揭示了Ded1p对双链RNA的解螺旋机制。
研究结果表明, 一旦Ded1p结合到RNA双链区域, Ded1p的构象就会发生改变, 只紧密地结合一条RNA单链, 从而与RNA双链不相容[15], 此外该构象改变的过程需要ATP结合才能实现[36-37]。这个过程就会导致局部双链被打开[15], 这与本试验所看到的高FRET的存在相一致。对晶体结构的研究发现, 起始解螺旋反应后, DExH/D-box家族的DEAD-box蛋白只能直接打开5~6个碱基对[38-40], 其余的碱基对会很快自发的解开, 不需要蛋白和ATP水解的参与[15]。由此, 我们认为本试验所显示的高FRET分布代表dsRNA完全解开时的状态。时间轨迹曲线同样显示dsRNA被一步解开。以上结果与局部双链打开模式一致。
本研究通过原核表达并纯化出有活性的Ded1p, 利用基于全内反射的mFRET试验, 直接证明DExH/D-box家族的DEAD-box蛋白Ded1p是以局部双链打开的模式发挥其解螺旋酶活性。
| [1] | Linder P, Jankowsky E. From unwinding to clamping:the DEAD box RNA helicase family[J]. Nat Rev Mol Cell Biol, 2011, 12(8): 505–516. DOI: 10.1038/nrm3154 |
| [2] | Yang Q, Campo M D, Lambowitz A M, et al. DEAD-box proteins unwind duplexes by local strand separation[J]. Molecular Cell, 2007, 28(2): 253–263. DOI: 10.1016/j.molcel.2007.08.016 |
| [3] | Tarn W Y, Chang T H. The current understanding of Ded1p/DDX3 homologs from yeast to human[J]. RNA Biol, 2009, 6: 17–20. DOI: 10.4161/rna.6.1.7440 |
| [4] | Sharma D, Jankowsky E. The Ded1/DDX3 subfamily of DEAD-box RNA helicases[J]. Critical Reviews in Biochemistry and Molecular Biology, 2014, 49(4): 343–360. DOI: 10.3109/10409238.2014.931339 |
| [5] | Kwong A D, Rao B G, Jeang K T. Viral and cellular RNA helicases as antiviral targets[J]. Nat Rev Drug Discov, 2005, 4(10): 845–853. DOI: 10.1038/nrd1853 |
| [6] | Rossler O G, Straka A, Stahl H. Rearrangement of structured RNA via branch migration structures catalysed by the highly related DEAD-box proteins p68 and p72[J]. Nucleic Acids Research, 2001, 29(10): 2088–2096. DOI: 10.1093/nar/29.10.2088 |
| [7] | Tsu C A, Kossen K, Uhlenbeck O C. The Escherichia coli DEAD protein DbpA recognizes a small RNA hairpin in 23S rRNA[J]. RNA, 2001, 7(5): 702–709. DOI: 10.1017/S1355838201010135 |
| [8] | Jankowsky E, Fairman M E. RNA helicases-one fold for many functions[J]. Current Opinion in Structural Biology, 2007, 17(3): 316–324. DOI: 10.1016/j.sbi.2007.05.007 |
| [9] | Theissen B, Karow A R, Gubaev A, et al. Cooperative binding of ATP and RNA induces a closed conformation in a DEAD box RNA helicase[J]. Proc Natl Acad Sci USA, 2008, 105(2): 548–553. DOI: 10.1073/pnas.0705488105 |
| [10] | Syed S, Pandey M, Patel S S, et al. Single-molecule fluorescence reveals the unwinding stepping mechanism of replicative helicase[J]. Cell Rep, 2014, 6(6): 1037–1045. DOI: 10.1016/j.celrep.2014.02.022 |
| [11] | del Campo M, Mohr S, Jiang Y, et al. Unwinding by local strand separation is critical for the function of DEAD-box proteins as RNA chaperones[J]. J Mol Biol, 2009, 389(4): 674–693. DOI: 10.1016/j.jmb.2009.04.043 |
| [12] | Yang Q S, Jankowsky E. The DEAD-box protein Ded1 unwinds RNA duplexes by a mode distinct from translocating helicases[J]. Nature Structural and Molecular Biology, 2006, 13(11): 981–986. DOI: 10.1038/nsmb1165 |
| [13] | Mallam A L, Campo M D, Gilman B, et al. Structural basis for RNA-duplex recognition and unwinding by the DEAD-box helicase Mss116p[J]. Nature, 2012, 490(7418): 121–125. DOI: 10.1038/nature11402 |
| [14] | Liu F, Putnam A, Jankowsky E. ATP hydrolysis is required for DEAD-box protein recycling but not for duplex unwinding[J]. Proc Natl Acad Sci USA, 2008, 105(51): 20209–20214. DOI: 10.1073/pnas.0811115106 |
| [15] | Chen Y F, Potratz J P, Tijerina P, et al. DEAD-box proteins can completely separate an RNA duplex using a single ATP[J]. Proc Natl Acad Sci USA, 2008, 105(51): 20203–20208. DOI: 10.1073/pnas.0811075106 |
| [16] | Jankowsky E, Fairman M E, Yang Q S. RNA helicases:versatile ATP-driven nanomotors[J]. Journal of Nanoscience and Nanotechnology, 2005, 5(12): 1983–1989. DOI: 10.1166/jnn.2005.508 |
| [17] | Rozen F, Edery I, Meerovitch K, et al. Bidirectional RNA helicase activity of eukaryotic translation initiation factor-4a and factor-4f[J]. Molecular and Cellular Biology, 1990, 10(3): 1134–1144. DOI: 10.1128/MCB.10.3.1134 |
| [18] | Huang Y, Liu Z R. The ATPase, RNA unwinding, and RNA binding activities of recombinant p68 RNA helicase[J]. J Biol Chem, 2002, 277(15): 12810–12815. DOI: 10.1074/jbc.M200182200 |
| [19] | Bizebard T, Ferlenghi I. Studies on three E.coli DEAD-box helicases point to an unwinding mechanism different from that of model DNA helicases[J]. Biochemistry, 2004, 43(24): 7857–7866. DOI: 10.1021/bi049852s |
| [20] | Yang Q, Fairman M E, Jankowsky E. DEAD-box-protein-assisted RNA structure conversion towards and against thermodynamic equilibrium values[J]. J Mol Biol, 2007, 368(4): 1087–1100. DOI: 10.1016/j.jmb.2007.02.071 |
| [21] | Schutz P, Bumann M, Oberholzer A E, et al. Crystal structure of the yeast eIF4A-eIF4G complex:an RNA-helicase controlled by protein-protein interactions[J]. Proc Natl Acad Sci USA, 2008, 105(28): 9564–9569. DOI: 10.1073/pnas.0800418105 |
| [22] | Putnam A A, Jankowsky E. DEAD-box helicases as integrators of RNA, nucleotide and protein binding[J]. Biochim Biophys Acta, 2013, 1829(8): 884–893. DOI: 10.1016/j.bbagrm.2013.02.002 |
| [23] | Beckham C, Hilliker A, Cziko A M, et al. The DEAD-box RNA helicase Ded1p affects and accumulates in Saccharomyces cerevisiae P-bodies[J]. Mol Biol Cell, 2008, 19(3): 984–993. |
| [24] | Struhl K. Nucleotide sequence and transcriptional mapping of the yeast Pet56-His3-Ded1 gene region[J]. Nucleic Acids Research, 1985, 13(23): 8587–8601. DOI: 10.1093/nar/13.23.8587 |
| [25] | Putnam A A, Gao Z F, Liu F, et al. Division of labor in an oligomer of the DEAD-Box RNA helicase Ded1p[J]. Mol Cell, 2015, 59(4): 541–552. DOI: 10.1016/j.molcel.2015.06.030 |
| [26] | Roy R, Hohng S, Ha T. A practical guide to single-molecule FRET[J]. Nat Methods, 2008, 5(6): 507–516. DOI: 10.1038/nmeth.1208 |
| [27] | Joo C, Ha T. Single-molecule FRET with total internal reflection microscopy[J]. Cold Spring Harb Protoc, 2012. DOI: 10.1101/pbd.top072058 |
| [28] | Ha T. Single-molecule fluorescence resonance energy transfer[J]. Methods, 2001, 25(1): 78–86. DOI: 10.1006/meth.2001.1217 |
| [29] | Karunatilaka K S, Solem A, Pyle A M, et al. Single-molecule analysis of Mss116-mediated group Ⅱ intron folding[J]. Nature, 2010, 467(7318): 935–939. DOI: 10.1038/nature09422 |
| [30] | Ha T, Kozlov A G, Lohman T M. Single-molecule views of protein movement on single-stranded DNA[J]. Annu Rev Biophys, 2012, 41: 295–319. DOI: 10.1146/annurev-biophys-042910-155351 |
| [31] | Bhattacharyya B, George N P, Thurmes T M, et al. Structural mechanisms of PriA-mediated DNA replication restart[J]. Proc Natl Acad Sci USA, 2014, 111(4): 1373–1378. DOI: 10.1073/pnas.1318001111 |
| [32] | Balci H, Arslan S, Myong S, et al. Single-molecule nanopositioning:structural transitions of a helicase-DNA complex during ATP hydrolysis[J]. Biophys J, 2011, 101(4): 976–984. DOI: 10.1016/j.bpj.2011.07.010 |
| [33] | Yang Q S, Jankowsky E. ATP-and ADP-dependent modulation of RNA unwinding and strand annealing activities by the DEAD-box protein DED1[J]. Biochemistry, 2005, 44(41): 13591–13601. DOI: 10.1021/bi0508946 |
| [34] | Liu F, Putnam A A, Jankowsky E. DEAD-box helicases form nucleotide-dependent, long-lived complexes with RNA[J]. Biochemistry, 2014, 53(2): 423–433. DOI: 10.1021/bi401540q |
| [35] | Cordin O, Banroques J, Tanner N K, et al. The DEAD-box protein family of RNA helicases[J]. Gene, 2006, 367: 17–37. DOI: 10.1016/j.gene.2005.10.019 |
| [36] | Lorsch J R, Herschlag D. The DEAD box protein eIF4A. 1. A minimal kinetic and thermodynamic framework reveals coupled binding of RNA and nucleotide[J]. Biochemistry, 1998, 37(8): 2180–2193. DOI: 10.1021/bi972430g |
| [37] | Polach K J, Uhlenbeck O C. Cooperative binding of ATP and RNA substrates to the DEAD/H protein DbpA[J]. Biochemistry, 2002, 41(11): 3693–3702. DOI: 10.1021/bi012062n |
| [38] | Sengoku T, Nureki O, Nakamura A, et al. Structural basis for RNA unwinding by the DEAD-box protein Drosophila Vasa[J]. Cell, 2006, 125(2): 287–300. DOI: 10.1016/j.cell.2006.01.054 |
| [39] | Andersen C B, Ballut L, Johansen J S, et al. Structure of the exon junction core complex with a trapped DEAD-box ATPase bound to RNA[J]. Science, 2006, 313(5795): 1968–1972. DOI: 10.1126/science.1131981 |
| [40] | Bono F, Ebert J, Lorentzen E, et al. The crystal structure of the exon junction complex reveals how it maintains a stable grip on mRNA[J]. Cell, 2006, 126(4): 713–725. DOI: 10.1016/j.cell.2006.08.006 |




