文章信息
- 徐志辉, 张慧慧, 张钰婷, 冯元韬, 张馨玉, 仇美华
- XU Zhihui, ZHANG Huihui, ZHANG Yuting, FENG Yuantao, ZHANG Xinyu, QIU Meihua
- Bacillomycin D突变体在生物膜形成中的转录组分析
- Transcriptome analysis of antibiotic bacillomycin D-deficient mutant on biofilm formation
- 南京农业大学学报, 2017, 40(5): 850-858
- Journal of Nanjing Agricultural University, 2017, 40(5): 850-858.
- http://dx.doi.org/10.7685/jnau.201704025
-
文章历史
- 收稿日期: 2017-04-17
2. 江苏省耕地质量与农业环境保护站, 江苏 南京 210036
2. Jiangsu Province Station of Farmland Quality and Agricultural Environment Protection, Nanjing 210036, China
解淀粉芽孢杆菌作为一类重要的植物根际促生细菌(plant growth-promoting rhizobacteria, PGPR), 具有易分离和培养, 生长速度快, 在植物根表易于存活和定殖, 能产生多种抗生素类代谢产物, 并且抗逆性强而有利于制剂生产和储存等优点, 是一种理想的生防微生物[1]。土壤中分离的大部分野生型解淀粉芽孢杆菌在实验室条件下都能形成稳定结构的生物膜。大量研究表明, 细菌在实验室条件下的生物膜形成能力与其在根际的定殖能力和根表的成膜能力紧密相关。Bais等[2]证实, 野生型芽孢杆菌Bacillus subtilis 6051的srf缺失突变株的生物膜形成能力及根际定殖能力与野生型相比均显著下降。Chen等[3]对几株野生的芽孢杆菌进行了生物膜形成相关基因的敲除, 发现突变体的成膜与定殖能力显著相关。
抗生素类物质的功能一般认为是微生物竞争的有效武器, 但近期报道证明抗生素类物质可以作为信号分子, 调节微生物群落的动态平衡[4]。Hoffman等[5]发现, 氨基糖苷类抗生素tobramycin在低于最低抑菌浓度时能促进细菌生物膜的形成。López等[6]报道, 脂肽类抗生素surfactin能诱导细胞膜穿孔, 导致细胞膜钾离子泄露, 这一信号被细胞膜激酶KinC感受并传导至SinI~Spo0A途径, 最终促进了枯草芽孢杆菌6051生物膜形成。前期研究表明, 同为脂肽类抗生素, bacillomycin D不能诱导钾离子泄露, 但其合成基因bamD缺失突变体显著影响其生物膜形成和根际定殖能力, 说明该抗生素影响芽孢杆菌生物膜形成, 且存在新的调控途径[7]。
解淀粉芽孢杆菌SQR9是本实验室从黄瓜种植发病区的健康植株根际分离的一株根际促生菌, 在盆栽和大田试验中表现出显著的促生和生防效果[8-9]。脂肽类抗生素bacillomycin D不仅是根际有益菌SQR9拮抗尖孢镰刀菌的主要拮抗物质, 还会影响SQR9生物膜的形成和根际定殖能力, 但其分子机制还不明确[7]。本研究拟采用转录组技术, 分析野生型SQR9与bacillomycin D突变体SQR9M1在生物膜形成过程中基因表达差异, 初步揭示bacillomycin D影响SQR9生物膜形成的机制。
1 材料与方法 1.1 材料 1.1.1 供试菌株解淀粉芽孢杆菌(Bacillus amyloliquefaciens)SQR9, 突变株SQR9M1(抗生素bacillomycin D基因簇bmyDABC中合成基因bmyD定点突变, 四环素抗性)。
1.1.2 试剂零霉素和氯霉素购自北京鼎国生物技术有限责任公司; Killing Buffer(20 mmol · L-1 Tris-HCl(pH7.5)、5 mmol · L-1 MgCl2、20 mmol · L-1 NaN3, 过滤除菌); RNA提取试剂盒购自美国Axygen公司; 其他试剂均系进口分装或国产分析纯。
1.1.3 供试培养基[10]LB培养基:酵母粉5 g, 蛋白胨10 g, 氯化钠5 g, 蒸馏水1 000 mL, pH7.0。MSgg培养基:磷酸钾缓冲液5 mmol · L-1(pH7.0), MOPS溶液100 mmol · L-1(pH7.0), 氯化镁2 mmol · L-1, 氯化钙700 μmol · L-1, 氯化锰50 μmol · L-1, 氯化铁50 μmol · L-1, 氯化锌1 μmol · L-1, 硫胺2 mmol · L-1, 甘油0.5%, 谷氨酸0.5%, 色氨酸50 μmol · L-1, 苯丙氨酸50 mg · mL-1。
1.1.4 引物所用引物均由上海英骏生物技术有限公司合成, 引物序列见表 1。
| 引物名称 Primers name | 引物序列 Oligonucleotide sequences |
| P1-feuB | 5′-GACAACTTCAATAGGCGTCTCAA-3′ |
| P2-feuB | 5′-CTCGTCGGATCGGACTACAGC-3′ |
| P3-yusV | 5′-CGACCAGATAATGGGCATAACG-3′ |
| P4-yusV | 5′-GCTTGACGAGCCGACAACCTA-3′ |
| P5-fhuB | 5′-GGGAATGATCCACCTGTAATCTACC-3′ |
| P6-fhuB | 5′-TGAAAGTAATCGGAATGTTTGTCGT-3′ |
| P7-yfmD | 5′-CGGTCAAGTCCGACATCCTGC-3′ |
| P8-yfmD | 5′-CGCCCTGACAAACAATGATTTCTAC-3′ |
| P9-sinI | 5′-TAGATAAGGAATGGGTTGAGC-3′ |
| P10-sinI | 5′-GATTTACGGTATGACTTCTGG-3′ |
| P11-yqxM | 5′-ATTTTTACGGCTTTCGTTCATT-3′ |
| P12-yqxM | 5′-GTCCGCTCTTTTCCCTTATTCT-3′ |
| P13-epsD | 5′-AGGACAACGGCTACGACATGA-3′ |
| P14-epsD | 5′-AGCGGGATAAACGAGAATAAAT-3′ |
| P15-kinC | 5′-AGAAATTTGTCGGAAGCCTGGG-3′ |
| P16-kinC | 5′-ATATGGTTGAGATGATGAGCGG-3′ |
生物膜的测定方法参照文献[11], 稍作修改。将过夜活化的SQR9和SQR9M1接种于新鲜LB培养基中, 37 ℃、170 r · min-1培养至D600约为1.0。4 ℃离心收集菌体后, 用无菌水洗涤并重悬于等体积的MSgg培养基中。生物膜定性试验采用24孔细胞培养板, 培养孔中加入2 mL MSgg培养基, 然后分别接入SQR9和SQR9M1各10 μL, 设置4个重复, 于37 ℃静置培养, 24 h后观察和监测SQR9和SQR9M1的成膜情况。为了探究外源添加铁离子后对野生型SQR9和突变体SQR9M1的影响, 设置了4个处理:1) SQR9M1+乳铁蛋白(lactoferrin, 20 μg · mL-1); 2) SQR9M1+氯化铁(Fe chloride, 10 μmol · L-1); 3) SQR9+乳铁蛋白(lactoferrin, 20 μg · mL-1); 4) SQR9+氯化铁(Fe chloride, 10 μmol · L-1)。每个处理重复3次。
生物膜定量测定时用移液器轻轻吸出生物膜下的培养基和游离的细胞, 加入漂洗缓冲液(0.15 mol · L-1硫酸铵、100 mmol · L-1磷酸二氢钾、34 mmol · L-1柠檬酸钠和1 mmol · L-1硫酸镁, pH7.0) 漂洗2次。自然风干后, 室温条件下用1%结晶紫染色20 min。小心吸出多余结晶紫, 然后用蒸馏水漂洗10次。自然风干后加入200 μL洗膜缓冲液(乙醇和丙酮, 体积比为4 : 1), 置于摇床上轻微振荡15 min。洗脱液稀释10倍后用分光光度计测定波长570 nm处的吸光值来定量生物膜。
1.2.2 菌株SQR9和SQR9M1生物膜形成24 h时的转录组分析SQR9和SQR9M1静态培养24 h后, 轻轻吸出生物膜下面的培养基, 每个孔里加入1.5 mL无菌水, 再加入1.5 mL预冷的Killing Buffer。将样品从孔中转移到离心管中, 6 000 r · min-1离心3 min后收集菌体, 弃上清液后放入液氮速冻, 保存于-70 ℃。将样品保存于干冰中, 送至国家人类基因组南方中心进行RNA提取、定量和Illumina测序。转录组数据与已知的SQR9全基因组序列进行比对, 统计每个基因中分别来自每个样品的序列数目, 转换成RPM值, 最后利用DEGseq程序包中的MARS(MA-plot-based method with Random Sampling model)模型, 计算每个基因在2组样品中的表达丰度差异, FDR值小于0.001的即被认定为具有显著性差异。
1.2.3 菌株SQR9和SQR9M1胞内铁离子含量的测定菌株SQR9和SQR9M1离心、风干后, 称量1 mg进行分析。加入5 mL硝酸和高氯酸(V(HNO3) : V(HClO4)=87 : 13)。用保鲜膜封住瓶口, 冷消煮(室温静置)至少12 h。按以下的温度梯度进行消煮:85 ℃, 30 min; 100 ℃, 30~60 min; 110 ℃, 30~60 min; 120 ℃, 30~60 min; 140 ℃, 30~60 min; 160 ℃, 30~60 min; 180 ℃, 持续煮, 快煮干时停止。消煮管冷却后加入10 mL 2.5%(体积分数)HNO3, 保鲜膜封住瓶口。用涡旋仪涡旋, 60 ℃水浴30 min, 回溶。静置一段时间, 待样品澄清时倒出上清液。采用等离子耦合光谱仪ICP-MS(Perkin Elmer Optima 2000 DV)测定样品中的铁离子含量。
1.2.4 菌株SQR9和SQR9M1生物膜菌体RNA的提取和反转录用RNA提取试剂盒(Axygen)分别提取SQR9和SQR9M1生物膜的菌体RNA。利用反转录试剂盒(TaKaRa)进行反转录, 得到的cDNA采用核酸定量仪Thermo scientific NANODROP 1000(NanoDrop Technologies, Wilmington, DE, USA)进行定量。
1.2.5 采用Real-time PCR定量分析生物膜和铁转运蛋白的相关基因采用Real-time PCR比较SQR9和SQR9M1在生物膜形成过程中各个基因的表达差异。生物膜和铁转运蛋白相关基因引物见表 1。采用SYBR® Premix Ex TaqTM(Perfect Real Time)试剂盒(宝生物工程有限公司, TaKaRa, 大连), 扩增仪为ABI(Applied BIO systems)PRISM® 7500 Real-time PCR System。反应体系为20 μL, 包括:SYBR® Premix Ex TaqTM(2×)10 μL, Forward Primer(10 μmol · L-1)0.4 μL, Reverse Primer(10 μmol · L-1)0.4 μL, ROX Reference DyeⅡ(50×)0.4 μL, DNA 2 μL, ddH2O 6.8 μL。反应程序:95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 30个循环; 95 ℃ 15 s, 60 ℃ 1 min; 95 ℃ 15 s。每个样品设3个重复, 以无菌水代替模板DNA为阴性对照。实时监测并记录荧光信号的变化。
1.3 数据统计与分析采用Microsoft ExcelTM2010统计软件进行数据统计和绘图。采用SPSS BASE ver.11.5软件进行方差分析, 显著性水平设定P < 0.05。
2 结果与分析 2.1 野生型SQR9和突变体SQR9M1的生物膜形成能力比较突变体SQR9M1在生物膜形成前期有明显的滞后现象, 最大差异发生在培养24 h左右, 因此我们选取培养后24 h进行转录组分析。图 1为突变体SQR9M1和野生型SQR9在MSgg培养基里静置培养24 h的定性和定量结果, 野生型SQR9生物膜的生物量为突变体SQR9M1的3倍以上。
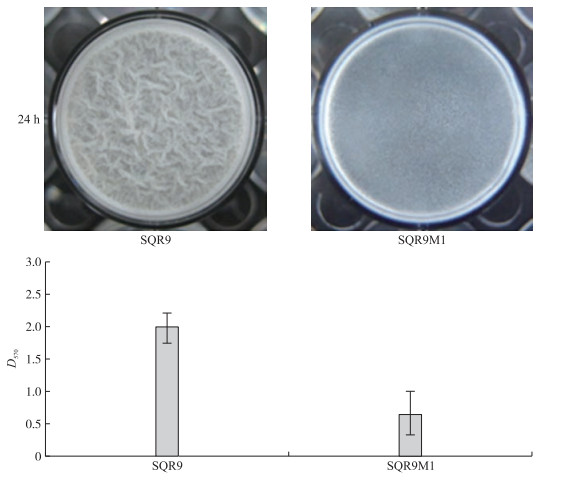
|
图 1 SQR9和突变体SQR9M1生物膜的形成能力 Figure 1 Microtitre plate assay of biofilm formation by wild-type SQR9 and mutant SQR9M1 at 24 h |
野生型SQR9和突变体SQR9M1生物膜菌体的cDNA经高通量测序后产生序列的平均长度约为100 bp, 样品的Reads数目都在1 000万左右, 每个样品的信息总量大约为10亿。本试验设定表达量的差异倍数在2以上即为显著差异。结果显示:共有1 261个基因表达量发生变化, 其中SQR9M1中有574个基因上调(P < 0.01), 687个基因下调(P < 0.01)。散点图更为清晰地展示了所有基因的表达差异情况, 图 2中绿色散点为SQR9M1中表达量升高超过50%的基因; 蓝色散点为2个样品之间差异不显著的基因; 红色散点为SQR9M1中表达量降低超过50%的基因。在上调的574个基因中, 上调33%以下的基因数为459个, 占上调基因总数的80%;在下调的687个基因中, 下调33%以下的基因数为435个, 占下调基因总数的63%。
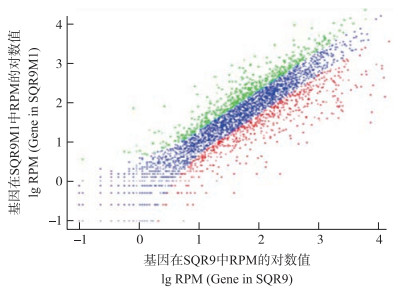
|
图 2 SQR9和SQR9M1生物膜形成中差异基因的散点图 Figure 2 Scatter diagram of gene expression in SQR9 and SQR9M1 RPM:每百万reads中来自某基因的reads数。 RPM:Reads per million reads. |
转录组分析圈图(图 3)体现了SQR9M1中所有基因在SQR9基因组上的连续变化。由图 3可知:SQR9M1在20~21 kb、38~39 kb和40~41 kb发生连续基因表达量上调, 其中包括三羧酸循环(TCA循环)、氨基酸代谢和膜蛋白运输相关基因。SQR9M1在6~7 kb、17~18 kb和25~26 kb发生连续基因表达量的下调, 其中包括次生产物代谢、碳水化合物类物质代谢和趋化与运动相关基因。
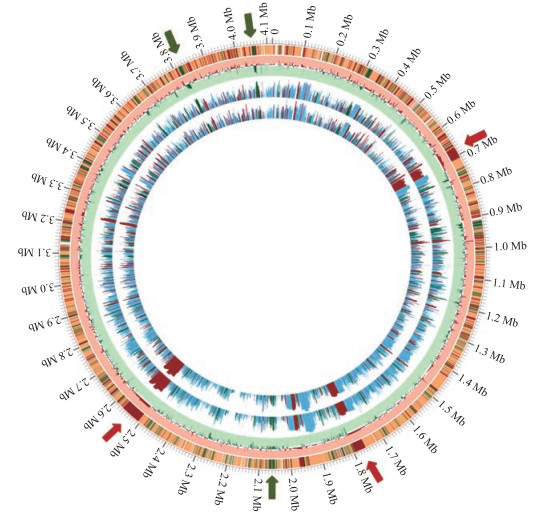
|
图 3 SQR9和SQR9M1生物膜形成中基因表达情况的圈图 Figure 3 Circular map of gene expression profiles between SQR9 and SQR9M1 圈图从外向内分别为:菌株SQR9基因组; 野生型SQR9和突变体SQR9M1中基因的相对表达差异; SQR9M1中的基因表达情况; SQR9中的基因表达情况。其中绿色为明显高表达, 红色为明显低表达。 Starting from the outermost circle going inwards:the outermost circles indicate genes in chromosome strands of SQR9;gene expression profiles SQR9 and SQR9M1;gene expression analysis in SQR9M1;gene expression analysis in SQR9. Up-regulated genes are green and down-regulated genes are red. |
将差异基因按照其功能分成19类, 图 4反映了差异基因在各个功能类别中的分布情况。整体看来, 功能明确的基因约占全部差异基因数量的70%。除去功能未知和猜测功能的基因, 变化最大的基因功能类群是无机离子转运和代谢。这部分基因中, 大量的ABC转运相关基因表达被显著调控, 特别是与铁离子转运相关的ABC转运基因。有9个基因被显著下调, 几乎包含了芽孢杆菌中已报道的所有铁运输相关基因, 其中包括FhuBGC、FeuABC和YfmCDEF这3个铁ABC转运系统。这说明bacillomycin D的突变体在形成生物膜的前期细胞内处于缺铁的状态, 可能与突变体SQR9M1生物膜形成延迟有密切联系。

|
图 4 SQR9和SQR9M1生物膜形成中差异基因的同源蛋白簇(COG)分析 Figure 4 Clusters of orthologous groups of proteins(COG)annotation of genes with significantly different mRNA transcript levels between SQR9 and SQR9M1 |
在信号转导方面, 有20个基因均显著下调, 其中包括一些与生物膜形成相关的基因, 如sinI、kinB和comP, 分别下降到野生型的50%、33%和50%。在次生代谢物合成方面, surfactin和difficidin都显著上调。与生物膜形成相关的胞外多糖EPS和胞外蛋白TasA的合成基因都有不同程度的下调, 但一些脂肽类、聚酮类和萜类化合物合成相关基因都显著上调, 如脂肽类化合物Surfactin的合成基因srfAC、Fengycin的合成基因fenA和fenB, 均上调了2倍, 与萜类物质合成过程相关的基因ispD(4-磷酸胞苷酸转移酶)、ispF(2-甲基赤藻糖醇)和atoB(乙酰基转移酶)都有一定程度的上调。最后一类与生物膜形成相关的基因类群是趋化和运动, 有关鞭毛合成和运动的4个基因都发生了显著下调, 包括fliJ、fliE、fliH、flgD(编码鞭毛各部分组成蛋白)等。
2.4 生物膜形成相关差异基因的Real-time PCR分析分别提取SQR9和SQR9M1在24 h时生物膜细胞的RNA, 进行反转录后, 验证部分铁ABC转运蛋白和与生物膜相关的一些基因。定量PCR结果(图 5)与转录组的结果一致。铁ABC转运蛋白相关的基因feuB、yusV、fhuB和yfmD均下降到野生型的50%, 与生物膜相关的基因sinI、yqxM和epsD分别下调到野生型的33%、12.5%和20%。

|
图 5 SQR9和SQR9M1在24 h时铁离子转运和生物膜相关基因的定量分析 Figure 5 Quantification of the genes related to iron ABC transporter and biofilm formation in SQR9 and SQR9M1 at 24 h |
根据转录组的结果, 我们猜测突变体SQR9M1形成生物膜的前期延迟可能与SQR9M1摄取的铁离子不足有关, 因此测定了SQR9和SQR9M1在实验室条件下培养时, 胞内的铁离子含量。由图 6可知:野生型SQR9在对数生长期时, 胞内铁离子(107细胞)含量迅速增加, 到30 h时能达到247 pmol, 而突变体SQR9M1相对于野生型, 在对数生长期时胞内铁含量增长的数量显著下降, 在30 h时, 胞内铁含量为129 pmol。外源加入用液相收集的bacillomycin D后, 突变体SQR9M1前期摄取铁离子的能力得到恢复。
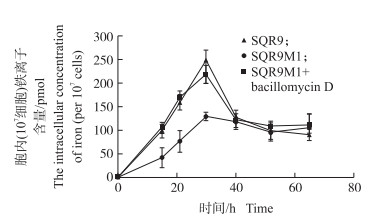
|
图 6 SQR9和SQR9M1胞内铁离子含量 Figure 6 Iron concentration of SQR9 and SQR9M1 |
从图 7可知:外源添加铁离子(终浓度100 μmol · L-1)后能恢复突变体SQR9M1的生物膜表型, 外源添加动物源乳铁蛋白lactoferrin后不能恢复SQR9M1的生物膜表型, 并且显著抑制了野生型SQR9的生物膜形成, 说明胞外铁离子浓度对菌株SQR9的生物膜形成紧密相关。收集24 h时各个处理的生物膜菌体, 提取RNA, 反转录成cDNA后进行定量PCR分析。由图 8可以看出:外源加入铁离子后能恢复SQR9M1生物膜相关基因yqxM、kinC和epsD的表达, 表明抗生素bacillomycin D很有可能参与了铁离子调控SQR9生物膜形成的通路。

|
图 7 添加氯化铁对SQR9和SQR9M1生物膜形成的影响 Figure 7 The effects of adding ferric chloride on biofilm formation of SQR9 and SQR9M1 1) SQR9M1+乳铁蛋白(20 μg · mL-1); 2) SQR9M1+氯化铁(10 μmol · L-1); 3) SQR9+乳铁蛋白(20 μg · mL-1); 4) SQR9+氯化铁(10 μmol · L-1)。 1) SQR9M1+lactoferrin(20 μg · mL-1); 2) SQR9M1+FeCl3(10 μmol · L-1); 3) SQR9+lactoferrin(20 μg · mL-1); 4) SQR9+FeCl3(10 μmol · L-1). |

|
图 8 SQR9M1生物膜相关基因(yqxM、epsD和kinC)相对野生型SQR9的转录分析 Figure 8 Transcriptional levels of yqxM, epsD and kinC in SQR9M1 relative to wild-type SQR9 |
根际有益微生物在植物根际的有效定殖是其发挥功能的重要前提[12]。实验室条件下能形成生物膜的土壤有益细菌, 多数能成功定殖于植物根际, 因此研究根际有益细菌生物膜形成的分子机制是研究其根际定殖机制的重要基础[13]。解淀粉芽孢杆菌SQR9是本实验室研究生物膜形成的模式菌株, 前期研究发现其代谢产物脂肽类抗生素bacillomycin D合成基因bamD突变体在生物膜形成前期显著滞后于野生型, 但其分子机制尚不明确[3]。近年来, 测序技术的发展为研究微生物的转录组和表达谱创造了有利条件[14]。Schembri等[15]研究了大肠杆菌在非生物附着物上形成生物膜后与浮游态细胞的基因转录变化情况, 结果表明整个基因组中9%的基因表达量上调, 4.5%的基因表达量下调, 差异基因主要是与黏附、趋化和胞外蛋白相关的基因。Stanley等[16]研究了枯草芽孢杆菌在静置培养形成生物膜的8、12和24 h的基因表达变化, 结果发现共有519个基因发生了变化, 其中超过55%的基因只在1个时间点里发生了变化。本试验利用转录组技术研究了突变体SQR9M1和野生型SQR9在生物膜形成前期的基因表达变化。结果表明:共1 261个基因发生了变化, 占SQR9基因组的30%, 其中574个基因上调, 687个基因下调, 发生变化2~3倍的基因数占变化基因总数的75%。主要变化的基因类群包括蛋白质翻译与调控、无机离子转运、信号转导、趋化与运动等。
突变体SQR9M1中与信号转导相关的很多基因发生的变化, 主要是两组分调控系统。ComPA是芽孢杆菌中重要的两组分调控系统, 主要调控芽孢杆菌感受态的形成(吸收外源DNA)和一些次生化合物的代谢, 如脂肽类抗生素surfactin和γ-多聚谷氨酸的产生[17]。这些生理活动对芽孢杆菌的生物膜形成具有重要影响。ComPA与另一个两组分调控系统DegSU密切相关, 全局性调控因子DegU的不同磷酸化水平能调控芽孢杆菌细胞从浮游状态向生物膜状态的改变[18]。本试验中degSU的表达并没有显著变化, 可能需要设置更多的时间点才能发现其变化规律。KinB是芽孢杆菌细胞膜上重要的组氨酸激酶, 它可以磷酸化芽孢形成中的重要调控因子, 如spo0A、spo0B、spo0 F等, 进而调控细胞的生物膜形成[19]。本试验中的kinB显著下调, 同时下游的一系列基因epsA-O和yqxM-sipW-tasA也显著下调, 推测可能是由于突变体SQR9M1的组氨酸激酶KinB被抑制, 导致芽孢杆菌生物膜形成的Spo0A通路被抑制, 进而引起SQR9M1生物膜形成前期的延迟。
突变体SQR9M1中与无机离子运输的相关基因显著下调, 其中包含了铁离子ABC转运蛋白相关基因。Ollinger等[20]证实枯草芽孢杆菌共有5个铁离子运输的转运系统, 即YwbMNL、YfmCDEF、FhuBGC、YfiYZ和FeuABC, 存在于SQR9基因组中。本研究中, 突变体SQR9M1中FeuABC、FhuBGC和YfmCDEF相关基因显著下调, 因此我们推测突变体SQR9M1在生物膜形成前期处于铁饥饿的生理状态, ICP-MS的结果验证了这一推测。
自然界中铁离子是生物体的必需元素, 所有的生命体都需要摄取铁来维持生命。细胞内铁离子的主要作用是:1) 各种酶的重要辅因子; 2) 参与细胞内电子传递; 3) 参与RNA和DNA的代谢。近几年, 越来越多的研究发现铁离子参与了微生物的生物膜调控[21]。Johnson等[22]发现在低铁的条件下, 金黄色葡萄球菌的铁ABC转运蛋白基因的缺失显著影响其生物膜的形成, 24 h形成生物膜的生物量仅为野生型的1/4, 说明铁离子ABC转运蛋白正向调控生物膜形成。另外, 铁离子ABC转运蛋白被证实可以正调控胞外黏附蛋白Emp和Eap, 这两个蛋白是参与金黄色葡萄球菌生物膜形成的重要组分[21]。Patriquin等[23]发现铜绿假单胞菌在低铁条件下, 细胞会产生类似抽搐的游动现象, 使其不能形成完整的生物膜。同时发现, 基因敲除铜绿假单胞菌中的铁载体pyoverdine后, 突变体只能形成非常微弱的生物膜, 即使外源加入高浓度的铁也不能恢复[24]。胞内铁离子浓度影响的不仅是生物膜的形成, 还有生物膜的维持, 所以铁离子对微生物生物膜的形成至关重要。本试验中, 外源加入铁离子后能恢复SQR9M1的表型以及生物膜相关基因yqxM、kinC和epsD的表达, 说明脂肽类抗生素bacillomycin D影响SQR9生物膜的形成与铁离子转运, 我们推测抗生素bacillomycin D能影响SQR9铁离子的吸收, 诱发了激酶kinC的表达, 从而经过SinI-Spo0A途径调控了生物膜的形成, 但其具体分子机制还需深入研究。另外, 铁离子与细菌群体感应因子有密切的联系, bacillomycin D能否作为群体感应因子调控SQR9的群体行为将是下一步研究的重点。
| [1] | Chen X H, Scholz R, Borriss M, et al. Difficidin and bacilysin produced by plant-associated Bacillus amyloliquefaciens are efficient in controlling fire blight disease[J]. Journal of Biotechnology, 2009, 140: 38–44. DOI: 10.1016/j.jbiotec.2008.10.015 |
| [2] | Bais H P, Fall R, Vicanco J M. Biocontrol of Bacillus subtilis against infection of Arabidopsis roots by Pseudomonas syringae is facilitated by biofilm formation and surfactin production[J]. Plant Physiol, 2004, 134: 307–319. DOI: 10.1104/pp.103.028712 |
| [3] | Chen Y, Yan F, Liu H X, et al. Biocontrol of tomato wilt disease by Bacillus subtilis isolates from natural environments depends on conserved genes mediating biofilm formation[J]. Environmental Microbiology, 2012, 15(3): 848–864. |
| [4] | Linares J F, Gustafsson I, Baquero F, et al. Antibiotics as intermicrobial signaling agents instead of weapons[J]. Proc Natl Acad Sci USA, 2006, 103: 19484–19489. DOI: 10.1073/pnas.0608949103 |
| [5] | Hoffman L R, D′Argenio D A, MacCoss M J, et al. Aminoglycoside antibiotics induce bacterial biofilm formation[J]. Nature, 2005, 436: 1171–1175. DOI: 10.1038/nature03912 |
| [6] | López D, Fischbach M A, Chu F, et al. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis[J]. Proc Natl Acad Sci USA, 2009, 106: 280–285. DOI: 10.1073/pnas.0810940106 |
| [7] | Xu Z, Shao J, Li B, et al. Contribution of bacillomycin D in Bacillus amyloliquefaciens SQR9 to antifungal activity and biofilm formation[J]. Applied and Environmental Microbiology, 2013, 79: 808–815. DOI: 10.1128/AEM.02645-12 |
| [8] | Cao Y, Zhang Z, Ling N, et al. Bacillus subtilis SQR9 can control Fusarium wilt in cucumber by colonizing plant roots[J]. Biology and Fertility of Soils, 2011, 47: 495–506. DOI: 10.1007/s00374-011-0556-2 |
| [9] | Qiu M, Zhang R, Xue C, et al. Application of bio-organic fertilizer can control Fusarium wilt of cucumber plants by regulating microbial community of rhizosphere soil[J]. Biology and Fertility of Soils, 2012, 48: 807–816. DOI: 10.1007/s00374-012-0675-4 |
| [10] | Branda S S, González-Pastor J E, Ben-Yehuda S, et al. Fruiting body formation by Bacillus subtilis[J]. Proc Natl Acad Sci USA, 2001, 98: 11621–11626. DOI: 10.1073/pnas.191384198 |
| [11] | Hamon M A, Lazazzera B A. The sporulation transcription factor Spo0A is required for biofilm development in Bacillus subtilis[J]. Molecular Microbiology, 2001, 42: 1199–1209. |
| [12] | Bais H P, Park S W, Weir T L, et al. How plants communicate using the underground information superhighway[J]. Trends in Plant Science, 2004, 9(1): 26–32. DOI: 10.1016/j.tplants.2003.11.008 |
| [13] | Zhang N, Wu K, He X, et al. A new bioorganic fertilizer can effectively control banana wilt by strong colonization of Bacillus subtilis N11[J]. Plant Soil, 2011, 344(1/2): 87–97. |
| [14] | 叶文武, 张萌, 曹明娜, 等. 大豆疫霉PsMPK1 沉默突变体的基因表达谱分析[J]. 南京农业大学学报, 2016, 39(3): 386–393. DOI: 10.7685/jnau.201509026 |
| [15] | Schembri M A, Kjærgaard K, Klemm P. Global gene expression in Escherichia coli biofilms[J]. Molecular Microbiology, 2003, 48: 253–267. DOI: 10.1046/j.1365-2958.2003.03432.x |
| [16] | Stanley N R, Britton R A, Grossman A D, et al. Identification of catabolite repression as a physiological regulator of biofilm formation by Bacillus subtilis by use of DNA microarrays[J]. Journal of Biotechnology, 2003, 185: 1951–1957. |
| [17] | Sullivan E R. Molecular genetics of biosurfactant production[J]. Current Opinion in Biotechnology, 1998, 9: 263–269. DOI: 10.1016/S0958-1669(98)80057-8 |
| [18] | Murray E J, Kiley T B, Stanley-Wall N R. A pivotal role for the response regulator DegU in controlling multicellular behaviour[J]. Microbiology, 2009, 155: 1–8. DOI: 10.1099/mic.0.023903-0 |
| [19] | Chastanet A, Vitkup D, Yuan G C, et al. Broadly heterogeneous activation of the master regulator for sporulation in Bacillus subtilis[J]. Proc Natl Acad Sci USA, 2010, 107: 8486–8491. DOI: 10.1073/pnas.1002499107 |
| [20] | Ollinger J, Song K B, Antelmann H, et al. Role of the Fur regulon in iron transport in Bacillus subtilis[J]. Journal of Bacteriology, 2006, 188(10): 3664–3673. DOI: 10.1128/JB.188.10.3664-3673.2006 |
| [21] | Johnson M, Cockayne A, Morrissey J A, et al. Iron-regulated biofilm formation in Staphylococcus aureus Newman requires ica and the secreted protein Emp[J]. Infection and Immunity, 2008, 76: 1756–1765. DOI: 10.1128/IAI.01635-07 |
| [22] | Johnson M, Cockayne A, Williams P H, et al. Iron-responsive regulation of biofilm formation in Staphylococcus aureus involves Fur-dependent and Fur-independent mechanisms[J]. Journal of Biotechnology, 2005, 187: 8211–8215. |
| [23] | Patriquin G M, Banin E, Gilmour C, et al. Influence of quorum sensing and iron on twitching motility and biofilm formation in Pseudomonas aeruginosa[J]. Journal of Biotechnology, 2008, 190: 662–671. |
| [24] | Banin E, Vasil M L, Greenberg E P, et al. Iron and Pseudomonas aeruginosa biofilm formation[J]. Proc Natl Acad Sci USA, 2005, 102: 11076–11081. DOI: 10.1073/pnas.0504266102 |




