文章信息
- 曹沛沛, 毛雅超, 刘涛, 陈发棣, 房伟民, 陈素梅, 蒋甲福
- CAO Peipei, MAO Yachao, LIU Tao, CHEN Fadi, FANG Weimin, CHEN Sumei, JIANG Jiafu
- 菊花Cm14-3-3v基因的克隆及表达分析
- Cloning and expression analysis of Cm14-3-3v gene in chrysanthemum
- 南京农业大学学报, 2017, 40(5): 820-826
- Journal of Nanjing Agricultural University, 2017, 40(5): 820-826.
- http://dx.doi.org/10.7685/jnau.201612048
-
文章历史
- 收稿日期: 2016-12-30
菊花(Chrysanthemum morifolium)是中国十大传统名花之一, 又是世界四大鲜切花之一, 用途和栽培地域极广, 具有很高的观赏和经济价值, 在花卉生产中具有十分重要的地位。在切花菊设施栽培中, 病害、虫害等生物胁迫, 干旱、涝害、热胁迫、寒害和盐胁迫等引起的非生物胁迫是制约菊花生产与园林应用的主要因子, 因此研究与此相关的基因是分子育种的重要基础[1]。
14-3-3蛋白是一种普遍存在于生物体内具有调节作用的酸性二聚体可溶性蛋白, 有多种同源或异源异构体, 具有高度保守性[2]。14-3-3蛋白的分布具有组织特异性, 主要分布于细胞质、质膜、细胞核、线粒体基质、叶绿体基质以及类囊体膜上[3-4]。研究发现14-3-3蛋白调控机制主要由所结合蛋白的磷酸化状态决定, 调控作用很大程度依赖蛋白激酶和蛋白磷酸酶所控制的磷酸化作用, 其亚细胞定位与其蛋白结构上的不同异构型有关, 能够特异性识别磷酸化序列; 通过调节靶蛋白的活性、改变靶标蛋白间的相互作用或亚细胞定位等方式发挥作用[5-6]。本研究根据14-3-3蛋白的保守性及菊花基因组信息克隆并鉴定了1个拟南芥14-3-3GF14υ同源基因Cm14-3-3υ。利用生物信息学技术分析了该基因编码蛋白的理化特性及进化特性, 分析了该基因在不同组织和非生物胁迫下的表达, 为进一步深入研究其响应非生物胁迫等功能奠定了基础。
1 材料与方法 1.1 试验材料供试菊花品种‘神马’由南京农业大学中国菊花品种资源保存中心提供。采集良好的插穗在砂床上扦插生根, 选取长势一致, 约8~10片展开叶的植株种植于底部打孔的一次性塑料杯中, 栽培基质为营养土和蛭石(质量比为1 : 1), 缓苗后于培养室(光照/黑暗时间为16 h/8 h, 温度为22 ℃/18 ℃, 相对湿度为68%~ 75%)培养2周, 用于胁迫处理。
1.2 植物处理方法参照文献[7]的方法, 处理时间有所调整, 从当天的09:00, 选用长势一致的菊花‘神马’幼苗进行非生物胁迫:盐(200 mmol · L-1 NaCl)、干旱(200 g · L-1 PEG6000)、低温(4 ℃)、伤害、50 μmmol · L-1脱落酸(ABA)、1 mmol · L-1茉莉酸甲酯(MeJA)分别处理0、1、3、6、12和24 h。盐胁迫和干旱胁迫的植株洗净基质后进行液体培养, 伤害处理切掉植株的第2片真叶; 植物激素处理:ABA、MeJA喷施在植物叶片上, 对照组喷水。除了盐、干旱、伤害胁迫取根外, ABA、MeJA、低温胁迫取材部位均为叶片。每个处理3个重复。
2016年秋季在大田中选取长势健壮、一致的蕾期菊花‘神马’植株, 取其根、茎、叶、舌状花、管状花, 立即放到液氮中, -80 ℃冰箱保存供实时荧光定量PCR(RT-qPCR)检测使用。
1.3 总RNA的提取和cDNA的合成采用Trizol(TaKaRa, Tokyo, Japan)法提取菊花材料的总RNA, 经检测合格后利用PrimeScript TM RT-PCR Kit反转录试剂盒(TaKaRa)合成第1链cDNA, -20 ℃保存备用。
1.3.1 菊花‘神马’Cm14-3-3υ基因的克隆及表达载体的构建根据菊花转录组库(SRP076366) 查找到Cm14-3-3υ DNA全长序列, 利用Primer Premier 5.0对所得到的序列进行RT-PCR扩增引物的设计。引物见表 1。利用高保真酶Ex Taq(TaKaRa)进行PCR扩增。PCR程序为:94 ℃ 2 min; 94 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 30 s, 共30个循环; 72 ℃ 7 min。以10 g · L-1琼脂糖凝胶电泳进行扩增产物的检测。当扩增产物与序列cDNA大小相同时, 使用Gel Extraction Kit纯化回收PCR扩增产物, 进行克隆并送南京思普金生物科技有限公司测序。采用Gateway技术进行载体构建, 使用pENTR1A作为入门载体, 将Cm14-3-3υ ORF产物用EcoRⅠ、BamHⅠ(TaKaRa)双酶切后与pENTR1A连接, 构建pENTR1A-Cm14-3-3υ入门载体。pENTR1A-Cm14-3-3υ质粒经过PvuⅠ单酶切线性化后与pMDC43载体进行LR重组并构建pMDC43-Cm14-3-3υ表达载体[8]。以洋葱表皮活体细胞作为亚细胞材料, 通过基因枪轰击进行瞬时转化, 35 S : : GFP质粒为阳性对照。
| 引物 Primer | 序列(5′→3′) Sequence | 用途 Usage |
| Cm14-3-3υ-QC-F/R | ATGACAAGAAAGAGGCAGCTGAT/GCAGCAGTAGAGGCCAATTGA | Cm14-3-3υ全长扩增 Amplication for full length of Cm14-3-3υ |
| Cm14-3-3υ-RTP-F/R | AACTCGCCTGACCGTGCTT/TTGATCTCATCCCCTGCCTC | Cm14-3-3υ荧光定量 PCRRT-qPCR primer of Cm14-3-3υ |
| ACTIN-P-F/R | CCATTCAAGCGACAGACTCA/TTTTGGTATCTGGTCCTGGAG | ACTIN定量扩增 Amplication for ACTIN |
对各处理样品的cDNA模板进行Cm14-3-3υ基因表达量的测定。RT-qPCR体系按照SYBR Premix Ex TaqTM(TaKaRa)操作指南进行。荧光定量使用Master cycler ep realplex 2S(Eppendorf)。使用Real Time PCR荧光定量仪进行相对实时荧光定量PCR。扩增反应体系为20 μL : 10 μL SYBR ® Premix Ex TaqTM Ⅱ, 5 μL(50 ng · L-1)cDNA模板, 正、反引物各1 μL, 3 μL ddH2O。反应条件为:95 ℃ 2 min; 95 ℃ 15 s, 55 ℃ 15 s, 72 ℃ 20 s, 40个循环。以菊花ACTIN作为内参基因, 每个样品RT-qPCR采用3次重复, 采用2-ΔΔCT算法分析各基因的表达量。
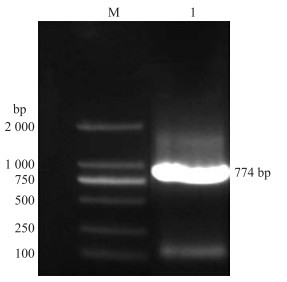
2 结果与分析 2.1 菊花‘神马’Cm14-3-3υ基因的克隆及编码蛋白的氨基酸序列分析通过转录组库对Cm14-3-3υ进行搜索, 得到Cm14-3-3υ的全长序列, 根据序列设计特异引物。以‘神马’cDNA为模板, 利用高保真酶Ex Taq进行PCR扩增。PCR产物经纯化加尾, 连接到pMD19-T上并转入大肠杆菌DH5α测序。测序结果利用BioXM 2.6软件比对分析可知与转录组的序列一致, 获得最大阅读框774 bp(图 1), 编码257个氨基酸。菊花14-3-3υ蛋白的相对分子质量为29.15×103, 理论等电点(pI)为4.70。根据Predict Protein和NetPhos 2.0 Server在线软件预测, 可知该蛋白定位于细胞质和细胞核, 含有33个潜在磷酸化位点, 即19个Ser磷酸化位点、7个Thr磷酸化位点和7个Tyr磷酸化位点(图 2); N-端含有5个蛋白结合区氨基酸残基位点, 分别为第1、20、40、45和78位(图 3)。
|
图 1 Cm14-3-3υ基因的PCR产物 Figure 1 The PCR product of Cm14-3-3υ gene M.DL2000 marker; 1. Cm14-3-3υ全长产物 The production full length of Cm14-3-3υ. |

|
图 2 Cm14-3-3υ蛋白的潜在磷酸化位点预测 Figure 2 The predicted potential phosphorylation sites of Cm14-3-3υ protein |

|
图 3 Cm14-3-3υ蛋白的结合位点预测 Figure 3 The predicted binding sites of Cm14-3-3υ protein |
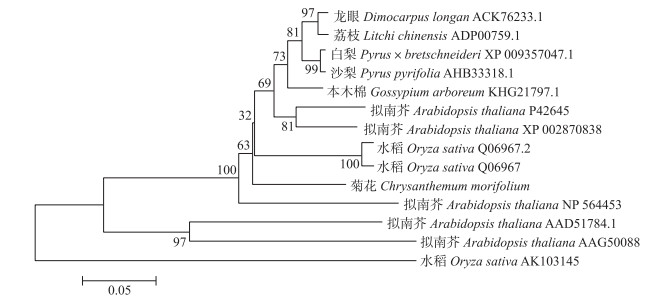
使用NCBI的BlASTx程序, 比对菊花Cm14-3-3υ氨基酸序列, 筛选出多个14-3-3υ同源蛋白。由图 4可见:龙眼(Dimocarpus longan)、荔枝(Litchi chinensis)、沙梨(Pyrus pyrifolia)、白梨(Pyrus×bretschneideri)、本木棉(Gossypium arboreum)、拟南芥(Arabidopsis thaliana)GF14ψ、拟南芥GF14υ、水稻(Oryza sativa)GF14a、水稻GF14f与Cm14-3-3υ蛋白的氨基酸序列相似性分别为87.36%、86.97%、86.59%、86.26%、87.79%、85.27%、82.90%、84.62%、84.62%, 表明从菊花中克隆得到的Cm14-3-3υ基因所编码的蛋白与其他物种的14-3-3蛋白具有高度的保守性。使用DNAMAN 6.0软件中的多重比对功能, 对菊花Cm14-3-3υ蛋白的氨基酸序列与其他11个物种的氨基酸序列进行多序列比对, 结果显示, 不同物种间该蛋白的中间区域相对保守, N-端和C-端差异相对较大(图 5)。
|
图 4 菊花Cm14-3-3υ与其他植物的氨基酸序列进化分析 Figure 4 Phylogenetic analysis of amino acid sequences encoded by Cm14-3-3υ and other plants |

|
图 5 Cm14-3-3υ蛋白与其他植物蛋白的氨基酸序列比对 Figure 5 Amino acid comparison of Cm14-3-3υ protein with those from other plants |
为了进一步明确Cm14-3-3υ功能, 对其在亚细胞水平上的分布进行了分析, 结果表明, Cm14-3-3υ定位在细胞核和细胞质中, 这与35S : : GFP的空载体的结果没有差别(图 6)。

|
图 6 Cm14-3-3υ蛋白的亚细胞定位 Figure 6 Subcellular localization of Cm14-3-3υ protein |
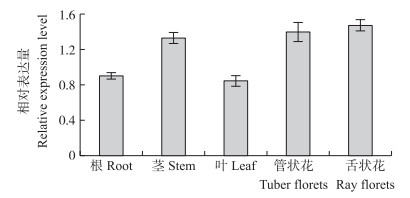
实时定量PCR分析Cm14-3-3υ基因在不同组织中的表达, 结果表明Cm14-3-3υ基因在根、茎、叶、舌状花、管状花中均有表达, 根和叶中表达量相对较少, 茎、管状花和舌状花中的表达量较高, 管状花和舌状花的表达量分别是叶片的1.56和1.63倍(图 7)。
|
图 7 菊花‘神马’Cm14-3-3υ在不同组织中的表达量 Figure 7 The expression level of Cm14-3-3υ in different tissues of'Jimba' |
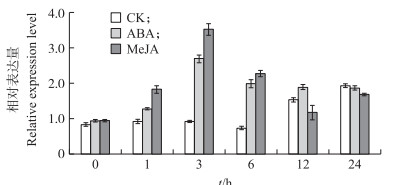
由图 8可见:Cm14-3-3υ表达均受脱落酸(ABA)和茉莉酸甲酯(MeJA)的诱导。在ABA和MeJA处理0~3 h时, Cm14-3-3υ基因表达量逐渐升高, 3 h时达到最高值, 分别是对照的3.80倍和2.97倍。ABA处理下, Cm14-3-3υ表达量在6~24 h时保持相对稳定, 且在6和12 h高于对照。MeJA处理下, Cm14-3-3υ表达量在12和24 h低于对照, 说明Cm14-3-3υ受MeJA早期诱导表达。
|
图 8 不同外源激素处理对菊花‘神马’Cm14-3-3υ基因表达量的影响 Figure 8 Effect of different exogenous hormones treatments on expression level of Cm14-3-3υ in'Jimba' |
由图 9可见:盐胁迫条件下, Cm14-3-3υ基因在胁迫开始1 h后明显升高, 3~24 h基因表达量有不同程度升高, 在3 h表达量达到最高值, 结果显示盐胁迫诱导Cm14-3-3υ基因的表达, 约为对照的1.8倍; 干旱和伤害胁迫下, 与对照相比表达变化没有显著差异, 表明Cm14-3-3υ基因不受此胁迫的诱导。在低温胁迫条件下, Cm14-3-3υ在0~1 h升高了1.6倍; 1~3 h表达量稳定在一个较高的水平; 6 h时由于受到周期节律影响, 对照和低温胁迫下表达量都下降且胁迫条件下下降更明显; 在24 h达到最大值, 为对照的6倍, 表明基因受低温胁迫显著诱导(图 10)。
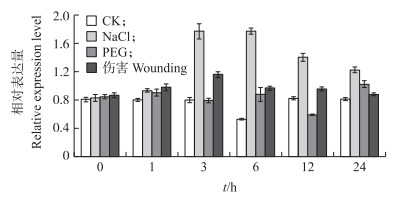
|
图 9 非生物胁迫对菊花‘神马’Cm14-3-3υ表达量的影响 Figure 9 Effect of abiotic stress on expression level of Cm14-3-3υ in'Jimba' |
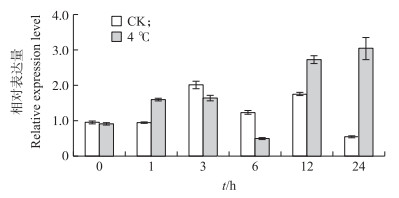
|
图 10 低温胁迫对菊花‘神马’Cm14-3-3υ表达量的影响 Figure 10 Effect of cold stress on expression level of Cm14-3-3υ in'Jimba' |
本研究从菊花‘神马’克隆了Cm14-3-3υ基因全长, 预测显示其有多个潜在的磷酸化位点, 并在N-端有5个蛋白结合区, 亚细胞定位结果显示该蛋白定位在细胞质与细胞核中, 这可能与其参与不同的生理生化代谢途径有关。
14-3-3蛋白参与植物生长发育、物质能量代谢、细胞周期、光与激素的信号传导、花期调控等许多重要的生理过程[2]。模式植物拟南芥中的GF 14ω、GF14χ、GF14Ф、GF14ψ、GF14υ、GF14ε、GF14k、GF14λ、GF14μ等基因已被克隆研究, 它们编码的产物可控制细胞的多种过程。如GF14μ、GF14υ与植物叶绿体的发育和根生长发育有关系, 拟南芥14-3-3υ突变体使根系变短[9]; GF14ω与Ca2+结合, 被质膜结合激酶磷酸化, 实现调控植物细胞周期的功能[10]。研究表明拟南芥14-3-3蛋白基因RCI1(AtGF14φ)和RCI2(AtGF14λ)受寒冷胁迫诱导[11]; 14-3-3蛋白可以与ABA信号转导途径中的AREB/ABF/ABI5-like和H+-ATPase相互作用, 而ABA信号转导途径与干旱、胁迫、温度相联系[12-13]。拟南芥14-3-3λ蛋白基因转入棉花体内可增强转基因棉花的抗旱性[14]; 胁迫刺激可以激活14-3-3蛋白自身磷酸化; 大麦在抵御白粉病菌的过程中, 14-3-3(pAS1-CYH/ 14-3-3a)蛋白的累积量增加[15]。水稻OsGFl4b和OsGFl4g几乎可被所有的胁迫和激素诱导[16]; 小麦(Triticum aestivum)Ta14R1和Ta14R2 基因均受ABA诱导呈现上调表达, 通过依赖ABA非生物胁迫响应途径发挥作用; 机械伤害或壳聚糖或MeJA胁迫下, 白云杉(Picea glauca)和杂交白杨(Populus tomentosa Carr.)中14-3-3EB9D的转录水平增强[17-18]。研究表明, 冷胁迫会导致油菜14-3-3基因与H+-ATPase基因表达量同时增加[19]。14-3-3基因参与植物的开花途径, 水稻中 14-3-3b作为Hd3a-OsFD1蛋白互作的桥梁和纽带, 14-3-3b蛋白一端与OsFD1互作, 一端与Hd3a互作, 调节OsHd3a与OsFD1互作, 调控水稻抽穗[20]。拟南芥中 14-3-3κ与PIF3互作激活光形态建成, 在响应光信号时 14-3-3κ与PHOT1、PHOT2及光周期途径中的开花调控因子CO互作, 调控下游基因FT、TFL表达[21]。长日照条件下, 14-3-3υ也受红光和远红光的影响, 调控植物根的生长, 突变体会导致植株晚花[22-23]。本研究表明, Cm14-3-3υ基因在菊花不同组织中均有表达, 在茎、舌状花、管状花中表达量较高, 该基因可能参与了植物的不同生长发育调控路径; Cm14-3-3υ基因还受ABA、MeJA、盐、冷胁迫的诱导表达, 在以上胁迫条件下表达量明显上调, 暗示了Cm14-3-3υ不仅参与开花和周期调控, 可能也参与胁迫响应过程, 但还需进一步证明。本研究为下一步的功能研究奠定了基础, 为进一步培育菊花抗逆新种质, 提供了候选基因。
| [1] | 成丽娜, 魏倩, ImtiazM, 等. 转基因育种技术在菊花性状改良中的应用进展[J]. 园艺学报, 2013, 40(9): 1813–1825. |
| [2] | Sehnke P C, Delille J M, Ferl R J. Consummating signal transduction:the role of 14-3-3 proteins in the completion of signal-induced transitions in protein activity[J]. Plant Cell, 2002, 14(81): S339–S354. |
| [3] | Chaudhri M, Scarabel M, Aitken A. Mammalian and yeast 14-3-3 isoforms form distinct patterns of dimers in vivo[J]. Biochemicaland Biophysical Research Communications, 2003, 300(3): 679–685. DOI: 10.1016/S0006-291X(02)02902-9 |
| [4] | Bunney T D, van Walraven H S, de Boer A H. 14-3-3 protein is a regulator of the mitochondrial and chloroplast ATP synthase[J]. Proc Natl Acad Sci USA, 2001, 98(7): 42–49. |
| [5] | Sehnke P C, Henry R, Cline K, et al. Interaction of a plant 14-3-3 protein with the signal peptide of a thylakoid-targeted chloroplast precursor protein and the presence of 14-3-3 isoforms in the chloroplast stroma[J]. Plant Physiology, 2000, 122(1): 235–242. DOI: 10.1104/pp.122.1.235 |
| [6] | Chen F, Li Q, Sun L, et al. The rice 14-3-3 gene family and its involvement in responses to biotic and abiotic stress[J]. DNA Res, 2006, 13(2): 53–63. DOI: 10.1093/dnares/dsl001 |
| [7] | Song A, Li P, Jiang J, et al. Phylogenetic and transcription analysis of chrysanthemum WRKY transcription factors[J]. International Journal of Molecular Sciences, 2014, 15(8): 14442–14455. DOI: 10.3390/ijms150814442 |
| [8] | Curtis M D, Grossniklaus U. A gateway cloning vector set for high-throughput functional analysis of genes in planta[J]. Plant Physiology, 2003, 133(2): 462–469. DOI: 10.1104/pp.103.027979 |
| [9] | Agrawal V D, Agrawal P. The 14-3-3 proteins of Arabidopsis regulate root growth and chloroplast development as components of the photosensory system[J]. Journal of Experimental Botany, 2012, 63(8): 3061–3070. DOI: 10.1093/jxb/ers022 |
| [10] | Fulgosi H, Soll J, Maraschin S D F, et al. 14-3-3 proteins and plant development[J]. Plant Molecular Biology, 2002, 50(6): 1019–1029. DOI: 10.1023/A:1021295604109 |
| [11] | Jarillo J A, Capel J, Leyva A, et al. Two related low-temperature-inducible genes of Arabidopsis encode proteins showing high homology to 14-3-3 proteins, a family of putative kinase regulators[J]. Plant Molecular Biology, 1994, 25(4): 693–704. DOI: 10.1007/BF00029607 |
| [12] | Merlot S, Leonhardt N, Fenzi F, et al. Constitutive activation of a plasma membrane H+-ATPase prevents abscisic acid-mediated stomatal closure[J]. Embo Journal, 2007, 26(13): 3216–3226. DOI: 10.1038/sj.emboj.7601750 |
| [13] | Schoonheim P J, Sinnige M P, Casaretto J A, et al. 14-3-3 adaptor proteins are intermediates in ABA signal transduction during barley seed germination[J]. Plant Journal, 2007, 49(2): 289–301. DOI: 10.1111/j.1365-313X.2006.02955.x |
| [14] | Gökirmak T, Paul A L, Ferl R J. Plant phosphopeptide-binding proteins as signaling mediators[J]. Current Opinion in Plant Biology, 2007, 13(5): 527–532. |
| [15] | Finni C, Andersen C H, Borch J, et al. Do 14-3-3 proteins and plasma membrane H+-ATPases interact in the barley epidermis in response to the barley powdery mildew fungus?[J]. Plant Molecular Biology, 2002, 49(2): 137–147. DOI: 10.1023/A:1014938417267 |
| [16] | Rosenquist M, Alsterfjord M, Larsson C, et al. Data mining the Arabidopsis genome reveals fifteen 14-3-3 genes. Expression is demonstrated for two out of five novel genes[J]. Plant Physiology, 2001, 127(1): 142–149. DOI: 10.1104/pp.127.1.142 |
| [17] | Lapointe G, Luckevich M D, Séguin A. Investigation on the induction of 14-3-3 in white spruce[J]. Plant Cell Reports, 2001, 20(1): 79–84. DOI: 10.1007/s002990000275 |
| [18] | Lapointe G, Luckevich M D, Cloutier M, et al. 14-3-3 gene family in hybrid poplar and its involvement in tree defence against pathogens[J]. Journal of Experimental Botany, 2001, 52(359): 1331–1338. |
| [19] | Seehaus K, Tenhaken R. Cloning of genes by mRNA differential display induced during the hypersensitive reaction of soybean after inoculation with Pseudomonas syringae pv. glycinea[J]. Plant Molecular Biology, 1998, 38(6): 1225–1234. DOI: 10.1023/A:1006036827841 |
| [20] | Tsuji H, Nakamura H, Taoka K, et al. Functional diversification of FD transcription factors in rice, components of florigen activation complexes[J]. Plant and Cell Physiology, 2013, 54(3): 385–397. DOI: 10.1093/pcp/pct005 |
| [21] | Kim H S, Oh J M, Luan S, et al. Cold stress causes rapid but differential changes in properties of plasma membrane H(+)-ATPase of camelina and rapeseed[J]. Journal of Plant Physiology, 2013, 170(9): 828–837. DOI: 10.1016/j.jplph.2013.01.007 |
| [22] | Mayfield J D, Folta K M, Paul A L, et al. The 14-3-3 proteins μ and υ influence transition to flowering and early phytochrome response[J]. Plant Physiology, 2007, 145(4): 1692–1702. DOI: 10.1104/pp.107.108654 |
| [23] | Adams E, Diaz C, Hong J P, et al. 14-3-3 proteins participate in light signaling through association with PHYTOCHROME INTERACTING FACTORs[J]. International Journal of Molecular Sciences, 2014, 15(12): 22801–22814. DOI: 10.3390/ijms151222801 |




