文章信息
- 王彤, 刘静, 郭月, 袁娜, 杜建厂
- WANG Tong, LIU Jing, GUO Yue, YUAN Na, DU Jianchang
- 基于matK基因和ITS序列的江苏地方豆类植物的亲缘关系研究
- Phylogenetic relationship among local legumes in Jiangsu Province based on analyses of matK gene and ITS sequence
- 南京农业大学学报, 2017, 40(5): 795-803
- Journal of Nanjing Agricultural University, 2017, 40(5): 795-803.
- http://dx.doi.org/10.7685/jnau.201612040
-
文章历史
- 收稿日期: 2016-12-26
2. 江苏省农业科学院农业生物技术研究所/江苏省农业生物学重点实验室, 江苏 南京 210014
2. Institute of Biotechnology/Jiangsu Key Laboratory of Agrobiology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China
豆类植物泛指能够产生豆荚的豆科作物, 在植物学分类上属于豆科(Leguminosae)中的蝶形花亚科(Papilionaceae)。豆类植物富含蛋白质、维生素、钙、磷、铁、钾、镁等, 具有很高的营养价值。目前, 大豆(Glycine max)[1]、菜豆(Phaseolus vulgaris)[2]、绿豆(Vigna radiata)[3]、百脉根(Lotus corniculatus)[4]、木豆(Cajanus cajan)[5]、野生大豆(Glycine soja)[6]、蒺藜苜蓿(Medicago truncatula)[7]、鹰嘴豆(Cicer arietinum)[8]和红小豆(Vigna angularis)[9]等植物已经先后完成了全基因组测序, 为豆类基因组学和遗传育种方面的深入研究提供了数据来源。
叶绿体基因组由于具有进化速率慢, 不存在基因重组和结构变异等特点, 为遗传进化的研究提供了便利条件[10]。叶绿体matK基因为长度1 500 bp的单拷贝编码基因, 位于叶绿体赖氨酸trnK基因的内含子中[11], 是叶绿体基因组蛋白编码基因中进化速率最快的基因之一[12]。matK基因的3′端相对保守, 而5′端变异较大, 但均可作为2个特征片段在不同分类水平上进行物种的分类[11]。2009年, matK基因被确定为DNA条形码, 并广泛应用于科内、属间的系统发育研究[13]。例如用于薯蓣科植物[14]、含羞草科金合欢属[15]、苔藓植物[16]以及伞形科和五加科[12]之间的亲缘关系分析等。
18S rRNA、5.8S rRNA和26S rRNA以串联重复的方式构成了高等植物的核糖体RNA。内转录间隔区(internal transcribed spacer, ITS区)包括ITS1(位于18S rRNA和5.8S rRNA之间)、ITS2(位于5.8S rRNA和26S rRNA之间)和介于它们之间长度非常保守的5.8S rRNA[17]。由于ITS序列两端的18S rRNA和26S rRNA相对保守, 这就为设计ITS序列的通用引物提供了可能。事实上, ITS序列现已广泛用于植物近缘属间、属内以及种内的多样性研究。例如, ITS序列现已作为目的序列成功用于豆科植物大豆属[18]、豇豆属[19]、虫草[20]等的亲缘关系的鉴定。此外, ITS序列在分析药用植物亲缘关系方面也具有很高的可靠性[21-22]。
近年来, 江苏省选育了许多地方豆类品种, 但是这些品种大多由不同的科研单位在不同的年代选育而成, 而且命名缺乏规范性, 最终导致各个地方品种之间亲缘关系不明确。本研究以14个江苏省选育的地方豆类品种为研究对象, 结合GenBank中大豆、菜豆、红小豆、绿豆和百脉根5种豆科植物的基因组信息, 通过对各地方品种matK基因和ITS序列的测定、序列比对、遗传参数计算, 以及构建系统进化树等, 研究不同物种(品种或品系)之间的分化时间和亲缘关系, 旨在为后续豆类植物的种质资源创新和利用, 以及豆类植物品种选育和遗传改良提供理论依据。
1 材料与方法 1.1 试验材料供试材料为14个地方豆类品种:2个大豆品种(‘Williams 82’‘通豆8号’), 4个红小豆品种(‘苏红豆1号’‘墨玉小豆’‘玛瑙小豆’‘黑金刚小豆’), 1个赤小豆品种(‘苏长粒饭豆’), 1个绿豆品种(‘通绿7号’), 6个菜豆品种(‘大白芸豆’‘苏水白饭豆’‘黑珍珠蚕豆’‘鞍纹大芸豆’‘奶花芸豆’‘红宝石芸豆’), 以及来源于GenBank中的5种豆科植物:大豆、红小豆、绿豆、菜豆、百脉根, 其matK基因序列号分别为:DQ317523.1、JN008218.1、DQ445950.1、NC_009259.1、NC_002694.1;ITS序列号分别为:FJ609734.1、JF430407.1、JX233505.1、JQ974390.1、KM372660.1。
1.2 基因组DNA的提取取14种豆类植物的新鲜叶片适量, 用植物基因组DNA提取试剂盒(TIANGEN)提取总DNA。取3 μL总DNA样品用12 g · L-1的琼脂糖凝胶电泳检测DNA提取效果, 剩余的保存于4 ℃冰箱备用。
1.3 matK基因和ITS序列的PCR扩增、纯化用于扩增14种豆类材料matK基因片段的引物为: 3F_KIM:5′-CGTACAGTACTTTTGTGTTTACGAG-3′, 3R_KIM:5′-ACCCAGTCCATCTGGAAATCTTGGTTC-3′[14]。PCR总体系为50 μL:10×PCR缓冲液5 μL, 0.025 mol · L-1 MgCl2 6 μL, 0.002 5 mol · L-1 dNTP mixture 4 μL, 0.01 mol · L-1引物各2 μL, 50 μg · μL-1模板DNA 1 μL, 5 U · μL-1 Taq酶0.25 μL, 加ddH2O至50 μL。反应程序:94 ℃ 3 min; 94 ℃ 45 s, 55 ℃ 30 s, 72 ℃ 1.5 min, 35个循环; 72 ℃ 10 min, 16 ℃保存。用于扩增ITS序列的引物为AB101:5′-ACGAATTCATGGTCCGGTGAAGTGTTCG-3′, AB102:5′-TAGAATTCCCCGGTTCGCTCGCCGTTAC-3′[23]。扩增ITS序列的PCR反应总体系与扩增matK基因的总体系相同。反应程序:95 ℃ 8 min; 94 ℃ 45 s, 62 ℃ 45 s, 72 ℃ 1 min, 30个循环; 72 ℃ 6 min, 16 ℃保存。经过12 g · L-1琼脂糖凝胶电泳检测PCR产物。检测合格后用Promega PCR产物纯化试剂盒进行纯化, 送南京金斯瑞生物科技有限公司测序。
1.4 序列分析和系统进化树的构建测序获得的DNA序列用SeqMan进行拼接, 用MEGA 6.0对18种豆类材料的matK基因序列和ITS序列进行比对后裁齐, 分析变异位点和信息位点, 并选择Maximum Composite Likelihood核酸距离模式, 计算遗传距离。以‘Williams 82’作为参考序列, 用BioEdit显示序列比对信息。以百脉根为外类群, 采用Neighbor-joining法构建分子进化树, 并用Maximum Likelihood法检验分类结果的准确性, 以1 000次bootstrap检测各分支的置信度。
1.5 分化时间计算利用matK基因并参照Yang等[9]的红小豆基因组分析结果, 用MEGA 6.0计算分化时间。将红小豆与菜豆的分歧点设置Calibration Name为name 1, MRCA Node Label设置为label 1, Min Divergence Time和Max Divergence Time分别为5.60和15.0[9]。将红小豆与大豆的分歧点设置Calibration Name为name 2, MRCA Node Label设置为label 2, Min Divergence Time和Max Divergence Time分别为16.9和29.0[9], 其余参数为MEGA 6.0默认值, 估算分化时间。
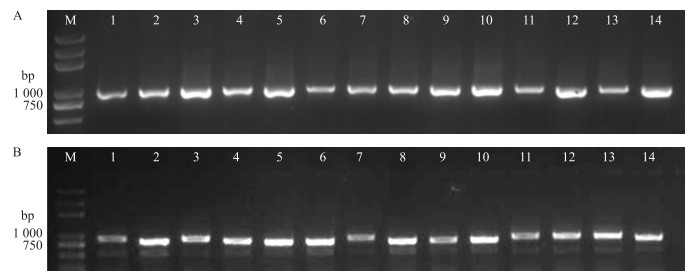
2 结果与分析 2.1 豆类植物的序列分析由图 1可见:PCR产物纯化后均得到清晰的目的片段。用SeqMan软件对测序所得序列进行拼接并进行人工校对, 并与GenBank中获得的5种其他豆类植物的同源序列进行比对。结果显示, matK基因序列长度为750 bp, ITS序列长度为795 bp。将18种豆类植物的matK基因序列和ITS序列分别用‘Williams 82’作为参考序列, 并导入BioEdit显示序列比对的变异位点(图 2和图 3)。
|
图 1 14种豆类植物matK基因序列(A)和ITS序列(B)的PCR扩增结果 Figure 1 PCR amplification of matK gene sequence(A) and ITS sequence(B) in 14 legumes M:DL5000 marker; 1:‘Williams 82’; 2:‘通豆8号’; 3:‘苏红豆1号’; 4:‘墨玉小豆’; 5:‘玛瑙小豆’; 6:‘黑金刚小豆’; 7:‘苏长粒饭豆’; 8:‘通绿7号’; 9:‘大白芸豆’; 10:‘苏水白饭豆’; 11:‘黑珍珠蚕豆’; 12:‘鞍纹大芸豆’; 13:‘奶花芸豆’; 14:‘红宝石芸豆’。 M:DL5000 marker; 1:'Williams 82';2:'Tongdou 8';3:'Suhongdou 1';4:'Moyuxiaodou'; 5:'Manaoxiaodou'; 6:'Heijingangxiaodou'; 7:'Suchanglifandou'; 8:'Tonglü 7';9:'Dabaiyundou'; 10:'Sushuibaifandou'; 11:'Heizhenzhucandou'; 12:'Anwen-dayundou'; 13:'Naihuayundou'; 14:'Hongbaoshiyundou'. The same as follows. |

|
图 2 18种豆类植物matK基因变异位点
Figure 2 Variable sites of matK gene in 18 legumes
15:大豆Glycine max(DQ317523.1);16:红小豆Vigna angularis(JN008218.1); 17:绿豆Vigna radiata(DQ445950.1);18:菜豆Phaseolus vulgaris(NC_009259.1). |

|
图 3 18种豆类植物ITS序列变异位点
Figure 3 Variable sites of ITS sequence in 18 legumes
15:大豆Glycine max(FJ609734.1);16:红小豆Vigna angularis(JF430407.1); 17:绿豆Vigna radiata(JX233505.1);18:菜豆Phaseolus vulgaris(JQ974390.1). |
利用MEGA 6.0统计18种豆类植物matK基因序列的变异位点数为85, 信息位点数为82。3个大豆品种之间和5个红小豆品种之间的matK基因序列完全一致; 2个绿豆品种之间存在1个碱基的INDEL; ‘苏长粒饭豆’与5个红小豆品种之间仅存在3个SNP位点, 小于其与菜豆、绿豆的差异。ITS序列的变异位点数为260, 信息位点数为242。3个大豆品种之间和5个红小豆品种之间的ITS序列也完全一致; 2个绿豆品种之间的ITS存在3个SNP和11个碱基的INDEL; ‘苏长粒饭豆’与5个红小豆品种之间仅存在14个SNP位点, 也小于其与菜豆、绿豆的差异。2条序列的比对结果显示, ‘苏长粒饭豆’与红小豆之间的亲缘关系比其与菜豆、绿豆之间的亲缘关系更近。
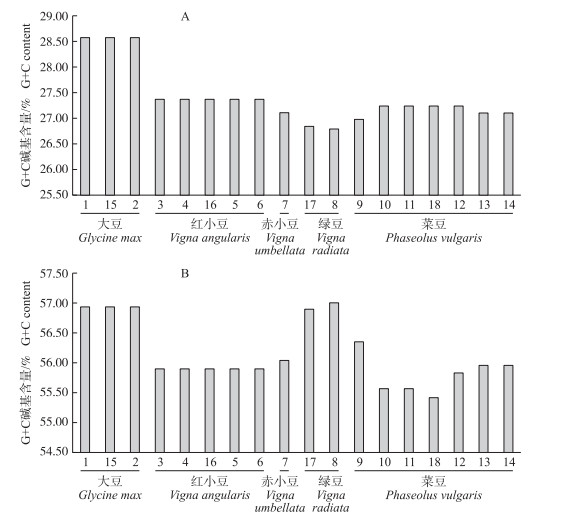
matK基因片段和ITS序列G+C含量分析结果(图 4)显示:matK基因的扩增片段中4种碱基的比例存在较大差异, A、C、G、T的平均含量为32.08%、14.28%、13.14%、40.50%, 其中胞嘧啶(C)和鸟嘌呤(G)的含量较低, 均小于14.69%, G+C的含量明显低于A+T的含量。由图 4-A可以看出:matK基因序列G+C含量最高的是3个大豆品种, 菜豆和红小豆的G+C含量差别不大, 最低的是2个绿豆品种。而ITS序列中A、C、G、T的平均含量为25.46%、28.48%、27.67%、18.38%, 其中胞嘧啶(C)和鸟嘌呤(G)的含量较高, 均大于27.00%, G+C含量较高, 均大于55.41%, G+C含量与A+T含量差别不大。由图 4-B可以看出:ITS序列G+C含量比较高的是3个大豆品种和2个绿豆品种, 菜豆的G+C含量相对比较低。总之, ITS序列的G+C含量明显高于matK基因序列的G+C含量。
|
图 4 matK基因序列(A)和ITS序列(B)G+C碱基含量 Figure 4 The G+C content of matK gene sequence(A) and ITS sequence(B) |
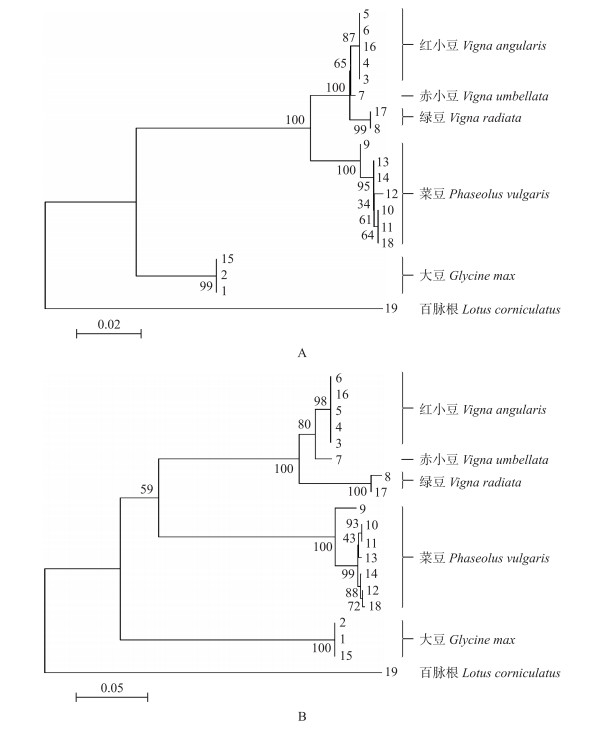
matK基因序列遗传距离计算结果显示, 18种豆类植物的遗传距离为0~0.099。属内遗传距离较小, 均小于0.009。属间遗传距离较大, 其中大豆与菜豆、大豆与红小豆、大豆与绿豆的遗传距离较大, 均大于0.093;绿豆与红小豆的遗传距离较小, 均小于0.009。ITS序列结果表明, 18种豆类植物的遗传距离为0~0.292。属内遗传距离较小, 均小于0.036。属间遗传距离较大, 其中大豆与菜豆、大豆与红小豆、大豆与绿豆的遗传距离较大, 均大于0.270;绿豆与红小豆的遗传距离较小, 均小于0.072。本研究分别用matK基因序列和ITS序列, 采用Neighbor-joining法构建系统进化树, 用百脉根作为外类群(图 5), 并用Maximum Likelihood法验证分类结果的准确性。结果表明:2种序列用上述2种方法构建的进化树关系基本一致, 18种豆科植物可分为3组, 红小豆和绿豆分为一组, 菜豆和大豆分别聚为一组, 这与遗传距离分析结果一致。其中, 红小豆分支和绿豆分支亲缘关系最近, 表明它们可能来自于同一个共同祖先。亲缘关系分析结果与形态学的结果基本一致, 表明matK基因和ITS序列可以用于豆科植物属间的亲缘关系分析。
|
图 5 matK基因序列(A)和ITS序列(B)Neighbor-joining法构建植物系统进化树 Figure 5 The phylogenetic tree of legumes constructed by matK gene sequence(A) and ITS sequence(B) |
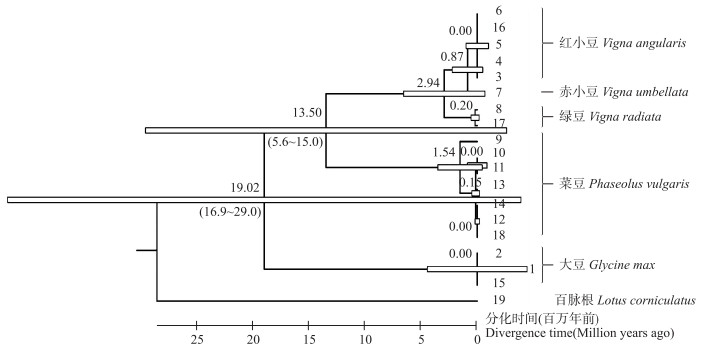
利用matK基因序列, 计算不同物种之间的分化时间。结果(图 6)显示:大豆与菜豆的分化时间为19.02百万年前, 菜豆与红小豆的分化时间为13.50百万年前。绿豆与红小豆的分化时间为2.94百万年前, 红小豆与‘苏长粒饭豆’之间的分化时间为0.87百万年前, 同一属内的豆类植物之间分化时间较小。
|
图 6 豆类分化时间 Figure 6 The divergence time of legumes |
matK基因和ITS序列都是DNA条形码鉴定的核心序列[24]。它们广泛用于植物的系统发育分析[12,14-16,18-22], 在科、属级水平类群的亲缘关系分析方面具有广泛的应用[16]。本研究将matK基因和ITS序列用于江苏地方豆类植物的亲缘关系研究, 结果显示同一属内的豆类植物之间matK基因序列和ITS序列的变异小, 遗传距离也较小, 它们之间的亲缘关系比较近, 而属间的遗传距离稍大于属内的遗传距离。经Neighbor-joining和Maximum Likelihood 2种方法构建进化树, 显示18种豆类的亲缘关系基本一致。由matK基因和ITS核苷酸序列构建的进化树可以看出, 18种豆科植物大致可以分为3个分支, 红小豆和绿豆分为一组, 菜豆和大豆分别聚为一组。说明红小豆与绿豆亲缘关系最近, 这与形态学分类结果基本一致。这与Yang等[9]用豆类植物全基因组测序结果构建的进化树的研究结果一致。
本研究结果还表明, 大白芸豆处于菜豆分支的最上游, 可能为菜豆进化过程中的早期品种。赤小豆与绿豆、红小豆、黑吉豆都属于菜豆族豇豆属, 它们共同构成了亚洲驯化的最重要的4种豇豆属作物[25]。由于‘苏长粒饭豆’属于豇豆属赤小豆种, 因此, 它与红小豆、绿豆的亲缘关系要近于菜豆品种。有研究也发现, 赤小豆与红小豆基因组间具有很高的共线性和同源性[26]。本研究也发现, ‘苏长粒饭豆’与红小豆matK基因和ITS序列的SNP数目小于其与菜豆、绿豆的SNP数目, 并且在进化树及分化时间分析中显示, ‘苏长粒饭豆’与红小豆的亲缘关系近于其与菜豆、绿豆的亲缘关系。‘苏水白饭豆’属于菜豆属菜豆种, 与‘苏长粒饭豆’不同, 我们的研究结果也证明了这一观点。由此可见, 仅从命名上, 并不能真正反映品种之间的从属关系。
本研究结果表明matK基因序列和ITS序列可以作为DNA条形码用来区分豆科不同属之间的豆类植物。存在差异的位点为变异位点, 而信息位点指的是至少存在2个不同核苷酸且每个不同核苷酸至少出现2次的变异位点。王德信[27]在天麻ITS序列的研究结果表明, 较丰富的变异位点和信息位点可用于近缘种的遗传多样性和亲缘关系的研究。本研究中采用同样的分析方法, 在18中豆科植物中, 检测到matK基因序列的变异位点数为85, 信息位点数为82, 表明18种豆科植物在matK基因片段上存在一定的分化。虽然matK基因是叶绿体中进化速率比较快的基因之一[12], 存在一定的分化, 但是由于叶绿体基因组的结构、基因的含量和基因的排列顺序都较为保守[28], 其进化速率远低于核基因的进化速率[29]。叶绿体基因的低进化速率, 限制了其在低级分类阶元(如属、亚属)中的应用[29], matK基因不能用于豆类属内的亲缘关系研究。ITS序列含有的变异位点数为260, 信息位点数为242, 其变异位点和信息位点所占序列总长度的比例明显大于matK基因的, 这与Johnson等[30]的观点一致, 但本研究结果表明, ITS序列不能用于鉴定豆类属内的亲缘关系。
随着基因组测序技术的发展, 对豆类的研究也越来越深入, 但对于一些地方品种亲缘关系的研究却很少。本研究就江苏地方培育的豆类展开了亲缘关系研究, 为以后的新品种培育、遗传改良等奠定基础, 也为江苏省地方豆类的进一步研究奠定了基础。此外, 本研究的试验材料只涉及本实验室收集的一些豆类品种, 今后需要扩大取材范围, 同时增加新的目的基因来对豆科植物的亲缘关系进行进一步的验证。
| [1] | Schmutz J, Cannon S B, Schlueter J, et al. Genome sequence of the palaeopolyploid soybean[J]. Nature, 2010, 463(7278): 178–183. DOI: 10.1038/nature08670 |
| [2] | Schmutz J, McClean P E, Mamidi S, et al. A reference genome for common bean and genome-wide analysis of dual domestications[J]. Nature Genetics, 2014, 46(7): 707–713. DOI: 10.1038/ng.3008 |
| [3] | Kang Y J, Kim S K, Kim M Y, et al. Genome sequence of mungbean and insights into evolution within Vigna species[J]. Nature Communications, 2014, 5: 5443. DOI: 10.1038/ncomms6443 |
| [4] | Sato S, Nakamura Y, Kaneko T, et al. Genome structure of the legume, Lotus japonicus[J]. DNA Research, 2008, 15(4): 227–239. DOI: 10.1093/dnares/dsn008 |
| [5] | Varshney R K, Chen W B, Li Y, et al. Draft genome sequence of pigeonpea(Cajanus cajan), an orphan legume crop of resource-poor farmers[J]. Nature Biotechnology, 2011, 30(1): 83–89. DOI: 10.1038/nbt.2022 |
| [6] | Kim M Y, Lee S, Van K, et al. Whole-genome sequencing and intensive analysis of the undomesticated soybean(Glycine soja Sieb. and Zucc.)genome[J]. Proc Natl Acad Sci USA, 2010, 107(51): 22032–22037. DOI: 10.1073/pnas.1009526107 |
| [7] | Young N D, Debelle F, Oldroyd G E, et al. The Medicago genome provides insight into the evolution of rhizobial symbioses[J]. Nature, 2011, 480(7378): 520–524. |
| [8] | Varshney R K, Song C, Saxena R K, et al. Draft genome sequence of chickpea(Cicer arietinum)provides a resource for trait improvement[J]. Nature Biotechnology, 2013, 31(3): 240–246. DOI: 10.1038/nbt.2491 |
| [9] | Yang K, Tian Z X, Chen C H, et al. Genome sequencing of adzuki bean(Vigna angularis)provides insight into high starch and low fat accumulation and domestication[J]. Proc Natl Acad Sci USA, 2015, 112(43): 13213–13218. DOI: 10.1073/pnas.1420949112 |
| [10] |
朱国忠, 戴伟民, 陈晓峰, 等. 基于基因组数据库的杂草稻叶绿体分子标记的开发[J].
南京农业大学学报, 2015, 38(2): 240–247.
Zhu G Z, Dai W M, Chen X F, et al. Development of molecular markers in weedy rice chloroplast based on genomic database[J]. Journal of Nanjing Agricultural University, 2015, 38(2): 240–247. DOI: 10.7685/j.issn.1000-2030.2015.02.010 (in Chinese with English abstract) |
| [11] | Hilu K W, Liang H P. The matK gene:sequence variation and application in plant systematics[J]. American Journal of Botany, 1997, 84(6): 830–839. DOI: 10.2307/2445819 |
| [12] | Plunkett G M, Soltis D E, Soltis P S. Clarification of the relationship between Apiaceae and Araliaceae based on matK and rbcL sequence data[J]. American Journal of Botany, 1997, 84(4): 565–580. DOI: 10.2307/2446032 |
| [13] | Cbol Plant Working Group. A DNA barcode for land plants[J]. Proc Natl Acad Sci USA, 2009, 106(31): 12794–12797. DOI: 10.1073/pnas.0905845106 |
| [14] | Sun X Q, Zhu Y J, Guo J L, et al. DNA barcoding the Dioscorea in China, a vital group in the evolution of monocotyledon:use of matK gene for species discrimination[J]. PLoS ONE, 2012, 7(2): e32057. DOI: 10.1371/journal.pone.0032057 |
| [15] | Steven G N, Subramanyam R. Testing plant barcoding in a sister species complex of pantropical Acacia(Mimosoideae, Fabaceae)[J]. Molecular Ecology Resources, 2009, 9(Suppl 1): 172–180. |
| [16] |
张安世, 司清亮, 张为民, 等. 基于matK基因的几种苔藓植物的亲缘关系分析[J].
河南农业科学, 2012, 41(1): 110–112.
Zhang A S, Si Q L, Zhang W M, et al. Analysis of the phylogenetic relationship among some bryophytes based on matK gene[J]. Journal of Henan Agricultural Sciences, 2012, 41(1): 110–112. (in Chinese with English abstract) |
| [17] |
于华会, 杨志玲, 杨旭, 等. 药用植物种质资源ITS序列研究进展[J].
中草药, 2010, 41(3): 491–496.
Yu H H, Yang Z L, Yang X, et al. Advances in studies on ITS sequence of medicinal plants germplasm resources[J]. Chinese Traditional and Herbal Drugs, 2010, 41(3): 491–496. (in Chinese with English abstract) |
| [18] | Kollipara K P, Singh R J, Hymowitz T. Phylogenetic and genomic relationships in the genus Glycine Willd. based on sequences from the ITS region of nuclear rDNA[J]. Genome, 1997, 40(1): 57–68. DOI: 10.1139/g97-008 |
| [19] | She C W, Jiang X H, Ou L J, et al. Molecular cytogenetic characterisation and phylogenetic analysis of the seven cultivated Vigna species(Fabaceae)[J]. Plant Biology, 2015, 17(1): 268–280. DOI: 10.1111/plb.12174 |
| [20] | 唐传红, 唐庆久, 张劲松, 等. 基于rDNA ITS序列和ERIC-PCR研究虫草真菌无性分离物的遗传多样性[J]. 南京农业大学学报, 2014, 37(2): 146–152. DOI: 10.7685/j.issn.1000-2030.2014.02.024 |
| [21] | Baraket G, Saddoud O, Chatti K, et al. Sequence analysis of the internal transcribed spacers(ITSs)region of the nuclear ribosomal DNA(nrDNA)in fig cultivars(Ficus carica L.)[J]. Scientia Horticulturae, 2009, 120(1): 34–40. DOI: 10.1016/j.scienta.2008.09.013 |
| [22] |
薛华杰, 闫茂华, 陆长梅, 等. 基于ITS序列的东亚当归属植物的分类学研究[J].
植物分类学报, 2007, 45(6): 783–795.
Xue H J, Yan M H, Lu C M, et al. Taxonomic study of Angelica from East Asia:inferences from ITS sequences of nuclear ribosomal DNA[J]. Acta Phytotaxonomica Sinca, 2007, 45(6): 783–795. (in Chinese with English abstract) |
| [23] | 黄明忠. 基于matK基因和ITS序列的海南野生兰科植物DNA条形码探讨[D]. 海口: 海南大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10589-2010183760.htm |
| [24] |
石林春, 刘金欣, 姚辉, 等. 种子植物matK引物通用性分析研究[J].
世界科学技术:中医药现代化, 2013, 15(3): 381–386.
Shi L C, Liu J X, Yao H, et al. Universality assessment of matK primer pairs in seed plants[J]. World Science and Technology:Modernization of Traditional Chinese Medicine and Materia Medica, 2013, 15(3): 381–386. (in Chinese with English abstract) |
| [25] |
王丽侠, 程须珍, 王素华. 中国饭豆种质资源遗传多样性及核心种质构建[J].
植物遗传资源学报, 2014, 15(2): 242–247.
Wang L X, Cheng X Z, Wang S H. Genetic diversity analysis and a core collection construction of rice bean(Vigna umbellata)in China[J]. Journal of Plant Genetic Resources, 2014, 15(2): 242–247. (in Chinese with English abstract) |
| [26] | Isemura T, Tomooka N, Kaga A, et al. Comparison of the pattern of crop domestication between two Asian beans, adzuki bean(Vigna angularis)and rice bean(V. umbellata)[J]. Japan Agricultural Research Quarterly, 2011, 45(1): 23–30. DOI: 10.6090/jarq.45.23 |
| [27] |
王德信. 天麻ITS-1测序及单核苷酸多态性变异位点分析[J].
生物技术, 2012, 22(5): 48–51.
Wang D X. ITS-1 sequencing and analysis of single nucleotide polymorphism variable site in Gastrodia elata[J]. Biotechnology, 2012, 22(5): 48–51. (in Chinese with English abstract) |
| [28] |
张韵洁, 李德铢. 叶绿体系统发育基因组学的研究进展[J].
植物分类与资源学报, 2011, 33(4): 365–375.
Zhang Y J, Li D Z. Advances in phylogenomics based on complete chlorplast genomes[J]. Plant Diversity and Resources, 2011, 33(4): 365–375. (in Chinese with English abstract) |
| [29] | Jin D P, Lee J H, Xu B, et al. Phylogeography of East Asian Lespedeza buergeri(Fabaceae)based on chloroplast and nuclear ribosomal DNA sequence variations[J]. Journal of Plant Research, 2016, 129(5): 793–805. DOI: 10.1007/s10265-016-0831-2 |
| [30] | Johnson L A, Soltis D E. Phylogenetic inference in Saxifragaceae sensu stricto and Gilia(Polemoniaceae)using matK sequences[J]. Annals of the Missouri Botanical Garden, 1995, 82(2): 149–175. DOI: 10.2307/2399875 |




