文章信息
- 房经贵, 朱旭东, 贾海锋, 王晨
- FANG Jinggui, ZHU Xudong, JIA Haifeng, WANG Chen
- 植物蔗糖合酶生理功能研究进展
- Research advances on physiological function of plant sucrose synthase
- 南京农业大学学报, 2017, 40(5): 759-768
- Journal of Nanjing Agricultural University, 2017, 40(5): 759-768.
- http://dx.doi.org/10.7685/jnau.201706003
-
文章历史
- 收稿日期: 2017-06-04
蔗糖是植物体内光合作用的产物, 是植物生长发育所必需的。它不仅是作物产量、纤维品质以及果实食用品质等的重要影响因素[1-4], 也可以作为一种信号分子调控植物的生长发育过程[5]。蔗糖合酶(sucrose synthase, SUS)是蔗糖代谢过程中的一类关键酶, 在生物体中广泛存在, 负责催化蔗糖分解与合成的可逆反应:蔗糖+二磷酸尿苷(uridine diphosphate, UDP)↔果糖+二磷酸尿苷-葡萄糖(uridine diphosphate glucose, UDPG)[6]。蔗糖合酶不同于转化酶、蔗糖磷酸合酶等其他蔗糖代谢相关酶的独特之处在于它是这些酶中唯一可以催化蔗糖代谢的可逆反应, 更重要的是, 蔗糖合酶能够以几乎不消耗能量的方式催化蔗糖的合成和分解, 允许蔗糖从“库”快速转化为“源”且不需要其他酶的参与[7]。这些特点使蔗糖合酶在植物的呼吸、碳水化合物的生物合成及利用等过程中发挥了重要的作用, 因此蔗糖合酶及其功能一直是揭示植物生长发育规律的重要研究内容, 而且已经取得了较大进展[8-10]。为了对蔗糖合酶有充分的了解, 以便更系统地研究其功能, 本文对植物蔗糖合酶的研究进展进行了较全面的概括和介绍。
1 植物蔗糖合酶的结构与定位 1.1 植物蔗糖合酶的结构蔗糖合酶是Cardini等[11]于1955年在小麦胚芽中首次发现。迄今为止, 已发现蔗糖合酶广泛存在于水稻、拟南芥等高等植物以及苔藓、藻类、细菌和古细菌中, 但在动物、真菌和病毒中还未发现其存在。蔗糖合酶是“保留型”GT-4糖基转移酶亚家族(GT-4 glycosyltransferase subfamily)的成员, 属于更大的金属不依赖型GT-B糖基转移酶超家族(metal-independent GT-B glycosyltransferase superfamily)。蔗糖合酶在结构和功能上类似于蔗糖磷酸合酶和糖原合酶的集合体。
Zheng等[12]和Schmölzer等[13]对拟南芥蔗糖合酶AtSUS1的研究表明:其具有典型的蔗糖合酶结构, 是由2个对称的四聚体组成(图 1-A), 共有35个α螺旋、13个β转角和13个β折叠等二级结构。每个蔗糖合酶单体由4个不同的结构域组成:N端的细胞靶向结构域(cellular targeting domain, CTD)和早期结瘤素(early nodulin, ENOD)40多肽结合结构域(ENOD 40 peptide-binding domain, EPBD), 两者组成了参与细胞靶向的“调控”结构域; C端的GT-BN结构域与GT-BC结构域, 两者组成了GT-B糖基转移酶结构域(图 1-B)。这4个结构域与C端延伸(C-terminal extension)构成一条完整的蔗糖合酶多肽链。蔗糖合酶的CTD结构域可以使酶与细胞内的细胞质、线粒体、叶绿体等相结合, EPBD结构域可以使酶与根瘤形成相关的激素相结合, GT-B结构域可以使蔗糖合酶发挥催化蔗糖代谢的作用。

|
图 1 蔗糖合酶四聚体中亚基间的相互作用示意图[14-15] Figure 1 Illustration of putative long-range cross-subunit interactions in sucrose synthase tetramers A.拟南芥AtSUS1蛋白(PDB code 3S29) 的结构; B.不同结构域的具体结构(CTD, 蓝色; EPBD, 灰色; Nα1螺旋, 橙色), 箭头表示AtSUS1中的主要磷酸化位点Ser13。 A.Tetrameric structure of AtSUS1 from Arabidopsis thaliana(PDB code 3S29);B. The specific structure of the different domains(CTD, blue; EPBD, gray; Nα1 helix, orange). The major phosphorylation site Ser13 in AtSUS1 is indicated by an arrow. |
蔗糖合酶的N端“调控”结构域内还存在2个丝氨酸的磷酸化位点(图 2), 分别位于其两侧。例如, 拟南芥与玉米中第1个磷酸化位点分别位于Ser13和Ser15, 第2个磷酸化位点分别位于Ser167和Ser170。其中, 第1个位点为起主要作用的磷酸化位点, 该位点的丝氨酸磷酸化减少了蔗糖合酶与肌动蛋白的结合, 增加了与膜的结合, 且在酸性条件下蔗糖合酶催化活性升高。而第2个位点的磷酸化增强了蔗糖合酶的泛素化而促进了蔗糖合酶定位与功能的转换。

|
图 2 蔗糖合酶的N-末端前20个氨基酸的部分序列比对[15] Figure 2 Partial sequence alignments of the first 20 N-terminal amino acids of plant sucrose synthases 粗体表示高度保守Ser残基磷酸化区。蓝色表示疏水残基; 黄色表示极性残留; 橙色表示带负电荷(酸性)残基; 绿色表示带正电(碱性)残基。X表示任意一种氨基酸残基。 Highly conserved motifs, which contain the putative phosphorylated Ser residue, are represented in bold. Blue, hydrophobic residues; yellow, polar residues; orange, negatively charged(acidic)residues; green, positively charged(basic)residues. X represents any residue. |
蛋白质的亚细胞与组织定位决定了其功能如何。植物蔗糖合酶主要以可溶性形式存在于细胞质中, 或者结合在质膜上。蔗糖合酶可以通过快速改变其在细胞中的存在形式而行使不同的功能。当蔗糖合酶以可溶性形式存在于细胞质时, 主要是为细胞的能量代谢和淀粉的合成提供蔗糖分解的产物, 当其结合在质膜上时, 则为纤维素和胼胝质的合成提供前体物质UDPG[14]。在大麦和黄瓜等植物中也发现了定位在细胞质和质膜中的蔗糖合酶[3,14]。
蔗糖合酶还可以结合于液泡膜[16]、肌动蛋白纤维[17]及线粒体[3,18]上。对这些结合方式的蔗糖合酶的功能研究还不多, 但在一定程度上可以根据其在细胞中的结合位置来推断其功能。例如, 蔗糖合酶结合在液泡膜上时催化从液泡运到细胞质的蔗糖分解, 向胞质释放UDPG和果糖[16]。蔗糖合酶在蔗糖浓度高时会结合肌动蛋白纤维, 分布在肌动蛋白细胞骨架和内膜系统上[19], 可以推断其参与细胞分化、细胞壁的合成、细胞器定位以及介导细胞质的流动等[17]。而对于结合在线粒体上的蔗糖合酶, 研究推断其可能不具有催化的功能, 不参与蔗糖代谢[18]。
另外, 通过蔗糖合酶的组织定位情况可以发现, 其参与果实、种子等多个组织和器官的发育过程。例如, 拟南芥蔗糖合酶在开花后依次出现在角果皮、种皮、珠柄和胚乳以及胚胎和糊粉层中, 说明蔗糖合酶在种子发育早期可能参与“瞬时”淀粉的积累[20], 而在后期参与胚和子叶组织中的蔗糖代谢活动。位于果皮伴胞中的蔗糖合酶可以通过其催化的反应为拟南芥果实中的韧皮部运输活动提供能量。
2 植物蔗糖合酶的生理功能蔗糖合酶的合成活性可以催化UDPG与果糖合成蔗糖, 参与果聚糖代谢、细胞壁构建和呼吸消耗等生物过程; 其分解活性可以催化蔗糖水解成UDPG与果糖, 参与淀粉、纤维素和半纤维素的合成等代谢途径, 这2种作用调节着动态与可逆的蔗糖代谢过程, 其功能归纳为图 3。每种植物蔗糖合酶活性的变化有其特征性, 对蔗糖代谢调控的方式也是不同的, 这2种活性的动态变化导致了植物组织中糖分组成与水平的改变, 进而影响植物的生长状态。

|
图 3 蔗糖合酶催化反应示意图 Figure 3 The schematic diagram of sucrose synthase catalyzed reaction |
淀粉是植物体内碳水化合物的主要储藏形式, 蔗糖合酶可以催化蔗糖分解成淀粉合成所需的葡萄糖和果糖。有研究发现, 当拟南芥和玉米等植物中缺失蔗糖合酶基因时, 种子的淀粉含量显著减少[21-22], 而超量表达蔗糖合酶基因则会使种子和块茎的淀粉、葡萄糖含量显著升高[23-24]。另外, 在水稻、马铃薯等植物中, 当淀粉含量显著增加时, 伴随着蔗糖合酶表达和分解活性的显著上升[21,25]。
2.2 参与纤维素的合成蔗糖合酶分解蔗糖形成的UDPG可以为纤维素合成提供大量底物, 促进纤维素的合成。在棉花、烟草和杨树中, 蔗糖合酶基因过表达会显著增加纤维素含量、纤维长度和强度[19,26-28]。反之, 抑制棉花中蔗糖合酶基因的表达会显著降低纤维质量[29]。
2.3 参与果实品质的形成果实中糖的种类与含量直接影响其营养价值、风味口感、色泽等品质性状[30]。蔗糖合酶通过调节果实内蔗糖、葡萄糖和果糖等的浓度平衡来影响果实的糖积累, 进而调控果实的甜度、风味等品质性状[31]。例如, 葡萄、桃、梨、柑橘和番茄等果实中的糖积累特征与蔗糖合酶的活性显著相关[32-37]。
2.4 提升植物固氮能力蔗糖合酶可以通过结合早期结瘤素(ENOD)多肽形成激素性多肽从而促进豆科植物的根瘤形成(图 4)。豆科模式植物百脉根和蒺藜苜蓿中蔗糖合酶基因发生突变或表达受到抑制时, 植株的根瘤生长延迟, 丛枝菌根发育受到影响, 菌根的氮吸收效率降低, 总氮含量降低等, 最终固氮能力下降, 说明蔗糖合酶基因在建立和维持豆科植物与根瘤的固氮共生方面有重要的作用[10,38-39]。
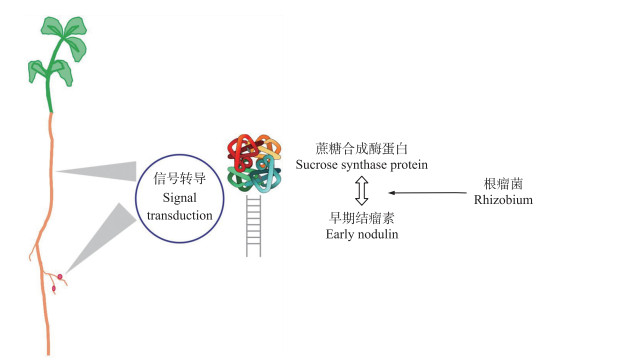
|
图 4 蔗糖合酶参与植物固氮的示意图 Figure 4 Schematic diagram of sucrose synthase participating in plant nitrogen fixation |
蔗糖合酶与植物逆境响应过程密切相关, 主要体现在植物对缺氧(低氧)胁迫的应答过程。由于蔗糖合酶催化蔗糖分解需要的能量更少, 因此植物在遭受缺氧胁迫而能量供给有限的情况下, 往往更倾向于依靠蔗糖合酶的催化反应来提供还原糖, 达到维持正常生长发育的目的。蔗糖合酶基因在缺氧条件下表达上调的特点反映其参与植物响应缺氧胁迫的过程[40-41], 从而提高植物对缺氧胁迫的抗性。例如, 玉米和木豆的耐涝性取决于根中是否有足够的糖储备, 而蔗糖合酶为糖酵解反应提供还原糖[42-43]。
蔗糖合酶也同样在甘蓝、葡萄等植物响应低温、病原侵染等胁迫反应的过程中发挥着重要作用[44-45]。
2.6 影响植物生殖生长基因表达分析表明:蔗糖合酶在任何组织与器官中都能表达, 蔗糖代谢在植物中是无处不在的, 但其在有些器官中扮演着更重要的角色。例如, 蔗糖合酶直接参与植物生殖生长, 这主要体现在蔗糖合酶可以影响植物花粉管的发育。在烟草花粉管的可溶性质膜和高尔基体中鉴定出含量丰富的蔗糖合酶蛋白, 说明蔗糖合酶在细胞外基质的构建和花粉管的形态形成中具有重要作用[8]。有研究报道指出, 当拟南芥、棉花等植物中蔗糖合酶基因过表达后, 其蔗糖合酶活性升高, 开花早, 败育种子数量减少而正常种子数量变多[29,46]; 当蒺藜苜蓿中蔗糖合酶基因的表达受到抑制时, 植株的花发育、开花时间及种子成熟等均受到影响[10,39]。
2.7 参与糖信号转导途径蔗糖作为一种重要的信号分子参与植物生长和发育。蔗糖合酶介导的蔗糖代谢反应本身释放的己糖也可以作为信号分子直接感知或通过能量和代谢物的变化间接感知上游信号从而触发信号转导[47], 与细胞周期、细胞壁信号转导、激素调节和程序性细胞死亡(programmed cell death, PCD)途径等发生相互作用来调控植物的叶、种子和纤维等发育过程(图 5)[5,29,48]。其中, 结合于质膜上的蔗糖合酶还可以作为特异的蔗糖信号传感器, 是与葡萄糖传感器及其信号传导机制不同的信号传导效应物[48]。
随着新一代基因组测序技术的出现和发展以及分子生物学和生物信息学技术的应用, 研究人员发现大多数植物蔗糖合酶家族是由多个成员构成的一个小的多基因家族。不同植物种类中蔗糖合酶基因家族成员的数量及其在染色体上的分布具有一定的差异[3,33,49-54]。不同植物中其家族成员数量从1个到31个不等, 如衣藻和卷柏只有1个蔗糖合酶基因, 葡萄中有5个, 巨尾桉中有15个。多数植物蔗糖合酶基因家族分布在少数几条染色体上, 如葡萄中蔗糖合酶基因分布在其19条染色体中的5条上, 巨尾桉蔗糖合酶基因分布在其11条染色体中的6条上。蔗糖合酶基因均具有2个结构域, 即蔗糖合酶结构域(sucrose synthase domain)和糖基转移酶结构域(glycosyl transferase domain), 这2个结构域分别对应蛋白质结构中的N端“调控”结构域和C端GT-B结构域。但不同蔗糖合酶基因的结构也有一定的差异, 主要体现在外显子和内含子的数量差异上。例如, 在水稻的6个蔗糖合酶基因(OsSUS1~6) 中, OsSUS5和OsSUS6编码的蛋白质在C端比其余4个基因编码蛋白质更长, 这是由于OsSUS 5有2个额外的外显子, OsSUS 6部分外显子合并以及最后1个外显子序列较长[55]。玉米中ZmSH1是ZmSUS1的同源基因, 两者差别在于ZmSH1缺失了最后1个内含子[22]。
3.2 植物中蔗糖合酶基因的进化对不同植物蔗糖合酶基因家族的大部分研究认为, 植物蔗糖合酶基因可分成3组, 但对每组的成员组成有不同认识。一种观点认为3组中仅有一个组可进一步分为单子叶植物亚组(monocot subgroup)和双子叶植物亚组(dicot subgroup)[3,53]; 也有观点认为3组中每组都包含单子叶植物和双子叶植物亚组[33,52], 说明单子叶植物和双子叶植物蔗糖合酶的功能存在共性。还有研究发现植物中蔗糖合酶基因被分成4组和5组的情况[56-57]。
尽管蔗糖合酶基因的类别不同, 但均表现出被子植物和裸子植物中蔗糖合酶成员分别聚为一类, 在单子叶植物和双子叶植物中又分别聚为一类的特点。所有这些发现都表明蔗糖合酶基因与植物的进化过程密切相关。不同植物中蔗糖合酶基因的进化与该植物进化过程中一些新功能的产生有着密切的联系, 因此各个植物中蔗糖合酶基因在结构和功能上不尽相同。
3.3 植物蔗糖合酶基因的转录调控机制蔗糖是植物生长发育以及应答各种胁迫中所必需的, 因此蔗糖合酶的表达在其作用过程中会受到精密调控, 从而呈现出“时空性”和“胁迫特异性”的转录调控机制的特点。
3.3.1 蔗糖合酶基因的“时空性”调控蔗糖合酶基因家族不同成员在植物生长发育过程中发挥的作用不同, 其转录表达呈现出一定程度的“时空性”。以水稻中的蔗糖合酶基因为例, OsSUS1主要在根、叶和节间伸长组织中表达, 表明OsSUS1在纤维素合成中具有潜在作用; OsSUS2在多个组织中均有表达, 说明其作用更加多样; OsSUS3和OsSUS4在颖果中有表达, 说明这2个基因在籽粒灌浆过程中具有调控碳分配的潜在作用; 而OsSUS5和OsSUS6的表达水平在所有组织中都很低[55]。类似地, 拟南芥AtSUS5和AtSUS6在叶片、花萼、花瓣、花药和根中表达, AtSUS2在不同发育期的种子里均表达, AtSUS3在种子发育中期表达, AtSUS1在花萼、成熟叶和果实中广泛表达, 而AtSUS4只在茎、根和发芽的种子中表达, 说明蔗糖合酶基因家族不同成员的功能具有多样化[58]。葡萄中蔗糖合酶基因家族也同样出现转录表达的“时空性”[32]。
3.3.2 蔗糖合酶基因的“胁迫特异性”调控植物蔗糖合酶基因家族不同成员在应答不同胁迫的过程中作用也不同, 其转录表达呈现出一定程度的“胁迫特异性”。例如, 在低氧(淹没)条件下的水稻幼苗中, OsSUS2表达水平显著增加, OsSUS5和OsSUS6表达则显著下降, 表明OsSUS2响应外界的环境胁迫从而促进蔗糖代谢活动[55]。烟草遭受干旱胁迫时NtSUS1和NtSUS2表达显著上升, 低温胁迫时NtSUS5表达水平升高6倍, 病毒侵染后NtSUS7表达也显著上升[53]。毛白杨和橡胶树遭受水分亏缺、低温和干旱胁迫后, PtSUS2.1、PtSUS3.1和HbSUS5等基因的表达显著升高, 这些都表明蔗糖合酶在胁迫响应过程中的重要作用[49,51]。
4 植物蔗糖合酶合成与分解活性的动态分布蔗糖合酶合成与分解活性的动态分布决定着其在特定时空下所能发挥的作用及其参与的生物过程。
4.1 “时空性”分布蔗糖合酶合成与分解活性呈现发育和组织器官特异性分布, 通过在不同发育阶段及不同组织器官中催化不同水平的蔗糖合成或分解反应而发挥作用。对梨、蜜瓜和脐橙等植物的研究表明, 果实发育不同阶段蔗糖合酶合成或分解活性大小不同。在果实发育早期, 蔗糖合酶以分解活性为主, 在发育后期合成活性则升高[36,59-61]。不同植物相同器官中其合成与分解活性的分布也不相同, 如桃、梨果实以蔗糖合酶合成活性为主, 与蔗糖积累呈正相关[61-62], 而在草莓、番木瓜中以蔗糖合酶分解活性为主, 且与蔗糖积累呈负相关[63-64]。
4.2 “胁迫特异性”分布由于植物响应逆境胁迫的过程同样伴随着蔗糖代谢的变化, 蔗糖合酶合成与分解2种活性的分布呈现出胁迫特异性的特点。例如在盐胁迫、低温、脱水和渗透胁迫下蔗糖合酶分解活性明显提高, 以增加糖分解的需求[65-67]。在适宜浓度的蔗糖溶液中培养番茄, 果实的蔗糖合酶活性明显提高, 但高浓度的蔗糖溶液则起抑制作用。二价金属离子也影响蔗糖合酶的活性, 如Mg2+能促进蔗糖合酶的活性, 而Zn2+、Hg2+、Cu2+、Fe2+、Ni2+和Co2+对蔗糖合酶活性均有抑制作用[65]。
4.3 “生物钟”分布在多种植物中的研究发现蔗糖合酶合成与分解活性也随昼夜呈规律性变化。一般来说, 蔗糖合酶分解活性较高, 且晚上较白天高, 而蔗糖合酶合成活性通常维持在较低水平[32,68-69]。但是也存在特殊情况, 如蜜瓜叶片中蔗糖合酶合成活性的昼夜变化呈双峰曲线, 中午11:00和下午17:00各有1个高峰, 且2个峰值相当。
4.4 其他影响因素蔗糖合酶的作用模式是一个较为复杂的系统。研究发现底物水平、环境因子(如温度和湿度等)、栽培措施、矿质营养和外源激素等多种因素都会导致蔗糖合酶的合成与分解活性的分布发生改变[70-75]。例如, 果糖和UDPG抑制蔗糖合酶的分解活性, 而UDP抑制蔗糖合酶的合成活性, 葡萄糖对合成和分解活性都有抑制作用[76-78], 这符合可逆反应的特点。另外, 施氮肥会使蔗糖合酶分解活性提高, 而使蔗糖合酶合成活性减弱[70]。低水平的铵态氮促进蔗糖合酶的合成活性, 而高水平的铵态氮则抑制蔗糖合酶合成活性, 促进分解活性[67]。
5 蔗糖合酶与遗传改良由于蔗糖合酶是催化蔗糖转化为淀粉的第一步, 而小麦、大麦和水稻等粮食作物产量的重要指标千粒质量主要取决于籽粒中的淀粉含量, 因此通过遗传改良, 提高蔗糖合酶的活性以使千粒质量增加, 最终使粮食作物的产量提高, 这已成为目前全球育种者的目标[79]。
目前小麦蔗糖合酶遗传改良的研究已经取得较大的进展, 研究人员利用小麦245份微核心种质、348份育成品种开展了小麦蔗糖合酶基因TaSUS 1、TaSUS2 与千粒质量的关联分析, 同时对小麦Ⅰ型与Ⅱ型蔗糖合酶的单元型(haplotype)进行了分析, 结果发现了4个蔗糖合酶基因的优异单元型, 这些优异单元型与高千粒质量显著相关, 在小麦育种过程中均受到正向选择。不同优异单元型的分子机制不同, 开发的分子标记可以快速识别优异单元型, 为分子辅助育种服务[79-80]。
6 研究展望随着对蔗糖合酶功能研究的逐渐深入, 人们对蔗糖合酶在植物发育中的作用有了更多认识; 同时发现蔗糖合酶具有多种多样的功能, 例如参与淀粉、纤维的合成以及固氮、逆境响应等, 这些都说明蔗糖合酶对植物的生长发育, 粮食作物产量的增加等有着极其重大的意义。但是, 与此同时也存在一些新的问题有待进一步研究:1) 蔗糖合酶家族成员之间相互协作的作用方式以及是否存在功能冗余问题仍不清楚; 2) 蔗糖合酶和其他蔗糖代谢相关酶、信号响应分子、环境胁迫因子等在分子水平上的相互作用以及调控的基础是什么?3) 蔗糖合酶与抗逆性的关系及该过程的信号转导通路是什么?4) 尚未在分子水平上研究各种理化因素以及昼夜周期对蔗糖合酶的合成与分解活性的调控机制等。这些研究对于全面认识蔗糖合酶具有很大的必要性。
相信今后通过功能基因组学与分子生物学等方法, 人们一定会更深入地了解植物蔗糖合酶的功能、蔗糖合酶代谢调控及其信号转导机制。这对人们认识植物生长发育的分子机制有重要意义, 也为利用蔗糖合酶基因改良作物品质奠定重要基础。
| [1] | Poovaiah C R, Mazarei M, Decker S R, et al. Transgenic switchgrass(Panicum virgatum L.)biomass is increased by overexpression of switchgrass sucrose synthase(PvSUS1)[J]. Biotechnology Journal, 2015, 10(4): 552–563. DOI: 10.1002/biot.v10.4 |
| [2] | Qazi H A, Paranjpe S, Bhargava S. Stem sugar accumulation in sweet sorghum-activity and expression of sucrose metabolizing enzymes and sucrose transporters[J]. Journal of Plant Physiology, 2012, 169(6): 605–613. DOI: 10.1016/j.jplph.2012.01.005 |
| [3] | Barrero-Sicilia C, Hernando-Amado S, González-Melendi P, et al. Structure, expression profile and subcellular localisation of four different sucrose synthase genes from barley[J]. Planta, 2011, 234(2): 391–403. DOI: 10.1007/s00425-011-1408-x |
| [4] |
冷翔鹏, 慕茜, 房经贵, 等. 葡萄浆果中的糖成分以及相关代谢研究的进展[J].
江苏林业科技, 2011, 38(2): 40–48.
Leng X P, Mu Q, Fang J G, et al. Advances of studies on sugar composition and related metabolisms in grape berries[J]. Journal of Jiangsu Forestry Science and Technology, 2011, 38(2): 40–48. (in Chinese with English abstract) |
| [5] | Ruan Y L. Signaling role of sucrose metabolism in development[J]. Molecular Plant, 2012, 5(4): 763–765. DOI: 10.1093/mp/sss046 |
| [6] | Geigenberger P, Stitt M. Sucrose synthase catalyses a readily reversible reaction in vivo in developing potato tubers and other plant tissuse[J]. Planta, 1993, 189(3): 329–339. DOI: 10.1007/BF00194429 |
| [7] | Fu H, Park W D. Sink-and vascular-associated sucrose synthase functions are encoded by different gene classes in potato[J]. The Plant Cell, 1995, 7: 1369–1385. DOI: 10.1105/tpc.7.9.1369 |
| [8] | Persia D, Cai G, Del C C, et al. Sucrose synthase is associated with the cell wall of tobacco pollen tubes[J]. Plant Physiology, 2008, 147(4): 1603–1618. DOI: 10.1104/pp.108.115956 |
| [9] | Kumutha D, Sairam R K, Ezhilmathi K, et al. Effect of waterlogging on carbohydrate metabolism in pigeon pea(Cajanus cajan L.):upregulation of sucrose synthase and alcohol dehydrogenase[J]. Plant Science, 2008, 175: 706–716. DOI: 10.1016/j.plantsci.2008.07.013 |
| [10] | Baier M C, Keck M, Gödde V, et al. Knockdown of the symbiotic sucrose synthase MtSucS1 affects arbuscule maturation and maintenance in mycorrhizal roots of Medicago truncatula[J]. Plant Physiology, 2010, 152(2): 1000–1014. DOI: 10.1104/pp.109.149898 |
| [11] | Cardini C E, Leloir L F, Chirboga J. The biosynthesis of sucrose[J]. Journal of Biological Chemistry, 1955, 214(1): 149–155. |
| [12] | Zheng Y, Anderson S, Zhang Y, et al. The structure of sucrose synthase-1 from Arabidopsis thaliana and its functional implications[J]. The Journal of Biological Chemistry, 2011, 286(41): 36108–36118. DOI: 10.1074/jbc.M111.275974 |
| [13] | Schmölzer K, Gutmann A, Diricks M, et al. Sucrose synthase:a unique glycosyltransferase for biocatalytic glycosylation process development[J]. Biotechnology Advances, 2016, 34(2): 88–111. DOI: 10.1016/j.biotechadv.2015.11.003 |
| [14] | Ruan Y L, Llewellyn D J, Furbank R T. Suppression of sucrose synthase gene expression represses cotton fiber cell initiation, elongation, and seed development[J]. The Plant Cell, 2003, 15(4): 952–964. DOI: 10.1105/tpc.010108 |
| [15] | Wang H, Sui X, Guo J, et al. Antisense suppression of cucumber(Cucumis sativus L.)sucrose synthase 3(CsSUS3) reduces hypoxic stress tolerance[J]. Plant, Cell and Environment, 2014, 37(3): 795–810. DOI: 10.1111/pce.2014.37.issue-3 |
| [16] | Etxeberria E, Gonzalez P. Evidence for a tonoplast-associated form of sucrose synthase and its potential involvement in sucrose mobilization from the vacuole[J]. Journal of Experimental Botany, 2003, 54(386): 1407–1414. DOI: 10.1093/jxb/erg148 |
| [17] | Duncan K A, Huber S C. Sucrose synthase oligomerization and F-actin association are regulated by sucrose concentration and phosphorylation[J]. Plant Cell Physiology, 2007, 48(11): 1612–1623. DOI: 10.1093/pcp/pcm133 |
| [18] | Subbaiah C C, Palaniappan A, Duncan K, et al. Mitochondrial localization and putative signaling function of sucrose synthase in maize[J]. The Journal of Biological Chemistry, 2006, 281(23): 15625–15635. DOI: 10.1074/jbc.M600355200 |
| [19] | Cai G, Faleri C, Del C C, et al. Distribution of callose synthase, cellulose synthase, and sucrose synthase in tobacco pollen tube is controlled in dissimilar ways by actin filaments and microtubules[J]. Plant Physiology, 2011, 155(3): 1169–1190. DOI: 10.1104/pp.110.171371 |
| [20] | Fallahi H, Scofield G N, Badger M R, et al. Localization of sucrose synthase in developing seed and siliques of Arabidopsis thaliana reveals diverse roles for SUS during development[J]. Journal of Experimental Botany, 2008, 59(12): 3283–3295. DOI: 10.1093/jxb/ern180 |
| [21] | Angeles-Núñez J G, Tiessen A. Arabidopsis sucrose synthase 2 and 3 modulate metabolic homeostasis and direct carbon towards starch synthesis in developing seeds[J]. Planta, 2010, 232(3): 701–718. DOI: 10.1007/s00425-010-1207-9 |
| [22] | Duncan K A, Hardin S C, Huber S C. The three maize sucrose synthase isoforms differ in distribution, localization, and phosphorylation[J]. Plant Cell Physiology, 2006, 47(7): 959–971. DOI: 10.1093/pcp/pcj068 |
| [23] | Li J, Baroja-Fernández E, Bahaji A, et al. Enhancing sucrose synthase activity results in increased levels of starch and ADP-glucose in maize(Zea mays L.)seed endosperms[J]. Plant Cell Physiology, 2013, 54(2): 282–294. DOI: 10.1093/pcp/pcs180 |
| [24] | Baroja-Fernández E, Muñoz F J, Montero M, et al. Enhancing sucrose synthase activity in transgenic potato(Solanum tuberosum L.)tubers results in increased levels of starch, ADPglucose and UDPglucose and total yield[J]. Plant Cell Physiology, 2009, 50(9): 1651–1662. DOI: 10.1093/pcp/pcp108 |
| [25] | Counce P A, Gravois K A. Sucrose synthase activity as a potential indicator of high rice grain yield[J]. Crop Science, 2006, 46(4): 1501–1507. DOI: 10.2135/cropsci2005.0240 |
| [26] | Jiang Y, Guo W, Zhu H, et al. Overexpression of GhSusA1 increases plant biomass and improves cotton fiber yield and quality[J]. Plant Biotechnology Journal, 2012, 10(3): 301–312. DOI: 10.1111/pbi.2012.10.issue-3 |
| [27] | Wei Z, Qu Z, Zhang L, et al. Overexpression of poplar xylem sucrose synthase in tobacco leads to a thickened cell wall and increased height[J]. PLoS ONE, 2015, 10(3): e0120669. DOI: 10.1371/journal.pone.0120669 |
| [28] | Coleman H D, Yan J, Mansfield S D. Sucrose synthase affects carbon partitioning to increase cellulose production and altered cell wall ultrastructure[J]. Proc Natl Acad Sci USA, 2009, 106(31): 13118–13123. DOI: 10.1073/pnas.0900188106 |
| [29] | Xu S M, Brill E, Llewellyn D J, et al. Overexpression of a potato sucrose synthase gene in cotton accelerates leaf expansion, reduces seed abortion, and enhances fiber production[J]. Molecular Plant, 2012, 5(2): 430–441. DOI: 10.1093/mp/ssr090 |
| [30] |
李晓颖, 谭洪花, 房经贵, 等. 果树果实的风味物质及其研究[J].
植物生理学报, 2011, 47(10): 943–950.
Li X Y, Tan H H, Fang J G, et al. Flavor compounds in fruits and research on them[J]. Plant Physiology Journal, 2011, 47(10): 943–950. (in Chinese with English abstract) |
| [31] |
房经贵, 刘崇怀.
葡萄分子生物学[M]. 北京: 科学出版社, 2014: 122-151.
Fang J G, Liu C H. Grape Molecular Biology[M]. Beijing: Science Press, 2014: 122-151. (in Chinese with English abstract) |
| [32] | Zhu X D, Zhang C B, Wu W M, et al. Enzyme activities and gene expression of starch metabolism provide insights into grape berry development[J]. Horticulture Research, 2017, 4: 17018. DOI: 10.1038/hortres.2017.18 |
| [33] | Islam M Z, Hu X M, Jin L F, et al. Genome-wide identification and expression profile analysis of citrus sucrose synthase genes:investigation of possible roles in the regulation of sugar accumulation[J]. PLoS ONE, 2014, 9(11): e113623. DOI: 10.1371/journal.pone.0113623 |
| [34] | Martínez-Esteso M J, Sellés-Marchart S, Lijavetzky D, et al. A DIGE-based quantitative proteomic analysis of grape berry flesh development and ripening reveals key events in sugar and organic acid metabolism[J]. Journal of Experimental Botany, 2011, 62(8): 2521–2569. DOI: 10.1093/jxb/erq434 |
| [35] | Morandi B, Corelli G L, Rieger M, et al. Carbohydrate availability affects growth and metabolism in peach fruit[J]. Physiology Plant, 2008, 133(2): 229–241. DOI: 10.1111/j.1399-3054.2008.01068.x |
| [36] | Tanase K, Shiratake K, Mori H, et al. Changes in the phosphorylation state of sucrose synthase during development of Japanese pear fruit[J]. Physiologia Plantarum, 2002, 114(1): 21–26. DOI: 10.1046/j.0031-9317.2001.10137.x |
| [37] | D'Aoust M A, Yelle S, Quoc B N. Antisense inhibition of tomato fruit sucrose synthase decreases fruit setting and the sucrose unloading capacity of young fruit[J]. The Plant Cell, 1999, 11: 2407–2418. DOI: 10.1105/tpc.11.12.2407 |
| [38] | Horst I, Welham T, Kelly S, et al. TILLING mutants of Lotus japonicus reveal that nitrogen assimilation and fixation can occur in the absence of nodule-enhanced sucrose synthase[J]. Plant Physiology, 2007, 144(2): 806–820. DOI: 10.1104/pp.107.097063 |
| [39] | Baier M C, Barsch A, Küster H, et al. Antisense repression of the Medicago truncatula nodule-enhanced sucrose synthase leads to a handicapped nitrogen fixation mirrored by specific alterations in the symbiotic transcriptome and metabolome[J]. Plant Physiology, 2007, 145(4): 1600–1618. DOI: 10.1104/pp.107.106955 |
| [40] | Biemelt S, Hajirezaei M R, Melzer M, et al. Sucrose synthase activity does not restrict glycolysis in roots of transgenic potato plants under hypoxic conditions[J]. Planta, 1999, 210(1): 41–49. DOI: 10.1007/s004250050652 |
| [41] | Ricard B, Toai T V, Chourey P, et al. Evidence for the critical role of sucrose synthase for anoxic tolerance of maize roots using a double mutant[J]. Plant Physiology, 1998, 116(4): 1323–1331. DOI: 10.1104/pp.116.4.1323 |
| [42] | Subbaiah C C, Sachs M M. Altered patterns of sucrose synthase phosphorylation and localization precede callose induction and root tip death in anoxic maize seedlings[J]. Plant Physiology, 2001, 125(2): 585–594. DOI: 10.1104/pp.125.2.585 |
| [43] | Kumutha D, Sairam R K, Ezhilmathi K, et al. Effect of waterlogging on carbohydrate metabolism in pigeon pea(Cajanus cajan L.):upregulation of sucrose synthase and alcohol dehydrogenase[J]. Plant Science, 2008(175): 706–716. |
| [44] | Sasaki H, Ichimura K, Imada S, et al. Sucrose synthase and sucrose phosphate synthase, but not acid invertase, are regulated by cold acclimation and deacclimation in cabbage seedlings[J]. Journal of Plant Physiology, 2001, 158(7): 847–852. DOI: 10.1078/0176-1617-00391 |
| [45] | Hren M, Ravnikar M, Brzin J, et al. Induced expression of sucrose synthase and alcohol dehydrogenaseⅠgenes in phytoplasma-infected grapevine plants grown in the field[J]. Plant Pathology, 2009, 58(1): 170–180. DOI: 10.1111/ppa.2009.58.issue-1 |
| [46] | Xu F Y, Joshi P C. Overexpression of aspen sucrose synthase gene promotes growth and development of transgenic Arabidopsis plants[J]. Advances in Bioscience and Biotechnology, 2010, 1: 426–438. DOI: 10.4236/abb.2010.15056 |
| [47] | Li L, Sheen J. Dynamic and diverse sugar signaling[J]. Current Opinion in Plant Biology, 2016, 33: 116–125. DOI: 10.1016/j.pbi.2016.06.018 |
| [48] | Lastdrager J, Hanson J, Smeekens S. Sugar signals and the control of plant growth and development[J]. Journal of Experimental Botany, 2014, 65(3): 799–807. DOI: 10.1093/jxb/ert474 |
| [49] | Xiao X, Tang C, Fang Y, et al. Structure and expression profile of the sucrose synthase gene family in the rubber tree:indicative of roles in stress response and sucrose utilization in the laticifers[J]. The FEBS Journal, 2014, 281(1): 291–305. DOI: 10.1111/febs.2013.281.issue-1 |
| [50] | Zou C, Lu C, Shang H, et al. Genome-wide analysis of the Sus gene family in cotton[J]. Journal of Integrative Plant Biology, 2013, 55(7): 643–653. DOI: 10.1111/jipb.12068 |
| [51] | An X, Chen Z, Wang J, et al. Identification and characterization of the Populus sucrose synthase gene family[J]. Gene, 2014, 539(1): 58–67. DOI: 10.1016/j.gene.2014.01.062 |
| [52] | Li F, Hao C, Yan L, et al. Gene structure, phylogeny and expression profile of the sucrose synthase gene family in cacao(Theobroma cacao L.)[J]. Journal of Genetics, 2015, 94(3): 461–472. DOI: 10.1007/s12041-015-0558-1 |
| [53] | Wang Z, Wei P, Wu M, et al. Analysis of the sucrose synthase gene family in tobacco:structure, phylogeny, and expression patterns[J]. Planta, 2015, 242(1): 153–166. DOI: 10.1007/s00425-015-2297-1 |
| [54] | Zhu X, Wang M, Li X, et al. Genome-wide analysis of the sucrose synthase gene family in grape(Vitis vinifera):structure, evolution, and expression profiles[J]. Genes, 2017, 8: 111. DOI: 10.3390/genes8040111 |
| [55] | Hirose T, Scofield G N, Terao T. An expression analysis profile for the entire sucrose synthase gene family in rice[J]. Plant Science, 2008, 174(5): 534–543. DOI: 10.1016/j.plantsci.2008.02.009 |
| [56] | Zhang D Q, Xu B H, Yang X H, et al. The sucrose synthase gene family in Populus:structure, expression, and evolution[J]. Tree Genetics and Genomes, 2011, 7(3): 443–456. DOI: 10.1007/s11295-010-0346-2 |
| [57] | Jiang S Y, Chi Y H, Wang J Z, et al. Sucrose metabolism gene families and their biological functions[J]. Scientific Reports, 2015, 5: 17583. DOI: 10.1038/srep17583 |
| [58] | Bieniawska Z, Paul Barratt D H, Garlick A P, et al. Analysis of the sucrose synthase gene family in Arabidopsis[J]. The Plant Journal, 2007, 49(5): 810–828. DOI: 10.1111/tpj.2007.49.issue-5 |
| [59] |
张中霞, 刘艳, 白立华, 等. 河套蜜瓜果实发育过程中糖积累与蔗糖代谢相关酶的关系[J].
西北植物学报, 2011, 31(1): 123–129.
Zhang Z X, Liu Y, Bai L H, et al. Relationship between sugar accumulation and its metabolizing enzymes during muskmelon fruit development[J]. Acta Botanica Boreali-Occidentalia Sinica, 2011, 31(1): 123–129. (in Chinese with English abstract) |
| [60] |
龚荣高, 张光伦, 吕秀兰, 等. 脐橙在不同生境下果实蔗糖代谢相关酶的研究[J].
园艺学报, 2004, 31(6): 719–722.
Gong R G, Zhang G L, Lü X L, et al. Studies on the sucrose-metabolizing enzymes in navel orange fruit from different habitats[J]. Acta Horticulturae Sinica, 2004, 31(6): 719–722. (in Chinese with English abstract) |
| [61] | Moriguchi T, Abe K, Sanada T, et al. Levels and role of sucrose synthase sucrose phosphate synthase and acid invertase in sucrose accumulation in fruit of Asian pear[J]. Journal of the American Society for Horticultural Science, 1992, 117(2): 274–278. |
| [62] | Bianco R L, Rieger M, Sung S J S. Effect of drought on sorbitol and sucrose metabolism in sinks and sources of peach[J]. Physiologia Plantarum, 2000, 108(1): 71–78. DOI: 10.1034/j.1399-3054.2000.108001071.x |
| [63] | Basson C E, Groenewald J H, Kossmann J, et al. Sugar and acid-related quality attributes and enzyme activities in strawberry fruits:invertase is the main sucrose hydrolysing enzyme[J]. Food Chemistry, 2010, 121(4): 1156–1162. DOI: 10.1016/j.foodchem.2010.01.064 |
| [64] | Zhou L, Paull R E. Sucrose metabolism during Papaya(Caica papaya)fruit growth and ripening[J]. Journal of the American Society for Horticultural Science, 2000, 126(3): 351–357. |
| [65] | Römer U, Schrader H, Günther N, et al. Expression, purification and characterization of recombinant sucrose synthase 1 from Solanum tuberosum L. for carbohydrate engineering[J]. Journal of Biotechnology, 2004, 107(2): 135–149. DOI: 10.1016/j.jbiotec.2003.10.017 |
| [66] | Klotz K L, Haagenson D M. Wounding, anoxia and cold induce sugar beet sucrose synthase transcriptional changes that are unrelated to protein expression and activity[J]. Journal of Plant Physiology, 2008, 165(4): 423–434. DOI: 10.1016/j.jplph.2007.02.001 |
| [67] | Haagenson D M, Klotz K L, McGrath J M. Sugar beet sucrose synthase genes differ in organ-specific and developmental expression[J]. Journal of Plant Physiology, 2006, 163(1): 102–106. DOI: 10.1016/j.jplph.2005.05.006 |
| [68] | Chen A, He S, Li F, et al. Analyses of the sucrose synthase gene family in cotton:structure, phylogeny and expression pattern[J]. BMC Plant Biology, 2012, 12(3): 1–17. |
| [69] |
蔡贵芳, 刘艳, 白立华, 等. 去果河套蜜瓜源叶碳水化合物及其相关酶昼夜变化特征[J].
西北植物学报, 2012, 32(9): 1774–1780.
Cai G F, Liu Y, Bai L H, et al. Day and night variations of carbohydrate contents and activities of related enzymes in source leaves of defruiting Cucumis melo L. cv. Hetao[J]. Acta Botanica Boreali-Occidentalia Sinica, 2012, 32(9): 1774–1780. (in Chinese with English abstract) |
| [70] | Ozawa K, Kawahigashi H. Positional cloning of the nitrite reductase gene associated with good growth and regeneration ability of calli and establishment of a new selection system for Agrobacterium mediated transformation in rice(Oryza sativa L.)[J]. Plant Science, 2006, 170: 384–393. DOI: 10.1016/j.plantsci.2005.09.015 |
| [71] | Pan Q H, Cao P, Duan C Q. Comparison of enzymes involved in sugar metabolism from Shang-24(Vinifera quinguangularis)and Cabernet Sauvignon(Vinifera vinifera)at veraison[J]. Australian Journal of Grape and Wine Research, 2009, 15: 9–17. DOI: 10.1111/ajgw.2009.15.issue-1 |
| [72] | Fortes A M, Teixeira R T, Agudelo-Romero P. Complex interplay of hormonal signals during grape berry ripening[J]. Molecules, 2015, 20(5): 9326–9343. DOI: 10.3390/molecules20059326 |
| [73] |
倪照君, 艾海提·阿米娜, 章镇, 等. 吲熟酯和钼酸铵处理对青种枇杷果实糖积累及相关酶活性的影响[J].
南京农业大学学报, 2010, 33(6): 33–37.
Ni Z J, Ai H T·A M N, Zhang Z, et al. Effects of ethychlozate and ammonium molybdate on sugar accumulation and related enzymes activities of Qingzhong loquat fruit[J]. Journal of Nanjing Agricultural University, 2010, 33(6): 33–37. DOI: 10.7685/j.issn.1000-2030.2010.06.007 (in Chinese with English abstract) |
| [74] |
李宁, 苏晓琼, 孙锦, 等. 外源24-表油菜素内酯对弱光胁迫下番茄叶片内源激素和碳水化合物代谢及果实的影响[J].
南京农业大学学报, 2014, 37(3): 51–56.
Li N, Su X Q, Sun J, et al. Effects of exogenous 24-epibrassinolide on endogenous hormone, carbohydrate metabolism and fruits of tomato under low light stress[J]. Journal of Nanjing Agricultural University, 2014, 37(3): 51–56. DOI: 10.7685/j.issn.1000-2030.2014.03.007 (in Chinese with English abstract) |
| [75] |
沈直, 唐设, 张海祥, 等. 灌浆期开放式增温对水稻强势粒和弱势粒淀粉代谢关键酶相关基因表达水平的影响[J].
南京农业大学学报, 2016, 39(6): 898–906.
Shen Z, Tang S, Zhang H X, et al. Effect of T-FACE high temperature on genes expression level of key enzymes involved in starch metabolism in superior spikelets and inferior spikelets of rice during grain filling period[J]. Journal of Nanjing Agricultural University, 2016, 39(6): 898–906. DOI: 10.7685/jnau.201604047 (in Chinese with English abstract) |
| [76] | Daloso D M, Williams T C, Antunes W C, et al. Guard cell-specific upregulation of sucrose synthase 3 reveals that the role of sucrose in stomatal function is primarily energetic[J]. New Phytologist, 2016, 209(4): 1470–1483. DOI: 10.1111/nph.13704 |
| [77] | Komastu A, Takanokura Y, Moriguchi T. Differential expression of three sucros-phosphate synthase isoforms during accumulation in citrus fruit(Citrus unshiu Marc.)[J]. Plant Science, 1999, 140: 169–178. DOI: 10.1016/S0168-9452(98)00217-9 |
| [78] | Koch K E, Nolte K D, Duke E R, et al. Sugar levels modulate differential expression of maize sucrose synthase genes[J]. The Plant Cell, 1992, 4: 59–69. DOI: 10.1105/tpc.4.1.59 |
| [79] | Hou J, Jiang Q, Hao C, et al. Global selection on sucrose synthase haplotypes during a century of wheat breeding[J]. Plant Physiology, 2014, 164(4): 1918–1929. DOI: 10.1104/pp.113.232454 |
| [80] | Jiang Q, Hou J, Hao C, et al. The wheat(T. aestivum)sucrose synthase 2 gene(TaSus2) active in endosperm development is associated with yield traits[J]. Functional and Integrative Genomics, 2011, 1(1): 49–61. |





