文章信息
- 董邦宁, 龙武华, 刘喜, 刘世家, 田云录, 陈亮明, 江玲, 王益华, 万建民
- DONG Bangning, LONG Wuhua, LIU Xi, LIU Shijia, TIAN Yunlu, CHEN Liangming, JIANG Ling, WANG Yihua, WAN Jianmin
- 水稻矮秆突变体htr的表型分析及基因克隆
- Phenotypic analysis and gene cloning of a dwarf mutant htr in rice
- 南京农业大学学报, 2017, 40(4): 573-583
- Journal of Nanjing Agricultural University, 2017, 40(4): 573-583.
- http://dx.doi.org/10.7685/jnau.201612014
-
文章历史
- 收稿日期: 2016-12-09
水稻是世界上最重要的粮食作物之一, 全球超过半数以上的人口以稻米为主粮, 而株高是影响水稻产量的重要农艺性状之一。20世纪60年代水稻半矮秆基因sd-1及其等位基因的利用使水稻单产提高20%~ 30%, 引发了全球水稻生产的第一次“绿色革命”[1-2], 20世纪70年代杂交稻的成功实现和近年来“超级稻”育种的兴起, 都是建立在矮化育种的成果之上[3]。
水稻矮秆基因的功能研究能够揭示调控水稻株型发育的分子机制, 并进一步阐述植物生长发育的机制。水稻植株矮化由主效基因控制, 同时也受到修饰基因、调节基因以及抑制基因等微效基因的调节[4]。矮秆基因能够直接导致水稻植株在形态学或细胞学上发生变化, 如茎秆节间缩短、细胞变小、细胞数目减少, 从而使植株出现矮化表型。Sunohara等[5]和马良勇等[6]根据水稻各节间的伸长模式, 将水稻矮秆突变体主要分为dn-、dm、d6-、nl-和sh-五种基本类型:dn-型各节间等比例缩短, dm-型倒二节间特异缩短, d6-型仅穗下倒一节间伸长, nl-型为上部节间缩短, 下部节间伸长, sh-型第一节间缩短最为严重。迄今为止, 水稻中已定位的矮秆基因超过130个, 主效基因大约有70个, 覆盖水稻12条染色体。已克隆的矮秆基因大都涉及激素代谢或信号传导, 例如, 赤霉素(gibberellins, GAs)合成或代谢途径基因, 如GID 2、DGL1、SUI1、SUI3 [7-9]; 油菜素内酯(brassinosteroids, BRs)合成或信号传导相关基因, 如D 11、BRD1、TUD1 [10-12]; 以及与生长素(indole acetic acids, IAAs)途径相关基因, 如IAA 1、IAA4、SAUR39 [13-15]。现有的研究结果表明:赤霉素、油菜素内酯、生长素、乙烯(ethylene)、细胞分裂素(cytokinin, CK)、脱落酸(abscisic acid, ABA)及独脚金内酯(strigolactones, SLs)均能影响水稻茎的生长和发育, 对水稻的株高具有调控作用, 其中以GAs和BRs与水稻茎秆伸长的关系最为密切[16]。
除受激素直接调控外, 株高同时也受到转录因子等其他因素的间接影响, 而AP2/ERF类转录因子就是其中重要一员[17]。AP2/ERF是植物所特有的一类庞大的转录因子家族, 参与植物的生长、发育以及多种生理生化反应的信号转导, 调控植物器官形成、激素(生长素、乙烯、脱落酸)响应等。AP2/ERF家族转录因子均包含由60~70个左右氨基酸组成的高度保守的DNA结合域, 即AP2/ERF结合域。根据其序列相似性和AP2/ERF结构域数量, 将该家族分为5个亚家族, 即AP2(APETALA2)、DREB(dehydration-responsive element binding proteins)、RAV(related to ABI3/VP1)、ERF(ethylene-responsive factor)和Soloist[18-19]。其中AP2亚家族包含2个重复的AP2结构域(AP2-R1和AP2-R2), 根据2个结构域是否有插入序列, 将AP2亚家族分为ANT和euAP2两组, 在ANT组中R1结构域有10个氨基酸插入, R2结构域有1个氨基酸插入; 而在euAP2组结构域不存在氨基酸的插入[20]。AP2/ERF各亚家族间的大部分成员具有相似功能, 其中AP2亚家族主要参与调控植物生长发育过程[21]。张计育等[22]报道水稻全基因组含有166个AP2/ERF家族成员, 其中26个属于AP2亚家族。目前水稻中已克隆一系列AP2/ERF类家族基因, 其中多个基因与株高调控相关, 如OsEATB[23]、OsAP2-39 [24]、OsCRL 5 [25]。OsEATB编码一个AP2家族转录因子, 调控GA生物合成相关贝壳杉烯合成酶A的基因表达, 进而影响植株体内GA的含量[23]。OsAP2-39含有一个AP2结构域, 通过调控ABA生物合成途径中关键基因OsNCE-1D和GA代谢相关基因EUI的表达, 从而调控ABA/GA在植株体内的平衡, 最终调节水稻植株的生长发育[24]。OsCRL 5通过诱导细胞分裂素A类应答调节子OsRR 1参与水稻不定根的形成, 同时也影响株高[25]。因此, 克隆并鉴定新的与株高相关的AP 2 /ERF类基因, 对研究该类转录因子调节水稻生长发育作用的分子机制具有重要意义。
本研究利用MNU化学诱变籼稻品种‘9311’获得1份稳定遗传的矮秆突变体htr, 对其表型进行了考察及节间细胞形态观察分析。遗传分析表明, htr矮秆性状由单隐性核基因控制, 并将其定位在第5染色体上。本研究初步揭示HTR控制株高的遗传和分子机制, 为水稻育种提供了新的矮秆基因资源。
1 材料与方法 1.1 材料及农艺性状考察矮秆突变体htr(height and tiller reduced)是籼稻品种‘9311’经MNU(N-甲烷-N-亚硝基脲)诱变处理获得。htr经南京农业大学土桥、牌楼和海南陵水实验基地多代自交, 其突变特征及各方面农艺性状均已稳定遗传。在成熟期对htr及野生型‘9311’进行农艺性状考察, 包括株高、分蘖数、各节间长度、剑叶长、剑叶宽, 粒长、粒宽、粒厚、穗长、千粒质量等指标。
1.2 定位群体构建和遗传分析配制htr与‘N22’杂交组合, 构建F2分离群体, 于成熟期时选取该群体中极端矮化单株用于基因的精细定位; 同时配制htr和野生型‘9311’的正反交组合, 分别统计正反交的F2群体中正常植株和矮化植株的株数, 计算分离比, 进行基因的遗传分析。
1.3 叶绿素含量测定随机选取抽穗时期htr和‘9311’各5株, 于剑叶中间部位剪取长、宽各0.5 cm片段, 称质量(0.03 g左右, 3个重复), 浸泡于2 mL 95%(体积分数)乙醇中, 室温避光浸泡48 h。取上清液, 用Molecular Devices M3分光光度计测量665、649和470 nm 3个波长下溶液的光密度值, 根据Lichtenthaler[26]的计算公式:
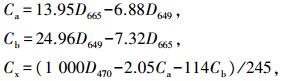
|
计算叶绿素a(Ca)、叶绿素b(Cb)和类胡萝卜素(Cx)等色素浓度。细胞色素含量(mg · g-1)=(叶绿素浓度×提取液体积×稀释倍数)/样品鲜质量。
1.4 茎秆振动切片观察于成熟期取htr和‘9311’茎秆的倒2节间0.5 cm长茎节, 纵向固定于振动切片机(Leica VT1200S)操作台上, 横向切取80 μm厚的切片; 同时取0.5 cm长茎节横向固定与操作台上, 纵向切取50 μm厚的切片, 将横切和纵切切片分别置于载玻片上, 利用体视显微镜(Nikon AZ100) 观察横切和纵切细胞大小及维管束分布情况。
1.5 赤霉素处理试验将htr和‘9311’种子用70%(体积分数)的84消毒液浸泡2 h, 灭菌水清洗3次后浸入到灭菌水中置于30 ℃培养箱培养48 h, 挑选露白一致的种子移至10 g · L-1琼脂培养基, 培养基含0、1×10-8、1×10-7、1×10-6、1×10-5 mol · L-1 GA3, 每个处理各放置htr和‘9311’发芽一致的种子15粒, 移至30 ℃培养箱中(光照14 h、黑暗10 h), 培养7 d后测定不同浓度GA3处理下水稻幼苗的株高。每个浓度均设置3个重复。
1.6 HTR基因定位利用本实验室的分子标记库筛选出‘9311’和‘N22’之间具有多态性的SSR和InDel标记, 从中挑选出均匀分布于12条水稻染色体的标记, 间距在10~15 cM之间, 并选择20个极端矮化个体的DNA进行连锁分析。确定连锁标记后, 在连锁标记所在染色体区利用Primer Premier 5.0软件开发新的标记(表 1), 共选取184个极端个体进行精细定位。
qRT-PCR引物采用金斯瑞在线软件设计(www.genscript.com/ssl-bin/app/primer), 使用Script® Green Master Mix(TaKaRa), 仪器为Applied Biosystems 7500 Real-time PCR System, 用水稻Ubiqutin基因作为内参, 引物序列见表 2。反应体系为cDNA模板2.0 μL, 2×SYBR Premix Ex Taq Ⅱ 10 μL, 前引物(10 μmol · L-1)0.8 μL, 后引物(10 μmol · L-1) 0.8 μL, 50×ROX Reference Dye 0.4 μL, ddH2O 6.0 μL。扩增程序:95 ℃预热变性30 s, 95 ℃变性5 s, 60 ℃退火扩增34 s, 共进行40次循环; 熔解曲线绘制过程:95 ℃变性15 s, 60 ℃退火1 min、95 ℃变性15 s, 使用2-ΔΔCT进行数据处理。
‘日本晴’幼苗在30 ℃培养箱生长至10 cm左右, 取幼苗叶鞘切成1 mm长, 放入50 mL灭菌锥形瓶, 1 g叶鞘加入酶解液2.5 mL(1.5%纤维素酶和0.3%离析酶), 抽真空45 min后置于恒温摇床(40 r · min-1, 28 ℃)避光酶解4 h。将酶解液过滤转移至50 mL离心管中, 加入等体积W5溶液(含150 mmol · L-1 NaCl, 125 mmol · L-1CaCl2, 5 mmol · L-1KCl, 2 mmol · L-1吗啉乙磺酸), 150 g离心5 min收集原生质体。配制20 mL蔗糖溶液(18 mL水+4 g蔗糖), 将原生质体悬浮于蔗糖溶液上面, 120~130 g离心6~7 min。收集原生质体, W5溶液洗净后用重悬液重悬备用[27]。
1.9 原生质体的转化和亚细胞定位在离心管中加10 μL质粒DNA(将野生型HTR基因的全长编码序列, 保留终止密码子, 采用双切、双连的方法连接到瞬时表达载体pAN580GFP上), 加入100 μL原生质体并混匀, 再加入220 μL 40%聚乙二醇, 混匀; 28 ℃放置15~20 min, 加入880 μL W5溶液, 小心混匀; 150 g离心3 min, 将沉淀用200 μL W5溶液重悬, 28 ℃黑暗培养过夜。次日使用Zeiss LSM780激光共聚焦显微镜观察。
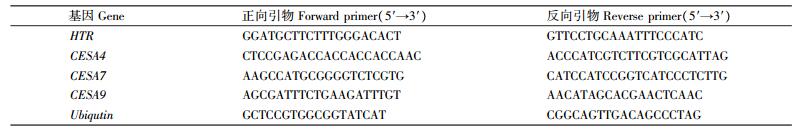
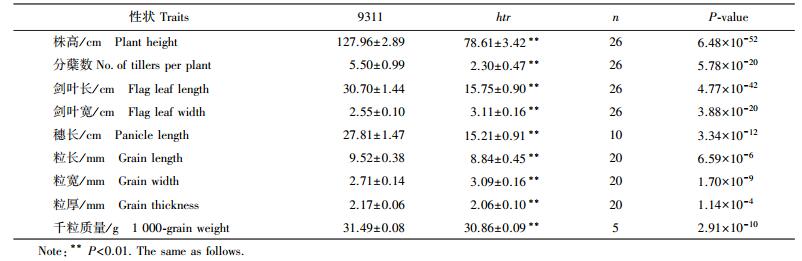
2 结果与分析 2.1 水稻矮秆突变体htr表型及农艺性状考察与‘9311’相比, htr植株出现明显矮化表型, 成熟期株高仅为野生型株高的61%(图 1-A, 表 3)。htr各节间及穗与‘9311’相比, 均有所缩短, 倒1、2、3、4、5节间分别为‘9311’长度的52.8%、60.5%、74.1%、94.9%、55.9%(图 1-B、C、D), 以倒1节间缩短最为严重, 因此htr为“sh-”类型矮秆突变体[6-7]。除了株高降低以外, htr其他农艺性状也发生改变, 分蘖数极显著降低, 仅为野生型的41.8%, htr粒长、粒宽、粒厚、千粒质量等指标均显著下降(表 3), htr的生育期与‘9311’基本一致。
|
图 1 野生型‘9311’和突变体htr成熟期的表型比较 Figure 1 Phenotypic comparison of the wild type'9311'and mutant htr at the mature stage A:‘9311’和htr成熟期植株形态比较, 标尺为15 cm; B:‘9311’和htr成熟期的穗和各节间长度比较, 标尺为4 cm; C, D:‘9311’和htr成熟期的穗及各节间长度模型, P代表穗, Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ、Ⅶ分别代表倒1、2、3、4、5、6、7节间(n=10)。 A:Gross morphology of'9311'and htr at the mature stage, Bar=15 cm; B:Gross length of the panicles and the internodes of'9311'and htr at the mature stage, Bar=4 cm; C and D:Diagram of the length of the panicles and internodes of'9311'and htr at the mature stage, P indicates the panicle, andⅠ, Ⅱ, Ⅲ, Ⅳ, Ⅴ, Ⅵ, Ⅶ indicate seven internodes, respectively(n=10). |
|
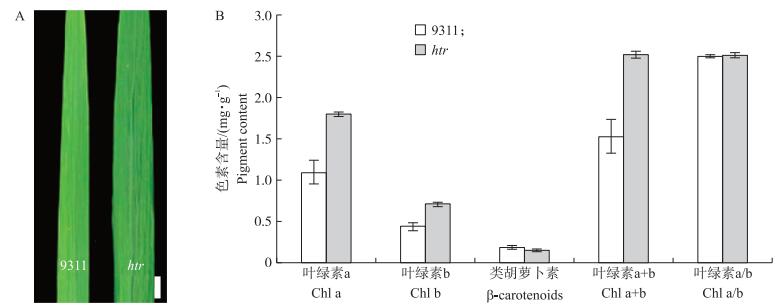
在抽穗期htr剑叶的宽度超过野生型, 并呈现深绿色(图 2-A, 表 3)。叶绿素含量测定表明, htr叶绿素a、叶绿素b含量显著高于野生型, 但叶绿素a/b比值在两者之间没有明显差异(图 2-B)。
|
图 2 野生型‘9311’和突变体htr抽穗时期的剑叶比较 Figure 2 Comparison of the wild type'9311'and mutant htr flag leaves at heading stage A:‘9311’和htr抽穗时期剑叶比较, 标尺为2 cm; B:‘9311’和htr抽穗时期剑色素含量(n=3)。 A:Gross flag leaf of'9311'and htr at heading stage, Bar=2 cm; B:Pigment content of'9311'and htr at heading stage(n=3). |
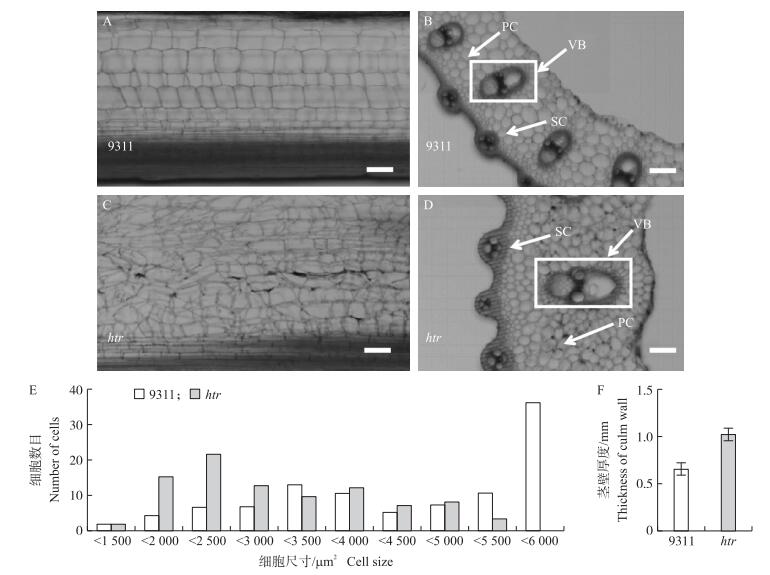
前人的研究表明, 水稻节间的伸长是居间分生组织的细胞分裂和细胞纵向伸长共同作用的结果。当植株从营养生长向生殖生长过渡后, 节间居间分生组织的细胞迅速分裂, 在纵向上形成细胞列, 细胞列中的细胞纵向伸长, 从而使节间伸长[28]。为了探索htr节间缩短的原因, 对htr和‘9311’成熟期倒二节间进行切片观察(图 3)。茎节的纵切面显示‘9311’细胞呈规则的长方形, 排列整齐(图 3-A), 而htr细胞形状不规则, 排列散乱, 未能形成规则的细胞排列, 且在茎的纵轴方向未能伸长(图 3-B), 这是htr茎间变短的直接原因。
|
图 3 ‘9311’和突变体htr的茎秆切片观察 Figure 3 Section analysis of culm cells of'9311'and mutant htr A, C:‘9311’和htr茎秆的纵切面, 标尺为100 μm; B, D:‘9311’和htr茎秆的横切面, 标尺为100 μm(SC:后壁细胞; VB:维管束; PC:薄壁细胞); E:‘9311’和htr横切面的薄壁细胞尺寸的统计(n=125);F:‘9311’和htr茎壁厚度(n=10)。 A, C:Longitudinal section of the internodes of'9311'and htr, Bars=100 μm; B, D:Transverse sections of the internodes of'9311'and htr, Bars=100 μm(SC:Sclerenchyma cells; VB:Vascular bundle; PC:Parenchyma cells); E:Statistics on the size of parenchyma cells of'9311'and htr(n=125);F:Thickness of the culm wall of the internodes of'9311'and htr(n=10). |
‘9311’的茎横切面由厚壁组织、维管束和薄壁组织构成, 薄壁细胞正常分布于维管束鞘周边(图 3-C), 但htr的薄壁细胞数增多, 细胞横截面积减小(图 3-D)。在htr和‘9311’相同部位茎的横切面随机选取各125个薄壁细胞进行统计, 结果显示htr横截面积变小的细胞数相比‘9311’增多, 说明htr薄壁细胞变小(图 3-E)。对倒二节间茎壁厚度进行测量, htr的茎壁厚度显著大于‘9311’(图 3-F)。这些结果表明htr茎壁厚度增加是由于薄壁细胞数在横向上增多, 最终表现为茎秆变粗。
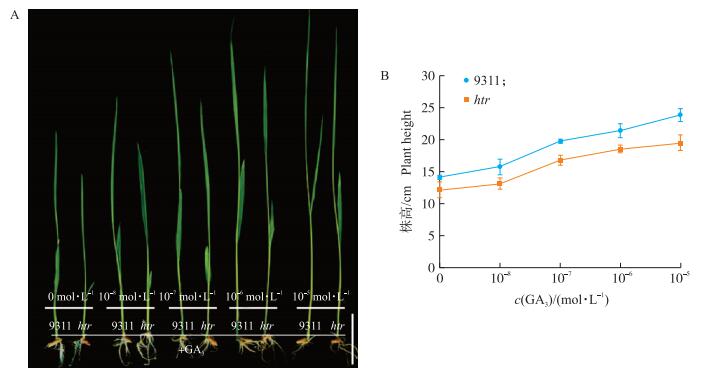
2.3 htr对赤霉素(GA)的敏感性已报道的水稻矮秆突变体, 大多涉及GA途径, 如sd 1、gid2、dgl1等。为了探究HTR是否参与GA途径, 对‘9311’和htr进行不同浓度外源GA处理。由图 4可以看出:10-8 mol · L-1外源GA3处理时, htr和‘9311’均有响应, 并且随着外源GA3浓度增加, htr和‘9311’的响应程度也随之增加, 且增加幅度基本一致。上述结果表明, 基因的突变不影响htr对外源GA的响应。因此, htr不是GA相关突变体。
|
图 4 GA3处理‘9311’和htr的幼苗表型(A)及株高(B) Figure 4 Seedling phenotype(A)and plant height(B)of '9311'and htr with GA3 treatment n=3, Bar=3 cm |
为了研究htr遗传特性, 将htr和‘9311’配制了正反交组合, F1代植株株高均正常。在正交和反交2个F2群体中植株正常与矮化分离符合3 : 1, 表明htr矮化受1对隐性核基因的控制(表 4)。

|
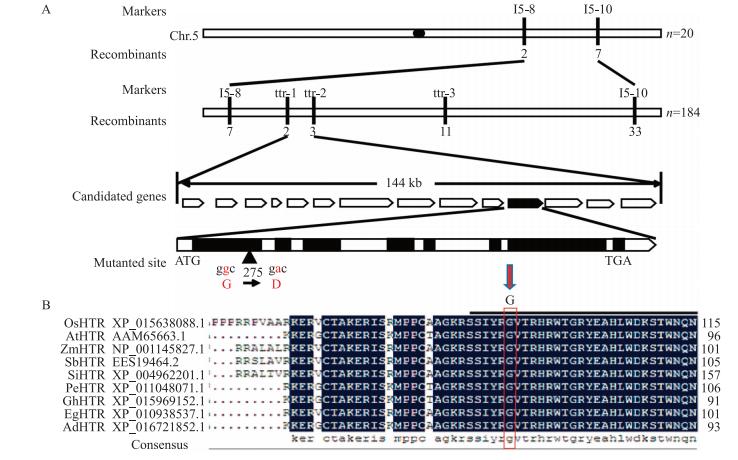
利用htr/‘N22’杂交组合获得F2群体, 挑选20个极端矮化个体进行初步连锁分析, 结果仅发现1处连锁位点, 第5染色体长臂上2个Indel分子标记I5-8和I5-10与矮秆基因连锁, 初步将基因定位在这2个标记之间, 物理距离为4 967.7 kb(图 5-A)。
|
图 5 htr的图位克隆 Figure 5 Map-based cloning of htr A:HTR的物理图谱和突变位点; B:不同物种中的HTR蛋白同源序列比对(水稻OsHTR:XP_015638088.1、拟南芥AtHTR:AAM65663.1、玉米ZmHTR:NP_001145827.1、高粱SbHTR:EES19464.2、狗尾草SiHTR:XP_004962201.1、胡杨PeHTR:XP_011048071.1、蔓花生GhHTR:XP_015969152.1、油棕EgHTR:XP_010938537.1、陆地棉AdHTR:XP_016721852.1)。 A:Physical mapping and mutation site of HTR; B:Comparison of amino acid sequences of HTR(Sequence ID:Oryza sativa HTR:XP_015638088.1, Arabidopsis thaliana HTR:AAM65663.1, Zea mays HTR:NP_001145827.1, Sorghum bicolor HTR:EES19464.2, Setaria italic HTR:XP_004962201.1, Populus euphratica HTR:XP_011048071.1, Arachis duranensis HTR:XP_015969152.1, Elaeis guineensis HTR:XP_010938537.1, Gossypium hirsutum HTR:XP_016721852.1). |
在初定位基础上, 利用‘N22’和‘9311’在初定位区间内序列之间的差异设计SSR和Indel标记进行精细定位, 最终将矮秆基因定位在ttr-1和ttr-2两个Indel标记之间, 其物理距离为144 kb(图 5-A)。通过NCBI和Gramene(http://www.gramene.org/)网站预测, 该区域包括14个ORFs。这14个ORFs分别进行测序, 发现htr中仅ORF11与野生型存在差异, 在编码区第275位核苷酸存在单碱基的替换, 由碱基G突变成碱基A, 导致相应编码蛋白的第92位甘氨酸突变成天冬氨酸。
ORF11是一个已经报道的矮秆基因。SMOS 1 (Os05g0389000)[29]。SMOS 1编码一个新的AP2亚家族转录因子, 作为一个依赖生长素型调节子, 通过调控下游与细胞伸长相关基因表达, 控制器官大小。鉴于此, 将HTR确定为SMOS 1等位基因。通过比对不同物种中HTR蛋白的同源序列发现htr突变位点位于高度保守的AP2结构域部分(图 5-B), 说明该突变位点在HTR功能上具有重要作用。
2.5 HTR的亚细胞定位利用SignalP软件(http://www.cbs.dtu.dk/services/SignalP/)对HTR进行信号肽预测, 发现其C端可能存在核定位信号。因此, 将HTR的N端与GFP融合, 构建由35S启动子驱动pAN580载体。将重组质粒转入水稻原生质体, 用激光共聚焦显微镜观察HTR蛋白在水稻细胞中的定位情况。结果(图 6)显示:GFP-HTR的绿色荧光信号定位在细胞核当中, 能够与细胞核标记(DAPI)荧光重合, 说明HTR在水稻中定位于细胞核。

|
图 6 HTR蛋白的亚细胞定位 Figure 6 Subcellular localization of HTR GFP:绿色荧光蛋白; Chl:叶绿素自发荧光; Bright:明场; Merged:GFP、Chl和Bright的荧光叠加; GFP-HTR:HTR与GFP的融合蛋白; DAPI:细胞核标记, 标尺为5 μm。 GFP:Green fluorescence protein; Chl:Chlorophyll auto-fluorescence; Bright:Bright-field; Merged:Merged image of GFP, Chl and Bright; GFP-HTR:Fluorescence of HTR; DAPI:Nucleus marker. Bar=5 μm. |
为了分析HTR表达模式, 取‘9311’的不同组织进行Real-time PCR定量分析, 结果表明HTR在水稻根、茎、鞘、节、叶和穗中都有表达, 呈组成型表达, 但主要在节中表达量最高(图 7-A)。同时比较了‘9311’和htr茎秆中HTR表达水平, htr茎秆中HTR的表达量极显著下降(图 7-B), 但测序发现启动子区域与野生型相比无差异, 猜测可能是由于HTR单碱基突变, 影响了mRNA的稳定性, 或者反馈调节抑制了HTR基因的表达等, 导致表达量下降。另外, htr茎秆中薄壁细胞排列紊乱, 细胞变小, 伸长受到抑制, 而纤维素是细胞壁的主要成分, 推测htr细胞壁中纤维素的合成可能受到影响, 对已报道的参与水稻茎中纤维素合成的相关基因(如CESA 4、CESA7、CESA9)[30]进行表达分析, 发现该3个纤维素合成酶基因的表达在htr中均极显著下调(图 7-C)。
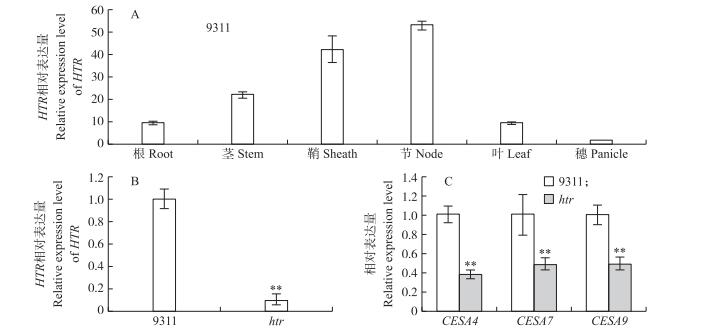
|
图 7 野生型‘9311’和htr中相关基因表达分析(n=3) Figure 7 Expression analysis of related genes in wild type'9311'and htr A:野生型不同组织中HTR的表达; B:野生型和htr茎秆中HTR表达; C:野生型与htr茎秆中纤维素合成相关基因(CESA 4、CESA7、CESA9)的表达。 A:Real-time RT-PCR analysis of the expression of HTR in different organs in wild type; B:Real-time RT-PCR analysis of the expression of HTR in stems of wild type and htr; C:Real-time RT-PCR analysis of genes(CESA 4、CESA7、CESA9)related to cellulose synthesis in stem of wild type and htr. |
目前生产上应用的矮秆基因多为sd1, 极少数利用其他矮秆基因, 如携带矮秆基因d 35 的Tan-Ginbozu[31]等, 而另外大多数矮秆基因则由于影响千粒质量、结实率和分蘖数等重要农艺性状指标, 在生产上难以直接应用, 而一些矮秆的弱等位基因突变体不良性状相对较缓和, 在水稻株型塑造上存在一定的应用价值。因此发掘新的矮秆基因并阐明影响水稻株高的分子机制, 对水稻育种而言是一项重要的工作。
本研究中, 我们通过筛选MNU化学诱变籼稻品种‘9311’的突变体库, 获得一份稳定遗传的矮秆突变体htr。经过细胞学观察, 发现其茎壁中在纵轴方向薄壁细胞变小, 未能正常伸长, 这可能是导致其株高降低的直接原因, 另外在茎壁的横切面发现薄壁细胞数目增多, 这直接导致htr的茎壁增厚, 使茎秆增粗。这些结果表明突变体茎秆中薄壁细胞的生长发育出现异常。
遗传分析发现htr矮秆性状受1对隐性单基因控制。进一步通过图位克隆, 我们发现htr突变体中编码1个已经报道的SMOS 1基因发生了单碱基替换, 导致编码的保守氨基酸发生改变。SMOS1编码1个不常见的AP2类转录因子, 带有1个不完整的AP2结构域, 通过生长素依赖形式调节细胞伸长, 从而调控器官大小[29]。此外, htr和smos 1在植株表型上很相似, 都出现株高显著降低, 穗及各节间均有不同程度缩短的现象, 因此, 我们推定HTR是SMOS1的等位基因。
通过对比基因的突变位点可以发现, smos1突变位点位于第7和6外显子上, 而这些突变最终都导致该基因翻译提前终止。本研究中, HTR在第1外显子上发生单碱基替换, 并没有导致翻译的提前终止。因此, htr和smos1在突变位点及突变后对转录造成的影响均存在差异。进一步利用水稻原生质体亚细胞定位发现, 有功能的带有GFP标签的HTR定位在细胞核里, 支持了HTR是转录因子。
前人研究中, AP2亚族转录因子主要参与植物发育调控, 如:拟南芥ANT转录因子和玉米中的IDS1转录因子都是AP2亚族的成员[32-33]。ANT参与调控花器官和胚珠的发育, ANT基因突变导致花器官数目减少, 形状改变或形成嵌合花器官, 珠壁不能正常形成, 胚囊的发育受到抑制[34]。IDS1决定玉米穗状花序分生组织的发育及成花的数量, IDS1基因突变导致玉米花的分生组织发育失控[35]。对拟南芥ant和玉米ids 1突变体进行了分析, 二者均存在花粉或柱头异常现象, 而本研究中htr突变体的花粉和柱头并无发育缺陷, 另外HTR的翻译提前终止突变体smos1也没有出现类似缺陷[29], 因此, 我们猜测AP2亚家族水稻中HTR与拟南芥ANT、玉米IDS1功能可能存在差异。
AP2亚族蛋白的DNA结合序列, 通过拟南芥的ANT蛋白得以阐明。利用体外筛选的方法(in vitro selection procedure), 证明ANT蛋白的DNA结合序列是5′-g CAC(A/G)N(A/T)Tc CC(a/g)ANG(c/t)-3′。ANT蛋白的2个AP2/ERF结构域必须同时作用于这一序列, 任何一个AP2/ERF结构域都不能与这一序列单独结合[34]。另外在AP2结构域的N-端部分存在一个碱性亲水区, 含有3个反向平行的β-折叠, 这3个折叠在识别顺式作用元件中具有极其重要的作用[35]。在本文中, 我们通过对编码HTR蛋白的氨基酸序列分析, 发现其包含2个AP2/ERF结构域, 而突变位点正好位于第1个AP2/ERF结构域部分, 这可能使其无法与目标DNA序列结合, 影响下游与细胞伸长相关基因表达, 进而导致细胞的形态发生改变。这与拟南芥中TINY突变引起细胞的形状和大小出现异常的情况类似[36]。结合前面细胞学观察结果:茎秆中薄壁细胞发育出现异常, 因此我们猜测突变体茎秆中纤维素合成可能受到影响, 于是对之前水稻中已经报道的次生壁生物合成基因CESA 4、CESA7和CESA9 [30]进行定量分析, 结果表明突变体中这3个基因表达均下调, 这可能使突变体茎秆中薄壁细胞的细胞壁形成和扩增受到影响, 导致薄壁细胞伸长受到抑制。
水稻高产育种的发展趋势是适当提高株高, 如近年来随着超级杂交稻的推广, 水稻的株高已从矮秆、半矮秆向高半矮秆发展, 增加生物学产量从而相应提高水稻产量, 但株高的提高增加了水稻植株倒伏的可能性[37]。茎秆的机械强度是重要的育种选择指标, 而茎秆的机械强度主要由茎秆直径、茎壁厚度和维管束所决定[38]。在本研究中, 虽然htr的一些主要农艺性状指标较野生型有所降低, 但结合其茎秆变粗、变短和茎壁变厚特征, 另外根据前人研究htr等位突变体smos1的茎秆抗倒伏能力提升结果[39], 最终我们认为htr茎秆机械强度增加, 提升了植株的抗倒伏能力。因此, 本研究对培育厚壁粗秆抗倒伏水稻新品种提供了重要的理论参考。
农业部长江中下游粳稻生物学与遗传育种重点实验室/长江流域杂交水稻协同创新中心/江苏省现代作物生产中心对本研究给予资助, 谨致谢意。
| [1] | Sasaki A, Ashikari M, Ueguchi-Tanaka M, et al. A mutant gibberellin-synthesis gene in rice[J]. Nature, 2002, 416(6882): 701–702. DOI: 10.1038/416701a |
| [2] | Peng J, Richards D E, Hartley N M, et al. 'Green revolution'genes encode mutant gibberellin response modulators[J]. Nature, 1999, 400(6741): 256–261. DOI: 10.1038/22307 |
| [3] |
朱立宏. 关于我国水稻高产育种的我见[J].
南京农业大学学报, 2007, 30(1): 129–135.
Zhu L H. Some critical consideration on rice high-yielding breeding in china[J]. Journal of Nanjing Agricultural University, 2007, 30(1): 129–135. DOI: 10.7685/j.issn.1000-2030.2007.01.026(in Chinese with English abstract) |
| [4] |
曹建, 孙学辉, 路铁刚. 一份水稻矮秆小粒突变体的形态特征和基因定位[J].
生物技术进展, 2014, 4(3): 186–191.
Cao J, Sun X H, Lu T G, et al. Morphological analysis and gene mapping of a rice small grain and dwarf mutant[J]. Current Biotechnology, 2014, 4(3): 186–191. (in Chinese with English abstract) |
| [5] | Sunohara H, Satoh H, Nagato Y. Mutations in panicle development affect culm elongation in rice[J]. Breeding Science, 2003, 53(2): 109–117. DOI: 10.1270/jsbbs.53.109 |
| [6] |
马良勇, 包劲松, 李西明, 等. 水稻矮生基因的克隆和功能研究进展[J].
中国水稻科学, 2009, 23(1): 1–11.
Ma L Y, Bao J S, Li X M, et al. Progress on cloning and functional analysis of dwarfism related genes in rice[J]. Chin J Rice Sci, 2009, 23(1): 1–11. (in Chinese with English abstract) |
| [7] | Sasaki A, Itoh H, Gomi K, et al. Accumulation of phosphorylated repressor for gibberellin signaling in an F-box mutant[J]. Science, 2003, 299(5614): 1896–1898. DOI: 10.1126/science.1081077 |
| [8] | Komorisono M. Analysis of the rice mutant dwarf and gladius leaf 1:aberrant katanin-mediated microtubule organization causes up-regulation of gibberellin biosynthetic genes independently of gibberellin signaling[J]. Plant Physiology, 2005, 138(4): 1982–1993. DOI: 10.1104/pp.105.062968 |
| [9] | Yin H, Gao P, Liu C, et al. SUI-family genes encode phosphatidylserine synthases and regulate stem development in rice[J]. Planta, 2013, 237(1): 15–27. DOI: 10.1007/s00425-012-1736-5 |
| [10] | Tanabe S. A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant, dwarf11, with reduced seed length[J]. Plant Cell, 2005, 17(3): 776–790. DOI: 10.1105/tpc.104.024950 |
| [11] | Hong Z, Ueguchi-Tanaka M, Shimizu-Sato S, et al. Loss-of-function of a rice brassinosteroid biosynthetic enzyme, C-6 oxidase, prevents the organized arrangement and polar elongation of cells in the leaves and stem[J]. Plant Journal, 2002, 32(4): 495–508. DOI: 10.1046/j.1365-313X.2002.01438.x |
| [12] | Hu X, Qian Q, Xu T, et al. The U-box E3 ubiquitin ligase TUD1 functions with a heterotrimeric G α subunit to regulate brassinosteroid-mediated growth in rice[J]. PLoS Genet, 2013, 9(3): e1003391. DOI: 10.1371/journal.pgen.1003391 |
| [13] | Song Y, You J, Xiong L. Characterization of OsIAA1 gene, a member of rice Aux/IAA family involved in auxin and brassinosteroid hormone responses and plant morphogenesis[J]. Plant Molecular Biology, 2009, 70(3): 297–309. DOI: 10.1007/s11103-009-9474-1 |
| [14] | Song Y, Xu Z F. Ectopic overexpression of an AUXIN/INDOLE-3-ACETIC ACID(Aux/IAA)gene OsIAA4 in rice induces morphological changes and reduces responsiveness to auxin[J]. International Journal of Molecular Sciences, 2013, 14(7): 13645–13656. DOI: 10.3390/ijms140713645 |
| [15] | Kant S, Bi Y M, Zhu T, et al. SAUR39, a small auxin-up RNA gene, acts as a negative regulator of auxin synthesis and transport in rice[J]. Plant Physiology, 2009, 151(2): 691–701. DOI: 10.1104/pp.109.143875 |
| [16] |
陈峰, 林红珍, 周起先, 等. 水稻矮秆基因的研究进展[J].
山东农业科学, 2013, 45(9): 127–133.
Chen F, Lin H Z, Zhou Q X, et al. Research progress of dwarf genes in rice[J]. Shandong Agricultural Sciences, 2013, 45(9): 127–133. (in Chinese with English abstract) |
| [17] | Dietz K J, Vogel M O, Viehhauser A. AP2/EREBP transcription factors are part of gene regulatory networks and integrate metabolic, hormonal and environmental signals in stress acclimation and retrograde signalling[J]. Protoplasma, 2010, 245(1/2/3/4): 3–14. |
| [18] | Sakuma Y, Liu Q, Dubouzet J G, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration-and cold-inducible gene expression[J]. Biochemical and Biophysical Research Communications, 2002, 290(3): 998–1009. DOI: 10.1006/bbrc.2001.6299 |
| [19] | Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiol, 2006, 140(2): 411–432. DOI: 10.1104/pp.105.073783 |
| [20] | Kim S, Soltis P S, Wall K, et al. Phylogeny and domain evolution in the APETALA2-like gene family[J]. Mol Biol Evol, 2006, 23(1): 107–120. |
| [21] | Shigyo M, Hasebe M, Ito M. Molecular evolution of the AP2 subfamily[J]. Gene, 2006, 366(2): 256–265. DOI: 10.1016/j.gene.2005.08.009 |
| [22] |
张计育, 王庆菊, 郭忠仁. 植物AP2/ERF类转录因子研究进展[J].
遗传, 2012, 34(7): 835–847.
Zhang J Y, Wang Q J, Guo Z R. Progresses on plant AP2/ERF transcription factors[J]. Hereditas, 2012, 34(7): 835–847. (in Chinese with English abstract) |
| [23] | Qi W, Sun F, Wang Q, et al. Rice ethylene-response AP2/ERF factor OsEATB restricts internode elongation by down-regulating a gibberellin biosynthetic gene[J]. Plant Physiol, 2011, 157(1): 216–228. DOI: 10.1104/pp.111.179945 |
| [24] | Yaish M W, El-Kereamy A, Zhu T, et al. The APETALA-2-like transcription factor OsAP2-39 controls key interactions between abscisic acid and gibberellin in rice[J]. PLoS Genet, 2010, 6(9): e1001098. DOI: 10.1371/journal.pgen.1001098 |
| [25] | Kitomi Y, Ito H, Hobo T, et al. The auxin responsive AP2/ERF transcription factor CROWN ROOTLESS5 is involved in crown root initiation in rice through the induction of OsRR1, a type-A response regulator of cytokinin signaling[J]. Plant Journal, 2011, 67(3): 472–484. DOI: 10.1111/j.1365-313X.2011.04610.x |
| [26] | Lichtenthaler H K. Chlorophylls and carotenoids:pigments of photosynthetic biomembranes[J]. Methods in Enzymology, 1987, 148(1): 350–382. |
| [27] |
蒋悦, 孙娟, 韩思迪, 等. 水稻黄叶突变体yl的遗传分析与基因定位[J].
南京农业大学学报, 2016, 39(6): 889–897.
Jiang Y, Sun J, Han S D, et al. Genetic analysis and gene mapping of a yellow leaf mutant yl in rice[J]. Journal of Nanjing Agricultural University, 2016, 39(6): 889–897. DOI: 10.7685/jnau.201604004(in Chinese with English abstract) |
| [28] | Wang Y, Li J. Molecular basis of plant architecture[J]. Annual Review of Plant Biology, 2008, 59(59): 253–279. |
| [29] | Aya K, Hobo T, Sato-Izawa K, et al. A novel AP2-type transcription factor, SMALL ORGAN SIZE1, controls organ size downstream of an auxin signaling pathway[J]. Plant Cell Physiol, 2014, 55(5): 897–912. DOI: 10.1093/pcp/pcu023 |
| [30] | Huang D, Wang S, Zhang B, et al. A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice[J]. Plant Cell, 2015, 27(6): 1681–1696. DOI: 10.1105/tpc.15.00015 |
| [31] | Itoh H, Tatsumi T, Sakamoto T, et al. A rice semi-dwarf gene, Tan-Ginbozu(D35), encodes the gibberellin biosynthesis enzyme, ent-kaurene oxidase[J]. Plant Mol Biol, 2004, 54(4): 533–547. DOI: 10.1023/B:PLAN.0000038261.21060.47 |
| [32] | Elliott R C, Betzner A S, Huttner E, et al. AINTEGUMENTA, an APETALA2-like gene of Arabidopsis with pleiotropic roles in ovule development and floral organ growth[J]. Plant Cell, 1996, 8(2): 155–168. DOI: 10.1105/tpc.8.2.155 |
| [33] | Chuck G, Meeley R B, Hake S. The control of maize spikelet meristem fate by the APETALA2-like gene indeterminate spikelet1[J]. Genes Dev, 1998, 12(8): 1145–1154. DOI: 10.1101/gad.12.8.1145 |
| [34] | Krizek B A. Ectopic expression of AINTEGUMENTA in Arabidopsis plants results in increased growth of floral organs[J]. Dev Genet, 1999, 25(3): 224–236. DOI: 10.1002/(ISSN)1520-6408 |
| [35] | Allen M D, Yamasaki K, Ohme-Takagi M, et al. A novel mode of DNA recognition by a beta-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA[J]. EMBO J, 1998, 17(18): 5484–5496. DOI: 10.1093/emboj/17.18.5484 |
| [36] | Wilson K, Long D, Swinburne J, et al. A dissociation insertion causes a semidominant mutation that increases expression of TINY, an Arabidopsis gene related to APETALA2[J]. Plant Cell, 1996, 8(4): 659–671. DOI: 10.1105/tpc.8.4.659 |
| [37] |
王燚, 魏中伟, 邵平, 等. 水稻茎秆特性与抗倒伏关系的研究现状[J].
湖北农业科学, 2015, 54(9): 2054–2057.
Wang Y, Wei Z W, Shao P, et al. Research status on relationship between culm characteristics and lodging resistance of rice[J]. Hubei Agricultural Sciences, 2015, 54(9): 2054–2057. (in Chinese with English abstract) |
| [38] | Duan C R, Wang B C, Wang P Q, et al. Relationship between the minute structure and the lodging resistance of rice stems[J]. Colloids and Surfaces B Biointerfaces, 2004, 35(3/4): 155–158. |
| [39] | Hirano K, Okuno A, Hobo T, et al. Utilization of stiff culm trait of rice smos1 mutant for increased lodging resistance[J]. PLoS ONE, 2014, 9(7): e96009. DOI: 10.1371/journal.pone.0096009 |