文章信息
- 吴蓓, 李梦瑶, 王广龙, 黄蔚, 熊爱生
- WU Bei, LI Mengyao, WANG Guanglong, HUANG Wei, XIONG Aisheng
- 芹菜肉桂酰辅酶A还原酶基因的克隆与表达分析
- Cloning and the expression profiles analysis of an AgCCR gene encoding cinnamoyl-CoA reductase from different cultivars of Apium graveolens
- 南京农业大学学报, 2016, 39(6): 907-914
- Journal of Nanjing Agricultural University, 2016, 39(6): 907-914.
- http://dx.doi.org/10.7685/jnau.201603028
-
文章历史
- 收稿日期: 2016-03-16
大肠杆菌菌种DH5α保存于本实验室; 总RNA提取试剂盒(RNA simple Total RNA Kit)和DNA回收试剂盒分别购于北京天根生化科技有限公司和杭州维特洁公司; 反转录试剂盒(PrimeScript RT reagent Kit)、聚合酶Ex Taq、DL2000 DNA marker、质粒载体pMD19-T vector simple、荧光定量SYBR Premix Ex Taq试剂盒等均购自大连TaKaRa公司。
试验材料‘六合黄心芹’和‘文图拉’种植于南京农业大学作物遗传与种质创新国家重点实验室人工气候室。待植株长到2个月时分别取‘六合黄心芹’和‘文图拉’幼苗根、叶柄、叶片等为试验材料, 同时将芹菜幼苗(5~6叶龄)在4 ℃、38 ℃条件下以及200 g · L-1的PEG6000溶液和200 mmol · L-1的NaCl溶液分别处理1、2、4、8、24 h后取样, 取植株顶部3~4片叶。取样后立即用液氮速冻后, 保存于-80 ℃冰箱中, 以ddH2O处理为对照组。
1.2 RNA的提取及cDNA的合成使用总RNA提取试剂盒提取试验材料的总RNA, 利用反转录试剂盒将总RNA反转录为cDNA。
1.3 芹菜AgCCR基因的克隆根据本实验室的芹菜‘六合黄心芹’和‘文图拉’转录组数据[18], 检索并拼接出芹菜AgCCR基因序列, 根据此段序列用Primer Premier 6.0设计1对引物, 引物分别为AgCCR-F:5′-ATGCCTTCTGTTTCAGGCCAGGTTG-3′和AgCCR-R:5′-CTAAGACTGGATATGAACGCGTTG-3′。以‘六合黄心芹’和‘美国西芹’的cDNA为模板以20 μL体系(2.5 ng · μL-1 cDNA 1 μL, Ex Taq DNA聚合酶10 μL, 10 pmol · μL-1引物各加1 μL, ddH2O 7 μL)进行扩增。PCR反应程序为:94 ℃ 5 min; 94 ℃ 30 s, 54 ℃ 30 s, 72 ℃ 60 s, 35个循环; 72 ℃ 10 min。用1.2 g · L-1琼脂糖凝胶对扩增产物进行电泳回收后, 连接到pMD19-T载体并转化至大肠杆菌DH5α, 抽提质粒后送南京金斯瑞公司测序。
1.4 序列分析同源性计算利用NCBI网站BLAST程序。利用DNAMAN 6.0进行氨基酸序列比对和蛋白质疏水性及亲水性分析, 使用MEGA 5软件邻接法(neighbor-joining method)对系统进化树进行编辑, 生成报告图形。氨基酸成分、蛋白质相对分子质量和等电点分析采用ProtParam软件。蛋白质结构分析中的三级结构分析通过Swiss-Model完成。
1.5 实时定量PCR反应qRT-PCR过程中, 内参基因用芹菜TUB-B基因[19], 引物分别为F:5′-TGGTGGCACTGGATCTGGTA-TGG-3′; R:5′-ACTTTCGGAGGAGGGAAGACTGAA-3′, 与目标基因一起扩增。qRT-PCR检测引物序列为BDAgCCR-F:5′-AAGCCAAAGTTCGTCGTGTTGTTT-3′和BDAgCCR-R:5′-ACCATAGCAATACCAGTTCCTA-GTGTT-3′。目标基因与芹菜TUB-B基因每个处理都设3个重复。相对定量使用参照基因的ΔΔCT法[20]。
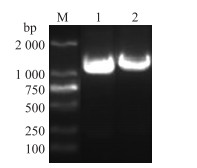
2 结果与分析 2.1 芹菜中AgCCR基因的克隆和序列分析分别以芹菜‘六合黄心芹’和‘文图拉’的cDNA为模板, 经过PCR获得了2个与预期一致的1 000 bp左右的片段(图 1)。序列测定与分析表明:源于‘六合黄心芹’和‘文图拉’的AgCCR基因均含有1个1 014 bp的开放阅读框(ORF)并编码337个氨基酸(图 2)。预测2个品种中该基因编码的氨基酸的蛋白质相对分子质量分别为37.05×103和37.00×103, 等电点分别为6.92和7.16。‘六合黄心芹’和‘文图拉’的AgCCR基因在核苷酸水平上的差异为3个位点, 分别是125位的a/g, 570位的a/g和684位的a/g。编码的氨基酸位点的差异是处于42位的D/G。
|
图 1 芹菜AgCCR基因cDNA的PCR扩增图谱 Figure 1 PCR amplification pattern of cDNA of AgCCR gene from celery M:DL2000 DNA marker; 1:六合黄心芹Liuhehuangxinqin; 2:文图拉Ventura |

|
图 2 芹菜AgCCR基因的核苷酸序列及其编码的氨基酸序列 Figure 2 Nucleotide acid and deduced amino acid sequences of AgCCR gene from celery 方框内序列为不同核苷酸酸位点及其编码的氨基酸位点。The sequences in the box are different nucleotide acid sites and the amino acid sites. |
利用BLASTp对‘六合黄心芹’和‘文图拉’AgCCR基因核苷酸序列推导的氨基酸序列进行同源性检索与比对, 结果显示AgCCR具有NADB_Rossmann保守结构域, 属于NADB_Rossmann超家族(图 3-A)。芹菜AgCCR基因编码的氨基酸序列与桉树(Eucalyptus grandis)、毛果杨(Populus trichocarpa)、葡萄(Vitis vinifera)、桃(Prunus persica)、大麦(Hordeum vulgare)、黑麦草(Lolium perenne)、水稻(Oryza sativa)、小麦(Triticumae stivum)、番茄(Solanum lycopersicum)、马铃薯(Solanum tuberosum)、茸毛烟草(Nicotiana tomentosiformis)、甜椒(Capsicum annuum)、拟南芥(Arabidopsis thaliana)、黄瓜(Cucumis sativus)、冈尼桉(Eucalyptus gunnii)的CCR蛋白氨基酸序列相似度较高。芹菜AgCCR基因编码的氨基酸序列与其他15种植物的CCR氨基酸序列进行多重比对, 结果表明芹菜与上述植物的CCR氨基酸序列具有高度保守的区域, 相似度达71.86%, 这些CCR蛋白在保守区域有4个活性位点(图 3-B)。

|
图 3 芹菜AgCCR基因编码的氨基酸序列保守域预测(A)和同源性比对(B) Figure 3 Prediction (A) and comparison (B) of the conserved domain of amino acid sequences encoded by AgCCR gene of celery 箭头所示为活性位点。The arrow marks active sites. |
对上述植物中的CCR基因编码的氨基酸组成成分及理化性质进行分析, 结果表明这些植物中肉桂酰辅酶A还原酶的氨基酸残基数为332~389, 理论相对分子质量为(36.57~40.48)×103, 理论等电点为5.89~7.52。碱性氨基酸数量多于酸性氨基酸数量, 脂肪族氨基酸数明显多于芳香族氨基酸数, 蛋白质可溶性预测中主要平均疏水特性(GRAVY)为-0.150左右(表 1)。氨基酸分析结果中出现的差异可能源于非保守区域的氨基酸差异。
| 植物 Plants | 氨基酸 残基数 Number of amino acid | 理论相对 分子质量/103 Theoretical relative molecular mass | 理论 等电点 Theoretical pI | 碱性氨基 酸比例/% Ratio of positive amino acid | 酸性氨基 酸比例/% Ratio of negative amino acid | 脂肪族氨基 酸比例/% Ratio of aliphatic amino acid | 芳香族氨基 酸比例/% Ratio of aromatics amino acid | 总平均 疏水性 Grand average of hydrophobicity |
| 六合黄心芹Liuhehuangxinqin | 337 | 37.05 | 6.92 | 13 | 11 | 24 | 7 | -0.161 |
| 文图拉Ventura | 337 | 37.00 | 7.16 | 13 | 11 | 24 | 7 | -0.152 |
| 桉树E.grandis | 375 | 40.48 | 7.16 | 13 | 11 | 23 | 6 | -0.206 |
| 毛果杨P.trichocarpa | 338 | 37.14 | 6.18 | 13 | 12 | 23 | 8 | -0.191 |
| 葡萄V.vinifera | 338 | 37.23 | 6.42 | 14 | 12 | 25 | 7 | -0.130 |
| 桃P.persica | 389 | 43.06 | 7.52 | 14 | 12 | 23 | 8 | -0.237 |
| 大麦H.vulgare | 348 | 37.72 | 6.59 | 14 | 12 | 24 | 7 | -0.111 |
| 黑麦草L.perenne | 362 | 39.14 | 5.89 | 13 | 13 | 24 | 6 | -0.101 |
| 水稻O.sativa | 361 | 38.66 | 5.94 | 12 | 12 | 23 | 6 | -0.034 |
| 小麦T.aestivum | 349 | 37.38 | 6.51 | 13 | 12 | 23 | 6 | -0.087 |
| 番茄S.lycopersicum | 332 | 36.79 | 7.10 | 14 | 12 | 24 | 8 | -0.180 |
| 马铃薯S.tuberosum | 332 | 36.80 | 7.39 | 14 | 12 | 24 | 8 | -0.175 |
| 茸毛烟草N.tomentosiformis | 332 | 36.80 | 6.92 | 14 | 12 | 24 | 8 | -0.191 |
| 甜椒C.annuum | 334 | 36.96 | 7.11 | 14 | 12 | 24 | 8 | -0.192 |
| 拟南芥A.thaliana | 332 | 36.62 | 6.86 | 13 | 12 | 24 | 8 | -0.166 |
| 黄瓜C.sativus | 338 | 36.99 | 6.24 | 13 | 12 | 24 | 7 | -0.140 |
| 冈尼桉E.gunnii | 336 | 36.57 | 6.17 | 13 | 12 | 25 | 7 | -0.098 |
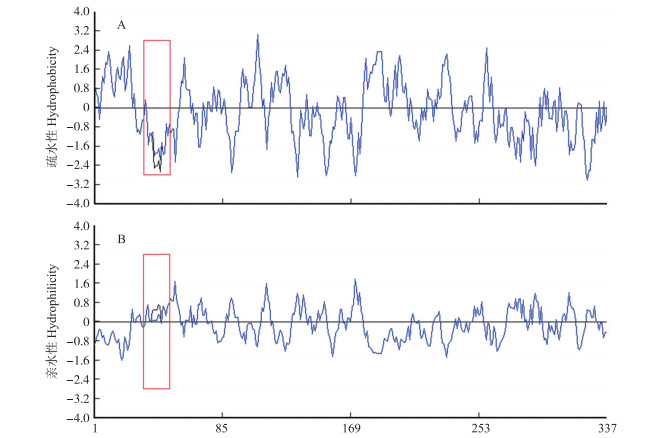
芹菜‘六合黄心芹’和‘文图拉’中克隆的AgCCR基因所推导的氨基酸序列进行了疏水性/亲水性分析。结果表明:2个品种AgCCR编码的氨基酸疏水性最强的位点均为位于第108位的谷氨酸(Glu), 亲水性最强的位点均为位于172位的甘氨酸(Gly), 2个品种AgCCR蛋白的氨基酸序列疏水性/亲水性基本一致, 均为疏水性蛋白(图 4)。
|
图 4 芹菜AgCCR氨基酸序列疏水性(A)和亲水性(B)比较 Figure 4 Comparison of hydrophobicity (A) and hydrophilicity (B) of amino acid sequences of AgCCR from celery 黑色曲线为‘六合黄心芹’; 蓝色曲线为‘文图拉’; 方框内为氨基酸差异位点。 Black bight represents'Liuhehuangxinqin'. Blue bight represents'Ventura'. The sequences in the box are different amino acid sites. |
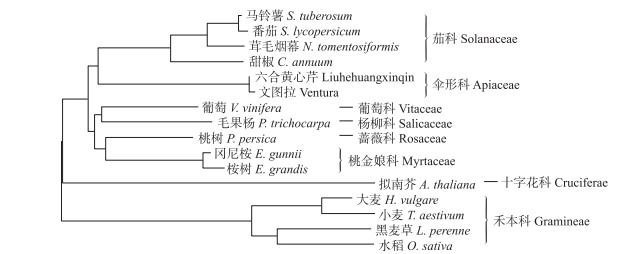
对序列比对中相似度高的14种植物的CCR蛋白和芹菜AgCCR蛋白进行同源进化分析, 并构建同源进化树, 结果(图 5)显示, 芹菜CCR蛋白与茄科植物的进化关系最近。同属禾本科的大麦、小麦、黑麦草和水稻以及同属于茄科的番茄、马铃薯、甜椒以及茸毛烟草各自聚集在同一分支中, 表明同科植物的CCR氨基酸序列相似度高, 该基因高度保守。
|
图 5 芹菜与其他植物CCR氨基酸序列的系统进化树 Figure 5 Phylogenetic tree of CCR among celery and other plant species |
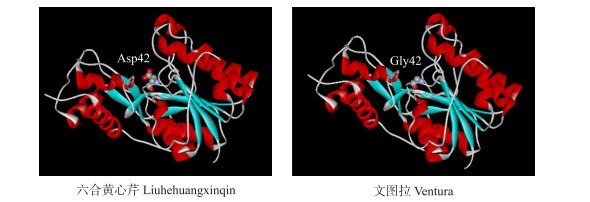
以矮牵牛肉桂酰辅酶A还原酶晶体结构(PDB, ID:4r1s.1)[21]为模型, 通过Swiss-Model对芹菜AgCCR进行三维结构同源建模。结果(图 6)显示, ‘六合黄心芹’和‘文图拉’肉桂酰辅酶A还原酶AgCCR与矮牵牛CCR在蛋白核心区域的氨基酸相似性分别为85.28%和84.97%。‘六合黄心芹’和‘文图拉’AgCCR氨基酸序列有1个氨基酸的差异, 该氨基酸位点的差异, 并没有明显改变芹菜AgCCR蛋白的三维结构。二者具有相似的三维空间结构, 均有10个α螺旋和9个β折叠。
|
图 6 芹菜AgCCR蛋白的三维结构 Figure 6 The three-dimension structure of AgCCR from celery |
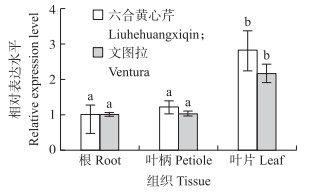
由图 7可见:AgCCR基因在‘六合黄心芹’和‘文图拉’的根、叶柄和叶片中均有表达, 呈现比较明显的组织特异性。AgCCR基因在‘六合黄心芹’和‘文图拉’叶片中表达水平最高, 而在根和叶柄中表达较少并且差异不显著。
|
图 7 芹菜AgCCR基因在不同组织中的表达水平 Figure 7 Expression level of AgCCR gene from celery in different tissues 同一品种不同字母代表在0.05水平上差异显著。 Different letters on the vertical bars indicate significant diffe-rence in the same cultivar at 0.05 level. The same as follows. |
由图 8可见:高温时, ‘六合黄心芹’中AgCCR基因表达水平在处理1 h后略有下降, 在处理8 h及以后表达水平显著增加; 而‘文图拉’中AgCCR基因在处理1 h后表达水平有所增加, 而后表达水平一直处在较低水平。低温处理下, ‘六合黄心芹’中AgCCR基因表达水平在前8 h没有显著变化, 而后显著增加; ‘文图拉’中AgCCR基因表达水平呈先升高再降低, 而后持续升高的过程。干旱处理时, ‘六合黄心芹’和‘文图拉’中AgCCR基因处理2和1 h后表达水平显著低于对照。盐胁迫下, AgCCR在2个芹菜品种中表达情况明显不同, ‘六合黄心芹’中AgCCR基因表达水平持续升高, 而‘文图拉’中AgCCR基因表达水平始终低于对照。

|
图 8 芹菜AgCCR基因在不同逆境条件下的表达水平 Figure 8 Expression level of AgCCR gene in celery under different adverse conditions |
肉桂酰辅酶A还原酶CCR是植物木质代谢中非常重要的酶, 木质素中3种单体的合成都必须经过CCR还原作用[11]。如果CCR活性受到影响, 木质素生物合成也会受到阻碍, 影响植株木质素含量[12]。多项研究表明CCR基因在植物抗逆抗病中起作用。Lauvergeat等[22]研究拟南芥中CCR基因时发现, 拟南芥被细菌感染的叶片中检测到AtCCR 2, 表明它与拟南芥的抗病有关。陈刚等[23]对不同抗白粉病南瓜品种接种白粉菌后发现高抗品种叶片中CCR基因的表达量增大。So等[24]认为大豆GmCCR基因与大豆抵抗非生物胁迫机制有关。本研究从芹菜中克隆AgCCR基因, 对其组织特异性和逆境胁迫后的表达分析对研究芹菜木质代谢和逆境响应机制有一定的参考价值。逆境胁迫会对植物的生长发育造成很大的影响, 转录因子和功能基因在植物逆境调控中起着重要的作用[5, 7, 25]。
本研究从‘六合黄心芹’和‘文图拉’中获得的芹菜AgCCR基因高度同源, 都含有1个编码337个氨基酸的开放阅读框。2个芹菜品种的核苷酸有3个位点的差异, 而所编码的氨基酸序列有1个位点的差异, 多数核苷酸位点突变属于同源突变。芹菜中AgCCR基因少数位点差异可能和芹菜某些特定生理功能有关, 也说明不同地域芹菜中AgCCR基因较为保守。
氨基酸序列同源性和进化树分析显示, ‘六合黄心芹’和‘文图拉’的AgCCR氨基酸序列与其他植物的肉桂酰辅酶A还原酶氨基酸序列高度保守, 同源性很高。进行同源比对的植物中普遍存在一段相同的保守结构域, 其序列为NWYCYGK, 可形成αβα的二级结构, 它可能与NADPH辅助因子结合, 也有可能是CCR的催化位点[26]。DNA保守域可以作为判断物种间亲缘系远近的依据[27]。在进化树上‘六合黄心芹’和‘文图拉’与茄科的茸毛烟草的进化关系最为接近, 同属茄科的马铃薯、番茄、茸毛烟草、甜椒处于同一分支, 同属禾本科的大麦、小麦、黑麦草和水稻在同一分支, 桃金娘科的2种桉树也在同一分支, 可以看出各科之间的进化关系是比较保守的。
本研究中, AgCCR基因在芹菜的根、叶柄和叶片组织中均有不同程度的表达, 其中在叶片组织中的表达明显高于其他检测组织, 表现出明显的组织特异性, 而品种间差异不大。玉米中的ZmCCR 1在茎、叶中表达量较高, 老茎的节间比嫩的节间中的ZmCCR 1基因的表达量高[28]。植物对外界环境信号的相应网络非常复杂, 逆境条件下植物可能会诱导核酸酶来修复逆境导致的基因突变[29]。经不同胁迫处理后2个芹菜品种AgCCR基因的相对表达水平有明显的差异, 其中盐处理下‘六合黄心芹’中AgCCR基因的相对表达水平显著高于对照, 而该基因在‘文图拉’中显著低于对照。本研究中, 200 g · L-1 PEG6000溶液模拟的干旱处理下2个芹菜中AgCCR基因表达均呈下降趋势, 而PEG6000处理下的玉米根部ZmCCR 2基因表达上升, 同时产生酚类物质以适应干旱胁迫[30]。干旱情况下CCR基因在芹菜和玉米中的表达差异可能与物种间差异有关, 也有可能是因为取样部位不同而产生了差异。不同逆境处理下不同芹菜品种AgCCR基因表达变化差异也可能和2种形态差异较大的芹菜品种特性有关。
| [1] | 方智远, 张武男. 中国蔬菜作物图鉴[M]. 南京: 江苏科学技术出版社, 2011 : 194 -197. Fang Z Y, Zhang W N. Pictorial Handbook of Vegetable Crops in China[M]. Nanjing: Jiangsu Science and Technology Press, 2011 : 194 -197. (in Chinese) |
| [2] | 宋小俊, 王怡婷. 芹菜药用功能研究概述[J]. 安徽农业科学, 2008, 36(15): 6360–6361. Song X J, Wang Y T. Research progress in the medicinal function of celery[J]. Journal of Anhui Agricultural Sciences, 2008,36(15): 6360–6361. (in Chinese) |
| [3] | 王武台, 古瑜, 韩启厚, 等. 芹菜种质资源亲缘关系的ISSR分析[J]. 中国蔬菜, 2011(8): 22–27. Wang W T, Gu Y, Han Q H, et al. ISSR analysis of celery germplasm genetic relationships[J]. China Vegetables, 2011(8): 22–27. (in Chinese) |
| [4] | 李梦瑶, 王枫, 侯喜林, 等. 芹菜过敏原蛋白Api g 1基因的克隆与表达分析[J]. 南京农业大学学报, 2013, 36(2): 13–19. Li M Y, Wang F, Hou X L, et al. Cloning and expression analysis of allergenic protein gene Api g 1 in celery[J]. Journal of Nanjing Agricultural University, 2013,36(2): 13–19. DOI: 10.7685/j.issn.1000-2030.2013.02.003 (in Chinese) |
| [5] | 田畅, 王枫, 蒋倩, 等. 芹菜核酸内切酶CELI基因的克隆及其表达分析[J]. 南京农业大学学报, 2014, 37(4): 39–45. Tian C, Wang F, Jiang Q, et al. Cloning and expression pattern analysis of CELI gene in celery[J]. Journal of Nanjing Agricultural University, 2014,37(4): 39–45. DOI: 10.7685/j.issn.1000-2030.2014.04.006 (in Chinese) |
| [6] | 王广龙, 王枫, 徐志胜, 等. 芹菜衔接蛋白基因AgMu2的克隆与表达分析[J]. 园艺学报, 2014, 41(7): 1369–1378. Wang G L, Wang F, Xu Z S, et al. Cloning and expression profile analysis of the AgMu2 gene of adaptor complex from celery[J]. Acta Horticulturae Sinica, 2014,41(7): 1369–1378. (in Chinese) |
| [7] | 李岩, 徐志胜, 谭国飞, 等. 芹菜热激转录因子基因AgHSFB2的克隆及不同温度处理下的表达响应[J]. 南京农业大学学报, 2015, 38(3): 360–368. Li Y, Xu Z S, Tan G F, et al. Cloning and expressional response analysis of AgHSFB2 under different temperature treatments in celery[J]. Journal of Nanjing Agricultural University, 2015,38(3): 360–368. DOI: 10.7685/j.issn.1000-2030.2015.03.002 (in Chinese) |
| [8] | 田畅, 蒋倩, 王枫, 等. 3个芹菜品种Agnp-G3PDH基因的克隆及其序列和表达特性分析[J]. 植物资源与环境学报, 2014, 23(2): 1–10. Tian C, Jiang Q, Wang F, et al. Cloning of Agnp-G3PDH gene from three cultivars of Apium graveolens and its sequence and expression characteristic analyses[J]. Journal of Plant Resources and Environment, 2014,23(2): 1–10. (in Chinese) |
| [9] | Hano C, Addi M, Bensaddek L, et al. Differential accumulation of monolignol-derived compounds in elicited flax (Linum usitatissimum) cell suspension cultures[J]. Planta, 2006,223(5): 975–989. DOI: 10.1007/s00425-005-0156-1 |
| [10] | 耿飒, 徐存拴, 李玉昌. 木质素的生物合成及其调控研究进展[J]. 西北植物学报, 2003, 23(1): 171–181. Geng S, Xu C S, Li Y C, et al. Advance in biosynthesis of lignin and its regulation[J]. Acta Botanica Boreali-Occidentalia Sinica, 2003,23(1): 171–181. (in Chinese) |
| [11] | Lacombe E, Hawkins S, Doorsselaere J V, et al. Cinnamoyl CoA reductase, the first committed enzyme of the lignin branch biosynthetic pathway:cloning, expression and phylogenetic relationships[J]. Plant Journal, 1997,11(3): 429–441. DOI: 10.1046/j.1365-313X.1997.11030429.x |
| [12] | Goujon T, Ferret V, Mila I, et al. Down-regulation of the AtCCR1 gene in Arabidopsis thaliana:effects on phenotype, lignins and cell wall degradability[J]. Planta, 2003,217(2): 218–228. |
| [13] | Piquemal J, Lapierre C, Myton K, et al. Down-regulation of cinnamoyl-CoA reductase induces significant changes of lignin profiles in transgenic tobacco plants[J]. Plant Journal, 2002,13(1): 71–83. DOI: 10.1046/j.1365-313X.1998.00014.x |
| [14] | Dobrev G, Zhekova B, Delcheva G, et al. Purification and characterization of cinnamoyl-coenzyme A:NADP oxidoreductase in Eucalyptus gunnii[J]. Plant Physiology, 1994,106(2): 625–632. DOI: 10.1104/pp.106.2.625 |
| [15] | Luderitz T, Schatz G, Grisebach H. Enzymic synthesis of lignin precursors. Purification and properties of 4-coumarate:CoA ligase from cambial sap of spruce (Picea abies L.)[J]. European Journal of Biochemistry, 1982,123(3): 583–586. |
| [16] | Sarni F, Grand C, Boudet A M. Purification and properties of cinnamoyl-CoA reductase and cinnamyl alcohol dehydrogenase from poplar stems (Populus×euramericana)[J]. European Journal of Biochemistry, 1984,139(2): 259–265. DOI: 10.1111/ejb.1984.139.issue-2 |
| [17] | Colrat S, Latche A, Guis M, et al. Purification and characterization of a NADPH-dependent aldehyde reductase from mung bean that detoxifies eutypine, a toxin from Eutypa lata[J]. Plant Physiology, 1999,119(2): 621–626. DOI: 10.1104/pp.119.2.621 |
| [18] | Jia X L, Wang G L, Xiong F, et al. De novo assembly, transcriptome characterization, lignin accumulation, and anatomic characteristics:novel insights into lignin biosynthesis during celery leaf development[J]. Scientific Reports, 2015,5: 8259. DOI: 10.1038/srep08259 |
| [19] | Li M Y, Wang F, Jiang Q, et al. Validation and comparison of reference genes for qPCR normalization of celery (Apium graveolens) at different development stages[J]. Frontiers in Plant Science, 2016,7: 313. |
| [20] | Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research, 2001,29(9): e45. DOI: 10.1093/nar/29.9.e45 |
| [21] | Haiyun P, Rui Z, Louie G V, et al. Structural studies of cinnamoyl-CoA reductase and cinnamyl-alcohol dehydrogenase, key enzymes of monolignol biosynthesis[J]. Plant Cell, 2014,26(9): 3709–3727. DOI: 10.1105/tpc.114.127399 |
| [22] | Lauvergeat V, Lacomme C, Lacombe E, et al. Two cinnamoyl-CoA reductase (CCR) genes from Arabidopsis thaliana are differentially expressed during development and in response to infection with pathogenic bacteria[J]. Phytochemistry, 2001,57(7): 1187–1195. DOI: 10.1016/S0031-9422(01)00053-X |
| [23] | 陈刚, 薛应钰, 白江平, 等. 美洲南瓜CCR基因表达特点的研究[J]. 西北植物学报, 2013, 33(5): 898–903. Chen G, Xue Y Y, Bai J P, et al. Express variation of CCR gene in different tissues of Cucurbita pepo[J]. Acta Botanica Boreali-Occidentalia Sinica, 2013,33(5): 898–903. (in Chinese) |
| [24] | So H A, Chung E, Cho C W, et al. Molecular cloning and characterization of soybean cinnamoyl CoA reductase induced by abiotic stresses[J]. Plant Pathology Journal, 2010,26(4): 380–385. DOI: 10.5423/PPJ.2010.26.4.380 |
| [25] | Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses[J]. Annual Review of Plant Biology, 2006,57: 781–803. DOI: 10.1146/annurev.arplant.57.032905.105444 |
| [26] | Lacombe E, Doorsselaere J V, Boerjan W, et al. Characterization of cis-elements required for vascular expression of the Cinnamoyl CoA Reductase gene and for protein-DNA complex formation[J]. Plant Journal, 2000,23(5): 663–676. DOI: 10.1046/j.1365-313x.2000.00838.x |
| [27] | Qin F, Masayuki K, Yoh S, et al. Regulation and functional analysis of ZmDREB2A in response to drought and heat stress in Zea mays L.[J]. Plant Journal, 2007,50(1): 54–69. DOI: 10.1111/j.1365-313X.2007.03034.x |
| [28] | Pichon M, Courbou I, Beckert M, et al. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase (CCR) and differential expression of the corresponding genes[J]. Plant Molecular Biology, 1998,38(4): 671–676. DOI: 10.1023/A:1006060101866 |
| [29] | Kazuhiko Y, Takanori K, Motoaki S, et al. DNA-binding domains of plant-specific transcription factors:structure, function, and evolution[J]. Trends in Plant Science, 2013,18(5): 267–276. DOI: 10.1016/j.tplants.2012.09.001 |
| [30] | Fan L, Linker R, Gepstein S, et al. Progressive inhibition by water deficit of cell wall extensibility and growth along the elongation zone of maize roots is related to increased lignin metabolism and progressive stelar accumulation of wall phenolics[J]. Plant Physiology, 2006,140(2): 603–612. DOI: 10.1104/pp.105.073130 |




