文章信息
- 李梦辉, 刘连瑞, 涂浩, 黄经纬, 张振超, 徐立新, 严若峰, 李祥瑞, 宋小凯. 2015.
- LI Menghui, LIU Lianrui, TU Hao, HUANG Jingwei, ZHANG Zhenchao, XU Lixin, YAN Ruofeng, LI Xiangrui, SONG Xiaokai. 2015.
- 利用gateway技术构建巨型艾美耳球虫孢子化卵囊cDNA表达文库
- Construction of a cDNA expression library of Eimeria maxima sporulated oocysts using gateway technology
- 南京农业大学学报, 38(4): 630-635
- Journal of Nanjing Agricultural University, 38(4): 630-635.
- http://dx.doi.org/10.7685/j.issn.1000-2030.2015.04.016
-
文章历史
- 收稿日期:2014-11-25
鸡球虫病是由顶复门(Apicomplexa)、孢子虫纲(Sporozoa)、艾美耳科(Eimeriidae)、艾美耳属(Eimeria)的7种球虫所引起的[1]。其中,巨型艾美耳球虫(Eimeria maxima)具有中等强度的致病性,是集约化养鸡场最常见的球虫之一[2]。目前,鸡球虫病的防治主要以药物为主,但耐药虫株的出现使药物预防效果明显降低,而且药物残留严重影响动物源性食品安全,这就需要人们去寻找新的防治途径[1, 3]。免疫预防是控制球虫病的理想方法,不仅能解决鸡球虫的耐药性问题,而且无药物残留,为生产绿色安全的肉制品提供了保障[1, 4]。
传统的球虫活疫苗对鸡有一定的潜在致病力,还有易散毒、成本高等缺点[4]。DNA疫苗作为第3代疫苗,具有安全性高、无残留、易保存、成本低等优点[5]。DNA疫苗除了能够诱导体液免疫反应外,还能诱导有效的细胞免疫反应,而细胞免疫在抗球虫免疫中起主导作用[4]。但要研制高效的抗球虫DNA疫苗,筛选具有良好免疫原性的候选抗原基因显得尤为迫切与重要[4, 5, 6]。在理论上,cDNA文库包括了生物体某一发育阶段的所有可表达的遗传信息,在研究具体某类特定细胞中基因组的表达状态及表达基因的功能鉴定方面具有特殊的优势[7]。因此,构建高质量的巨型艾美耳球虫孢子化卵囊cDNA表达文库有助于寻找到有效的免疫保护性候选基因。
gateway技术是利用细菌λ噬菌体的整合酶-切除酶反应系统所创建的一种体外特异位点重组反应技术[8, 9],其在构建cDNA文库方面已经得到了广泛应用[10, 11, 12, 13]。在鸡球虫方面,张建哲等[14]利用SMART技术构建了巨型艾美耳球虫cDNA文库并分析其表达序列标签,但未见利用gateway技术构建巨型艾美耳球虫cDNA表达文库的相关报道。本试验以巨型艾美耳球虫孢子化卵囊为材料,利用gateway技术构建cDNA初级文库,再将cDNA重组到真核表达载体pVAX1.0上,从而构建了巨型艾美耳球虫孢子化卵囊cDNA表达文库,为进一步筛选巨型艾美耳球虫免疫保护性基因奠定了基础,对于研究抗巨型艾美耳球虫疫苗具有重要意义。
1 材料与方法 1.1 材料 1.1.1 虫株巨型艾美耳球虫江苏株,由南京农业大学动物医学院寄生虫学教研室单卵囊分离建株,保存于25 g · L-1重铬酸钾溶液中,每3个月复壮1次。
1.1.2 主要试剂Trizol试剂、FastTrack® MAG mRNA提取试剂盒、CloneMinerTM Ⅱ cDNA文库构建试剂盒(含载体pDONRTM222和Electro MAXTMDH10B感受态细胞等)、PureLink® HiPure Plasmid Filter Midiprep Kit、真核表达载体pVAX1.0均购于Invitrogen公司。卡那霉素购于Sigma公司。
1.2 方法 1.2.1 卵囊分离及其种类鉴定将实验室保存的巨型艾美耳球虫孢子化卵囊感染鸡,收集感染后144~192 h的粪便,用饱和NaCl溶液浓度漂浮法分离卵囊。得到的卵囊加入25 g · L-1重铬酸钾溶液,25~29 ℃下培养至少30 h,进行孢子化。待孢子化率达80%以上时,参照麦克马斯特法[15]计算卵囊的总量。对100个孢子化卵囊进行形态观察和大小测定。随机从卵囊液中取出约105个卵囊,用Stool DNA试剂盒提取DNA以进行ITS1-PCR鉴定[16],其余卵囊置于4 ℃保存。
1.2.2 总RNA提取及mRNA分离取约1×108个孢子化卵囊,用含1 g · L-1 DEPC(焦碳酸二乙酯Diethy pyrocarbonate)的蒸馏水离心洗涤,去除重铬酸钾,卵囊沉淀由液氮研磨成粉末,随后转入50 mL RNase free离心管中,再加入20 mL的Trizol试剂(Trizol与粉末体积比大于4 ∶ 1),按照Trizol法提取卵囊总RNA。用mRNA提取试剂盒对总RNA进行分离纯化。用10 g · L-1的甲醛变性胶电泳检测所提取RNA的完整性和mRNA的大小分布范围,用Thermo nanodrop核酸分析仪检测总RNA和mRNA的浓度。
1.2.3 cDNA初级文库的构建cDNA双链的合成及接头的连接:取纯化的mRNA,加入biotin-attB 2 -Oligo(dT)Primer、5×First Strand Buffer、DTT、dNTPs、SuperScript Ⅲ反转录酶,合成cDNA第一链,之后向第一链合成反应液中加入5×Second Strand Buffer、Escherichia coli DNA连接酶、E.coli DNA聚合酶Ⅰ、E.coli RNaseH等试剂合成cDNA第二链,接着加入5×Adapter Buffer、attB 1 Adapter、DTT、T4 DNA连接酶等试剂连接上attB 1 重组接头。
cDNA分级分离及收集:待cDNA分级分离柱中储存液流尽,用0.8 mL TEN Buffer重复清洗柱子3次。准备好3个新的1.5 mL离心管,标记1~3。将连接上重组接头的cDNA产物于70 ℃、10 min灭活连接酶,然后,将其混匀后加入柱子中,先收集滤液于1号管,滴完后再加入100 μL TEN Buffer,继续收集滤液于1号管。滴完后,再加入240 μL TEN Buffer,收集滤液于2号管。全部滴完后,再加入80 μL TEN Buffer,收集滤液于3号管。
BP(attB×attP)重组反应:取收集到的大片段cDNA,加入pDONRTM 222和Clonase® Ⅱ enzyme Mix,进行BP重组反应,然后将重组反应产物用电击转化法转入大肠杆菌DH10B感受态细胞,然后在37 ℃、250 r · min-1的条件下培养1 h,即得到初级文库菌液。从初级文库菌液中取10 μL,按照1 ∶ 1 000的比例稀释后,取50 μL稀释的菌液涂布于直径90 mm含氨苄青霉素抗性的LB平板上,14 h后计数平板上的克隆数用于库容量的鉴定,剩余菌液加入甘油(终质量浓度200 g · L-1)保存于-80 ℃。文库滴度的计算公式:文库滴度=(平板上克隆数×稀释倍数)/涂板体积。文库滴度再乘以文库总体积得出文库的总容量。随机挑取24个克隆进行PCR鉴定。引物(pDONR222-F/R)为:5′-TCCCAGTCACGACGTTGTAAAACGACGGCCAGTCTT-3′/5′-AGAG-CTGCCAGGAAACAGCTATGACCATGTAATACGACTC-3′。
PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,35个循环;72 ℃ 5 min。反应结束后用10 g · L-1的琼脂糖凝胶电泳检测插入片段大小和阳性克隆的重组率。
1.2.4 cDNA表达文库的构建pVAX1.0载体的gateway技术改造:将载体pVAX1.0加入重组位点元件ccdB基因,改造成为gateway兼容载体pVAX1-DEST。
LR(attL×attR)重组反应:取包含有5×106~1×107个阳性克隆的初级文库菌液接种到100 mL含有卡那霉素(终质量浓度50 μg · mL-1)的肉汤培养液中,在30 ℃、250 r · min-1的条件下摇菌培养至A600为1.0。使用PureLink® HiPure Plasmid Filter Midiprep Kit提取初级文库质粒,将得到的初级文库质粒稀释至300 ng · μL-1,然后取出1 μL向其中加入pVAX1-DEST、LR Clonase® Ⅱ Mix、ddH2O进行LR重组反应。将重组反应产物用电击转化法转入大肠杆菌DH10B感受态细胞,在37 ℃、250 r · min-1的条件下培养1 h,即得到 表达文库菌液。参照初级文库的检测方法对表达文库进行检测。PCR引物(CMV-F/BGH)为:5′-CGCAAAT- GGGCGGTAGGCGTG-3′/5′-TGGCAACTAGAAGGCACAGTCGAGG-3′。
PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 2 min,35个循环;72 ℃ 5 min。反应结束后用10 g · L-1的琼脂糖凝胶电泳检测插入片段大小和阳性克隆的重组率。
1.2.5 巨型艾美耳球虫已知基因的扩增将表达文库菌液混匀后,从中取出10 μL接种到5 mL含有卡那霉素(终质量浓度50 μg · mL-1)的肉汤培养液中,在30 ℃、250 r · min-1条件下培养至A600为1.0。提取质粒作为模板,以已知的7个巨型艾美耳球虫基因MIC 3-1、MIC3-2、MIC3-3、MIC2、MIC7、MIC5、AMA1 的特异性引物进行PCR扩增。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 7 min。反应结束后用10 g · L-1的琼脂糖凝胶进行电泳检测。
2 结果与分析 2.1 卵囊分离及其种类鉴定收集卵囊总量约为2×108个,显微镜下孢子化卵囊呈卵圆形或椭圆形,卵囊长25.0~42.5 μm、宽22.5~35.0 μm,形状指数为1.20。以提取的孢子化卵囊基因组DNA为模板,分别用堆型艾美耳球虫(E.acervulina)、布氏艾美耳球虫(E.brunetti)、巨型艾美耳球虫(E.maxima)、和缓艾美耳球虫(E.mitis)、毒害艾美耳球虫(E.necatrix)、早熟艾美耳球虫(E.praecox)和柔嫩艾美耳球虫(E.tenella)的ITS1特异性引物[16]进行扩增,结果只扩增出了与巨型艾美耳球虫ITS1大小相符(144 bp)的条带(图 1),其他6个种均未扩增出相应条带,表明收集到的卵囊为巨型艾美耳球虫纯种卵囊。
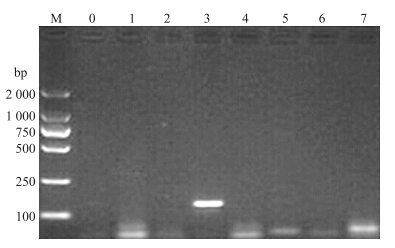 | 图 1 巨型艾美耳球虫的ITS1-PCR产物鉴定 Fig. 1 PCR product amplification of ITS1 of Eimeria maxima M.DNA marker;0.阴性对照Negative control;1.E.acervulina; 2.E.brunetti;3.E.maxima;4.E.mitis;5.E.necatrix;6.E.praecox;7.E.tenella |
提取的总RNA经10 g · L-1甲醛变性胶电泳鉴定,可看到清晰的28S、18S和5S条带;28S条带明显亮于18S条带,比例约为2 ∶ 1(图 2)。总RNA样品经核酸分析仪检测,质量浓度为0.762 μg · μL-1,A260/A280为1.92。由总RNA分离得到的mRNA经10 g · L-1甲醛变性胶电泳鉴定,可看到分布于300~3 000 bp的一片模糊拖带(图 2)。样品经Thermo nanodrop核酸分析仪检测,A260/A280为2.32,质量浓度为44.3 ng · μL-1,共得到总RNA 8.86 μg。
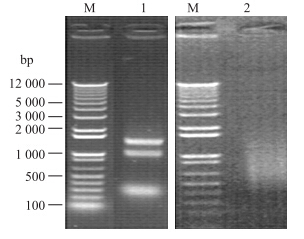 | 图 2 提取的总RNA和分离的mRNA Fig. 2 Total RNA and isolated mRNA M.DNA marker;1.提取的总RNA Total RNA;2.分离的mRNA Isolated mRNA |
取50 μL稀释的初级文库菌液(1 ∶ 1 000稀释)涂布于含氨苄青霉素的抗性LB平板上,14 h后平板上的克隆数为230(图 3)。按照公式,计算出初级文库滴度为4.6×106 CFU · mL-1,文库总容量为0.92×107 CFU。
 | 图 3 初级文库的库容量鉴定(1 ∶ 1 000稀释) Fig. 3 Identification of entry library total clones (1 ∶ 1 000 dilution ratio) |
随机挑取24个克隆进行PCR鉴定,结果全部为阳性克隆,分别有不同大小的片段插入。表明初级文库的重组率为100%,插入片段大小范围为0.9~2.8 kb,平均插入片段大小为1.63 kb(图 4)。
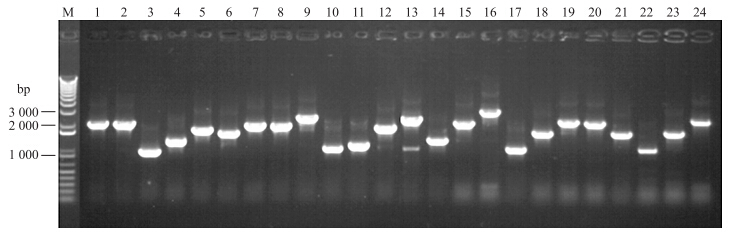 | 图 4 初级文库的PCR鉴定 Fig. 4 CR identification of entry library M.DNA marker;1~24.随机挑选24个克隆的PCR产物PCR products of randomly selected 24 clones |
取50 μL稀释的表达文库菌液(1 ∶ 1 000)涂布于含卡那霉素的抗性LB平板上,14 h后平板上的克隆数为580(图略)。按照公式计算出表达文库滴度为1.16×107 CFU · mL-1,文库总容量为2.32×107 CFU。
2.4.2 插入片段大小和重组率鉴定随机挑取24个克隆进行PCR鉴定,结果全部为阳性克隆,分别有不同大小的片段插入,表明表达文库的重组率为100%。插入片段大小范围为1~3 kb,平均插入片段大小为1.64 kb(图 5)。
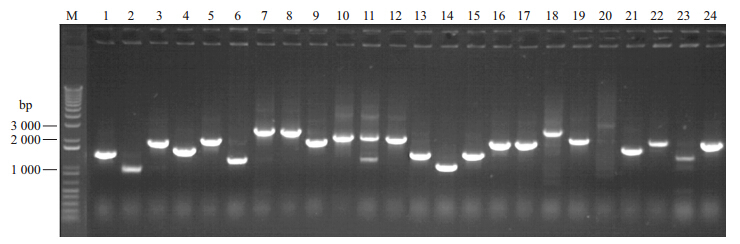 | 图 5 表达文库PCR的鉴定 Fig. 5 PCR identification of expression library M.DNA marker;1~24.随机挑选24个克隆的PCR产物PCR products of randomly selected 24 clones |
以文库质粒为模板,用7个已知的巨型艾美耳球虫基因MIC 3-1、MIC3-2、MIC3-3、MIC5、MIC7、MIC2和AMA1 的特异性引物进行PCR扩增。已知巨型艾美耳球虫基因的片段大小分别为450、684、336、1 275、519、888和1 422 bp。扩增结果(图 6)显示从构建的文库中能够扩增出上述已知基因的条带。
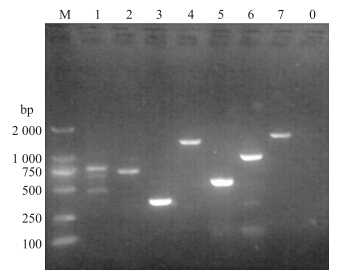 | 图 6 表达文库对已知基因的PCR扩增结果 Fig. 6 PCR amplification of expression library M.DNA marker;1.MIC 3-1 ;2.MIC 3-2 ;3.MIC 3-3 ;4.MIC 5 ;5.MIC 7 ;6.MIC 2 ;7.AMA 1 ;0.阴性对照Negative control |
为了构建高质量的cDNA文库,本试验根据卵囊的形态和大小并结合之前的报道[17]对虫种进行一个初步的鉴定后,再利用ITS-PCR进行鉴定,两种方法相结合检验就确保了试验材料的纯度。本试验采取的文库构建方法为gateway技术,与标准文库构建法相比,不需要使用限制性内切酶切割等步骤,因此不会对任何基因产生剪切,同时Super Script Ⅲ反转录酶因能特异性降低RNA酶活性而充分保证了合成的cDNA第一链的长度和产量,产生的cDNA文库的全长比例优于标准cDNA文库;与gateway技术相结合的重组酶可以将构建好的初级cDNA文库穿梭于任何表达载体间,保证文库的库容量无明显损失,构建的未剪切cDNA文库的库容量等均优于标准cDNA文库[18, 19, 20]。
高质量的cDNA文库是研究特定组织基因表达情况和筛选目的基因的有利工具。一般从3个方面来判断一个文库的质量:库容量、平均插入片段长度和重组率。库容量可以说明该文库是否有代表性,即cDNA种类的完整性,一个高质量的cDNA文库,其库容量应大于5×106 CFU;平均插入片段的长度可以说明是否插入了全长的cDNA序列,只有大部分克隆含有完整性插入的cDNA,文库才能体现完整的遗传信息[18, 19, 20, 21, 11]。本试验所构建的巨型艾美耳球虫孢子化卵囊cDNA表达文库重组率为100%,库容量为2.32×107 CFU,能用特异性引物扩增出7个已知的巨型艾美耳球虫基因,说明此文库代表性强;插入片段长度为1~3 kb,平均大小为1.64 kb,充分保证了大分子cDNA的完整性,而且插入片段大小各不相同,说明多样性好。综合来看,该文库达到了高质量文库的标准。
| [1] | Blake D P,Tomley F M. Securing poultry production from the ever-present Eimeria challenge[J]. Trends in Parasitology,2014,30(1):12-19 |
| [2] | Reid A J,Blake D P,Ansari H R,et al. Genomic analysis of the causative agents of coccidiosis in domestic chickens[J]. Genome Research,2014,24(10):1676-1685 |
| [3] | Williams R B. A compartmentalised model for the estimation of the cost of coccidiosis to the world's chicken production industry[J]. International Journal for Parasitology,1999,29(8):1209-1229 |
| [4] | Dalloul R A,Lillehoj H S. Poultry coccidiosis:recent advancements in control measures and vaccine development[J]. Expert Review of Vaccines,2006,5(1):143-163 |
| [5] | Ivory C,Chadee K. DNA vaccines:designing strategies against parasitic infections[J]. Genetic Vaccines and Therapy,2004,2(1):17 |
| [6] | Witcombe D M,Smith N C. Strategies for anti-coccidial prophylaxis[J]. Parasitology,2014,141(11):1379-1389 |
| [7] | 闫晓菲,韩红玉,岳城,等. cDNA文库及其在原虫研究中的应用[J]. 生物技术通报,2008(1):52-60 [Yan X F,Han H Y,Yue C,et al. Application of cDNA library in protozoo[J]. Biotechnology Bulletin,2008(1):52-60(in Chinese with English abstract)] |
| [8] | Walhout A J,Sordella R,Lu X,et al. Protein interaction mapping in C.elegans using proteins involved development[J]. Science,2000,287:116-122 |
| [9] | Hartley J L,Temple G F,Brasch M A. DNA cloning using in vitro site-specific recombination[J]. Genome Res,2000,10(11):1788-1795 |
| [10] | Ohara O,Temple G. Directional cDNA library construction assisted by the in vitro recombination reaction[J]. Nucleic Acids Research,2001,29(4):1-8 |
| [11] | Ohara O,Nagase T,Mitsui G,et al. Characterization of size-fractionated cDNA libraries generated by the in vitro recombination-assisted method[J]. DNA Res,2002,9(2):47-57 |
| [12] | 李晨,沈海涛,张煜星,等. 利用gateway技术构建天山雪莲cDNA表达文库[J]. 石河子大学学报:自然科学版,2010,28(5):534-536 [Li C,Shen H T,Zhang Y X,et al. Construction of a cDNA library of Saussurea involucrata Kar.et Kir using gateway technology[J]. Journal of Shihezi University:Natural Science Edition,2010,28(5):534-536(in Chinese with English abstract)] |
| [13] | 张玉平,王顺清,王彩霞,等. gateway非放射标记法构建小鼠破骨细胞前体细胞cDNA文库[J]. 中国组织工程研究,2012,16(2):215-219 [Zhang Y P,Wang S Q,Wang C X,et al. Construction of an osteoclast precursor cell cDNA library using gateway non-radiolabeling technology in mice[J]. Chinese Journal of Tissue Engineering Research,2012,16(2):215-219(in Chinese with English abstract)] |
| [14] | 张建哲. 巨型艾美耳球虫孢子化卵囊cDNA文库的构建及筛选[D]. 上海:上海师范大学,2008 [Zhang J Z. The construction and screening of cDNA library of Eimeria maxima sporulated oocysts[D]. Shanghai:Shanghai Normal University,2008(in Chinese with English abstract)] |
| [15] | 索勋,李国清. 鸡球虫病学[M]. 北京:中国农业大学出版社,1998:254-255 [Suo X,Li G Q. Coccidia and Coccidiosis of Domestic Fowl[M]. Beijing:China Agricultural University Press,1998:254-255(in Chinese)] |
| [16] | Jenkins M C,Miska K,Klopp S. Application of polymerase chain reaction based on ITS1 rDNA to speciate Eimeria[J]. Avian Diseases,2006,50:110-114 |
| [17] | 黄兵,史天卫,吴薛忠,等. 巨型艾美耳球虫纯种鉴定和致病性研究[J]. 中国兽医寄生虫病,1995,3(4):12-14 [Huang B,Shi T W,Wu X Z,et al. Studies on the identification and pathogenicity of pure species of Eimeria maxima[J]. Chinese Journal of Veterinary Parasitology,1995,3(4):12-14(in Chinese with English abstract)] |
| [18] | 王顺清,林蔡弟,毛平,等. 小鼠巨噬细胞cDNA酵母表达文库的构建与鉴定[J]. 中华生物医学工程杂志,2010,16(4):292-297 [Wang S Q,Lin C D,Mao P,et al. Construction and identification of yeast expression cDNA library of mouse macrophage[J]. Chinese Journal of Biomedical Engineering,2010,16(4):292-297(in Chinese with English abstract)] |
| [19] | 邱爱连,刘林林,蔡汉阳,等. gateway技术构建酵母双杂分析用辣椒cDNA文库[J]. 江西农业大学学报,2010,32(4):791-796 [Qiu A L,Liu L L,Cai H Y,et al. Construction of a cDNA library of pepper for yeast two-hybrid analysis using gateway technology[J]. Acta Agriculturae Universitatis Jiangxiensis,2010,32(4):791-796(in Chinese with English abstract)] |
| [20] | 佟春玲,陆涛峰,余露露,等. cDNA文库构建方法及在细胞移植等领域的应用[J]. 中国组织工程研究与临床康复,2009,13(44):8705-8708 [Tong C L,Lu T F,Yu L L,et al. cDNA library construction methods and its application in cell transplantation[J]. Journal of Clinical Rehabilitative Tissue Engineering Research,2009,13(44):8705-8708(in Chinese with English abstract)] |
| [21] | 邢俊杰,陶小平,李玲龙,等. 小粒野生稻酵母双杂交cDNA文库的构建[J]. 杂交水稻,2010,25(1):67-69 [Xing J J,Tao X P,Li L L,et al. Construction of yeast two-hybrid cDNA library of Oryza minuta[J]. Hybrid Rice,2010,25(1):67-69(in Chinese with English abstract)] |




