文章信息
- 韩小红, 卢赐鼎, 华银, 林浩宇, 是雨霏, 吴松青, 张飞萍, 梁光红.
- Han Xiaohong, Lu Ciding, Hua Yin, Lin Haoyu, Shi Yufei, Wu Songqing, Zhang Feiping, Liang Guanghong.
- 星天牛转录组及三大解毒酶家族相关基因系统发育分析
- Phylogenetic Analysis of Transcriptome and Three Detoxification Enzyme Families Related Genes in Anoplophora chinensis (Coleoptera: Cerambycidae)
- 林业科学, 2019, 55(5): 104-113.
- Scientia Silvae Sinicae, 2019, 55(5): 104-113.
- DOI: 10.11707/j.1001-7488.20190512
-
文章历史
- 收稿日期:2018-08-27
- 修回日期:2019-03-13
-
作者相关文章
2. 生态公益林重大有害生物防控福建省高校重点实验室 福州 350002
2. Key Laboratory of Integrated Pest Management in Ecological Forests, Fujian Province University, Fujian Agriculture and Forestry University Fuzhou 350002
星天牛(Anoplophora chinensis)在国内各地均有分布,在国外主要分布于韩国、日本、缅甸、马来西亚、菲律宾以及一些欧洲国家,是世界公认的重要蛀干害虫,近年被欧洲植保组织(EPPO)列为重要检疫对象(魏建荣等,2011)。其食性极为广泛,可危害柑(Citrus reticulata)、梨(Pyrus spp.)、苹果(Malus pumila)、桃(Amygdalus persica)、木麻黄(Casuarina equisetifolia)、苦楝(Melia azedarach)、垂柳(Salix babylonica)、梧桐(Firmiana platanifolia)、台湾相思(Acacia confusa)等上百种寄主植物。成虫取食细枝皮层,幼虫蛀食树干韧皮部和木质部,形成枯枝或断枝,影响树木健康生长,严重时导致树木死亡,甚至造成重大经济损失(魏建荣等,2011)。
显然,星天牛能够广泛适应不同种类的寄主植物,而不同寄主植物内的营养物质特别是植物次生物质的种类与含量各不相同,表明其消化道的解毒酶体系也具有极强的适应和调节能力; 另一方面,我国近年来除采用花绒寄甲(Dastarcus helophoroides)(李孟楼等,2009)、川硬皮肿腿蜂(Scleroderma sichuanensis)(杜开书等,2006)等天敌昆虫、病原真菌(蔡守平,2017)等生物防治方法外,也常采取化学防治措施控制其种群数量,取得了较好的防治效果; 但也为该虫提供了杀虫剂选择压力,可能导致其产生抗药性并不断增强(潘志萍等,2006; 张丽阳等,2016),防治难度不断加大。
已有研究发现,昆虫在取食及适应不同寄主植物或面临各种杀虫剂胁迫时,通常依靠体内解毒酶系来调节自身的免疫机制,从而表现一定程度的解毒能力,且这种能力是由其基因所调控的,并具有可遗传性(蔡守平,2017)。随着昆虫抗药性研究的深入,大量研究已经明确了细胞色素P450酶系(CYP)、羧酸酯酶(CarE)、谷胱甘肽S-转移酶(GST)三大解毒酶在昆虫抗药性代谢中的关键作用(彭建新,2006)。昆虫面临上述胁迫后,这些酶系都会产生应激作用,通过增加对杀虫剂或有毒物质的转化和降解作用,以降低毒性或通过阻隔作用保护自己的靶标位点,对有毒物质进行转化,从而降低或弱化毒性(Wu et al., 2016)。但迄今为止,星天牛基因数据极少,相关转录组研究主要涉及其嗅觉表达谱的分析以及化学感受蛋白基因的识别与表达(Wang et al., 2017; Sun et al., 2018),暂无对杀虫剂的抗药性分子机制等方面的研究报道。
本研究采用高通量测序技术对星天牛进行转录组测序,获得其转录本信息,试图探明星天牛体内的三大解毒酶系的表达谱情况,并通过构建系统进化树,探析这3种酶系基因家族的类别及其系统演化关系,为星天牛对多寄主种类的适应性与杀虫剂的解毒机制研究提供依据,也为农林害虫防治提供参考。
1 材料与方法 1.1 样虫收集于2015年,在福建农林大学校内的垂柳上捕捉星天牛成虫,置于养虫笼内,以新鲜的柳枝喂食。挑选出形态完整的星天牛(雌雄各3头,2组重复,共12头),饥饿处理至不再产生排泄物的第2天,混合用于转录组测序,放入液氮2 min后,置于研钵搅碎,将其组织装入离心管,密封,置于-80 ℃冰箱中冷冻保存。
1.2 RNA提取及质量检测使用TRIzoI Reagent(Invitrogen, Carlsbad, USA)试剂盒提取样品总RNA,1%琼脂糖凝胶电泳检测总RNA质量; 其纯度(OD260/OD280)的检测使用Nanodrop分光光度计(IMPLEN,CA,USA),并按照Qubit® RNA Assay Kit in Qubit® 2.0 Flurometer(Life Technologies, CA, USA)定量检测总RNA浓度; 最后用Agilent 2100(Agilent Technologies,CA,USA)精确检测RNA的完整性。
1.3 测序、组装及注释将满足测序质量要求的星天牛总RNA样品送至北京百迈客生物科技有限公司进行文库构建与RNA-Seq测序。测序完成后,经处理获得clean reads,并得到Q20、Q30,GC含量以及相关重复序列情况,然后对其进行组装,得到unigenes序列。分别与Nr、Pfam、KOG、COG、GO、KEGG、Swiss-Prot数据库进行BLAST程序比对并进行功能注释(杨帆等,2014; Zhang et al., 2018)。
1.4 星天牛解毒酶基因的系统发育分析以星天牛3种解毒酶基因(CYP、CarE、GST)的蛋白序列为基础,在NCBI数据库里下载鞘翅目中全部注册近缘物种的3种同源蛋白序列,并采用MEGA5.1分析软件进行比对分析。将CYP基因中>1 700 bp、CarE基因和GST基因中>1 800 bp的基因序列使用邻接法(neighbor-joining, NJ)构建进化树,在建树前进行遗传距离、模型的估算和选择,Bootstrap值为1 000。
2 结果与分析 2.1 测序及组装星天牛转录组测序样品数据量超过了6G,总的GC含量为42.44%,数据中所有reads的质量参数Q20为94.5%,表明测序质量好,结果可靠。组装后用最长的转录本作为unigene,得到32 247条unigenes,其总长为30 430 883 bp,平均长度为943.68 bp。大于1 000 bp的unigenes数量有9 325条(图 1),占全部unigenes的28.92%。表明该转录组文库的测序和组装结果较好,能够进行后续生物信息学分析。
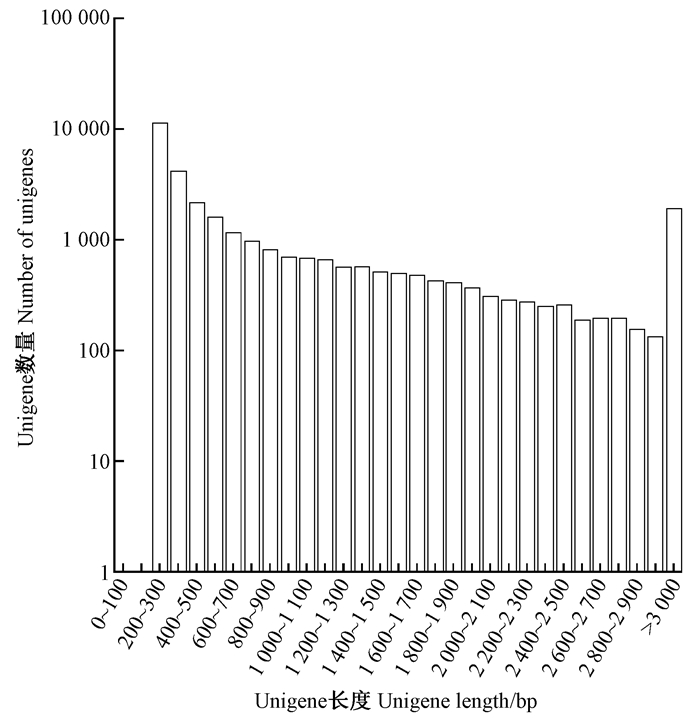
|
图 1 星天牛转录组unigenes长度分布 Fig. 1 Unigenes length distribution of transcriptome in A. chinensis |
将组装得到的unigenes与7个数据库进行比对,得到注释信息,共有1 489条基因被7个数据库同时注释成功,占总unigenes数的8.09%(表 1)。在Swiss-Prot注释成功的unigenes的数量最多(99.11%),其后依次是Nr(96.60%),Pfam(64.46%),KOG(60.65%),GO(47.15%),COG(28.70%)和KEGG(26.83%)。Unigene注释到Nr数据库中的物种分布图(图 2)显示:赤拟谷盗(Tribolium castaneum)所占比例最高(60.25%),其后依次是山松甲虫(Dendroctonus ponderosae)(17.58%)、豌豆蚜(Acyrthosiphon pisum)(1.56%)、毕氏粗角猛蚁(Cerapachys biroi)(1.49%)、红火蚁(Solenopsis invicta)(1.37%),其他物种占14.85%。共有248条抗性相关基因注释成功(139条P450酶系基因、72条CarE酶系基因、37条GST酶系基因)。
|
|

|
图 2 星天牛转录组unigenes在Nr数据库中的物种分布 Fig. 2 Species distribution of A. chinensis unigenes in Nr database |
在GO注释的8 083条unigenes中,注释到分子功能的基因最多(11 868),其次是生物学过程(11 466),细胞功能的最少(4 859)。47个亚类中,蛋白结合和催化活性在分子功能分类中最多,代谢过程和细胞进程在生物过程中最多,细胞成分和细胞在细胞组分中最多(图 3)。
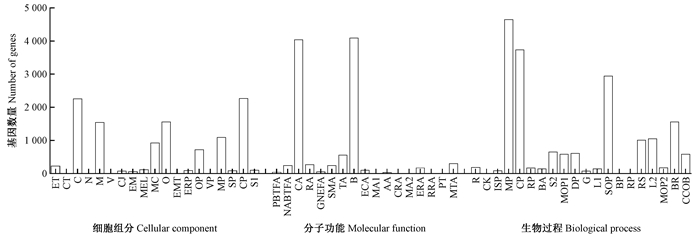
|
图 3 星天中转录组unigenes GO分类 Fig. 3 GO categorization of unigenes in A. chinensis 细胞组分Cellular cpmponenet ER:胞外区Cellular component;CT:胶原蛋白三聚物Collagen trimer;C:细胞Cell;N:拟核Nucleoid;M:细胞膜Membrane;V:病毒Virion;CJ:细胞连接Cell junction;EM:细胞外基质Extracellular matrix;MEL:膜封闭腔Membrane-enclosed lumen;MC:大分子复合物Macromolecular complex;O:细胞器Organelle;EMP:部分细胞外基质Extracellular matrix part;ERP:部分胞外区Extracellular region part;OP:部分细胞器Organelle part;VP:部分病毒Virion part;MP:部分细胞膜Membrane part;SP:部分突触Synapse part;CP:部分细胞Cell part;S:突触Synapse. 分子功能Molecular function PBTFA:蛋白结合转录活性因子Protein binding transcription factor activity;NABTFA:核酸结合转录活性因子Nucleic acid binding transcription factor activity;CA:催化活性Catalytic activity;RA:受体活性Receptor activity;GNEFA:鸟苷核苷酸交换活性因子Guanyl-nucleotide exchange factor activity;SMA:结构活性分子Structural molecule activity;TA:转运活性Transporter activity;B:组装Binding;ECA:电子载体活性Electron carrier activity;MA1:形态发生活性Morphogen activity;AA:抗氧化活性Antioxidant activity;CRA:通道调节Channel regulator activity;MA2:金属辅酶Metallochaperone activity;ERA:酶活性调节剂Enzyme regulator activity. 生物过程Biological process R:复制Reproduction;CK:细胞杀伤Cell killing;ISP:免疫反应过程Immune system process;MP:代谢过程Metabolic process;CP:细胞过程Cellular process;RP:生殖过程Reproductive process;BA:生物粘附Biological adhesion;S:发送信号Signaling;MOP:多细胞生物过程Multicellular organismal process;DP:发展过程Developmental process;G:发育过程Growth;L:运动过程Locomotion;SOP:单一生物过程Single-organism process;BP:生物相Biological phase;RP:有节奏的过程Rhythmic process;RS:刺激反应Response to stimulus;L:定位Localization;MOP:多机体过程Multi-organism process;BR:生物调节Biological regulation;RRA:受体活性调节剂Receptor regulator activity;PT:蛋白质标签Protein tag;MTA:分子活性转换器Molecular transducer activity;CCOB:细胞组成或生物发生Cellular component organization or biogenesis. |
在COG数据库中,共有4 920个unigenes得到了注释,同时被分到25个功能组中(图 4)。其中一般功能预测是最大功能组,约占总体24.95%;其次是复制、重组和修饰约占总体9.83%;碳水化合物转运和代谢约占总体7.02%。
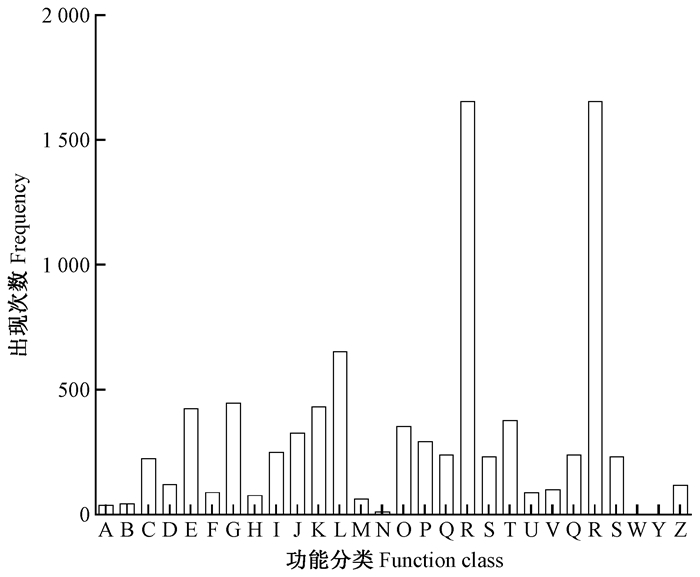
|
图 4 星天牛转录组unigenes COG分类 Fig. 4 COG categorization of unigenes in A. chinensis A: RNA加工与修饰RNA processing and modification; B:染色质结构与动力Chromatin structure and dynamics; C:能量生产和转换Energy production and conversion; D:细胞周期控制、细胞分裂、染色体分裂Cell cycle control, cell division, chromosome partitioning; E:氨基酸转运和代谢Amino acid transport and metabolism; F:核苷酸转运和代谢Nucleotide transport and metabolism; G:碳水化合物转运和代谢Carbohydrate transport and metabolism; H:辅酶转运和代谢Coenzyme transport and metabolism; I:脂质转运和代谢Lipid transport and metabolism; J:翻译、核糖体结构和生物发生Translation, ribosomal structure and biogenesis; K:转录Transcription; L:复制、重组、修复Replication, recombination and repair; M:细胞壁/膜/包膜生物发生Cell wall/membrane/envelope biogenesis; N:细胞运动Cell motility; O:翻译后修饰、蛋白质周转、伴侣Posttranslational modification, protein turnover, chaperones; P:无机离子转运与代谢Inorganic ion transport and metabolism; Q:次级代谢产物的生物合成、转运、分解代谢Secondary metabolites biosynthesis, transport and catabolism; R:仅通用功能预测General function prediction only; S:功能未知Function unknown; T:信号转导机制Signal transduction mechanisms; U:细胞内运输、分泌、水泡运输Intracellular trafficking, secretion, and vesicular transport; V:防御机制Defense mechanisms; W:细胞外结构Extracellular structures; Y:核结构Nuclear structure; Z:细胞骨架Cytoskeleton. |
将转录组数据与KEGG数据库进行结合分析,结果表明,4 600条unigenes参与了162个代谢通路,其中嘌呤代谢和RNA运输代谢通路注释最多(144),其次是内质网蛋白质加工(141)、剪接(126)、氧化磷酸化(110)、泛素介导的蛋白水解(104)(表 2)。
|
|
CYP共有139条基因注释成功,在抗性基因中比例较大,其总长度为127 130.11 bp,介于203~3 938.21 bp之间,平均长度为921.23 bp。其中,基因ID长度大于1 700 bp的有23条,可分别注释到CYP4、CYP6、CYP9、CYP12等家族,并且注释到NADPH-细胞色素C还原酶(NADPH-cytochrome P450 reductase,CPR)基因1条。将这23条序列与已知鞘翅目昆虫的CYP蛋白序列比对分析并构建进化树(图 5),星天牛的大部分CYP基因都至少与一种鞘翅目近缘物种的CYP基因聚类在一起,且序列一致性都高于70%,特别是星天牛CYP6基因家族与鞘翅目其他物种的同源数最多。星天牛与光肩星天牛(A. glabripennis)被聚为一类的最多,这说明其在CYP的分化地位上,两物种之间的亲缘关系最近,与传统分类学上的亲缘关系一致。

|
图 5 星天牛与其鞘翅目近缘物种CYP基因的系统发生关系 Fig. 5 The phylogeny of cytochrome P450 genes of A.chinensis and the known closely related species of Coleoptera species A. chinensis:星天牛Anoplophora chinensis; A. glabripennis:光肩星天牛Anoplophora glabripennis; T. castaneum:赤拟谷盗Tribolium castaneum; T. molitor:黄粉虫Tenebrio molitor; A. tumida:小蜂甲Aethina tumida; L. decemlineata:马铃薯甲虫Leptinotarsa decemlineata; D. ponderosae:山松甲虫Dendroclonus ponderosae; A. planipennis:白蜡窄吉丁Agrilus planipennis. |
有72条CarE基因注释成功,筛选出19条序列长度大于1 800 bp的基因序列,将其与鞘翅目已知近缘物种的CarE蛋白序列比对并构建进化树(图 6),CarE可聚为4类,分别为:分泌的β-酯酶(Secreted β-esterases),主要参与一些激素以及信息素的加工,与光肩星天牛的同源性最高; 神经趋化蛋白(Neurotactins),参与星天牛的神经和发育过程,星天牛的神经趋化蛋白可与松褐天牛(Monochamus alternatus)以及光肩星天牛聚在一起; 外源性代谢酶(Xenobiotic metabolising enzymes),对昆虫本身起到消化与解毒的功能,为数量最多的一类; 还有一类为未知功能(Unknown functions),其功能暂不明确。

|
图 6 星天牛与其近缘物种CarE基因的系统发生关系 Fig. 6 The phylogeny of xyloesterase genes of A.chinensis and the known closely related species of Coleoptera species A. chinensis:星天牛Anoplophora chinensis; A. glabripennis:光肩星天牛Anoplophora glabripennis; T. castaneum:赤拟谷盗Tribolium castaneum; T. molitor:黄粉虫Tenebrio molitor; A. tumida:小蜂甲Aethina tumida; L. decemlineata:马铃薯甲虫Leptinotarsa decemlineata; D. ponderosae:山松甲虫Dendroclonus ponderosae; M. alternatus:松褐天牛Monochamus alternatus. |
根据Nr注释结果,GST共有37条基因注释成功,其平均长度为1 509 bp。采用较长(>1 800 bp)的36条星天牛GST基因序列与鞘翅目近缘物种的GST蛋白序列比对并构建进化树(图 7),星天牛等多种鞘翅目昆虫可以互相聚为一类,其中星天牛表达的GST主要为Microsomal GST、Sigma GST、Omega GST、Delta GST、Theta GST等亚家族,具有保护生物大分子免受氧化损伤、催化活力等功能,这些亚家族的GST都同时与光肩星天牛为直系同源关系; 相比于前2类解毒酶系,GST还具有较高的保守性。
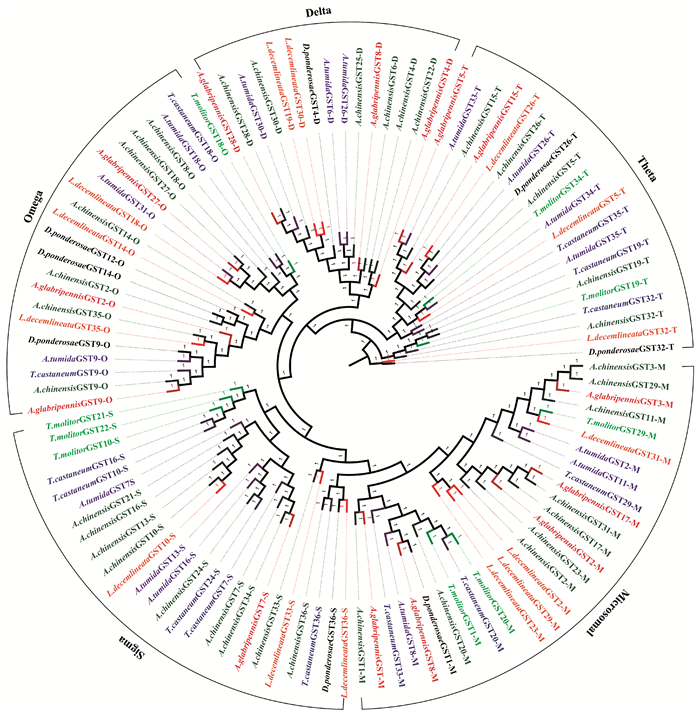
|
图 7 星天牛与其近缘物种GST基因的系统发生关系 Fig. 7 The phylogeny of glutathione S-transferase genes of A.chinensis and the known closely related species of Coleoptera species A. chinensis:星天牛Anoplophora chinensis; A. glabripennis:光肩星天牛Anoplophora glabripennis; T. castaneum:赤拟谷盗Tribolium castaneum; T. molitor:黄粉虫Tenebrio molitor; A. tumida:小蜂甲Aethina tumida; L. decemlineata:马铃薯甲虫Leptinotarsa decemlineata; D. ponderosae:山松甲虫Dendroclonus ponderosae. |
转录组分析为挖掘昆虫功能基因提供了有效方法,从而为研究昆虫的食性转化、生理适应、亲缘关系以及物种的协同进化与互作等复杂问题提供了更有意义的方向(Hamilton et al., 2014)。将获得的序列与近缘物种进行同源比对发现,还有23 013条unigenes(41.65%)未被注释成功,可能与注释信息的缺乏、序列本身片段较短或者一些新基因的出现有关(Hedengren et al., 2000; 胡俊杰等,2017); Nr数据库中,星天牛与赤拟谷盗的同源序列最多,可能与其已注册的基因信息较多以及两者的亲缘关系较近有关(孟翔等,2016); 通过GO、COG数据库对星天牛的unigenes进行功能分类,较为全面地描述了星天牛的基因属性。在KEGG数据库中,星天牛的unigenes匹配了162个代谢通路,其中嘌呤代谢和RNA运输代谢通路注释最多(144),这可能与生物体细胞的生理功能、RNA的定位和基因表达的水平与RNA运输有关,因为RNA参与转录、翻译过程,对转录产物、蛋白质与酶的形成至关重要,从而对生物体的相关生命活动造成影响。
目前,化学杀虫剂是全球植物保护的主体(张丽阳等,2016),大量频繁地使用化学杀虫剂,使得昆虫抗药性日益增强,这对于维持昆虫个体的生命活动及种群的繁衍非常重要,其体内的解毒酶系对其抗药性的产生和进化具有非常重要的作用(Vogel et al., 2011)。本研究发现,星天牛体内3种主要解毒酶系的基因数量与其他已知测序结果的昆虫的基因数量存在差异,这可能是昆虫物种个体及生物学特性差异所致,也可能与其对外源物质解毒功能差异有关(刘长莉等,2013); 除了部分基因与近缘昆虫物种直系同源聚类外,星天牛体内还有一些基因单独聚为一支,但基因功能尚无法明确,需要进一步研究。
P450基因数量和种类的多样性决定了功能的多样性,其主要功能包括对外源有毒物质的降解和对内源物质的合成和代谢的调节(Chen et al., 2018)。值得关注的是,在星天牛P450转录组注释结果的139条基因中,共有47条unigenes注释为CYP6(33.81%),前人已经研究并证实了CYP6家族的多个基因与昆虫的抗药性有关(张丽阳等,2016; Kim et al., 2017),且基因表达量上调是昆虫CYP介导多种药剂代谢抗性的主要机制(Lee et al., 2018),由此推测星天牛也可能具有类似的解毒机制。针对其他鞘翅目昆虫,也表现出其特定的聚类关系,比如小蜂甲(CYP6-23、CYP6-25)、光肩星天牛(CYP6-6、CYP6-25)、赤拟谷盗(CYP6-3、CYP6-26),它们在进化树中都形成了单独一支,这表明在昆虫种类分化后也可能出现了各种类昆虫体内CYP的分化。
CarE在有机磷、氨基甲酸酯、拟除虫菊酯类等杀虫剂代谢中起重要作用。当酯酶基因扩增时,可使该基因表达的酯酶数量增多,从而引起酯酶活力增高,加速酯键的化学农药代谢,使昆虫具有解毒功能(Zhang et al., 2013; 张丽阳等,2016)。比如当酯酶E4和FE4中任一种酶活力增高、表达量增大或者量增多,都可导致烟蚜(Myzus persicae)体内抗性的产生(彭建新,2006); 在抗药性家蝇中其解毒作用的产生主要是由于一个或多个CarE基因发生上调(Feng et al., 2018)。星天牛CarE基因的数量约占CYP基因的50%,且表达量较高; CarE解毒酶蛋白基因主要为β-酯酶、神经趋化蛋白、外源性代谢酶和未知等4种类型,分别参与星天牛的一些激素以及信息素的加工、神经和发育过程、消化与解毒等功能,一部分基因功能还未探明,也值得深入研究。
GST也是昆虫体内重要的代谢酶之一,在害虫体内参与有机磷、除虫菊酯等类杀虫剂降解,反式作用调节基因的分离或扩增导致GST表达增强,从而加速了对GST的特异杀虫剂底物的代谢,进而对外源化合物的解毒代谢起重要作用(Lumjuan et al., 2005; 彭建新,2006)。其参与解毒外源物质的分子机制虽然研究较多,但多限于埃及伊蚊(Aedes aegypti)等热门研究的昆虫(Zhou et al., 2018),其对杀虫剂DDT和氯菊酯的抗性研究中也曾发现过体内GST的过表达(Lumjuan et al., 2005); 到目前为止,关于具体基因调控过程尚不明确(尤福洋,2016),值得进一步探索; 同时,针对研究较少的非模式昆虫重要物种的研究也值得尝试。本研究发现,在星天牛体内所表达的GST的数量最少,仅约CarE的50%,且与光肩星天牛为直系同源关系,表明星天牛与其在面临同类化学杀虫剂时可能具有相似的GST解毒酶功能。
4 结论本研究获得了星天牛的转录组数据,对其进行了基因功能注释和功能分类,并对三大解毒酶基因在星天牛体内的表达情况进行了分析,在此基础上对3种解毒酶的系统进化分析,探析星天牛与鞘翅目近缘物种之间解毒酶系基因的分化情况。研究结果所挖掘的相关基因可为星天牛对杀虫剂的适应性、抗药性产生机制、抗药性遗传研究奠定基础,同时为减弱昆虫抗药性以及研究新型可持续、环境友好型杀虫剂提供思路。
蔡守平. 2017.星天牛高致病力绿僵菌菌株筛选、应用及免疫响应的转录组分析.南京: 南京林业大学博士学位论文. (Cai S P. 2017. Researches on screening, application of high pathogenicity Metarhizium anisopliae against A. chinensis larvae and transcriptome analysis of immune response to the infection. Nanjing: PhD thesis of Nanjing Forestry University.[in Chinese]) http://cdmd.cnki.com.cn/Article/CDMD-10298-1018800005.htm |
杜开书, 周祖基, 杨伟. 2006. 川硬皮肿腿蜂防治柳树星天牛试验初报. 安徽农业科学, 34(13): 3104-3105. (Du K S, Zhou Z J, Yang W. 2006. Experiment in the control of Anoplophora chinensis(Forster)with Scleroderma sichuanensis Xiao. Journal of Anhui Agricultural Sciences, 34(13): 3104-3105. DOI:10.3969/j.issn.0517-6611.2006.13.082 [in Chinese]) |
胡俊杰, 孟翔, 周佳滨, 等. 2017. 扶桑绵粉蚧转录组分析. 昆虫学报, 60(1): 9-17. (Hu J J, Meng X, Zhou J B, et al. 2017. Transcriptome analysis of the cotton mealybug, Phenacoccus solenopsis(Hemiptera:Pseudococcidae). Acta Entomologica Sinica, 60(1): 9-17. [in Chinese]) |
李孟楼, 李有忠, 雷琼, 等. 2009. 释放花绒寄甲卵对光肩星天牛幼虫的防治效果. 林业科学, 45(4): 78-82. (Li M L, Li Y Z, Lei Q, et al. 2009. Biocontrol of Asian longhorned bettle larva by releasing eggs of Dastarcus helophoroides(Coleoptera:Bothrideridae). Scientia Silvae Sinicae, 45(4): 78-82. DOI:10.3321/j.issn:1001-7488.2009.04.013 [in Chinese]) |
刘长莉, 卢利霞, 许艳丽, 等. 2013. 灰飞虱唾液腺三大解毒酶家族的转录组分析. 昆虫学报, 56(12): 1509-1515. (Liu C L, Lu L X, Xu Y L, et al. 2013. Transcriptional analysis of the three major detoxifying enzymes families in the salivary glands of the small brown planthopper, Laodelphax striatellus(Hemiptera:Delphacidae). Acta Entomologica Sinica, 56(12): 1509-1515. [in Chinese]) |
孟翔, 胡俊杰, 刘慧, 等. 2016. 荔枝蒂蛀虫转录组及嗅觉相关基因分析. 昆虫学报, 59(8): 823-830. (Meng X, Hu J J, Liu H, et al. 2016. Analysis of the transcriptome and olfaction-related genes of Conopomorpha sinensis Bradley(Lepidoptera:Gracilariidae). Acta Entomologica Sinica, 59(8): 823-830. [in Chinese]) |
潘志萍, 李敦松. 2006. 昆虫抗药性监测与检测技术研究进展. 应用生态学报, 17(8): 1539-1543. (Pan Z P, Li D S. 2006. Research advances in monitoring and detecting insect pestcide resistance. Journal of Applied Ecology, 17(8): 1539-1543. DOI:10.3321/j.issn:1001-9332.2006.08.036 [in Chinese]) |
彭建新. 2006. 昆虫分子遗传学. 武汉: 华中师范大学出版社. (Peng J X. 2006. Molecular genetics of insects. Wuhan: Huazhong Normal University Publishing House. [in Chinese]) |
魏建荣, 赵文霞, 张永安. 2011. 星天牛研究进展. 植物检疫, 25(5): 81-85. (Wei J R, Zhao W X, Zhang Y A. 2011. Research progress of Anoplophora chinensis. Plant Quarantine, 25(5): 81-85. [in Chinese]) |
杨帆, 黄立华, 张爱兵. 2014. 高通量转录组测序技术及其在鳞翅目昆虫上的应用. 昆虫学报, 57(8): 991-1000. (Yang F, Huang L H, Zhang A B. 2014. High-throughput transcriptome sequencing technology and its applications in Lepidoptera. Acta Entomologica Sinica, 57(8): 991-1000. [in Chinese]) |
尤福洋. 2016.灰飞虱(Laodelphax striatellus)谷胱甘肽S-转移酶基因的分子克隆及表达量分析.南京: 南京农业大学硕士学位论文. (You F Y. 2016. Molecular cloning and expression analysis of glutathione S-transferase from small brown planthopper(Laodelphax striatellus). Nanjing: MS thesis of Nanjing Agricultural University.[in Chinese]) http://cdmd.cnki.com.cn/Article/CDMD-10307-1017259973.htm |
张丽阳, 刘承兰. 2016. 昆虫抗药性机制及抗性治理研究进展. 环境昆虫学报, 38(3): 640-647. (Zhang L Y, Liu C L. 2016. Research progress on mechanism of insect resistance to insecticides and its management. Journal of Environmental Entomology, 38(3): 640-647. [in Chinese]) |
Chen C, Wang C C, Liu Y, et al. 2018. Transcriptome analysis and identification of P450 genes relevant to imidacloprid detoxification in Bradysia odoriphaga. Sci Rep, 8(1): 2564. DOI:10.1038/s41598-018-20981-2 |
Feng X C, Li M, Liu N N. 2018. Carboxylesterase genes in pyrethroid resistant house flies, Musca domestica. Insect Biochemistry & Molecular Biology, 92: 30-39. |
Hamilton P T, Leong J S, Koop B F, et al. 2014. Transcriptional responses in a Drosophila defensive symbiosis. Molecular Ecology, 23(6): 1558-1570. DOI:10.1111/mec.12603 |
Hedengren M, Borge K, Hultmark D. 2000. Expression and evolution of the Drosophila attacin/diptericin gene family. Biochemical & Biophysical Research Communications, 279(2): 574-581. |
Kim H S, Han J, Kim H J, et al. 2017. Identification of 28 cytochrome P450 genes from the transcriptome of the marine rotifer Brachionus plicatilis and analysis of their expression. Comparative Biochemistry & Physiology Part D Genomics & Proteomics, 23: 1-7. |
Lee B Y, Kim D H, Kim H S, et al. 2018. Identification of 74 cytochrome P450 genes and co-localized cytochrome P450 genes of the CYP2K, CYP5A, and CYP46A subfamilies in the mangrove killifish Kryptolebias marmoratus. Bmc Genomics, 19(1): 7-18. DOI:10.1186/s12864-017-4410-2 |
Lumjuan N, Mccarroll L, Prapanthadara L A, et al. 2005. Elevated activity of an Epsilon class glutathione transferase confers DDT resistance in the dengue vector, Aedes aegypti. Insect Biochemistry & Molecular Biology, 35(8): 861-871. |
Sun L, Zhang Y N, Qian J L, et al. 2018. Identification and Expression Patterns of Anoplophora chinensis(Forster)chemosensory receptor genes from the Antennal transcriptome. Frontiers in Physiology, 9: 90-102. DOI:10.3389/fphys.2018.00090 |
Vogel H, Altincicek B, Glockner G, et al. 2011. A comprehensive transcriptome and immune-gene repertoire of the lepidopteran model host Galleria mellonella. BMC Genomics, 12(26): 1-19. |
Wang J, Hu P, Gao P, et al. 2017. Antennal transcriptome analysis and expression profiles of olfactory genes in Anoplophora chinensis. Sci Rep, 7(1): 15470. DOI:10.1038/s41598-017-15425-2 |
Wu S, Zhu X, Liu Z, et al. 2016. Identification of genes relevant to pesticides and biology from global transcriptome data of Monochamus alternatus Hope(Coleoptera:Cerambycidae)larvae. PLoS One, 11(1): e0147855. DOI:10.1371/journal.pone.0147855 |
Zhang X, Kang X, Wu H, et al. 2018. Transcriptome-wide survey, gene expression profiling and exogenous chemical-induced transcriptional responses of cytochrome P450 superfamily genes in migratory locust(Locusta migratoria). Insect Biochemistry & Molecular Biology, 100: 66-77. |
Zhang Y, Li S, Xu L, et al. 2013. Overexpression of carboxylesterase-1 and mutation(F439H)of acetylcholinesterase-1 are associated with chlorpyrifos resistance in Laodelphax striatellus. Pesticide Biochemistry & Physiology, 106(1/2): 8-13. |
Zhou C, Yang H, Wang Z, et al. 2018. Comparative transcriptome analysis of Sogatella furcifera(Horváth)exposed to different insecticides. Scientific Reports, 8(1): 8773. DOI:10.1038/s41598-018-27062-4 |
Zhou J, Zhao L L, Yu H Y, et al. 2017. Comparative analysis of the Monochamus alternatus immune system. Insect Science, 25(4): 581-603. |
 2019, Vol. 55
2019, Vol. 55

