文章信息
- 卢惠君, 李子义, 梁瀚予, 岳远志, 周天畅, 杨玉璋, 王玉成, 及晓宇.
- Lu Huijun, Li Ziyi, Liang Hanyu, Yue Yuanzhi, Zhou Tianchang, Yang Yuzhang, Wang Yucheng, Ji Xiaoyu.
- 刚毛柽柳NAC24基因的表达及抗逆功能分析
- Expression and Stress Tolerance Analysis of NAC24 from Tamarix hispida
- 林业科学, 2019, 55(3): 54-63.
- Scientia Silvae Sinicae, 2019, 55(3): 54-63.
- DOI: 10.11707/j.1001-7488.20190306
-
文章历史
- 收稿日期:2018-04-24
- 修回日期:2018-06-25
-
作者相关文章
盐碱和干旱等一些常见的非生物性胁迫可能导致植物发生形态学、生理学、生物化学和分子水平等方面的变化,严重影响植物的生长发育。应用基因工程手段对植物进行遗传改良已成为提高植物抗逆性的有效手段。因此,筛选抗逆能力优良的基因,并研究其抗逆机理,从而培育抗逆改良农林新品种,已成为现代植物分子育种的主流,将在很大程度上缓解和改善我国的土地荒漠化和盐碱化的现状,推进生态文明建设,具有重要的生态、经济和社会意义。
NAC类转录因子是植物特有的最大的转录因子家族之一,广泛参与植物的生长发育过程,并在植物响应盐、干旱等多种非生物胁迫的过程中发挥至关重要的调控作用(Hao et al., 2011; Kjaersgaard et al., 2011; Yang et al., 2011; Zhong et al., 2010; Nakashima et al., 2012; Tran et al., 2010; Xia et al., 2010a; 2010b)。NAC转录因子N端的结构域高度保守,由150~160个氨基酸组成,分为A-E 5个亚结构域;C端转录调控区却高度变异,具有激活和抑制转录的功能(Shen et al., 2009; Ooka et al., 2003; Wang et al., 2013)。近年来,研究发现NAC在逆境胁迫的响应中起到了重要作用(Huang et al., 2015)。研究发现拟南芥(Arabidopsis thaliana)中的3个基因ANAC019、ANAC055和ANAC072,受盐、干旱等胁迫的诱导,过表达这些基因提高了转基因拟南芥的胁迫耐受性(Tran et al., 2004)。水稻(Oryza sativa) SNAC1基因受逆境胁迫诱导,参与水稻中大量胁迫相关基因的表达调控,过量表达SNAC1基因,显著提高了水稻的抗旱和耐盐能力(Hu et al., 2006)。小麦(Triticum aestivum)的TaNAC2基因能够响应盐、干旱、冷害和ABA胁迫,在拟南芥中过表达TaNAC2基因,提高了植株对干旱、盐和冷害的耐受力(Mao et al., 2012)。陆地棉(Gossypium hirsutum) GhNAC6受低温、高温、高盐的逆境胁迫诱导,参与植株生长发育及逆境响应过程(杨勇等, 2017)。可见,NAC转录因子的非生物胁迫研究大多集中于拟南芥、水稻、小麦及陆地棉等草本植物中,而在木本植物中相关研究则较少。
柽柳属(Tamarix)植物是盐生木本植物,广泛分布于各种不同程度的盐渍化土壤,是中亚生态系统中的关键种,在抗逆研究方面具有重要的价值(尹林克, 1995)。因此,柽柳属植物是研究木本植物耐盐分子机制,分离重要耐盐功能基因的理想物种之一。
本研究从刚毛柽柳(Tamarix hispida)NaHCO3胁迫转录组数据库中分离获得1个NAC转录因子基因, 命名为ThNAC 24。通过qRT-PCR技术分析盐、干旱胁迫条件下ThNAC24基因在不同胁迫时间点及不同组织的表达情况,初步鉴定其是否响应盐、干旱胁迫。通过农杆菌介导的高效瞬时遗传转化体系(Ji et al., 2014)获得ThNAC24基因瞬时过表达、抑制表达及对照刚毛柽柳植株。在盐、干旱胁迫下对ThNAC24基因瞬时表达及对照刚毛柽柳植株进行抗逆生理指标测定,进一步鉴定ThNAC24基因的耐盐、抗旱能力。以期为研究木本植物NAC转录因子的抗逆功能提供理论基础。
1 材料与方法 1.1 植物材料刚毛柽柳种子采自中国科学院吐鲁番沙漠植物园。将刚毛柽柳种子播种于沙子与草炭土(体积比为3 :1)混合物中,置于温度22(±2) ℃、相对湿度65%~75%、光照强度400 μmol ·m-2s-1、光周期16 h光照/8 h黑暗的人工气候室培养。选取8周苗龄的刚毛柽柳用于试验研究。
1.2 质粒与菌株大肠杆菌(Escherichia coli)Top10、根癌农杆菌(Agrobacterium tumefaciens)EHA105及植物表达载体pROKⅡ、pFGC5941均为东北林业大学林木遗传育种国家重点实验室保存。
1.3 刚毛柽柳ThNAC24基因的克隆取8周苗龄刚毛柽柳幼苗6株,用改良的CTAB法(王玉成等, 2003)提取刚毛柽柳的总RNA,并利用TransScript One-Step gDNA Removal and cDNA Synthesis SuperMIX(北京全式金生物技术有限公司)反转录试剂盒合成第1链cDNA,作为模板备用。
在实验室前期的研究工作中,根据刚毛柽柳转录组数据,筛选获得一个NAC转录因子基因,将其命名为ThNAC24 (GenBank登陆号:KF031949)。根据此序列设计克隆引物ThNAC24-F和ThNAC24-R(表 1),以刚毛柽柳cDNA为模板进行PCR扩增,将获得的片段连接到pMD18-T载体上,转化大肠杆菌,并进行测序验证。
|
|
通过DNAMAN软件,获得ThNAC24的氨基酸序列。在NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载9个不同物种的NAC序列数据,通过MUSCLE(https://www.ebi.ac.uk/Tools/msa/muscle/)进行多序列比对分析。在UniPort(http://www.uniport.org/)数据库下载拟南芥105条NAC家族序列数据,用MEGA6.0软件构建系统进化树。
1.5 非生物胁迫处理选取长势良好且大小一致的8周苗龄刚毛柽柳进行抗逆试验,分别用水、300 mmol ·L-1 NaCl溶液和400 mmol ·L-1甘露醇(Mannitol)溶液从根部浇灌刚毛柽柳,进行盐和干旱胁迫,其中水处理为对照组。在胁迫6、12、24、48 h后分别取刚毛柽柳根及叶组织,液氮冷冻后,置于-80 ℃保存,用于ThNAC24基因表达模式分析,试验进行3次重复。
1.6 ThNAC24基因表达模式分析用改良的CTAB法分别提取不同胁迫时间刚毛柽柳根及叶组织的总RNA,并反转录成cDNA。根据ThNAC24基因设计q-ThNAC-F和q-ThNAC-R引物(表 1),选取刚毛柽柳Actin (GenBank登陆号: FJ618517)和β-tubulin (GenBank登录号: FJ618519)为内参基因,引物序列见表 1。利用SYBR Green Realtime PCR Master mix(Toyobo,Japan)试剂盒,在Qtower3G(Analytik Jena,German)仪器上进行qRT-PCR。反应体系为10 μL 2×SYBR Green Realtime PCR Master mix,上下游引物各0.5 μL(20 μmol ·L-1),2 μL模板(100 ng),7 μL RNAase-free ddH2O。反应条件为:94 ℃预变性30 s;94 ℃变性12 s,60 ℃退火30 s,72 ℃延伸40 s,79 ℃读板1 s,45个循环,试验进行3次生物学重复。用2-ΔΔCt法进行基因的相对定量分析(Livak et al., 2001)。
1.7 植物过表达载体pROKⅡ-ThNAC24的构建设计引物pROK Ⅱ-ThNAC24-F和pROK Ⅱ-ThNAC24-R(表 1),引入BamH I和Sac I酶切位点,以刚毛柽柳cDNA为模板进行PCR扩增ThNAC24基因,用BamH I和Sac I对pROKⅡ质粒和纯化的ThNAC24基因进行双酶切,将ThNAC24连接到pROKⅡ载体上,转化大肠杆菌,并进行测序验证。经测序比对正确后提取重组pROKⅡ-ThNAC24载体质粒,将其转化农杆菌EHA105感受态,保存菌种备用。
1.8 植物抑制表达载体pFGC5941-ThNAC24的构建根据ThNAC24序列分别设计引物pFGC5941-ThNAC24-CIS-F和pFGC5941-ThNAC24-CIS-R(表 1),引入Nco I和Asc I酶切位点。以刚毛柽柳cDNA为模板,进行PCR扩增获得ThNAC24干扰片段,用Nco I和Asc I对pFGC5941质粒和纯化的ThNAC24基因片段进行双酶切,将ThNAC24干扰片段正向连接到pFGC5941载体上,转化大肠杆菌,并进行测序验证。经测序比对正确后提取重组载体质粒,将其命名为pFGC5941-ThNAC24-CIS。设计引物pFGC5941-ThNAC24-anti-F和pFGC5941-ThNAC24-anti-R,引入Xba I和BamH I酶切位点。用上述方法将ThNAC24干扰片段反向构建到pFGC5941-ThNAC24-CIS载体上。经测序比对正确后提取重组载体质粒,将其命名为pFGC5941-ThNAC24,将其转化农杆菌EHA105感受态,保存菌种备用。
1.9 ThNAC24基因瞬时表达刚毛柽柳的获得及胁迫处理将pROKⅡ-ThNAC24-EHA105、pFGC5941-ThNAC24-EHA105和pROKⅡ-EHA105按照农杆菌介导的高效瞬时转化系统瞬时侵染4周大小的刚毛柽柳组培苗,获得ThNAC24基因瞬时过表达(OE)、抑制表达(IE)及对照(Control)植株。为了研究ThNAC24基因在瞬时表达刚毛柽柳植株中的表达情况,将上述刚毛柽柳分别移入含100 mmol ·L-1 NaCl、150 mmol ·L-1 Manntiol的1/2 MS培养基中胁迫24 h。分别提取盐、干旱胁迫及正常条件下ThNAC24基因过表达、抑制表达及对照刚毛柽柳植株的总RNA,进行qRT-PCR分析。
1.10 ThNAC24基因瞬时表达刚毛柽柳的抗逆生理指标分析将ThNAC24基因瞬时过表达、抑制表达及对照刚毛柽柳植株分别移入含100 mmol ·L-1 NaCl、150 mmol ·L-1 Manntiol的1/2 MS培养基中胁迫处理2 h。分别取瞬时表达和对照刚毛柽柳植株叶片,置于2 mL离心管中,加入1.5 mL二氨基联苯胺(DAB)或氯化硝基四氮唑蓝(NBT)染色液,室温染色过夜。染色结束后,用75%乙醇+5%甘油沸水浴脱色,拍照。
将ThNAC24基因瞬时过表达、抑制表达及对照刚毛柽柳植株分别移入含100 mmol ·L-1 NaCl、150 mmol ·L-1 Manntiol的1/2 MS培养基中胁迫24 h,取各胁迫处理的瞬时表达和对照刚毛柽柳植株,依次测定各植株内的过氧化物酶(POD)及超氧化物歧化酶(SOD)活性,电解质渗透率、失水率(失水率分别在胁迫处理0.5、1、1.5、2、3、5、6 h测定)及丙二醛(MDA)含量,方法参照王关林等(2016)。
2 结果与分析 2.1 ThNAC24基因保守序列比对及系统进化树分析ThNAC24基因的开放阅读框为1 023 bp,编码340个氨基酸。在NCBI网站上对ThNAC24编码的氨基酸序列进行同源性搜索BLAST,发现该序列与菠菜(Spinacia oleracea) SoNAC和藜麦(Chenopodium quinoa) CqNAC的相似性分别为71%和66%。
通过多序列比对及系统进化树分析ThNAC24氨基酸序列与其他9个物种的NAC蛋白相似性,多序列比对结果显示,ThNAC24在N端的氨基酸序列相似度比较高,具有NAC家族的序列特征(图 1A)。与拟南芥105个NAC蛋白进化树分析结果显示,ThNAC24与ANAC103和ANAC082蛋白质序列亲缘关系较近(图 1B)。
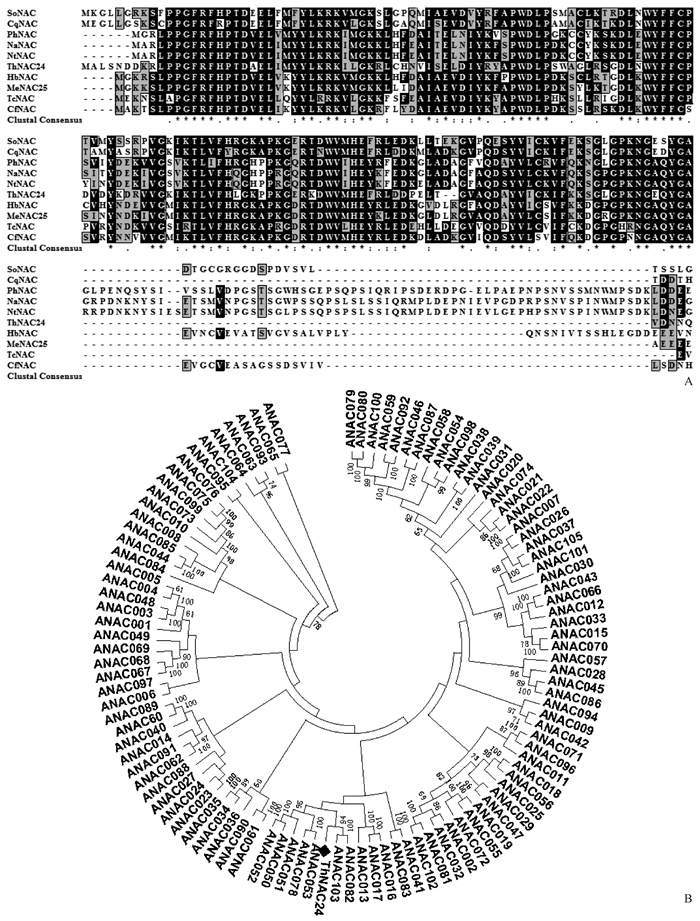
|
图 1 ThNAC24多序列比对及系统进化树分析 Fig. 1 Multiple sequence alignment and phylogenetic analysis of ThNAC24 SoNAC:菠菜Spinacia oleracea (XP_021862009.1); HbNAC:橡胶树Hevea brasiliensis (XP_021680837.1); MeNAC25:木薯Manihot esculenta (ALC79002.1); CqNAC:藜麦Chenopodium quinoa (XP_021723260.1); PhNAC:矮牵牛Petunia hybrida (AAM34765.1); NaNAC:烟草Nicotiana attenuata (XP_019259094.1); NtNAC:烟草Nicotiana tabacum (XP_016446782.1);TcNAC:可可Theobroma cacao (XP_017978285.1);CfNAC:土瓶草Cephalotus follicularis (GAV87791.1). |
为研究盐、干旱胁迫条件下ThNAC24基因的表达情况,分别提取胁迫0、6、12、24、48 h刚毛柽柳根、叶组织的总RNA,进行qRT-PCR分析。结果显示:盐胁迫下,ThNAC24基因上调表达,在根组织中胁迫12 h表达量最高,而叶组织中胁迫24 h的表达量最高(图 2)。干旱胁迫下,ThNAC24基因上调表达,在根组织中胁迫6 h表达量最高,在叶组织中胁迫12 h的表达量最高(图 2)。上述结果表明:ThNAC24基因在刚毛柽柳根和叶组织中均有表达且响应盐和干旱胁迫。

|
图 2 盐、干旱胁迫条件下ThNAC24基因的表达模式 Fig. 2 Expression of ThNAC24 in Tamarix hispida plants under salt or drought stress |
通过qRT-PCR对ThNAC24过表达(OE)、抑制表达(IE)及对照(Control)刚毛柽柳植株中ThNAC24基因的表达水平进行研究,以转化后48 h的对照刚毛柽柳植株ThNAC24的表达量作为基准值(记作1)来均一化ThNAC24基因的表达。qRT-PCR结果表明,在非胁迫条件下(1/2 MS培养基),过表达植株中ThNAC24基因的表达量明显高于对照和抑制表达植株;在盐、干旱胁迫条件下(含100 mmol ·L-1 NaCl或150 mmol ·L-1 Mannitol的1/2 MS培养基),过表达及对照植株中ThNAC24基因的表达量相对于对照显著上升,表明该基因在瞬时转化刚毛柽柳植株中已经成功过表达。在抑制表达植株中,其表达量相对于对照显著降低,说明ThNAC24在瞬时转化刚毛柽柳植株中已经成功被抑制表达(图 3)。上述结果表明,在过表达和抑制表达植株中ThNAC24的表达量显著的上升或下降,表明已经得到了功能获得和缺失的瞬时表达刚毛柽柳植株,可用于后续的功能获得及缺失研究。

|
图 3 盐、干旱胁迫下ThNAC24基因在瞬时过表达、抑制表达及对照刚毛柽柳植株中的表达情况 Fig. 3 Expression of ThNAC24 in OE, IE and Control T. hispida plants under salt or drought stress Control:对照植株; OE:瞬时过表达植株; IE:瞬时抑制表达植株。*:显著性差异(P < 0.05)。下同。 Control: Control plant; OE: Transient overexpression plant; IE: Transient inhibitory expression plant. Asterisks indicate significant difference (P < 0.05). The same below. |
选用DAB和NBT染色检测刚毛柽柳体内H2O2和超氧阴离子的含量,研究ThNAC24基因瞬时表达及对照刚毛柽柳植株的ROS水平。分别取盐、干旱胁迫处理2 h的瞬时表达和对照刚毛柽柳植株叶片(3次重复)进行DAB和NBT染色。DAB染色结果表明:在非胁迫条件下(0 h),过表达、抑制表达及对照植株棕色均较浅且无明显差异,说明各植株中H2O2含量基本相同;盐、干旱胁迫下,过表达植株叶片上的棕色明显浅于抑制表达及对照植株叶片,说明过表达植株叶片内的H2O2的含量较抑制表达及对照植株叶片的少(图 4)。NBT染色结果表明:在非胁迫条件下(0 h),过表达、抑制表达及对照植株蓝色均较浅且无明显差异,说明各植株中超氧阴离子含量基本相同;盐、干旱胁迫下,过表达植株叶片上的蓝色明显浅于抑制表达及对照植株叶片,说明过表达植株叶片内的超氧阴离子的含量较抑制表达及对照植株叶片的少(图 4)。上述结果说明过表达ThNAC24基因能够降低刚毛柽柳体内H2O2和超氧阴离子含量,从而增强刚毛柽柳的ROS清除能力。
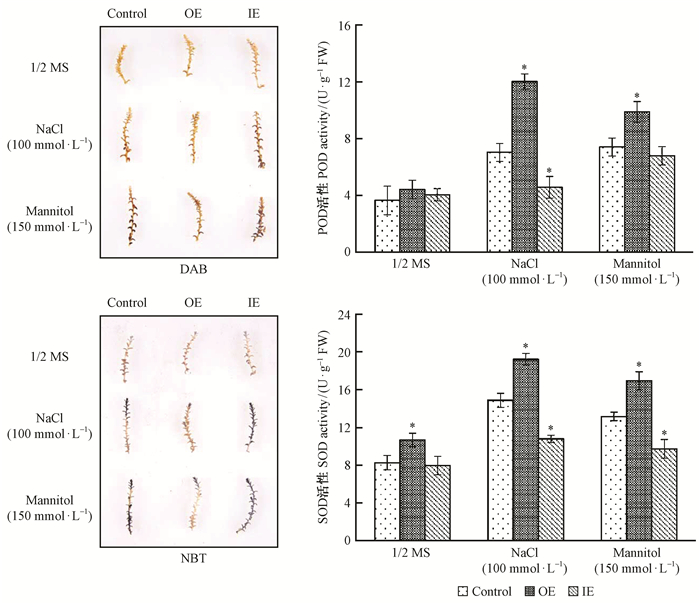
|
图 4 盐、干旱胁迫下瞬时过表达、抑制表达及对照刚毛柽柳植株ROS水平及POD、SOD活性分析 Fig. 4 Analysis of ROS accumulation and the activities of POD and SOD in OE、IE and Control T. hispida plants under salt or drought stress |
进一步研究瞬时表达及对照刚毛柽柳植株在盐、干旱胁迫条件下POD和SOD活性。分别取各胁迫处理24 h的瞬时表达和对照刚毛柽柳植株(3次重复)测定POD和SOD活性。结果如图 4所示:在正常生长条件下,ThNAC24基因过表达、抑制表达及对照植株的POD、SOD活性无明显差异;在盐、干旱胁迫条件下,ThNAC24基因过表达植株的POD、SOD活性明显高于对照及抑制表达植株。上述结果表明过表达ThNAC24基因增加了POD和SOD活性、提高了刚毛柽柳的ROS清除能力,进而提高了刚毛柽柳的耐盐、抗旱能力。
2.5 ThNAC24基因瞬时表达及对照刚毛柽柳植株细胞受损情况、失水率及MDA含量分析电解质渗透率是衡量细胞膜损伤情况的一个重要生理指标,逆境胁迫下细胞膜受损导致电解质外渗增加,破坏细胞膜结构的完整性,直接影响植物的抗逆能力。分别取各胁迫处理24 h的瞬时表达和对照刚毛柽柳植株叶片(3个重复)进行电解质渗透率测定,结果见图 5。正常生长条件下,瞬时表达和对照刚毛柽柳植株的电解质渗透率基本一致;盐、干旱胁迫条件下,抑制表达和对照植株的电解质渗透率明显高于过表达植株,其中抑制表达植株的值最高。该结果说明ThNAC24基因的过表达能降低刚毛柽柳在逆境胁迫下的电解质渗透率,从而保护细胞膜结构的完整性。
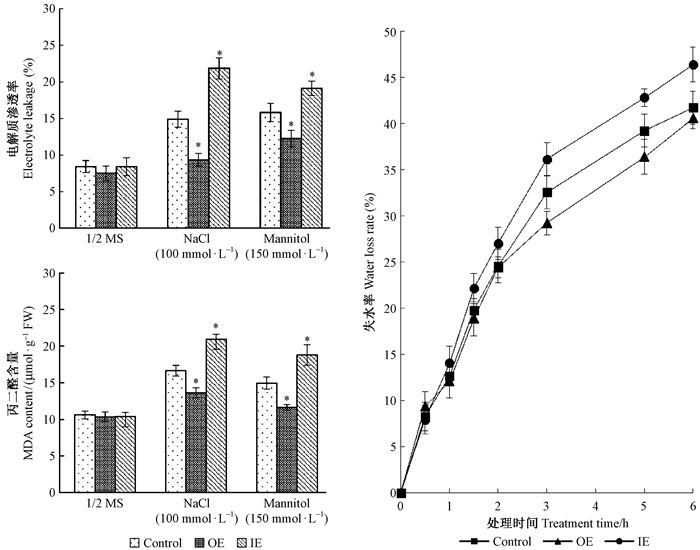
|
图 5 盐、干旱胁迫下瞬时过表达、抑制表达及对照刚毛柽柳植株电解质渗透率、失水率及MDA含量分析 Fig. 5 Electrolyte leakage, water loss rates and MDA contents analyses in OE, IE and Control T. hispida plants under salt or drought stress |
失水率是植物应对逆境生长的重要生理指标之一,抗逆能力越强则失水率越低。分别取瞬时表达和对照刚毛柽柳植株叶片(3次重复)进行失水率测定,结果如图 5所示:瞬时表达和对照刚毛柽柳植株在0.5 h内失水率无明显变化;在1~2 h内失水率均逐渐发生变化;3~6 h内失水率差异逐渐增加,其中过表达植株的失水率最低。结果表明ThNAC24基因的表达与失水率呈负相关,即ThNAC24基因的过表达可以降低植物在逆境条件下的失水率,从而增加刚毛柽柳的抗逆性。
丙二醛(MDA)是膜脂过氧化作用的产物之一,其含量可以反映植物遭受逆境伤害的程度。MDA积累越多,表明细胞膜脂损伤的程度越高。分别取瞬时表达和对照刚毛柽柳植株叶片(3次重复)进行MDA测定,结果如图 5所示:在正常生长条件下,瞬时表达和对照刚毛柽柳植株的MDA含量无明显差异;而在盐、干旱胁迫条件下,抑制表达及对照植株的MDA含量明显高于过表达植株,过表达植株的MDA含量最低,而抑制表达植株的值最高。ThNAC24基因的过表达能显著降低刚毛柽柳在逆境胁迫下MDA的积累,从而增强其抗逆性。
3 讨论本研究以盐生木本植物刚毛柽柳为研究材料,选取ThNAC24转录因子基因进行抗逆功能分析。大多数NAC蛋白质都有1个保守的N端DNA结合结构域和多样化的C端结构域(Duval et al., 2002; Ooka et al., 2003)。通过与9个物种的NAC蛋白进行多序列比对,结果显示ThNAC24具有1个保守的N端DNA结合结构域,该基因具有NAC家族的序列特征(图 1A)。与拟南芥105个NAC蛋白进化树分析结果显示,ThNAC24与ANAC103和ANAC082蛋白质序列亲缘关系较近(图 1B)。
本研究对ThNAC24基因在不同胁迫时间、不同组织中的表达情况进行分析。结果表明ThNAC24基因在刚毛柽柳根和叶组织中均有表达,且响应盐、干旱胁迫。以往的研究也表明NAC转录因子基因能够响应盐、干旱等非生物胁迫。如:拟南芥ANAC 019、ANAC055和ANAC072基因(Tran et al., 2004),水稻SNAC1基因(Hu et al., 2006),小麦TaNAC2基因(Tang et al., 2012),青杄(Picea wilsonii) PwNAC42基因(袁义杭等,2018),番茄(Solanum lycopersicum)SlNAC 11基因(Wang et al., 2017a)等NAC类转录因子基因对盐和干旱胁迫均有响应。
本研究通过农杆菌介导的高效瞬时遗传转化系统获得了ThNAC24基因瞬时过表达、抑制表达及对照刚毛柽柳植株。qRT-PCR结果表明:在过表达和抑制表达植株中ThNAC24基因的表达量显著的上升或下降,说明上述瞬时表达刚毛柽柳可用于后续的功能获得和缺失的研究。为了进一步验证刚毛柽柳ThNAC24基因的抗逆功能,分别对ThNAC24基因过表达、抑制表达及对照刚毛柽柳植株的抗逆生理指标进行了测定。根据DAB和NBT染色(图 4),POD、SOD活性结果(图 4)检测刚毛柽柳体内的ROS水平。盐、干旱胁迫下过表达植株的POD、SOD活性显著高于对照和抑制表达植株。因此,推断可能是胁迫后ThNAC24基因的表达调控POD、SOD酶相关基因的表达增加了POD和SOD的活性, 从而提高了刚毛柽柳的ROS清除能力。ThNAC24基因的过表达能降低刚毛柽柳在逆境胁迫下的电解质渗透率、失水率及MDA含量(图 5),从而增加植物的抗逆性。研究发现SNAC 3基因在水稻中过量表达,能够调节转基因水稻的ROS清除能力,从而增加其对于高温、干旱和氧化的耐受能力(Fang et al., 2015; Chen et al., 2017)。欧洲油菜(Brassica napus) NAC56和NAC55基因受逆境胁迫诱导,参与大量与胁迫相关基因的表达调控,通过增强ROS清除能力,显著提高油菜的非生物胁迫耐受性(Niu et al., 2016)。Wang等(2017b)研究发现过表达刚毛柽柳ThNAC13基因能够增强转基因拟南芥、瞬时表达刚毛柽柳的ROS清除能力,进而提高植物的耐盐和抗旱能力。这些研究表明,NAC转录因子基因能够通过增强ROS清除能力来提高植株非生物胁迫耐受性。这与本研究中过量表达ThNAC24基因能够提高刚毛柽柳耐盐和抗旱能力的结果相一致。本研究为系统研究刚毛柽柳NAC转录因子基因的抗逆分子机制奠定了基础,进一步丰富了木本植物NAC转录因子基因家族的抗逆功能研究成果。
4 结论本研究从刚毛柽柳中克隆获得了ThNAC24转录因子基因,该基因开放阅读框为1 023 bp,编码340个氨基酸。ThNAC24转录因子具有1个保守的N端DNA结合结构域,该基因具有NAC家族的序列特征,与拟南芥ANAC103和ANAC082蛋白亲缘关系较近。该基因在刚毛柽柳叶、根组织中均有表达,且响应盐、干旱胁迫。盐、干旱胁迫下过表达ThNAC24基因通过增强POD和SOD活性,进而提高ROS清除能力,减少细胞受损或死亡,从而提高刚毛柽柳的耐盐及抗旱能力。在后续的研究中,将进一步研究ThNAC24基因的抗逆分子机制。
王关林, 方宏筠. 2016. 植物基因工程实验技术指南. 2版. 北京: 科学出版社, 273-274, 277-278. (Wang G L, Fang H J. 2016. Laboratory guide for plant genetic engineering. 2nd ed. Beijing: Science Press, 273-274, 277-278. [in Chinese]) |
王玉成, 薄海侠, 杨传平. 2003. 胡杨、柽柳总RNA提取方法的建立. 东北林业大学学报, (5): 99-100. (Wang Y C, Bo H Y, Yang C P. 2003. A method for rapid isolation of total RNA from Tamarix and Populus euphratica Oliv. Journal of Northeast Forestry University, (5): 99-100. DOI:10.3969/j.issn.1000-5382.2003.05.033 [in Chinese]) |
杨勇, 田新权, 刘会利, 等. 2017. 陆地棉NAC转录因子基因GhNAC6的克隆、表达和耐盐性分析. 棉花学报, 29(2): 138-146. (Yang Y, Tian X Q, Liu H L, et al. 2017. Cloning, expression and salt-tolerance analysis of the NAC transcription factor gene GhNAC6 in Upland Cotton. Cotton Science, 29(2): 138-146. [in Chinese]) |
尹林克. 1995. 中亚荒漠生态系统中的关键种-柽柳(Tamarix spp). 干旱区研究, 12(3): 43-47. (Yin L K. 1995. Tamarix spp.-The key species of desert ecosystem. Arid Zone Research, 12(3): 43-47. [in Chinese]) |
袁义杭, 张鹤华, 游韩莉, 等. 2018. 青杄PwNAC42基因的克隆及表达模式分析. 生物技术通报, 34(4): 1-6. (Yuan Y H, Zhang H H, You H L, et al. 2018. Cloning and expression analysis of PwNAC42 in Picea wilsonii. Biotechnology Bulletin, 34(4): 1-6. [in Chinese]) |
Chen Q, Niu F, Yan J, et al. 2017. Oilseed rape NAC56 transcription factor modulates reactive oxygen species accumulation and hypersensitive response-like cell death. Physiol Plant, 160(2): 209-221. DOI:10.1111/ppl.2017.160.issue-2 |
Duval M, Hsieh T F, Kim S Y, et al. 2002. Molecular characterization of AtNAMa:member of the Arabidopsis NAC domain superfamily. Plant Molecular Biology, 50(2): 237-248. DOI:10.1023/A:1016028530943 |
Fang Y, Liao K, Du H, et al. 2015. A stress-responsive NAC transcription factor SNAC3 confers heat and drought tolerance through modulation of reactive oxygen species in rice. J Exp Bot, 66(21): 6803-6817. DOI:10.1093/jxb/erv386 |
Hao Y J, Wei W, Song Q X, et al. 2011. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants. The Plant Journal, 68(2): 302-313. DOI:10.1111/j.1365-313X.2011.04687.x |
Hu H H, Dai M Q, Yao J L, et al. 2006. Overexpressing a NAM, ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice. Proc Natl Acad Sci USA, 103(35): 12987-12992. DOI:10.1073/pnas.0604882103 |
Huang D, Wang S, Zhang B, et al. 2015. A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice. Plant Cell, 27(6): 1681-1696. DOI:10.1105/tpc.15.00015 |
Ji X Y, Zheng L, Liu Y J, et al. 2014. A transient transformation system for the functional characterization of genes involved in stress response. Plant Mol Biol Rep, 32(3): 732-739. DOI:10.1007/s11105-013-0683-z |
Kjaersgaard T, Jensen M K, Christiansen M W, et al. 2011. Senescence-associated barley NAC (NAM, ATAF1, 2, CUC) transcription factor interacts with radical-induced cell death 1 through a disordered regulatory domain. Journal of Biological Chemistry, 286(41): 35418-35429. DOI:10.1074/jbc.M111.247221 |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
Mao X, Zhang H, Qian X, et al. 2012. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances in Arabidopsis. Journal of Experimental Botany, 63(8): 2933-2946. DOI:10.1093/jxb/err462 |
Nakashima K, Takasaki H, Mizoi J, et al. 2012. NAC transcription factors in plant abiotic stress responses. Biochimica et Biophysica Acta-Gene Regulatory Mechanisma, 1819(2): 97-103. DOI:10.1016/j.bbagrm.2011.10.005 |
Niu F, Wang C, Yan J, et al. 2016. Functional characterization of NAC55 transcription factor from oilseed rape (Brassica napus L.) as a novel transcription alactivator modulating reactive oxygen species accumulation and cell death. Plant Mol Biol, 92(1/2): 89-104. |
Ooka H, Satoh K, Doi K, et al. 2003. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana. DNA Research, 10(6): 239-247. DOI:10.1093/dnares/10.6.239 |
Shen H, Yin Y, Chen F, et al. 2009. A bioinformatic analysis of NAC genes for plant cell wall development in relation to lignocellulosic bioenergy production. Bioenergy Research, 2(4): 217-232. DOI:10.1007/s12155-009-9047-9 |
Tang Y M, Liu M Y, Gao S Q, et al. 2012. Molecular characterization of novel TaNAC genes in wheat and overexpression of TaNAC2a confers drought tolerance in tobacco. Physiol Plant, 114(3): 210-224. |
Tran L S, Nakashima K, Sakuma Y, et al. 2004. Isolation and functional analysis of Arabidopsis stress inducible NAC transcription factors that bind to a drought responsive cis-element in the early responsive to dehydration stress 1 promoter. Plant Cell, 16(9): 2481-2498. DOI:10.1105/tpc.104.022699 |
Tran L S, Nishiyama R, Yamaguchi-Shinozaki K, et al. 2010. Potential utilization of NAC transcription factors to enhance abiotic stress tolerance in plants biotechnological approach. GM Crops, 1(1): 32-39. DOI:10.4161/gmcr |
Wang L, Li Z, Lu M, et al. 2017b. ThNAC13, a NAC transcription factor from Tamarix hispida, confers salt and osmotic stress tolerance to transgenic Tamarix and Arabidopsis. Front Plant Sci, 8: 635. DOI:10.3389/fpls.2017.00635 |
Wang L L, Hu Z L, Zhu M K, et al. 2017a. The abiotic stress-responsive NAC transcription factor SlNAC11 is involved in drought and salt response in tomato (Solanum lycopersicum L.). Plant Cell Tissue and Organ Culture, 129(1): 161-174. DOI:10.1007/s11240-017-1167-x |
Wang Z, Dane F. 2013. NAC (NAM/ATAF/CUC) transcription factors in different stresses and their signaling pathway. Acta Physiologiae Plantarum, 35(5): 1397-1408. DOI:10.1007/s11738-012-1195-4 |
Xia N, Zhang G, Liu X Y, et al. 2010a. Characterization of a novel wheat NAC transcription factor gene involved in defense response against stripe rust pathogen infection and abiotic stresses. Molecular Biology Reports, 37(8): 3703-3712. DOI:10.1007/s11033-010-0023-4 |
Xia N, Zhang G, Sun Y F, et al. 2010b. TaNAC8, a novel NAC transcription factor gene in wheat, responds to stripe rust pathogen infection and abiotic stresses. Physiological and Molecular Plant Pathology, 74(5/6): 394-402. |
Yang S D, Seo P J, Yoon H K, et al. 2011. The Arabidopsis NAC transcription factor VNI2 integrates abscisic acid signals into leaf senescence via the COR/RD genes. The Plant Cell, 23(6): 2155-2168. DOI:10.1105/tpc.111.084913 |
Zhong R, Lee C, Ye Z H. 2010. Global analysis of direct targets of secondary wall NAC master switches in Arabidopsis. Molecular Plant, 3(6): 1087-1103. DOI:10.1093/mp/ssq062 |
 2019, Vol. 55
2019, Vol. 55

