文章信息
- 于涛, 张宇阳, 高健, 柯蕾, 马文宝, 李俊清.
- Yu Tao, Zhang Yuyang, Gao Jian, Ke Lei, Ma Wenbao, Li Junqing.
- 极小种群濒危植物盐桦叶绿体基因组特征分析
- Complete Chloroplast Genome Sequence of Betula halophila, a Plant Species with Extremely Small Populations
- 林业科学, 2019, 55(2): 41-49.
- Scientia Silvae Sinicae, 2019, 55(2): 41-49.
- DOI: 10.11707/j.1001-7488.20190205
-
文章历史
- 收稿日期:2018-03-05
- 修回日期:2018-06-26
-
作者相关文章
2. 包头师范学院资源与环境学院 包头 014030;
3. 四川省林业科学研究院 国家林业和草原局四川森林生态与资源环境重点实验室 成都 610081
2. Faculty of Resources and Environment, Baotou Teachers'College Baotou 014030;
3. Key Laboratory of National Forestry and Grassland Administration on Sichuan Forest Ecology and Resources and Environment Sichuan Academy of Forestry Chengdu 610081
叶绿体基因组的结构和基因组成在高等植物比较保守,为双链环状结构(王玲等,2012)。遗传方式以单亲遗传为主,通常在植物中只有1个单倍型遗传,重组率低,后代遗传稳定(Birky,1995)。因此,叶绿体基因作为研究植物各个分类单元系统进化的首选分子标记(Clegg et al., 1994;张韵洁等,2011)。以往系统发育学研究所使用的叶绿体基因片段较短,不能全面系统地解析物种之间的系统发育关系,而如今测序技术的发展,为进行大批量叶绿体基因组的测序提供了可能。植物叶绿体基因组测序的完成,可以为物种系统发育重建等相关研究提供大量的分子标记片段,此外,叶绿体基因组信息还可应用于植物的遗传转化、分子育种等方面,可为重要经济作物、药用植物、濒危植物的资源保护和利用提供基础资料(Nie et al., 2012;桂腾琴等,2009)。
盐桦(Betula halophila)为桦木科(Betulaceae)桦木属(Betula)植物,落叶小乔木,由秦仁昌教授在新疆阿勒泰巴里巴盖采集并命名,新疆特有的濒危种(张立运等,2000),国家二级珍稀濒危植物并选入《极小种群野生植物拯救保护工程规划(2011—2015)》名录(汪智军等,2003;国政等,2013)。陶玲等(2001)以分布区、分类学地位、生物学特性和利用价值4个指标对中国50种荒漠植物进行珍稀濒危程度的综合定量评价,盐桦的综合评价值为第2位,仅次于四合木(Tetraena mongolica)。近年来,对于盐桦的研究主要集中在组织培养与耐盐机理方面(梅新娣等,2006;李宏等,2009),而对于分子系统发育研究较少,仅梅新娣等(2008)和Wang等(2016)使用ITS片段区序列对其进行系统发育研究,尚未见利用叶绿体基因序列信息对盐桦系统发育进行研究的报道。
本文首次报道了盐桦叶绿体全基因组基本特征信息,并与其近缘种欧洲矮桦进行了生物信息学比较分析,有助于桦木属植物进化模式与系统发育研究,也为极小种群野生植物盐桦的保护工作提供了遗传背景信息。
1 材料与方法 1.1 试验材料盐桦样品由中国林业科学研究院森林生态环境与保护研究所提供,采集实生苗(种子采集于新疆阿勒泰巴里巴盖,播种育苗)幼叶使用硅胶干燥后保存。全基因组DNA的提取使用植物基因组DNA提取试剂盒(TIANGEN Beijing China)。DNA样品经上海欧易信息科技有限公司检测合格后,使用Illumina Xten测序仪,建库插入片段长度350 bp,采用双末端150 bp测序策略进行测序。
1.2 叶绿体基因组组装通过HiSeq Xten测序平台对样品进行测序,为了提高后续分析的可靠性,对原始数据使用NGSQC软件(Patel et al., 2012),参数设置为去除Q20比例低于70%的短序列(reads)。截取3′端质量低于Q20的碱基,去除包含N碱基的reads。去除截取后总长小于70 bp的reads。得到测序有效数据(clean data)30 000 000条。以发表的欧洲矮桦(Betula nana)(NC_033978)叶绿体基因组序列为参考,利用bwa+samtools软件(Li et al., 2009a;2009b)从clean data中筛选叶绿体基因组reads,通过MITObim v1.8 (Hahn et al., 2013)和NOVOplasty (Dierckxsens et al., 2016)进行叶绿体基因组的组装。SAMtools软件(Li et al., 2009b)查看覆盖度信息对组装结果进行修正,得到最终的盐桦叶绿体基因组。
1.3 叶绿体基因注释和比对分析CpGAVAS (Liu et al., 2012)对盐桦叶绿体基因组蛋白编码基因、tRNA和rRNA进行注释;BLAST (McGinnis et al., 2004)比对NCBI数据库对基因注释结果进行修正;将注释的结果用NCBI网站指定的Sequin软件提交盐桦叶绿体基因组序列数据和注释信息。通过OGDRAW软件(Lohse et al., 2013)呈现叶绿体基因组物理图谱;盐桦和欧洲矮桦的叶绿体基因组序列通过MAFFT软件(Katoh et al., 2002)比对后,使用DnaSP v.5 (Librado et al., 2009)进行核酸多态性的滑动框检测。设置滑动框为600 bp,步长为200 bp。
1.4 SSR分析Perl程序MISA (MIcroSAtellite identification tool) (Parida et al., 2010)分析盐桦叶绿体基因组中简单重复序列(simple sequence repeats,SSRs),软件参数设置为1-10、2-4、3-4、4-3、5-3和6-3,即单核苷酸重复数≥10,二核苷酸重复数≥4,三核苷酸重复数≥4,四核苷酸≥3,五核苷酸重复数≥3,六核苷酸重复数≥3。并且设置2个SSR之间的最小距离若小于100 bp,认定为一个复合微卫星。
1.5 基因受选择分析基因正选择分析使用盐桦与欧洲矮桦蛋白质编码基因,采用MAFFT对基因序列进行两两比对,利用DnaSP v.5对比对后序列进行受选择分析,计算非同义替换率ka, 同义替换率ks与ka/ks值。以ka/ks >1为受正选择标准。
1.6 系统发育分析系统发育分析使用14个物种的叶绿体基因组,选择拟南芥(Arabidopsis thaliana)NC000932、银白杨(Populus alba)AP008956为外类群,使用MAFFT对序列进行比对,PAPU (Swofford,2003)与MEGA (Tamura et al., 2007)软件分别构建最大简约法(Maximum parsimony,MP)和邻近法(Neighbor-Joining,NJ)系统发育树。
2 结果与分析 2.1 盐桦叶绿体基因组结构分析盐桦叶绿体基因组为闭合的双链环状分子,包含大单拷贝区(LSC)、小单拷贝区(SSC)和反向重复区a、b(IRs: IRa和IRb)4个区段(图 1)。序列全长160 648 bp,鸟嘌呤(G)和胞嘧啶(C)所占的比率(GC)为36.1%。其中LSC区段长89 553 bp,GC含量33.7%;SSC区段长19 027 bp,GC含量为29.7%;2个IRs区段均为26 034 bp,GC含量均为42.5% (表 1)。
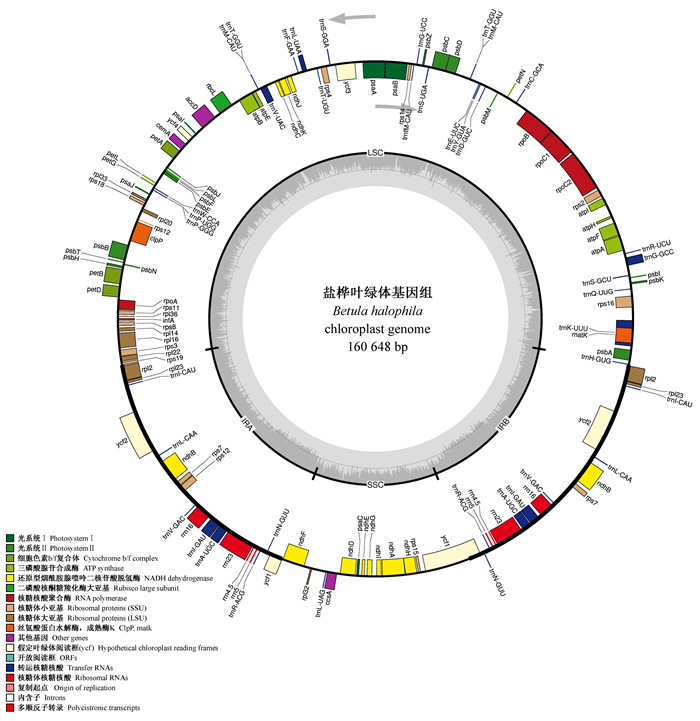
|
图 1 盐桦叶绿体基因组物理图谱 Fig. 1 Schematic representation of the Betula halophila chloroplast genome |
|
|
根据CpGAVAS软件及BLAST比对结果,成功注释了盐桦叶绿体基因组共114个基因,包括79个蛋白编码基因,31个tRNA基因和4个rRNA基因,其中20个基因含有2个拷贝,包括4个rRNA基因(rrn4.5、rrn5、rrn16和rrn23),9个tRNA基因(trnA-UGC、trnI-CAU、trnI-GAU、trnL-CAA、trnM-CAU、trnN-GUU、trnR-ACG、trnT-GGU、trnV-GAC)和7个蛋白编码基因(ndhB、rpl2、rpl23、rps7、rps12、ycf1和ycf2)(表 2)。
|
|
蛋白编码基因序列总长81 576 bp,占叶绿体基因组总长的50.8%,GC含量37.0%;tRNA基因序列总长3 002 bp,占叶绿体基因组序列的1.9%,GC含量为49.7%;rRNA基因序列总长9 049 bp,占叶绿体基因组序列的5.63%,GC含量为55.2%。
在蛋白编码基因中,具有内含子(intron)的基因共有12个,其中9个基因(rps16、atpF、rpoC1、petB、petD、rpl16、rpl2、ndhB和ndhA)含有1个内含子,其余3个基因(ycf3、rps12和clpP)则含有2个内含子;tRNA中有6个含有内含子,分别为trnK-UUU、trnG-GCC、trnL-UAA、trnV-UAC、trnI-GAU和trnA-UGC(表 3)。对比欧洲矮桦和盐桦叶绿体基因组发现,序列长度上欧洲矮桦(160 579 bp)略短于盐桦(160 648 bp),主要是盐桦IRs区长度较长导致,二者碱基GC含量相似,基因构成和次序一致。
|
|
盐桦叶绿体基因组筛选得到91个SSR位点,其中33个SSRs均由A或T组成,其中单核苷酸重复类型33个,二核苷酸重复类型41个,三核苷酸重复类型7个,四核苷酸重复类型5个,五核苷酸重复类型5个(图 2)。LSC区段、SSC区段和IRs区域分别有56个、13个和22个SSRs。
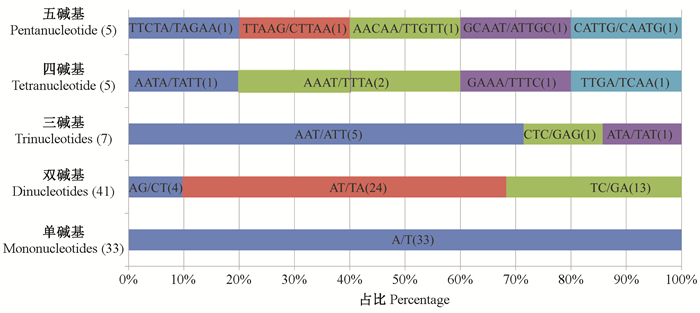
|
图 2 盐桦叶绿体基因组物理图谱SSRs位点类型及数量 Fig. 2 Simple sequence repeats (SSRs) sequences in the Betula halophila chloroplast genomes |
盐桦和欧洲矮桦的叶绿体基因组序列经MAFFT比对后通过DnaSP软件分析,结果显示,ndhC-trnV、petA-psbJ、rpl22-rps19区域的核酸变异度(nucleotide variability, π>0.02)显著高于其他区域,高变区都位于LSC区,IRs区和SSC区内2个种的变异度较小(图 3),说明叶绿体基因组中各区段的核酸变异度存在不同,LSC区是变异发生的高频区域。3个高变区的筛选和不同区域核酸变异度的检测,都为今后盐桦谱系地理和桦木属植物系统发育学等研究提供了基础。
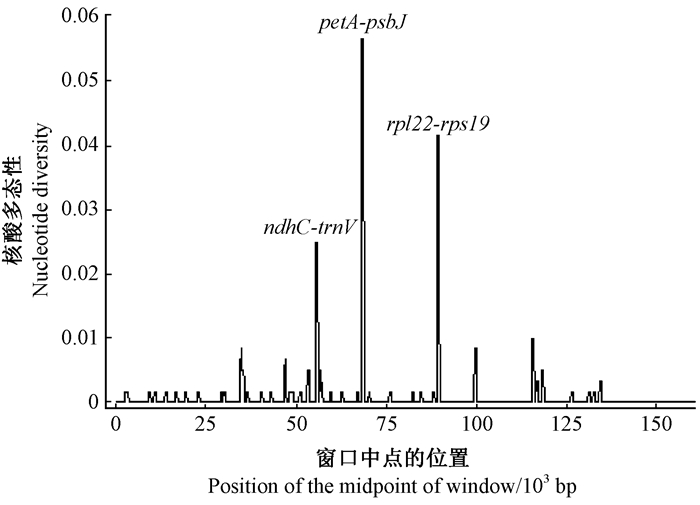
|
图 3 滑动框分析盐桦和欧洲矮桦叶绿体基因组核酸多样性 Fig. 3 Sliding window analysis for the nucleotide diversity (π) of the whole chloroplast genomes between two Betula species 框长:600 bp;步长:200 bp。Window length: 600 bp; Step size: 200 bp. |
将盐桦和欧洲矮桦叶绿体编码基因通过DnaSP v.5进行基因受选择分析(图 4),基因同义替换率ks值较低,基因在图中分布集中于左侧,显示出盐桦与欧洲矮桦的分化时间并不久远,没有大量的突变累计下来。4个基因的ka/ks值大于1,显示出强烈的受选择信号,ka/ks值大小顺序依次为ycf1 (4.5), rpoC1 (3.29), rpl2 (1.44), ndhA (1.32)。
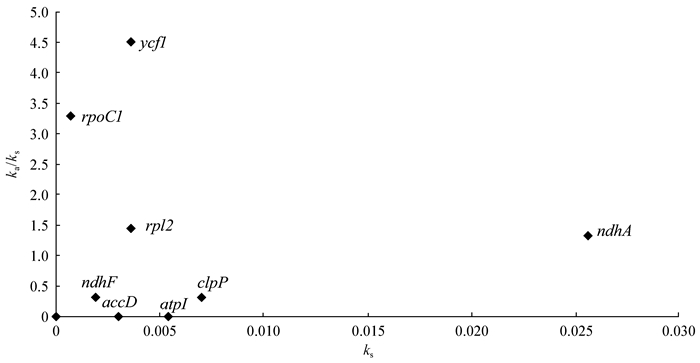
|
图 4 盐桦和欧洲矮桦叶绿体基因ks与ka/ks分析 Fig. 4 Gene-specific ks and ka/ks values between the chloroplast genomes of two Betula species (B. halophila and B. nana) ka:非同义替换率; ks:同义替换率。 ka: Nonsynonymous rate; ks: Synonymous rate. |
将盐桦和欧洲矮桦与其他双子叶植物共14条叶绿体基因组序列通过MP、NJ方法进行聚类分析,MP、NJ树11个节点中有9个支持度为100%,支持盐桦和欧洲矮桦亲缘关系最近,桦木属物种与铁木属天目铁木(Ostrya rehderiana)在同一分支(图 5)。
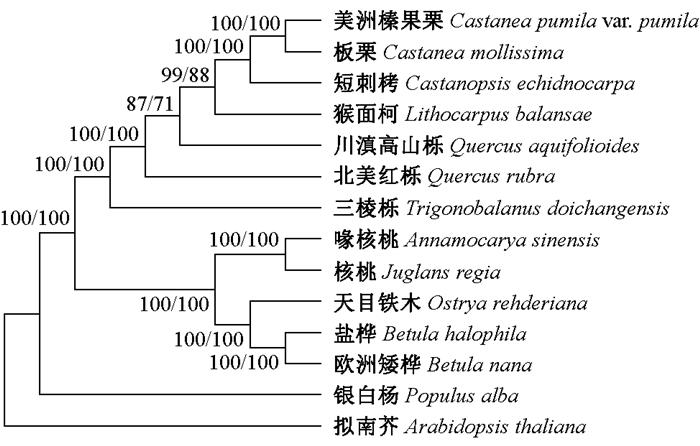
|
图 5 基于叶绿体全基因组的14个植物的NJ和MP聚类 Fig. 5 Phylogenetic tree reconstructed by the Neighbour-Joining and maximum parsimonious methods according to complete chloroplast genomes of 14 species |
本研究利用二代测序技术对盐桦叶绿体基因组进行测序,并组装得到完整的叶绿体基因组序列,是继欧洲矮桦后公布的第2个桦木属植物叶绿体基因组。通过比对分析,目前公布的桦木属2个种的叶绿体基因组碱基GC含量相似,基因构成和次序一致,体现了叶绿体基因组结构稳定,总体进化速率较低(Dong et al., 2016)。因为叶绿体基因组在总体上的保守性,在利用叶绿体基因组信息时,通常会筛选其中的高变区如matK、trnH-psbA、rbcL等常用的叶绿体片段。利用叶绿体全基因组信息,可全面准确地筛选出叶绿体基因组中高变区。紫薇属(Lagerstroemia)植物中利用叶绿体全基因组信息寻找高变区,并利用高变区信息重构系统发育关系,显示利用筛选出的片段信息可以得出与全基因组信息一致的结果(Xu et al., 2017)。在盐桦和欧洲矮桦的比对研究中,通过滑动框分析序列的核酸多态性发现,常用的叶绿体分子条形码区段,在盐桦和欧洲矮桦的比对中变异不明显。同时ndhC-trnV、petA-psbJ、rpl22-rps19片段的核酸变异度明显高于其他区域,在今后桦木属植物系统发育学和谱系地理学的研究中叶绿体分子条形码的选择应该优先考虑。SSRs是一种高效的分子标记,在群体水平和个体水平的遗传差异分析得到了广泛的应用。盐桦叶绿体基因组的SSRs位点分析发现,SSRs的碱基组成偏向使用A/T碱基,与其他叶绿体基因组SSRs分析结果(Xu et al., 2017)类似,盐桦和欧洲矮桦叶绿体基因组的SSRs构成具有很高的相似性,rpoC2、rpoC1、ycf2、ndhB都含有二核苷酸重复序列,四核苷酸重复序列在rpl32和ndhE中都有存在,其余大部分都位于基因间区。
对于盐桦和欧洲矮桦SSRs的比对研究,有助于寻找桦木属叶绿体基因组中的通用SSRs,可应用于后续桦木属植物的深入研究中。盐桦和欧洲矮桦叶绿体基因组基因受选择研究显示,共有4个基因(ycf1, rpoC1, rpl2, ndhA)受到强烈的正选择信号,但ycf1与rpoC1基因ka值较大可能因其为假基因,这2个基因在系统发育的研究之中都作为DNA分子条形码使用(Dong et al., 2015;Parveen et al., 2017);没有特定功能或功能缺失而受到放松的进化压力,如崖柏属(Thuja)物种叶绿体基因组中chlL与chlN基因丧失功能,其ka值明显高于其近缘物种(Kusumi et al., 2006)。NdhA属于NADH脱氢酶亚基成熟酶基因, 该功能基因对强光敏感,在栎属(Quercus)的研究中存在正选择(Peng et al., 2011; Yang et al., 2016)。Rpl2 为参与核糖体蛋白编码基因,核糖体蛋白质的研究显示,其在植物抵御胁迫等方面有重要的作用,如黑麦(Secale cereale)rps7基因与水稻(Oryza sativa)rpl14基因分别响应低温与干旱的胁迫(Berberich et al., 2000;杜志如等,2008)。盐桦生境处于高盐分胁迫的条件下,可能对核糖体蛋白质编码基因rpl2存在正选择作用。
由于已报道研究所选用的片段信息量不足,致使杂交频繁的桦木属系统发育关系并没有得到较好的解决(Wang et al., 2016),而叶绿体基因组信息是对桦木属系统发育关系解释较好的补充。本研究利用欧洲矮桦作为参考,对盐桦进行叶绿体全基因组的拼接,得到全长160 648 bp的盐桦叶绿体基因组序列,但受限于桦木科其他物种的叶绿体基因组尚未被测序,不能整体性地重建桦木科整体的系统发育关系。随着测序技术的发展,细胞器基因组测序正在成为解决系统发育学研究复杂类群的方法,特别是在属下分类学水平上。利用叶绿体全基因组信息,先后在北美箭竹族(Arundinarieae)和蔷薇科(Rosaceae)等系统发育关系复杂、重建难度高的类群研究中起到了很好的效果(Ma et al., 2014;Zhang et al., 2017)。在不久的将来,细胞器基因组的大规模测序将有利于打破目前使用短序列进行系统发育研究的局限性(Parks et al., 2009;Nock et al., 2011),并结合核基因组中的遗传信息可更好地解决桦木属系统发育研究问题。
4 结论随着高通量测序技术的发展和叶绿体基因组数据的增加,采用对植物全基因组DNA进行测序的方法,利用近缘种叶绿体基因组信息进行拼接组装叶绿体基因组是如今较为经济和准确的研究叶绿体基因组的方式。本文对极小种群植物盐桦进行叶绿体基因组组装和注释,对比分析了其叶绿体基因组结构、基因组成和SSRs分布,发现研究结果都与欧洲矮桦相似,通过比对发现二者存在3个差异性较高的片段区域,可作为桦木属植物叶绿体基因组分子条形码选择的基础。并对盐桦与欧洲矮桦的叶绿体基因进行正选择分析,利用ka/ks筛选出4个具有正选择信号的基因。本研究丰富了桦木属的叶绿体基因组数据库,为今后极小种群盐桦的保护与利用奠定了基础。
杜志如, 席江, 万佳, 等. 2008. 水稻核糖体蛋白(OsRPL14)基因的克隆及表达分析. 中国农学通报, 24(4): 130-134. (Du Z R, Xi J, Wan J, et al. 2008. Cloning and expression analysis of a ribosomal protein from rice(OsRPL14). Chinese Agricultural Science Bulletin, 24(4): 130-134. [in Chinese]) |
桂腾琴, 孙敏. 2009. 叶绿体基因工程的研究进展. 安徽农业科学, 37(8): 3429-3431. (Gui T Q, Sun M. 2009. Progress of studies on chloroplast genetic engineering. Journal of Anhui Agricultural Sciences, 37(8): 3429-3431. DOI:10.3969/j.issn.0517-6611.2009.08.037 [in Chinese]) |
国政, 臧润国. 2013. 中国极小种群野生植物濒危程度评价指标体系. 林业科学, 49(6): 10-17. (Guo Z, Zang R G. 2013. Evaluation index system of endangered levels of the wild plants with tiny population in China. Scientia Silvae Sinicae, 49(6): 10-17. DOI:10.3969/j.issn.1672-8246.2013.06.002 [in Chinese]) |
李宏, 邓江宇, 张红, 等. 2009. NaCl胁迫对盐桦幼苗生理特性的影响. 西北植物学报, 29(11): 2281-2287. (Li H, Deng J Y, Zhang H, et al. 2009. Physiological characteristics of Betula halophila seedlings under NaCl stress. Acta Botanica Boreali-Occidentalia Sinica, 29(11): 2281-2287. DOI:10.3321/j.issn:1000-4025.2009.11.020 [in Chinese]) |
梅新娣, 马纪, 张富春. 2006. 新疆濒危植物盐桦试管苗生根培养的研究. 新疆农业科学, 43(3): 218-223. (Mei X D, Ma J, Zhang F C. 2006. Rooting cultivation of rare endangered plant Betula halophila in vitro. Xinjiang Agricultural Sciences, 43(3): 218-223. DOI:10.3969/j.issn.1001-4330.2006.03.013 [in Chinese]) |
梅新娣, 张富春. 2008. 应用ITS序列分析新疆濒危植物盐桦的系统发育. 生物技术, 18(6): 4-6. (Mei X D, Zhang F C. 2008. Phylogeny of endangered plant species-Betula halophila in Xinjiang inferred from ITS sequence data. Xinjiang Agricultural Sciences, 18(6): 4-6. [in Chinese]) |
陶玲, 李新荣, 刘新民, 等. 2001. 中国珍稀濒危荒漠植物保护等级的定量研究. 林业科学, 37(1): 52-57. (Tao L, Li X R, Liu X M, et al. 2001. Quantitative study of conservation grading of rare and endangered desert plants in China. Scientia Silvae Sinicae, 37(1): 52-57. DOI:10.3321/j.issn:1001-7488.2001.01.008 [in Chinese]) |
王玲, 董文攀, 周世良. 2012. 被子植物叶绿体基因组的结构变异研究进展. 西北植物学报, 32(6): 1282-1288. (Wang L, Dong W P, Zhou S L. 2012. Structural mutations and reorganizations in chloroplast genomes of flowering plants. Acta Botanica Boreali-Occidentalia Sinica, 32(6): 1282-1288. DOI:10.3969/j.issn.1000-4025.2012.06.031 [in Chinese]) |
汪智军, 李行斌, 郭仲军, 等. 2003. 新疆14种珍稀濒危植物资源现状及保护. 中国野生植物资源, 22(2): 15-16. (Wang Z J, Li X B, Guo Z J, et al. 2003. 14 rare and endangered plant resources in Xinjiang and their protection. Chinese Wild Plant Resources, 22(2): 15-16. DOI:10.3969/j.issn.1006-9690.2003.02.006 [in Chinese]) |
张立运, 潘伯荣. 2000. 新疆植物资源评价及开发利用. 干旱区地理, 23(4): 334-335. (Zhang L Y, Pan B R. 2000. Evaluation on exploitation and utilization of plant resources in Xinjiang. Arid Land Geography, 23(4): 334-335. [in Chinese]) |
张韵洁, 李德铢. 2011. 叶绿体系统发育基因组学的研究进展. 植物分类与资源学报, 33(4): 365-375. (Zhang Y J, Li D Z. 2011. Advances in phylogenomics based on complete chloroplast genomes. Plant Diversity and Resources, 33(4): 365-375. [in Chinese]) |
Berberich T, Uebeler M, Feierabend J. 2000. cDNA cloning of cytoplasmic ribosomal protein S7 of winter rye (Secale cereale) and its expression in low-temperature-treated leaves. Biochimica Et Biophysica Acta, 1492(1): 276-279. DOI:10.1016/S0167-4781(00)00103-2 |
Birky C W. 1995. Uniparental inheritance of mitochondrial and chloroplast genes:mechanisms and evolution. Proc Natl Acad Sci USA, 92(25): 11331-11338. DOI:10.1073/pnas.92.25.11331 |
Clegg M T, Gaut B S, Learn G H, et al. 1994. Rates and patterns of chloroplast DNA evolution. Proc Natl Acad Sci USA, 91(15): 6795-6801. DOI:10.1073/pnas.91.15.6795 |
Dierckxsens N, Mardulyn P, Smits G. 2016. NOVOPlasty: de novo assembly of organelle genomes from whole genome data. Nucleic Acids Research, doi: 10.1093/nar/gkw955.
|
Dong W P, Xu C, Li C H, et al. 2015. Ycf1, the most promising plastid DNA barcode of land plants. Scientific Reports, 5: 8348. DOI:10.1038/srep08348 |
Dong W P, Xu C, Li D L, et al. 2016. Comparative analysis of the complete chloroplast genome sequences in psammophytic Haloxylon species (Amaranthaceae). PeerJ, 4(2): e2699. |
Hahn C, Bachmann L, Chevreux B. 2013. Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads-a baiting and iterative mapping approach. Nucleic Acids Res, 41(13): e129. DOI:10.1093/nar/gkt371 |
Katoh K, Misawa K, Kuma K I, et al. 2002. MAFFT:a novel method for rapid multiple sequence alignment based on fast Fourier transform. Nucleic Acids Res, 30(14): 3059-3066. DOI:10.1093/nar/gkf436 |
Kusumi J, Sato A, Tachida H. 2006. Relaxation of functional constraint on light-independent protochlorophyllide oxidoreductase in Thuja. Molecular Biology & Evolution, 23(5): 941-948. |
Li H, Durbin R. 2009a. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 25: 1754-1760. DOI:10.1093/bioinformatics/btp324 |
Li H, Handsaker B, Wysoker A, et al. 2009b. The sequence Alignment/Map format and SAMtools. Bioinformatics, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 |
Librado P, Rozas J. 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
Liu C, Shi L, Zhu Y, et al. 2012. CpGAVAS, an integrated web server for the annotation, visualization, analysis, and GenBank submission of completely sequenced chloroplast genome sequences. Bmc Genomics, 13(1): 715. DOI:10.1186/1471-2164-13-715 |
Lohse M, Drechsel O, Kahlau S, et al. 2013. Organellar Genome DRAW-a suite of tools for generating physical maps of plastid and mitochondrial genomes and visualizing expression data sets. Nucleic Acids Research, 41: W575-581. DOI:10.1093/nar/gkt289 |
Ma P F, Zhang Y X, Zeng C X, et al. 2014. Chloroplast phylogenomic analyses resolve deep-level relationships of an intractable bamboo tribe Arundinarieae(Poaceae). Systematic Biology, 63(6): 933. DOI:10.1093/sysbio/syu054 |
McGinnis S, Madden T L. 2004. BLAST:at the core of a powerful and diverse set of sequence analysis tools. Nucleic Acids Res, 32: W20-25. DOI:10.1093/nar/gkh435 |
Nie X J, Lu S Z, Zhang Y X, et al. 2012. Complete chloroplast genome sequence of a major invasive species, Crofton weed (Ageratina adenophora). PLoS One, 7(5): e36869. DOI:10.1371/journal.pone.0036869 |
Nock C J, Waters D L, Edwards M A, et al. 2011. Chloroplast genome sequences from total DNA for plant identification. Plant Biotechnol J, 9: 328-333. DOI:10.1111/pbi.2011.9.issue-3 |
Parida S K, Yadava D K, Mohapatra T. 2010. Microsatellites in Brassica unigenes:relative abundance, marker design, and use in comparative physical mapping and genome analysis. Genome, 53(1): 55-67. DOI:10.1139/G09-084 |
Parks M, Cronn R, Liston A. 2009. Increasing phylogenetic resolution at low taxonomic levels using massively parallel sequencing of chloroplast genomes. BMC Biology, 7: 84. DOI:10.1186/1741-7007-7-84 |
Parveen I, Singh H K, Malik S, et al. 2017. Evaluating five different loci (rbcL, rpoB, rpoC1, matK and ITS) for DNA barcoding of Indian orchids. Genome, 60(8): 665-671. DOI:10.1139/gen-2016-0215 |
Patel R K, Jain M. 2012. QC Toolkit:A Toolkit for quality control of next generation sequencing data. PLoS One, 7(2): e30619. DOI:10.1371/journal.pone.0030619 |
Peng L W, Yamamoto H, Shikanai T. 2011. Structure and biogenesis of the chloroplast NAD(P)H dehydrogenase complex. Biochimica et Biophysica Acta(BBA)-Bioenergetics, 1807(8): 945-953. DOI:10.1016/j.bbabio.2010.10.015 |
Swofford D L. 2003. PAUP*: Phylogenetic analysis using parsimony (*and other methods), Version 4. Sunderland Massachusetts, USA: Sinauer Associates.
|
Tamura K, Dudley J, Nei M, et al. 2007. MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4. 0. Molecular Biology and Evolution, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
Wang N, Mcallister H A, Bartlett P R, et al. 2016. Molecular phylogeny and genome size evolution of the genus Betula (Betulaceae). Annals of Botany, 117(6): 1023-1035. DOI:10.1093/aob/mcw048 |
Xu C, Dong W P, Li W Q, et al. 2017. Comparative analysis of six Lagerstroemia complete chloroplast genomes. Frontiers in Plant Science, 8: article 15.
|
Yang Y C, Zhou T, Duan D, et al. 2016. Comparative analysis of the complete chloroplast genomes of five Quercus species. Frontiers in Plant Science, 7: article 959.
|
Zhang S D, Jin J J, Chen S Y, et al. 2017. Diversification of Rosaceae since the Late Cretaceous based on plastid phylogenomics. New Phytologist, 214(3): 1355-1367. DOI:10.1111/nph.14461 |
 2019, Vol. 55
2019, Vol. 55

