文章信息
- 杨安娜, 陆云峰, 张俊红, 吴家森, 徐金良, 童再康.
- Yang Anna, Lu Yunfeng, Zhang Junhong, Wu Jiasen, Xu Jinliang, Tong Zaikang.
- 杉木人工林土壤养分及酸杆菌群落结构变化
- Changes in Soil Nutrients and Acidobacteria Community Structure in Cunninghamia lanceolata Plantations
- 林业科学, 2019, 55(1): 119-127.
- Scientia Silvae Sinicae, 2019, 55(1): 119-127.
- DOI: 10.11707/j.1001-7488.20190114
-
文章历史
- 收稿日期:2017-10-08
- 修回日期:2018-03-05
-
作者相关文章
2. 浙江省宁波市林业局林特种苗繁育中心 宁波 315012;
3. 浙江省开化县林场 开化 324300
2. The Seeding Breeding Center of Ningbo Forestry Bureau Ningbo 315012;
3. Kaihua Forestry Farm, Zhejiang Province Kaihua 324300
杉木(Cunninghamia lanceolata)是我国特有速生针叶树种,在我国南方山区栽培面积最大。杉木根系分泌物中的大量有机酸及酚酸类物质导致土壤酸化(陈龙池等,2002),且杉木凋落物量少、分解周期长,导致林地土壤养分下降(何友军等,2006),自20世纪70年代以来,大面积杉木人工林营建及其在同一地块连续多代种植,导致土壤酸化和贫瘠化,林分生产力下降(张志才等,2012;Li et al., 2015; 王士亚,2016)。研究表明,土壤pH值、土壤碱解氮、有效磷、速效钾以及有机碳能反映土壤养分的储存与供应,是评价土壤肥力的主要指标(陈永亮等,2002;彭新华等,2004;李胜蓝等,2014)。微生物在调控土壤生态系统中发挥着关键作用(Schnitzer et al., 2011)。杉木人工林取代阔叶林后,土壤中细菌数量和多样性水平降低,且随连栽代数增加呈持续降低趋势(夏志超等,2012;刘丽等,2013)。此外,土壤酸性环境及低营养条件最适合酸杆菌(Acidobacteria)生存,可作为土壤贫瘠的评价指标(Ward et al., 2009)。然而,杉木人工林酸杆菌群的群落结构研究未见报道。
酸杆菌最初由Kishimoto等(1993)分离获得,认定为新嗜酸菌属。随着新菌株的不断发现及深入研究,该类细菌被定义为一种新细菌门,即酸杆菌门(Ludwig et al., 1997; Barns et al., 1999)。酸杆菌存在于各种生境,其中土壤中的数量约占细菌总量20%~50%,对土壤生态系统的构建具重要作用,与富营养细菌变形杆菌(Proteobacteria)相当(Jones et al., 2009)。但目前对杉木人工林土壤中酸杆菌的群落结构与多样性模式研究较少。因此,研究杉木人工林连栽导致的土壤性质改变及其对酸杆菌群落结构的影响,对阐明杉木人工林长期生产力维持机制及发展土壤酸杆菌细菌生态学具有重要科学意义。
宏基因组测序是对特定环境样品中所有微生物基因组进行高通量测序的方法,是研究土壤微生物的前沿工具,其中以16S rDNA基因扩增的测序技术最为常用。王文波等(2016)利用宏基因组测序发现轮作可增加杨树(Populus)人工林土壤中氮素代谢相关细菌数量。Hu等(2016)研究表明在喀斯特地区种植人参果(Solanum muricatum)可以增加土壤细菌多样性及其丰度。常安然等(2017)对烟草(Nicotiana tabacum)土壤细菌16S rDNA进行高通量测序结果表明不同烟草根际土壤微生物多样性水平较高,共检测出25个菌门,其中变形菌门占明显优势,而土壤速效氮、含水量和pH对其根际土壤细菌影响较大。本研究基于16S rDNA高通量测序,研究杉木人工1代林、连栽2代林以及原造林迹地马尾松(Pinus massoniana)林的酸杆菌群落结构、土壤理化性质,分析各酸杆菌群落的相对丰度与土壤性质的相关性,旨在为构建健康杉木林土壤生态系统及林地可持续经营提供理论依据。
1 材料与方法 1.1 试验林概况试验林位于浙江省开化县林场,属中亚热带北缘季风气候,年均温16.4 ℃,7月均温27.5 ℃,1月均温4.7 ℃,年均降水量1 805 mm,年均无霜期257天(李婷婷, 2011)。土壤类型为红壤。本研究选择同一山坡的马尾松林改植杉木造林试验地进行土壤样品采集。于1987年砍伐马尾松林,营建杉木无性系试验林,于2000年将杉木砍伐,再次营建杉木无性系试验林,从而形成二代连栽林(CS,Second plantation with C. lanceolata);一代林(CF,First plantation with C. lanceolata,)为2000年砍伐马尾松后营建的杉木无性系林;以马尾松林(林龄约50年)为对照(CK),共3个处理,以3个处理为大区、无性系为裂区进行裂区试验设计,重复3次。砍伐方式为全伐,采伐后进行炼山。
1.2 土壤测试样品采集于2016年8月在CF、CS的3个无性系裂区(F24×那1-1,13号,B44-1)内各设置3个样地,同时在CK大区设置3个样地,每个样地按五点取样法“S”形路线取样,每块样地土壤按四分法均匀混为1份,并选取0~20 cm和20~40 cm 2个土壤深度采集样品,共计土样42份。利用GPS记录其经、纬度与海拔高度,采样地及林分生长概况如表 1。将样品放入装满冰袋的泡沫箱后带回实验室,一部分保存于-40 ℃冰箱用于提取土壤DNA,另一部分自然风干后用于土壤理化指标测定。
|
|
1) 土壤理化性质测定经自然干燥后研磨的土壤过2 mm和0.25 mm筛,用于土壤理化性质的测定。pH计测定土壤pH值,烘干法测定束缚水含量,碱解扩散法测定碱解氮,Olsen法测定有效磷,火焰光度法测定速效钾,重铬酸钾容量法测定有机碳(鲁如坤, 2000)。
2) 土壤细菌测定取过1 mm筛后的0.5 g土壤,采用E.Z.N.A.TMsoil DNA Kit试剂盒(Omega Biotek,USA)提取土壤DNA,Nanodrop检验DNA纯度和浓度,储存于-20 ℃冰箱。16S rDNA测序使用高可变区的PCR扩增产物建库,依照Illumina测序仪文库构建方法。16S rDNA测序以V3和V4为目标区域进行引物设计,引物序列为319F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)。使用Phusion酶扩增相应高可变区,PCR反应体系(25 μL)为:DNA模板50 ng、Pusion Hot start flex 2X Master Mix 12.5 μL、上下游引物各2.5 μL、ddH2O补足至25 μL。反应程序为:98 ℃预变性30 s;35个循环(98 ℃变性10 s,54 ℃复性30 s,72 ℃延伸45 s);最后72 ℃延伸10 min。PCR扩增产物通过2%琼脂糖凝胶电泳进行检测,并对目标片段进行回收。
1.4 数据处理与分析土壤理化性质的试验数据用平均值±标准偏差(X ±SD)表示,采用SPSS软件(Version:19.0)进行以土壤理化性质指标为变量,不同林地及不同土层为自变量的双因素方差分析,试样均值比较采用5%显著性水平的最小显著差异检验。并以主成分分析方法对土壤肥力进行评价,对土壤pH值、束缚水含量、碱解氮、有效磷、速效钾、有机碳6个因子进行主成分分析的取样适合性检定(KMO),计算得KMO值>0.7,说明适合主成分分析。
土壤细菌测定中扩增完成的PCR产物纯化后用AxyPrepTM Mag PCR Normalizer(AxyPrep,USA)做归一化处理,构建好的文库上样到cBot或簇生成系统,序列于RDP数据库、Greengenes数据库及NCBI 16SMicrobial数据库进行比对,针对Illumina MiSeq 2x 300 bp paired-end测序数据进行信息分析。
2 结果与分析 2.1 不同林地的土壤性质比较以土壤理化性质指标为变量,以不同类型林地为自变量进行方差分析。结果表明(表 2),不同类型林地间的pH值、束缚水含量、碱解氮、有效磷、速效钾及有机碳在无显著差异。为此,在后续分析中以3个林型均值进行分析。
|
|
不同林地、土层的土壤理化性质测定结果(表 3)表明,马尾松改植杉木纯林后,其土壤上层(0~20 cm,下同)的pH值显著降低,而下层(20~40 cm,下同)土壤中pH略有下降,但均未达到显著差异;CS上层土壤的碱解氮含量与有效磷含量分别显著低于CK与CF相应土层的含量,且CK的上层与下层土壤的速效钾含量均显著高于CF、CS相应土层的含量,CK上层土壤的有机碳含量也显著高于CF、CS相应土层的含量;仅CF林地上层束缚水含量显著高于相应土层CS与CK林地。从上下层差异看,3种林地上层的碱解氮、有效磷、有机碳和速效钾含量均显著高于下层土壤的含量。从不同林地看,杉木纯林的营建致使土壤pH降低,土壤速效养分降低,且随连栽代数增加呈下降趋势。
|
|
由主成分分析知,第1主成分(特征值3.117)与第2主成分(特征值1.095)的累计贡献率达70.201%(表 4),且第1主成分综合了束缚水含量、碱解氮、有效磷、速效钾与有机碳等相关因子信息,而第2主成分则仅为pH值信息(表 5)。
|
|
|
|
用F1、F2分别表示1、2个主成分的综合指标,并用X1,X2……X6依次表示为土壤pH值、束缚水含量、碱解氮、有效磷、速效钾、有机碳,对因子载荷换算得到特征向量及以下2个主成分方程:F1=-0.075X1+ 0.296X2 + 0.215X3 + 0.241X4 + 0.269X5 +0.257X6;F2=0.863 X1-0.207X2 + 0.190X3 + 0.071X4 + 0.128X5-0.229X6。
利用以上方程计算主成分得分后,以各主成分的方差贡献率为权数进行加权求和,不同林地、不同土层的土壤肥力综合得分、排名(表 6)可知:CF(0~20 cm)>CS(0~20 cm)>CF(20~40 cm)>CS(20~40 cm)。将3种林地不同土层得分分别累加,得到3个林地的综合得分为CK(92.34)>CF(80.72)>CS(71.19)。可见,马尾松林改植杉木纯林后,其林地土壤肥力显著下降,且杉木采伐后连栽杉木纯林,其林地土壤肥力下降更快,尤其在杉木根系集中分布的上层土(0~20 cm)更为明显。
|
|
可操作分类单元(OTU, operational taxonomy unit)是用于物种分类及物种相对丰度分析的基本元。采用CD-HIT将序列相似性大于97%的clean tags定为一个OTU,即每个OTU对应一个细菌种。在3种林地土壤中共检测出6 594条OTU,其中酸杆菌门共检测出2 269条OTU。分析表明,3种林地2个土层的细菌门水平上酸杆菌门均占有最大优势(32.68%~49.17%),是土壤细菌的重要成员。由3种林地2个土层的酸杆菌数所占细菌数的比例(图 1)可知,CS上层土壤的酸杆菌门相对丰度显著高于CK同层土壤,但3种林地土壤下层的酸杆菌相对丰度无显著差异,且同一林地上下层土壤的酸杆菌相对丰度也无显著差异。

|
图 1 不同林地类型土壤酸杆菌所占百分比 Fig. 1 Proportion of Acidobacteria groups in different plantations 不同字母表示差异显著(P < 0.05)。 The different letters mean the significant difference(P < 0.05). |
所有土壤样品中共检测出18个酸杆菌属,分别为:Gp1、Gp2、Gp3、Gp4、Gp5、Gp6、Gp7、Gp10、Gp12、Gp13、Gp15、Gp16、Gp17、Granulicella、Acidicapsa、Edaphobacter、Candidatus Koribacter和Candidatus Solibacter。不同的酸杆菌属在不同林地类型土壤中相对丰度不同,可聚为3个大类(图 2)。其中,Granulicella、Gp15、Gp5、Gp6、Gp13、Edaphobacter、Gp1、Gp3、Gp2和Gp12相对丰度在CF和CS林地土壤中高于CK,而Gp10、Candidatus Koribacter、Candidatus Solibacter、Gp16和Acidicapsa相对丰度在CF与CK林地土壤中高于CS;Gp4、Gp17和Gp7相对丰度在CK和CS下层土中较高。
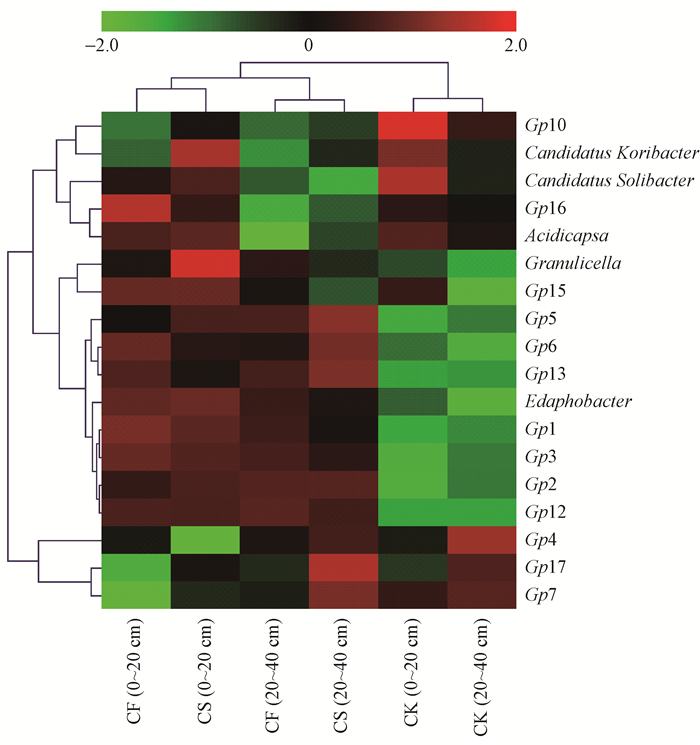
|
图 2 3种林分类型及2种土层土壤的酸杆菌属相对丰度聚类 Fig. 2 Relative abundance clustering map of different Acidobacteria genus in distinct forests and soil depths |
优势酸杆菌属(相对占比大于0.05%)相对丰度排名为:Gp2> Gp1> Gp3> Gp6>Gp5>Edaphobacter> Gp13>Granulicella> Gp10。其中Gp2在3种林地类型及2种土壤深度中均为绝对优势菌群(图 3),占酸杆菌群的47.74%~68.80%,Gp1占21.69%~29.72%,其次为Gp3占13.30%~22.41%。
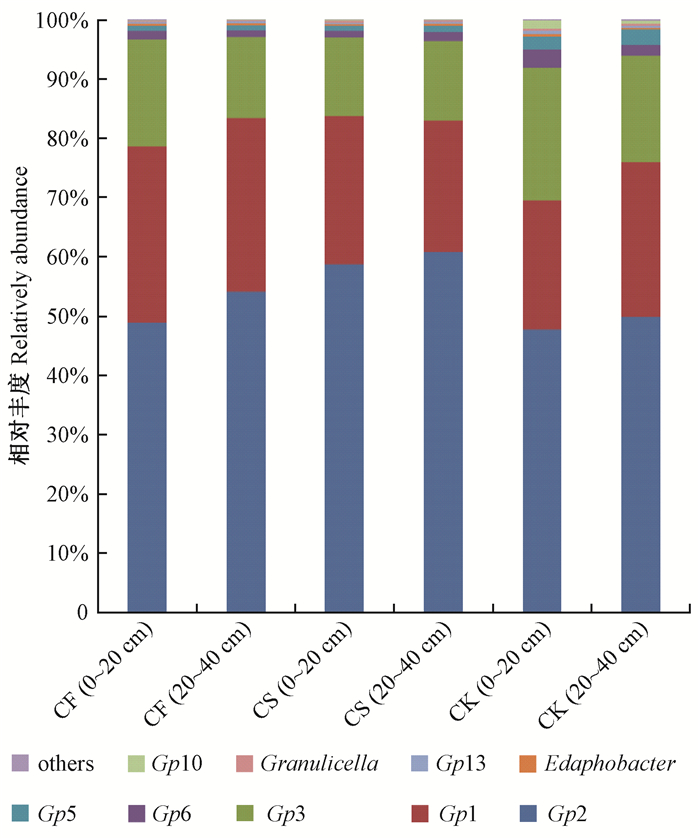
|
图 3 优势酸杆菌类群在3种林分类型及2种土层的百分比 Fig. 3 Relatively abundance of dominant Acidobacteria genera in distinct forest and soil depths “others”包括非优势酸杆菌属及未能归类的酸杆菌。 Non-dominant Acidobacteria genera and the sequences that could not be classified into any known Acidobacteria genera were labeled "others". |
不同酸杆菌类群对土壤理化因子的响应具有明显差异(表 7)。相关分析表明,Gp1与pH值呈显著负相关,Gp10与pH值呈显著正相关;Gp2和Gp3与有效磷分别呈显著负相关与显著正相关,而Edaphobacter与含水量、碱解氮均呈显著负相关。
|
|
通过16S rDNA基因扩增的测序技术在所获得序列中共检测出18个酸杆菌属,通过系统发育进化分析可以看出共分为3个大类(图 4),其中Gp1、Candidatus Koribacter归为一类,Gp15、Edaphobacter、Granulicella、Acidicapsa归为一类,Gp2、Gp5、Gp13、Gp12、Gp3、Candidatus Solibacter、Gp7、Gp10、Gp16、Gp4、Gp17、Gp6归为一类。

|
图 4 酸杆菌属系统发育进化树 Fig. 4 Phylogenetic analysis of Acidobacteria |
3种林地土壤理化因子均具有表聚性特征,主要因土壤表层受枯落物的影响,其分解后释放营养元素,其中碱解氮及磷元素多以难溶或固定形式存在,导致土壤养分难以沉降(程瑞梅等, 2010)。在马尾松林改植杉木人工纯林后土壤碱解氮、速效钾、有机碳含量显著降低,该结果与何斌等(2002)的研究结果相似。连栽杉木纯林导致林地土壤肥力持续下降,这与杉木自身生物学特性相关,其枯枝落叶不易分解,循环滞缓,导致人工纯林土壤趋于贫瘠化(杨玉盛等,2004)。在本研究3种林地土壤细菌中酸杆菌所占比例均最高,且杉木连栽林0~20 cm土层的酸杆菌占比显著高于对照马尾松林0~20 cm土壤,这可能与杉木根系分泌酚酸类物质有利于酸杆菌一些菌群定植与繁殖有关。此外,马尾松林土壤养分优于杉木纯林及杉木连栽纯林,而Gp1、Gp2等菌群的丰度在对照马尾松林土壤中较低,因此杉木纯林栽培印证了酸杆菌在贫瘠土壤中丰富度和多样性更高,可作为判断土壤环境瘠薄的指标这一结论(Ward et al., 2009)。酸杆菌虽然在土壤环境中广泛存在,但由于其难分离培养的特点,酸杆菌的生理特性及生态功能还知之甚少,对有限分离到的菌株研究结果表明,酸杆菌对植物残体具有降解功能,Pankratov等(2011)研究发现酸杆菌降解功能虽不如其他已知纤维素降解菌,但其抗逆性强,能够在寒冷北方酸性湿地中生存,对该条件下的纤维素降解起到重要作用。此外,酸杆菌Gp1的多个分离株可能参与了铁循环(Lu et al., 2010)及单碳化合物的代谢过程(Radajewski et al., 2002)。这些研究暗示在生境恶劣的情况下,寡营养细菌酸杆菌群落将替代部分富营养细菌维持土壤细菌基本生态功能。
有研究表明土壤酸杆菌多样性与植物群落类型有关(Liles et al., 2003; Navarrete et al., 2013),在草原土壤中Gp6与Gp4为优势菌群,在农田土壤中Gp4、Gp6和Gp7含量较高,在森林土壤中Gp1为优势菌群。不同自然环境下土壤酸杆菌门最主要的类群均是Gp1、Gp4与Gp6(Naether et al., 2012),本研究发现Gp2、Gp1、Gp3在3类林地条件下均为优势菌群,该结果与对神农架森林土壤酸杆菌的研究结果(Zhang et al., 2014)相一致。本研究发现Gp1和Gp10相对丰度与土壤pH分别呈显著负相关(r=-0.326, P=0.035)和显著正相关(r=0.326, P=0.035),而其他优势酸杆菌属与pH值无显著相关性。该结果与东北黑土区研究结果Gp1相对丰度与土壤pH呈显著负相关(Liu et al., 2015)相一致,同样,对北美与南美的土壤样品的分析表明Gp1与Gp10相对丰度与土壤pH分别呈显著负相关与显著正相关(Jones,2009)。且该结果同样符合Barns(1999)与Jones等(2009)的观点,即pH值对不同酸杆菌类群的影响不完全相同,即不是所有酸杆菌类群都嗜酸。
酸杆菌门具有系统发育多样性及普遍性(Quaiser et al., 2003),Barns等(2007)通过16S rRNA基因序列的系统发育分析定义了26个主要酸杆菌类群,但针对更细层次分类单元还缺乏系统研究。本文通过16S rRNA基因扩增的测序技术将酸杆菌属分为3个大类(图 4),这一结果与土壤酸杆菌属在不同林分类型不同土层深度中相对丰度聚类(图 2)不完全一致。其中Gp2、Gp5、Gp13、Gp12、Gp3、Gp6在2次聚类中都聚为一类,同样地Gp7、Gp4、Gp17,Gp15、Edaphobacter、Granulicella以及Candidatus Solibacter、Gp10、Gp16在2次聚类中都分别聚为一类,该结果可能预示以上序列相似的酸杆菌属其生态功能也相近,而不同的酸杆菌类群的具体生理特性、生态功能仍需进一步研究。后续研究应针对优势菌群系统地构建酸杆菌门的分离培养体系,研究其生理特性及生态功能多样性,进而探讨土壤酸杆菌优势菌群、功能菌群与杉木人工纯林生产力的关系,这对推动微生物学发展、促进杉木林地力恢复均具有重要意义。
4 结论宏基因组高通量测序结果包含大部分弱势细菌种群,能够更全面反映细菌多样性的真实水平,目前,对不同林地土壤微生物进行宏基因组测序仍少见报道。本文分析了浙江开化林场杉木人工纯林土壤肥力及酸杆菌群落结构,并以相同立地条件的马尾松林土壤作为对照,结果表明杉木人工纯林的连续栽植致使土壤肥力水平降低,而土壤寡营养细菌酸杆菌占比显著升高。
常安然, 李佳, 张耸, 等. 2017. 基于宏基因组学16S rDNA测序对烟草根际土壤细菌群落组成分析. 中国农业科技导报, 19(2): 43-50. (Chang A R, Li J, Zhang S, et al. 2017. Analysis of bacterial community structure in rhizosphere soil of tobacco based on the metagenomics 16S rDNA sequencing technology. Journal of Agricultural Science and Technology, 19(2): 43-50. [in Chinese]) |
陈龙池, 廖利平, 汪思龙, 等. 2002. 香草醛和对羟基苯甲酸对杉木幼苗生理特性的影响. 应用生态学报, 13(10): 1291-1294. (Chen L C, Liao L P, Wang S L, et al. 2002. Effect of vanillin and P-hydroxybenzoic acid on physiological characteristics of Chinese fir seedlings. Chinese Journal of Applied Ecology, 13(10): 1291-1294. DOI:10.3321/j.issn:1001-9332.2002.10.020 [in Chinese]) |
陈永亮, 韩士杰, 周玉梅, 等. 2002. 胡桃楸、落叶松纯林及其混交林根际土壤有效磷特性的研究. 应用生态学报, 13(7): 790-794. (Chen Y L, Han S J, Zhou Y M, et al. 2002. Characteristics of available P in the rhizosphere soil pure Juglans mandshurica and Larix gmelinii and their mixed plantation. Chinese Journal of Applied Ecology, 13(7): 790-794. DOI:10.3321/j.issn:1001-9332.2002.07.006 [in Chinese]) |
程瑞梅, 肖文发, 王晓荣, 等. 2010. 三峡库区植被不同演替阶段的土壤养分特征. 林业科学, 46(9): 1-6. (Cheng R M, Xiao W F, Wang X R, et al. 2010. Soil nutrient characteristics in different vegetation successional stages of three gorges reservoir area. Scientia Silvae Sinicae, 46(9): 1-6. [in Chinese]) |
何斌, 梁伟克, 陈文军, 等. 2002. 湿地松、杉木林取代马尾松林后土壤肥力的差异. 东北林业大学学报, 30(6): 11-13. (He B, Liang W K, Chen W J, et al. 2002. Difference of soil fertilities following replacement of Pinus massoniana plantation by Pinus elliottii plantation and Cunninghamia lanceolata plantation. Journal of Northeast Forestry University, 30(6): 11-13. DOI:10.3969/j.issn.1000-5382.2002.06.003 [in Chinese]) |
何友军, 王清奎, 汪思龙, 等. 2006. 杉木人工林土壤微生物生物量碳氮特征及其与土壤养分的关系. 应用生态学报, 17(12): 2292-2296. (He Y J, Wang Q K, Wang S L, et al. 2006. Characteristics of soil microbial biomass carbon and nitrogen and their relationships with soil nutrients in Cunninghamia lanceolata plantations. Chinese Journal of Applied Ecology, 17(12): 2292-2296. DOI:10.3321/j.issn:1001-9332.2006.12.012 [in Chinese]) |
李胜蓝, 方晰, 项文化, 等. 2014. 湘中丘陵区4种森林类型土壤微生物生物量碳氮含量. 林业科学, 50(5): 8-16. (Li S L, Fang X, Xiang W H, et al. 2014. Soil microbial biomass carbon and nitrogen concentrations in four subtropical forests in hilly region of central Hunan province, China. Scientia Silvae Sinicae, 50(5): 8-16. [in Chinese]) |
李婷婷.2011.浙江开化县林场杉木理论出材率表编制方法研究.北京: 北京林业大学硕士学位论文. (Li T T.2011.Construction of theoretical merchantable volume tables of Cunninghamia lanceolata in Kaihua forest farm.Beijing: MS thesis of Beijing Forestry University.[in Chinese]) http://cdmd.cnki.com.cn/Article/CDMD-10022-1011134650.htm |
刘丽, 徐明恺, 汪思龙, 等. 2013. 杉木人工林土壤质量演变过程中土壤微生物群落结构变化. 生态学报, 33(15): 4692-4706. (Liu L, Xu M K, Wang S L, et al. 2013. Effect of different Cunninghamia lanceolata plantation soil qualities on soil microbial community structure. Acta Ecologica Sinica, 33(15): 4692-4706. [in Chinese]) |
鲁如坤. 2000. 土壤农业化学分析方法. 北京: 中国农业科技出版社. (Lu R K. 2000. Soil argrochemistry analysis protocoes. Beijing: China Agriculture Science Press. [in Chinese]) |
彭新华, 张斌, 赵其国. 2004. 土壤有机碳库与土壤结构稳定性关系的研究进展. 土壤学报, 41(4): 618-623. (Peng X H, Zhang B, Zhao Q G. 2004. A review on relationship between soil organic carbon pools and soil structure stability. Acta Pedologica Sinica, 41(4): 618-623. DOI:10.3321/j.issn:0564-3929.2004.04.019 [in Chinese]) |
王士亚.2016.连栽障碍地杉木无性系土壤酶活性及微生物功能多样性分析.福州: 福建农林大学硕士学士论文. (Wang S Y.2016.Analysis of the soil enzyme activity and microbial functional diversity of Cunninghamia lanceolata clones in continuous planting obstacles soil of Cunninghamia lanceolata.Fuzhou: MS thesis of Fujian Agriculture & Forestry University.[in Chinese]) http://cdmd.cnki.com.cn/Article/CDMD-10389-1016129231.htm |
王文波, 王延平, 王华田, 等. 2016. 杨树人工林连作与轮作对土壤氮素细菌类群和氮素代谢的影响. 林业科学, 52(5): 45-54. (Wang W B, Wang Y P, Wang H T, et al. 2016. Effects of different continuous cropping and rotation of poplar plantation on soil nitrogen bacteria community and nitrogen metabolism. Scientia Silvae Sinicae, 52(5): 45-54. [in Chinese]) |
夏志超, 孔垂华, 王朋, 等. 2012. 杉木人工林土壤微生物群落结构特征. 应用生态学报, 23(8): 2135-2140. (Xia Z C, Kong C H, Wang P, et al. 2012. Characteristics of soil microbial community structure in Cunninghamia lanceolata plantation. Chinese Journal of Applied Ecology, 23(8): 2135-2140. [in Chinese]) |
杨玉盛, 陈银秀, 何宗明, 等. 2004. 福建柏和杉木人工林凋落物性质的比较. 林业科学, 40(1): 2-10. (Yang Y S, Chen Y X, He Z M, et al. 2004. Comparatively study on litter properties between plantations of Fokienia hodginsii and Cunninghamia lanceolata.. Scientia Silvae Sinicae, 40(1): 2-10. DOI:10.3321/j.issn:1001-7488.2004.01.001 [in Chinese]) |
张志才. 2012. 第1代与第2代9年生杉木林分生产力及土壤肥力比较. 福建林业科技, 39(3): 83-87. (Zhang Z C. 2012. Comparison of productivity and soil properties between the first and second generations of 9-year-old Cunninghamia lanceolata plantations. Journal of Fujian Forestry Science and Technology, 39(3): 83-87. DOI:10.3969/j.issn.1002-7351.2012.03.19 [in Chinese]) |
Barns S M. 1999. Wide distribution and diversity of members of the bacterial kingdom Acidobacterium in the environment. Applied & Environmental Microbiology, 65(4): 1731-1737. |
Barns S M, Cain E C, Sommerville L, et al. 2007. Acidobacteria Phylum sequences in uranium-contaminated subsurface sediments greatly expand the known diversity within the phylum. Applied & Environmental Microbiology, 73(9): 3113-6. |
Hu J, Yang H, Long X, et al. 2016. Pepino (Solanum muricatum) planting increased diversity and abundance of bacterial communities in karst area. Scientific Reports, 6: 21938. DOI:10.1038/srep21938 |
Jones R T, Robeson M S, Lauber C L, et al. 2009. A comprehensive survey of soil Acidobacterial diversity using pyrosequencing and clone library analyses. Isme Journal, 3(4): 442. DOI:10.1038/ismej.2008.127 |
Kielak A M, van Veen J A, Kowalchuk G A. 2010. Comparative analysis of Acidobacterial genomic fragments from terrestrial and aquatic metagenomic libraries, with emphasis on Acidobacteria subdivision 6. Applied & Environmental Microbiology, 76(20): 6769-6777. |
Kielak Veen A, Pijl A S, van Veen J A, et al. 2009. Phylogenetic diversity of Acidobacteria in a former agricultural soil. Isme Journal, 3(3): 378-382. DOI:10.1038/ismej.2008.113 |
Kishimoto N, Kosako Y, Tano T. 1993. Acidiphilium aminolytica sp. nov.:An acidophilic chemoorganotrophic bacterium isolated from acidic mineral environment.Current Microbiology, 27(3): 131-136. |
Li H, Wang X, Liang C, et al. 2015. Aboveground-belowground biodiversity linkages differ in early and late successional temperate forests. Scientific Reports, 5(X): 12234. |
Liles M R, Manske B F, Bintrim S B, et al. 2003. A census of rRNA genes and linked genomic sequences within a soil metagenomic library. Appl Environ Microbiol, 69(5): 2684-2691. DOI:10.1128/AEM.69.5.2684-2691.2003 |
Liu J J, Sui Y Y, Yu Z H, et al. 2015. Soil carbon content drives the biogeographical distribution of fungal communities in the black soil zone of northeast China. Soil Biology & Biochemistry, 83(1): 29-39. |
Ludwig W, Bauer S H, Bauer M, et al. 1997. Detection and in situ identification of representatives of a widely distributed new bacterial phylum. Fems Microbiology Letters, 153(1): 181-190. DOI:10.1111/j.1574-6968.1997.tb10480.x |
Lu S P, Gischkat S, Reiche M, et al. 2010. Ecophysiolog y of Fe-cycling bacteria in acidic sediments. Applied and Environmental Microbiology, 76: 8174-8183. DOI:10.1128/AEM.01931-10 |
Naether A, Foesel B U, Naegele V, et al. 2012. Environmental factors affect Acidobacterial communities below the subgroup level in grassland and forest soils. Applied & Environmental Microbiology, 78(20): 7398-406. |
Navarrete A A, Kuramae E E, de Hollander M, et al. 2013. Acidobacterial community responses to agricultural management of soybean in Amazon forest soils. Fems Microbiology Ecology, 83(3): 607-621. DOI:10.1111/fem.2013.83.issue-3 |
Pankratov T A, Ivanova A O, Dedysh S N, et al. 2011. Bacterial populations and environmental factors controlling cellulose degradation in an acidic Sphagnum peat. Environmental Microbiology, 13: 1800-1814. DOI:10.1111/j.1462-2920.2011.02491.x |
Quaiser A, Ochsenreiter T, Lanz C, et al. 2003. Acidobacteria form a coherent but highly diverse group within the bacterial domain:evidence from environmental genomics. Molecular Microbiology, 50(2): 563-575. DOI:10.1046/j.1365-2958.2003.03707.x |
Radajewski S, Webster G, Reay D S, et al. 2002. Identification of active methylotroph populations in an acidic forest soil by stable-isotope probing. Microbiology, 148: 2331-2342. DOI:10.1099/00221287-148-8-2331 |
Schnitzer S A, Klironomos J N, Hillerislambers J, et al. 2011. Soil microbes drive the classic plant diversity-productivity pattern. Ecology, 92(2): 296-303. DOI:10.1890/10-0773.1 |
Ward N L, Challacombe J F, Janssen P H, et al. 2009. Three genomes from the phylum Acidobacteria provide insight into the lifestyles of these microorganisms in soils. Applied & Environmental Microbiology, 75(7): 2046. |
Zhang Y G, Cong J, Lu H, et al. 2014. Community structure and elevational diversity patterns of soil Acidobacteria. Journal of Environmental Sciences (China), 26(8): 1717-1724. DOI:10.1016/j.jes.2014.06.012 |
 2019, Vol. 55
2019, Vol. 55

