文章信息
- 王海波, 龚明, 郭俊云, 辛胡, 唐利洲, 刘潮, 高永, 代冬琴
- Wang Haibo, Gong Ming, Guo Junyun, Xin Hu, Tang Lizhou, Liu Chao, Gao Yong, Dai Dongqin
- 麻疯树AP2/ERF基因家族孤儿基因Soloist的克隆与原核表达分析
- Molecular Cloning and Prokaryotic Expression of Orphan Gene Soloist of AP2/ERF Gene Family in Jatropha curcas
- 林业科学, 2018, 54(9): 60-69.
- Scientia Silvae Sinicae, 2018, 54(9): 60-69.
- DOI: 10.11707/j.1001-7488.20180908
-
文章历史
- 收稿日期:2018-01-04
- 修回日期:2018-02-24
-
作者相关文章
2. 云南师范大学生命科学学院 昆明 650500;
3. 曲靖师范学院生物资源与 食品工程学院 曲靖 655011;
4. 西南林业大学林学院 昆明 650224
2. School of Life Sciences, Yunnan Normal University Kunming 650500;
3. College of Biological Resource and Food Engineering, Qujing Normal University Qujing 655011;
4. College of Forestry, Southwest Forestry University Kunming 650224
转录因子作为一类调控反式作用因子,可以结合靶基因启动子区域特定顺式作用元件,从而调节靶基因以特定的强度与时空进行转录表达。与植物抗逆性密切相关的转录因子主要有AP2/ERF、bZIP、WRKY、NAC、MYB等。AP2/ERF转录因子家族在参与植物生长发育及抗逆性等过程中发挥重要作用。Sakuma等(2002)依据AP2结构域内氨基酸序列的同源性及AP2结构域的数目,将AP2/ERF超家族分为AP2(APETALA2)、ERF(ethylene responsive factor)、CBF/DREB(C-repeat binding factor/dehydration-responsive element binding factor)或DREB、RAV(related to ABI3/VP1)及Soloist 5个亚家族(Sakuma et al., 2002)。其中,AP2亚家族包含2个AP2结构域,且2个结构域之间通过保守的25个氨基酸残基连接(Riechmann et al., 1998; Licausi et al., 2010);ERF亚家族包含1个AP2结构域(第2个β-折叠包含保守Val14、Glu19)(Sakuma et al., 2002)与1个WLG基序,识别GCC盒(AGCCGCC)(Hao et al., 1998);DREB亚家族也包含1个AP2结构域(第2个β-折叠包含保守Ala14、Asp19)(Sakuma et al., 2002),结合CRT/DRE元件(A/GCCGAC)(Sakuma et al., 2002; Hong et al., 2009);RAV亚家族包含1个AP2结构域与1个B3结构域(由2段α-螺旋与7段β-折叠组成)(Sakuma et al., 2002; Hu et al., 2011);Soloist亚家族也包含1个AP2结构域。AP2/ERF家族基因在植物界成员较多,参与植物大部分的生理过程。其中,AP2亚家族主要调控植物花器官的形成与发育过程(Byzova et al., 1999; Li et al., 2014a);ERF亚家族主要响应病原、昆虫入侵等生物胁迫及依赖/不依赖植物激素如乙烯的非生物胁迫(Lai et al., 2014);DREB亚家族主要功能是响应低温、干旱、高温及盐害等非生物胁迫(Agarwal et al., 2006; Khan, 2011);RAV亚家族主要负责调控植物的开花过程、芽原体形成及叶片衰老等(Matías-Hernández et al., 2014);作为AP2/ERF家族的孤儿基因,大部分植物只有1个Soloist基因,且其功能研究还较少,如拟南芥(Arabidopsis thaliana)的Soloist基因At4g13040正调控水杨酸的生物合成从而参与抗病过程(Giri et al., 2014)。
麻疯树(Jatropha curcas)是目前世界上最主要的生物柴油植物,也是我国可再生能源发展纲要中规划的重点树种(Makkar et al., 2009)。麻疯树隶属大戟科(Euphorbiaceae)麻疯树属(Jatropha),为多年生落叶小型乔木。广布于世界热带亚热带地区,在我国滇、黔、川、桂、闽、粤及琼等地有较多野生资源分布(林娟等, 2004)。喜光热气候、易于繁殖、不择土壤,种子含油量高,亦可作为干热河谷地带的绿化树种(Yang et al., 2012),兼具良好的经济效益与生态效应,具有极为广阔的开发前景。麻疯树起源于热带及亚热带地区,温度是影响其地理分布的主要因素。随着麻疯树栽培面积的扩大和向高海拔山区的拓展,低温冷害将成为限制麻疯树产业发展的主要环境因子。与模式植物拟南芥及主要农作物如玉米(Zea mays)、水稻(Oryza sativa)等相比,麻疯树抗冷性的研究还非常有限,深入研究麻疯树抗冷性的生理生化及分子机制,有助于通过分子育种手段来培育抗冷新品种。AP2/ERF家族成员是参与植物多种抗逆性的重要转录因子。Jofuku等(1994)首先分离到了拟南芥的APETALA2(AP2)基因。之后大量植物的AP2/ERF家族基因也被克隆并进行了功能研究,目前,已经对大豆(Glycine max)(魏海超等, 2015)、雷蒙德氏棉(Gossypium raimondii)(卢合均, 2014)、麻疯树(唐跃辉, 2013)、毛果杨(Populus trichocarpa)(赵金玲等, 2015)、桑(Morus alba)(祝娟娟, 2013)及毛竹(Phyllostachys edulis)(吴惠俐, 2015)等植物在全基因组范围内进行了AP2/ERF基因家族的鉴定,而对于麻疯树Soloist孤儿基因的克隆、分子进化、表达模式分析,以及与麻疯树抗冷性的关系还未见报道。2011年,麻疯树基因组由日本Kazusa DNA研究所首次测序完成(Sato et al., 2011),2014年,中国科学院对麻疯树基因组进行了高通量重测序,为从基因组水平全面鉴定与分析麻疯树AP2/ERF基因家族,并更好地应用于麻疯树遗传改良奠定了基础。另外,前期研究通过麻疯树低温转录组与数字基因表达谱(DGE)测序(Wang et al., 2013)也表明AP2/ERF基因家族的Soloist基因参与麻疯树低温胁迫响应过程。本研究基于麻疯树基因组数据,利用生物信息学方法鉴定到1个麻疯树Soloist基因,并对其进行了基因克隆、低温表达及原核表达分析。随着更多植物基因组的公布以及Soloist基因的克隆,对Soloist转录因子进行系统研究,将有助于了解Soloist亚家族基因的分类、功能、进化历程及逆境胁迫调控作用,也为今后Soloist基因的应用奠定理论基础。
1 材料与方法 1.1 试验材料及处理麻疯树种子采自云南省楚雄州元谋县干热河谷地区。选取饱满的麻疯树种子,用1.5%CuSO4消毒20 min,无菌水漂洗5次,于26 ℃的恒温培养箱中吸涨24 h。将吸涨的种子在无菌水中漂洗3次,置于垫有5层用无菌水湿润滤纸的白磁盘中,于相对湿度75%、26 ℃、16 h/8 h光周期的恒温培养箱中萌发5天。将发芽的种子播于消毒的培养土中并于同上条件的恒温培养箱中生长15天至第2片真叶展开,每天用无菌水浇灌培养土。将生长15天的麻疯树幼苗置于相对湿度75%、12 ℃、16 h/8 h光周期的低温培养箱中进行低温处理,分别取低温处理0.5、3、12、24、48 h与对照(CK,正常培养)的第2片真叶、茎及根,用铝箔纸包好,液氮速冻后保存于-80 ℃冰箱中用于RNA的提取。
1.2 试验方法 1.2.1 麻疯树AP2/ERF基因家族的鉴定及JcSoloist基因序列分析通过Pfam(http://pfam.xfam.org/)下载AP2/ERF结构域的隐马尔可夫模型(PF00847),同时,从GenBank下载麻疯树最新注释蛋白质数据库。利用Hmmer工具的Hmmsearch程序对数据库进行序列相似性比对(阈值E<1e-10,序列相似性>50%),获得初步筛选的麻疯树AP2/ERF蛋白序列。通过Excel程序去除重复序列,之后利用Pfam与GenBank CDD在线工具对AP2/ERF结构域做进一步鉴定,得到最终的非冗余麻疯树AP2/ERF家族蛋白序列。将鉴定到的麻疯树AP2/ERF蛋白序列利用ClustalX(Version2.0)进行序列相似性比对,然后用MEGA6.0软件通过邻接法(NJ)构建系统进化树,根据进化树聚类数据并结合拟南芥(At04g13040)与水稻(Os02g29550)Soloist蛋白序列的BlastP结果确定麻疯树JcSoloist蛋白序列,进而通过GenBank得到其基因、CDS及mRNA序列。利用ProtParam工具(http://web.expasy.org/protparam/)分析麻疯树JcSoloist蛋白序列的氨基酸长度、理论分子量(Mw)及等电点(pI)等基本参数。根据JcSoloist的CDS序列检索得到该基因起始密码子ATG上游1 500 bp的启动子序列,进而通过PlantCARE工具对其顺式作用元件进行鉴定。利用在线GSDS(Gene Structure Display Server,http://gsds.cbi.pku.edu.cn/)工具进行JcSoloist基因mRNA序列与基因序列比对以确定基因内含子与外显子的结构并绘制基因结构图。另外,利用MEME(http://meme-suite.org/tools/meme)在线分析工具对麻疯树AP2/ERF基因家族及JcSoloist蛋白序列进行motif分析。
1.2.2 麻疯树JcSoloist基因编码框全长cDNA的克隆采用TriZol试剂(Invitrogen公司)提取麻疯树对照与不同低温处理下的叶片、茎及根的总RNA,通过DNase I(TaKaRa公司)消化其中的残余基因组DNA,得到纯化的总RNA。分别取3 μg总RNA,利用PrimeScriptTM RT reagent Kit with gDNA Eraser(TaKaRa公司)合成第1链cDNA。以叶片cDNA为模板,使用高保真DNA聚合酶KOD FX Neo DNA Polymerase(TOYOBO公司)及JcSoloist_F引物(表 1)进行PCR扩增,扩增条件为:98 ℃预变性5 min;98 ℃变性10 s,60 ℃退火30 s,68 ℃延伸2 min,30个循环;68 ℃后延伸5 min。电泳检测PCR产物质量,利用OMEGA Gel Extraction Kit(Omega公司)回收目的基因条带。将目的基因片段构建至克隆载体pMD18-T,命名为pMD18-T-JcSoloist,热激法转化大肠杆菌(Escherichia coli)Trans1-T1感受态细胞(TransGene公司),涂LB抗性(Amp)平板,过夜生长,进行蓝白斑筛选。菌落PCR验证的阳性克隆,测序做进一步的鉴定。
|
|
以18S rRNA为内参,利用1.2.2中提取麻疯树各组织材料cDNA为模板,进行JcSoloist基因的实时荧光定量PCR(quantitative real-time PCR,qPCR)表达分析(Zhang et al., 2013),引物序列见表 1,20 μL反应体系,每个样品重复3次,仪器为LightCycler96(Roche公司)。扩增条件为:94 ℃预变性30 s;94 ℃变性10 s,53.5 ℃退火15 s,72 ℃延伸15 s,45个循环,之后增加溶解曲线程序(95 ℃ 10 s,65 ℃ 60 s,97 ℃ 1 s,连续检测信号)。采用2-ΔΔCt法进行基因相对定量表达分析。器官差异表达分析以叶片的表达量为参照,低温差异表达分析以对照(CK)的表达量为参照。
1.2.4 原核表达载体的构建与目的蛋白的诱导表达根据pMD18-T-JcSoloist的测序结果,设计带酶切位点的引物JcSoloist_E(表 1)扩增麻疯树JcSoloist基因的全长编码框序列,扩增产物与原核表达载体pET-32a经EcoRⅠ与XhoⅠ双酶切,切胶回收后利用T4 DNA连接酶16 ℃连接12 h,获得重组质粒,将其转化大肠杆菌Trans1-T1感受态细胞,涂布LB抗性(含50 mg·L-1的Amp)平板,37 ℃过夜培养。挑选阳性克隆经EcoR Ⅰ与XhoⅠ双酶切鉴定,送深圳华大基因公司进行测序,经酶切与测序验证正确的重组载体命名为pET-32a-JcSoloist。
将构建的麻疯树原核表达载体pET-32a-JcSoloist通过热激法转化大肠杆菌BL21(DE3)菌株,同时转化pET-32a空载体作为对照,挑选阳性单克隆接种至5 mL LB液体培养基中(含50 mg·L-1的Amp),37 ℃ 220 r·min-1过夜振荡培养。之后按体积比1:100接种于100 mL LB液体培养基中(含50 mg·L-1的Amp),37 ℃ 220 r·min-1振荡培养至OD600为0.6,取1 mL菌液至1.5 mL EP管中,4 ℃保存。加入IPTG至终浓度为1 mmol·L-1,37 ℃诱导表达,分别取诱导1、3、6、12 h的菌液于4 ℃保存备用。将未诱导与诱导不同时间的菌液12 000 r·min-1离心5 min,弃上清,将菌体沉淀中加入200 μL PBS缓冲液(pH7.4)重悬浮,加入50 μL 5×SDS上样缓冲液,100 ℃煮沸10 min,冷却至室温,12 000 r·min-1离心5 min,取20 μL上清蛋白样品上样,进行SDS-PAGE电泳(5%浓缩胶,12%分离胶),考马斯亮蓝染色检测蛋白质的表达情况。
2 结果与分析 2.1 麻疯树AP2/ERF基因家族及JcSoloist基因的鉴定利用AP2/ERF结构域的隐马尔可夫模型检索麻疯树蛋白质数据库,并通过Pfam与CDD去冗余,共鉴定到119个麻疯树AP2/ERF家族成员,与唐跃辉(2013)的报道一致。同时结合拟南芥与水稻的Soloist蛋白序列的BlastP结果,筛选到1个麻疯树Soloist基因(JcAP2/ERF118,命名为JcSoloist)(图 1方框所示)。结构域分析显示麻疯树JcSoloist蛋白序列包含1个AP2结构域,位于135-190位之间(图 1)。

|
图 1 麻疯树AP2/ERF家族进化树构建及保守结构域分析 Figure 1 Phylogenetic relationship and conserved domain analysis of AP2/ERF proteins in Jatropha curcas 方框表示麻疯树JcSoloist基因,3号块表示AP2结构域,其他数字块表示AP2/ERF家族的保守基序。 JcSoloist gene of J. curcas is marked by pane. AP2 domain and conserved motifs for AP2/ERF family are presented by No.3 lump and other numerical lumps, respectively. |
以麻疯树叶片cDNA为模板,利用JcSoloist_F引物,扩增JcSoloist基因编码框全长序列。经过测序,克隆的麻疯树JcSoloist基因cDNA序列790 bp,ORFfinder工具分析显示包含完整的编码框699 bp,编码232个氨基酸,且在编码框的5′端、3′端分别鉴定到274 bp与270 bp的5′-UTR与3′-UTR非编码区。预测JcSoloist蛋白分子量为26.3 kDa,理论等电点为9.65。氨基酸组成分析显示,极性氨基酸如酸性、碱性氨基酸含量较高,分别占12.5%、20.7%,另外,丝氨酸(Ser)含量也较高,占11.6%,预示JcSoloist可能受磷酸化/去磷酸的共价修饰调节。
2.3 麻疯树JcSoloist基因结构分析鉴定到的119个麻疯树AP2/ERF家族基因,仅有30个包含内含子,63个ERF亚家族中有13个含有1个内含子,AP2与Soloist亚家族17个基因都包含4~9个内含子(唐跃辉, 2013)。通过GSDS工具对麻疯树、拟南芥及水稻Soloist的基因结构分析发现,麻疯树JcSoloist基因全长5 383 bp,较拟南芥(1 591 bp)与水稻(4 903 bp)都长,包含6个外显子与5个内含子,与水稻数量一致,而拟南芥Soloist基因包含7个外显子与6个内含子(图 2)。另外,3个物种Soloist基因的终止密码子也不同,单子叶植物水稻为TAA,而双子叶植物麻疯树与拟南芥为TGA,预示Soloist基因终止密码子使用可能存在物种与分类特异性。

|
图 2 麻疯树(Jc)与拟南芥(At)、水稻(Os)Soloist基因结构 Figure 2 Gene structure of Soloist from Jatropha curcas(Jc), Arabidopsis thaliana(At) and Oryza sativa(Os) |
利用麻疯树、拟南芥与水稻的基因组序列通过GenBank Blast检索分别获得Soloist基因起始密码子ATG上游1 500 bp的启动子序列,并利用PlantCARE工具对其进行顺式作用元件分析(图 3)。结果表明,麻疯树、拟南芥及水稻Soloist基因的调控元件存在物种特异性与分布位置特异性,都鉴定到了多个TATA-box(RNA聚合酶Ⅱ结合位点)、CAAT-box(调控转录起始频率)必须作用元件。另外,也都鉴定到了不同的激素与逆境响应元件,其中拟南芥Soloist基因响应元件分布较为分散,包括乙烯、赤霉素、生长素、水杨酸及高温响应元件,水稻Soloist基因响应元件分布较为集中,包括脱落酸、赤霉素、茉莉酸甲酯、干旱及高温响应元件,而麻疯树JcSoloist基因鉴定到的响应元件较拟南芥与水稻少,包括赤霉素、水杨酸及干旱元件,说明麻疯树JcSoloist基因可能更多参与激素信号系统的调节过程。
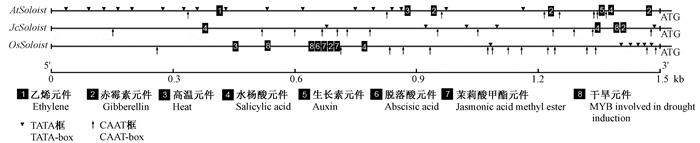
|
图 3 麻疯树与拟南芥、水稻Soloist基因启动子顺式作用元件 Figure 3 Cis-acting elements of Soloist promoter from Jatropha curcas with Arabidopsis thaliana and Oryza sativa |
将拟南芥、水稻与麻疯树的Soloist蛋白序列利用ClustalX进行多序列比对发现,N端保守性较高,C端保守性较低,而中间部位除AP2结构域外,其他序列保守性也较低。根据AP2结构域氨基酸序列及预测的二级结构,鉴定到麻疯树Soloist蛋白AP2结构域的典型3条反平行β-折叠与1条α-螺旋结构模型,其中,3条β-折叠氨基酸残基数目分别为4、8、8,而1条α-螺旋氨基酸残基数目为16(图 4),且该数目在不同物种中相对恒定(Sakuma et al., 2002)。

|
图 4 麻疯树JcSoloist基因推导氨基酸序列与拟南芥、水稻Soloist的多重序列比对及保守AP2结构域 Figure 4 Sequence alignment of Soloist from Jatropha curcas with Arabidopsis thaliana and Oryza sativa AP2结构域的3个β-折叠与1个α-螺旋用虚线表示。 3 β-sheets and one α-helix of AP2 domain are marked by dotted lines. |
通过PlantCARE工具在麻疯树JcSoloist基因启动子区域鉴定到水杨酸与赤霉素等激素响应元件,而包含较少的非生物胁迫调控元件。但DGE数据分析显示,麻疯树JcSoloist基因是低温锻炼上调表达较大的基因。为弄清JcSoloist基因与麻疯树抗冷性的关系,本研究利用qPCR技术对JcSoloist基因的器官表达与低温差异表达特性进行了分析。结果表明,麻疯树JcSoloist基因存在显著的器官表达特异性,在根中表达量较高(较叶片中高2.78倍),其次为茎,而在叶片中表达量较低(图 5)。另外,在叶片中JcSoloist基因受低温诱导表达更为显著,与对照相比,随着低温处理时间的延长,在低温锻炼3 h时达到最大表达量,较对照提高34.38倍,之后在24 h基本维持对照表达水平(图 6A);在表达量较高的根中,同样受低温诱导,且分别在低温锻炼0.5、24 h时出现2次表达峰值,表达量分别较对照提高2.57倍、3.83倍(图 6B)。虽然在麻疯树JcSoloist基因启动子区域未鉴定到低温响应元件,但JcSoloist属于低温高响应基因,与麻疯树的低温胁迫响应或低温信号转导直接相关,预示低温条件下JcSoloist基因的表达可能受到其他调控元件或转录因子的激活。

|
图 5 麻疯树JcSoloist基因在不同器官中的差异表达分析 Figure 5 Differential expression of JcSoloist gene in different organs of Jatropha curcas |
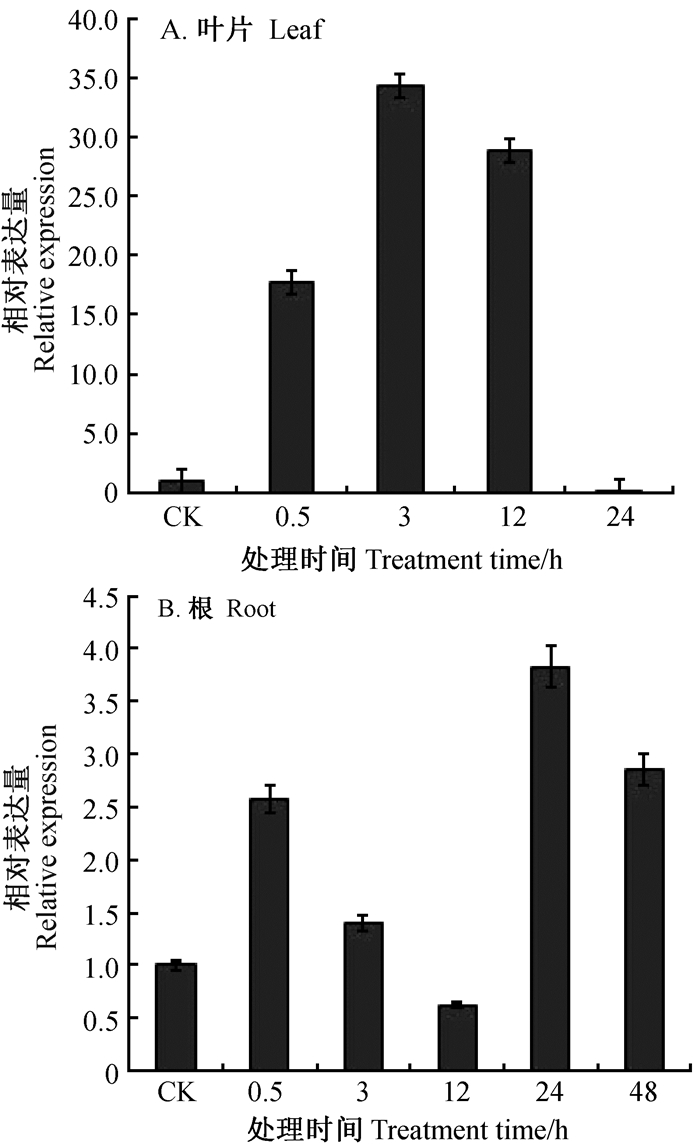
|
图 6 低温处理下麻疯树JcSoloist基因的表达模式分析 Figure 6 Expression profiles of Jatropha curcas JcSoloist gene under chilling treatment |
通过EcoRⅠ与XhoⅠ双酶切,将JcSoloist基因编码框连接至pET-32a表达载体的多克隆位点,构建了原核表达重组载体pET-32a-JcSoloist,经过酶切与测序验证正确,未出现突变。
将大肠杆菌BL21(DE3)菌株、转pET-32a空载体与pET-32a-JcSoloist重组载体的菌液裂解后进行SDS-PAGE表达蛋白的电泳分析(图 7)。pET-32a载体的表达标签为20.0 kDa,麻疯树JcSoloist蛋白预测分子量为26.3 kDa,在IPTG诱导的不同时间都出现1条约46.0 kDa的蛋白条带,与理论融合蛋白的分子量一致,表明JcSoloist基因已经在大肠杆菌中成功表达。另外,目的蛋白在诱导1 h后即开始高效表达,其表达量随着诱导时间的延长(3、6、12 h)也在快速提高。而在同一蛋白分子量位置,大肠杆菌BL21(DE3)菌株(CK-1)、转pET-32a空载体(CK-2)及转pET-32a-JcSoloist重组载体未诱导(0 h)的菌株都未出现相应分子量的条带。
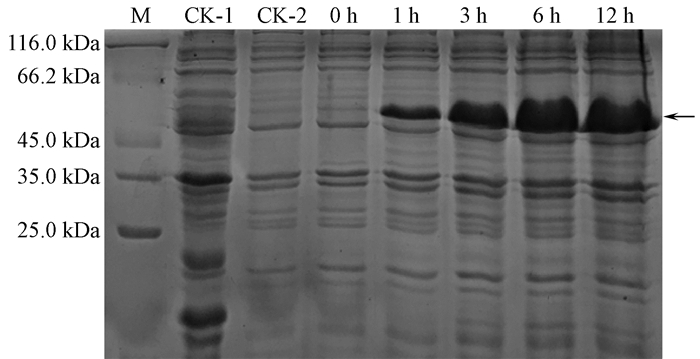
|
图 7 麻疯树JcSoloist基因表达蛋白的SDS-PAGE电泳 Figure 7 SDS-PAGE electrophoresis of prokaryotic expression protein of JcSoloist gene in Jatropha curcas CK-1表示大肠杆菌BL21(DE3)菌株;CK-2表示转空pET-32a表达载体;0 h表示转重组表达载体未诱导;1 h、3 h、6 h、12 h表示转重组表达载体并经过IPTG分别诱导1 h、3 h、6 h、12 h;M表示蛋白质Marker。 CK-1 represents the supematant from Escherichia coil BL21(DE3) strain; CK-2 represents the supematant from E. coil BL21(DE3) strain containing pET-32a; 0 h means the supematant from E. coil BL21(DE3) strain with recombinant vector but no IPTG induction; 1 h, 3 h, 6 h, and 12 h indicate the supematant from E. coil BL21(DE3) strain with recombinant vector and induced by IPTG for 1 h, 3 h, 6 h, and 12 h; M represents protein marker. |
研究发现,AP2/ERF基因家族主要分布于植物与低等原核生物中,而其他真核生物如动物基因组中分布较少,最初被认为是植物特有的转录因子,但最近在病毒Enterbacteria phage RB49(Magnani et al., 2004)、束毛藻(Trichodesmium erythraeum)、嗜热四膜虫(Tetrahymena thermophila)(Wuitschick et al., 2004)及线虫(Magnani et al., 2004)等中也发现了AP2基因。如束毛藻AP2蛋白能结合与植物AP2蛋白DNA结合序列相似的富含GC的顺式作用元件。基于此,Magnani等(2004)推测植物中的AP2/ERF家族基因可能起源于细菌或病毒基因的横向转移,如原始蓝细菌(Cyanobacteria)在进化成叶绿体的共生进化过程中,原始的AP2/ERF基因整合进入植物基因组,现在大多数植物的AP2/ERF基因没有内含子也证明AP2/ERF基因可能来自早期原核细胞,如拟南芥145个AP2/ERF基因只有23个含有内含子。本研究鉴定到的119个麻疯树AP2/ERF基因仅有30个含有内含子(13个有1个内含子,17个有4~9个内含子),而JcSoloist基因含有5个内含子,属麻疯树AP2/ERF家族基因中含内含子数量较多的基因之一,且内含子的总长度(4 684 bp)远大于外显子及对应编码框(699 bp)的长度,也与本研究在原核细胞中表达的JcSoloist蛋白大小(26.3 kDa)一致,推测这些包含了内含子的AP2/ERF家族基因可能是在原始不含内含子AP2/ERF基因基础上进化而来。另外,单子叶植物和双子叶植物的AP2/ERF基因并没有明显进化上的差异。基于系统发生以及基因结构分析,本研究中麻疯树与同属双子叶植物的拟南芥的Soloist基因长度差异明显,而与单子叶植物水稻的Soloist基因长度相近,但外显子分布却差异较大,说明在单子叶和双子叶植物分化之前,AP2/ERF基因的分化就已经出现(Gutterson et al., 2004)。
AP2/ERF基因家族的成员都包含AP2结构域。AP2结构域由50~70个氨基酸残基组成,从N端到C端AP2结构域包含YRG(约20个氨基酸残基组成3个反向β-折叠)与RAYD(约40个氨基酸残基,包含由约16~18个氨基酸残基组成的1个双亲α-螺旋)2个基序结构,其中YRG基序富含碱性氨基酸与极性亲水氨基酸,负责结合DNA区域,而RAYD基序主要负责调节YRG基序与DNA的结合强度。序列分析显示,麻疯树JcSoloist的AP2结构域中,YRG基序内部的3个β-折叠分别包含1(Lys)、2(Lys、Lys)、3(Lys、Lys、His)个碱性氨基酸,同时富含芳香族氨基酸如Tyr、Phe及Trp。另外,据报道,在AP2/ERF家族的DREB亚家族中,CBF转录因子YRG基序的第2个β-折叠在与基因启动子CCGAC序列结合中发挥重要作用,而其中的Glu是核心氨基酸残基(Okamuro et al., 1997; Allen et al., 1998; 王海波等, 2015);本研究中麻疯树JcSoloist在同样位置被Gln取代,推测此变化决定麻疯树JcSoloist转录因子可能结合不同的启动子序列。
AP2/ERF家族的CBF/DREB亚家族基因是响应植物非生物胁迫的主要基因(Agarwal et al., 2006),此类转录因子可以识别低温响应基因的特异顺式作用元件(CRT/DRE)的CCGAC序列,如准噶尔无叶豆(Eremosparton songoricum)EsDREB2B基因(Li et al., 2014b)、新疆野苹果(Malus sieversii)MsDREB2C基因(Zhao et al., 2013)、宽叶独行菜(Lepidium latifolium)LlCBF基因(Akhtar et al., 2013)及麻疯树JcCBF基因(王海波等, 2015)等都受到低温胁迫的诱导表达。而目前Soloist亚家族基因的功能研究还报导较少,Giri等(2014)鉴定到拟南芥的1个Soloist基因At4g13040,该基因能够促进拟南芥水杨酸的积累进而参与抗病过程;杜磊等(2013)报道了橡胶树(Hevea brasiliensis)中的Soloist类似基因HbSoloist,并通过半定量PCR分析显示,该基因被低温、乙烯及茉莉酸诱导表达,且表达量显著增加都发生在处理后期的24、48、72 h。而本研究中,麻疯树JcSoloist属于低温高响应基因,在低温处理的0.5~3 h即开始上调表达。以上结果表明Soloist作为AP2/ERF家族的孤儿基因,可能主要参与植物非生物胁迫及激素信号响应等过程。
4 结论本研究从麻疯树中克隆到与抗冷性密切相关的AP2/ERF家族的JcSoloist基因编码框cDNA序列,其包含6个外显子与5个内含子,编码框长度699 bp,编码232个氨基酸。该基因在麻疯树根中表达量较高,而在叶片中表达量相对较低,但在叶片与根中都属于低温诱导表达,与麻疯树的低温胁迫响应直接相关。通过构建原核表达载体pET-32a-JcSoloist,JcSoloist蛋白在大肠杆菌中进行了稳定而高效的表达。本研究结果为进一步揭示JcSoloist基因的生物学功能奠定了基础。
杜磊, 张德春, 邓治, 等. 2013. 巴西橡胶树一个AP2/EREBP转录因子的克隆及表达分析[J]. 植物生理学报, 49(11): 1238-1244. (Du L, Zhang D C, Deng Z, et al. 2013. Cloning and expression analysis of an AP2/EREBP gene in Hevea brasiliensis Muell[J]. Arg.. Plant Physiology Journal, 49(11): 1238-1244. [in Chinese]) |
林娟, 周选围, 唐克轩, 等. 2004. 麻疯树植物资源研究概况[J]. 热带亚热带植物学报, 12(3): 285-290. (Lin J, Zhou X W, Tang K X, et al. 2004. A survey of the studies on the resources of Jatropha curcas L[J]. . Journal of Tropical and Subtropical Botany, 12(3): 285-290. DOI:10.3969/j.issn.1005-3395.2004.03.018 [in Chinese]) |
卢合均. 2014.棉花AP2/EREBP基因家族的全基因组分析.北京: 中国农业科学院硕士学位论文. (Lu H J. 2014. Genome-wide analysis of AP2/ERF gene family in cotton. Beijing: MS thesis of Chinese Academy of Agricultural Sciences. [in Chinese]) http://cdmd.cnki.com.cn/Article/CDMD-82101-1014326389.htm |
唐跃辉. 2013.麻疯树AP2/ERF家族生物信息学和表达谱分析.北京: 中国科学院大学硕士学位论文. (Tang Y H. 2013. Bioinformatics and expression profile analysis of AP2/ERF transcription factor family in Jatropha curcas L.. Beijing: MS thesis of University of Chinese Academy of Sciences. [in Chinese]) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2368710 |
王海波, 王莎莎, 邹竹荣, 等. 2015. 小桐子转录因子CBF基因的克隆及酵母表达载体的构建[J]. 植物生理学报, 51(7): 1157-1166. (Wang H B, Wang S S, Zou Z R, et al. 2015. Molecular cloning and yeast expression vector construction of the transcription factor encoding CBF from Jatropha curcas[J]. Plant Physiology Journal, 51(7): 1157-1166. [in Chinese]) |
魏海超, 刘媛, 豆明珠, 等. 2015. 大豆AP2/ERF基因家族的分子进化分析[J]. 植物生理学报, 51(10): 1706-1718. (Wei H C, Liu Y, Dou M Z, et al. 2015. Molecular evolution of AP2/ERF gene family in Glycine max[J]. Plant Physiology Journal, 51(10): 1706-1718. [in Chinese]) |
吴惠俐. 2015.毛竹AP2/ERF基因家族分析及DREB类转录因子表达研究.北京: 中国林业科学研究院硕士学位论文. (Wu H L. 2015. Analysis of AP2/ERF gene family and expression of DREB transcription factors in Phyllostachys edulis. Beijing: MS thesis of Chinese Academy of Forestry. [in Chinese]) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2903932 |
赵金玲, 姚文静, 王升级, 等. 2015. 杨树AP2/ERF转录因子家族生物信息学分析[J]. 东北林业大学学报, 43(10): 21-29. (Zhao J L, Yao W J, Wang S J, et al. 2015. AP2/ERF gene family in Populus trichocarpa by bioinformatics[J]. Journal of Northeast Forestry University, 43(10): 21-29. DOI:10.3969/j.issn.1000-5382.2015.10.005 [in Chinese]) |
祝娟娟. 2013.桑树DREB基因家族生物信息学分析及功能研究.重庆: 西南大学硕士学位论文. (Zhu J J. 2013. Bioinformatics analysis and functional study of DREB genes from Morus spp.. Chongqing: MS thesis of Southwest University. [in Chinese]) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2310634 |
Agarwal P K, Agarwal P, Reddy M K, et al. 2006. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants[J]. Plant Cell Reports, 25(12): 1263-1274. DOI:10.1007/s00299-006-0204-8 |
Akhtar M, Jaiswal A, Jaiswal J P, et al. 2013. Cloning and characterization of cold, salt, and drought inducible C-repeat binding factor gene from a highly cold adapted ecotype of Lepidium latifolium L[J]. . Physiology and Molecular Biology of Plants, 19(2): 221-230. DOI:10.1007/s12298-012-0154-2 |
Allen M D, Yamasaki K, Ohme-Takagi M, et al. 1998. A novel mode of DNA recognition by a β-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA[J]. EMBO Journal, 17(18): 5484-5496. DOI:10.1093/emboj/17.18.5484 |
Byzova M V, Franken J, Aarts M G, et al. 1999. Arabidopsis STERILE APETALA, a multifunctional gene regulating inflorescence, flower, and ovule development[J]. Genes & Development, 13(8): 1002-1014. |
Giri M K, Swain S, Gautam J K, et al. 2014. The Arabidopsis thaliana At4g13040 gene, a unique member of the AP2/EREBP family, is a positive regulator for salicylic acid accumulation and basal defense against bacterial pathogens[J]. Journal of Plant Physiology, 171(10): 860-867. DOI:10.1016/j.jplph.2013.12.015 |
Gutterson N, Reuber T L. 2004. Regulation of disease resistance pathways by AP2/ERF transcription factors[J]. Current Opinion in Plant Biology, 7(4): 465-471. DOI:10.1016/j.pbi.2004.04.007 |
Hao D Y, Ohme-Takagi M, Sarai A. 1998. Unique mode of GCC box recognition by the DNA-binding domain of ethylene responsive element-binding factor (ERF domain) in plants[J]. Journal of Biological Chemistry, 273(41): 26857-26861. DOI:10.1074/jbc.273.41.26857 |
Hong B, Ma C, Yang Y J, et al. 2009. Over-expression of AtDREB1A in chrysanthemum enhances tolerance to heat stress[J]. Plant Molecular Biology, 70(3): 231-240. DOI:10.1007/s11103-009-9468-z |
Hu L, Liu S. 2011. Genome-wide identification and phylogenetic analysis of the ERF gene family in cucumbers[J]. Genetics & Molecular Biology, 34(4): 624-633. |
Jofuku K D, den Boer B G, Van M M, et al. 1994. Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J]. Plant Cell, 6(9): 1211-1225. DOI:10.1105/tpc.6.9.1211 |
Khan M S. 2011. The role of DREB transcription factors in abiotic stress tolerance of plants[J]. Biotechnology & Biotechnological Equipment, 25(3): 2433-2442. |
Lai Y, Dang F F, Lin J, et al. 2014. Overexpression of a pepper CaERF5 gene in tobacco plants enhances resistance to Ralstonia solanacearum infection[J]. Functional Plant Biology, 41(7): 758-767. DOI:10.1071/FP13305 |
Li D M, Lü F B, Zhu G F, et al. 2014a. Identification of warm day and cool night conditions induced flowering-related genes in a Phalaenopsis orchid hybrid by suppression subtractive hybridization[J]. Genetics & Molecular Research, 13(3): 7037-7051. |
Li X S, Zhang D Y, Li H Y, et al. 2014b. EsDREB2B, a novel truncated DREB2-type transcription factor in the desert legume Eremosparton songoricum, enhances tolerance to multiple abiotic stresses in yeast and transgenic tobacco[J]. BMC Plant Biology, 14(1): 44. DOI:10.1186/1471-2229-14-44 |
Licausi F, Giorgi F M, Zenoni S, et al. 2010. Genomic and transcriptomic analysis of the AP2/ERF superfamily in Vitis vinifera[J]. BMC Genomics, 11(1): 719. DOI:10.1186/1471-2164-11-719 |
Magnani E, Sjolander K, Hake S. 2004. From endonucleases to transcription factors: evolution of the AP2 DNA binding domain in plants[J]. Plant Cell, 16(9): 2265-2277. DOI:10.1105/tpc.104.023135 |
Makkar H P S, Becker K. 2009. Jatropha curcas, a promising crop for the generation of biodiesel and value-added coproducts[J]. European Journal of Lipid Science & Technology, 111(8): 773-787. |
Matías-Hernández L, Aguilar-Jaramillo A E, Marín-González E, et al. 2014. RAV genes: regulation of floral induction and beyond[J]. Annals of Botany, 114(7): 1459-1470. DOI:10.1093/aob/mcu069 |
Okamuro J K, Caster B, Villarroel R, et al. 1997. The AP2 domain of APETALA2 defines a large new family of DNA binding proteins in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 94(13): 7076-7081. DOI:10.1073/pnas.94.13.7076 |
Riechmann J L, Meyerowitz E M. 1998. The AP2/EREBP family of plant transcription factors[J]. Biological Chemistry, 379(6): 633-646. |
Sakuma Y, Liu Q, Dubouzet J G, et al. 2002. DNA binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration and cold-inducible gene expression[J]. Biochemical and Biophysical Research Communications, 290(3): 998-1009. DOI:10.1006/bbrc.2001.6299 |
Sato S, Hirakawa H, Isobe S, et al. 2011. Sequence analysis of the genome of an oil-bearing tree, Jatropha curcas L[J]. . DNA Research, 18(1): 65-76. DOI:10.1093/dnares/dsq030 |
Shafeinie A, Mohammadi V, Alizadeh H, et al. 2014. Overexpression of Arabidopsis dehydration-responsive element-binding protein 2A confers tolerance to salinity stress to transgenic canola[J]. Pakistan Journal of Biological Sciences, 17(5): 619-629. DOI:10.3923/pjbs.2014.619.629 |
Wang H B, Zou Z R, Wang S S, et al. 2013. Global analysis of transcriptome responses and gene expression profiles to cold stress of Jatropha curcas L[J]. . PLoS One, 8(12): e82817. DOI:10.1371/journal.pone.0082817 |
Wuitschick J D, Lindstrom P R, Meyer A E, et al. 2004. Homing endonucleases encoded by germline-limited genes in Tetrahymena thermophila have APETELA2 DNA binding domains[J]. Eukaryotic Cell, 3(3): 685-694. DOI:10.1128/EC.3.3.685-694.2004 |
Yang C Y, Fang Z, Li B, et al. 2012. Review and prospects of Jatropha biodiesel industry in China[J]. Renewable & Sustainable Energy Reviews, 16(4): 2178-2190. |
Zhang L, He L L, Fu Q T, et al. 2013. Selection of reliable reference genes for gene expression studies in the biofuel plant Jatropha curcas using real-time quantitative PCR[J]. International Journal of Molecular Sciences, 14(12): 24338-24354. DOI:10.3390/ijms141224338 |
Zhao K, Shen X J, Yuan H Z, et al. 2013. Isolation and characterization of dehydration-responsive element-binding factor 2C (MsDREB2C) from Malus ieversii Roem[J]. Plant and Cell Physiology, 54(9): 1415-1430. DOI:10.1093/pcp/pct087 |
 2018, Vol. 54
2018, Vol. 54

