文章信息
- 魏明科, 俞金健, 黄晓龙, 刘琼瑶, 黄华宏, 林二培, 童再康
- Wei Mingke, Yu Jinjian, Huang Xiaolong, Liu Qiongyao, Huang Huahong, Lin Erpei, Tong Zaikang
- 杉木NAC转录因子基因ClNAC1的克隆、表达及单核苷酸多态性分析
- Cloning, Expression and Single Nucleotide Polymorphisms Analysis of NAC Transcription Factor Gene ClNAC1 in Cunninghamia lanceolata
- 林业科学, 2018, 54(9): 49-59.
- Scientia Silvae Sinicae, 2018, 54(9): 49-59.
- DOI: 10.11707/j.1001-7488.20180907
-
文章历史
- 收稿日期:2017-11-20
- 修回日期:2018-04-09
-
作者相关文章
木材是自然界非常重要的可再生资源,具有不可替代的经济和生态价值。纤维素、木质素、半纤维素是木材(即次生木质部)的重要组成成分,这些成分的合成对木材品质有重要的影响。近年来,许多参与木材成分生物合成的相关酶基因和转录因子相继被分离和鉴定,如纤维素合成酶(cellulose synthase, CesA)(Wu et al., 2000)、4-香豆酰CoA连接酶(4-coumarate coenzyme A ligase, 4CL)(Kalluri et al., 2004)、苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL)(徐莉莉等, 2013)等编码基因,MYB(V-myb avian myeloblastosis viral oncogene homolog)(Groover et al., 2006)、HD-Zip Ⅲ(Class Ⅲ homeodomain leucine zipper)(Groover, 2005)、NAC(no apicalmeristem/Arabidopsis thaliana transcription activator factor/CUP-shaped cotyledon)(Demura et al., 2007)等转录因子。其中NAC是该领域备受关注的一类转录因子家族,其家族成员在N端具有高度保守的NAC结构域。近年的一些研究证实NAC家族的一些成员在植物次生壁形成中发挥重要的调控作用。拟南芥(Arabidopsis thaliana)中一组NAC转录因子NST1(NAC secondary wall thickening promoting factor 1)、NST2、NST3/SND1(secondary wall-associated NAC domain protein 1)、VND6(vascular-related NAC-domain 6)和VND7被证实是作为次生壁合成调控网络中的主开关因子,并协同下游转录因子MYB46、MYB83等的表达来调控纤维和花药药室内壁中次生壁的形成(Zhong et al., 2010)。毛果杨(Populus trichocarpa)PtrWND2B/6B(wood-associated NAC domain transcription factors 2B/6B)与SND1直系同源,是调控次生壁生物合成的主开关(Zhong et al., 2010);PtrNAC154是次生壁形成的正调控因子,在杂交杨717-1B4(Populus alba × P. tremula)中超表达该基因导致植株高生长下降,且茎的皮层比例相对提高(Grant et al., 2010)。可见,NAC家族的一些成员在木材的形成中具有重要的调控作用。
SNP(single nucleotide polymorphism,单核苷酸多态性)是指由单个核苷酸的变异引起的基因组水平上DNA序列多态性,具有数量多、分布广等特点,是目前广泛应用的第3代分子标记(谢媛等,2012)。近年的研究发现,与杨树目标性状相关的候选基因在不同基因型中存在丰富的SNP变异。毛白杨(Populus tomentosa)中干细胞决定基因PtoWus1和PtoWus2、蔗糖合成酶基因PtoSUS1在不同个体间存在许多变异SNP位点(杨晓慧等,2009;潘炜等,2011)。而且候选基因中的一些SNP位点与目标性状变异有重要的关联。毛白杨S-腺苷高半胱氨酸水解酶基因PtoSAHHA的3个SNP位点与木材品质性状(纤维长度和微纤丝角)显著关联,可作为优良木材品质性状早期选择的重要标记(巩琛锐等,2013)。因此,分析目标性状候选基因的SNP变异,可为深入解析该基因功能和研发分子辅助育种技术提供重要线索和依据。
杉木(Cunninghamia lanceolata)是我国重要的针叶用材树种之一,材质改良已成为现阶段杉木育种的重要内容,其木材形成分子机制的研究日益受到人们的重视。高燕(2011)构建了杉木木材形成特异表达基因文库,并从中分离克隆了2个扩展蛋白ClEXPA基因。Huang等(2012)建立了杉木转录组数据库,克隆了参与纤维素和木质素生物合成的ClCesA、ClCCoAOMT等18个基因,并分析了其组织表达特异性。徐莉莉等(2013)利用RACE技术分离得到3个ClPAL基因的全长cDNA,系统分析了这些基因在不同激素和应压木处理下的表达差异。然而,迄今为止,尚未有杉木NAC转录因子的相关研究报道。为此,本研究以选育出的优良杉木无性系为材料,克隆与木材次生壁形成相关的ClNAC1基因,并对其进行序列特征、组织表达特异性分析;分析该基因的SNP及其连锁不平衡(LD),以期为深入解析该基因功能和连锁不平衡作图奠定重要基础。
1 材料与方法 1.1 植物材料试验材料为栽植于浙江农林大学林木良种繁育基地的杉木速生无性系(2015-2),在2017年3月初至6月中旬间,采集不同的器官和组织。其中雌球花(female cone, FC)、雄球花(male cone, MC)、嫩芽(bud, BD)取自树冠上部;嫩叶(young leaf, YL)、成熟叶(mature leaf, ML)均取自树冠上部的当年生枝;在同一无性系的水培植株上取幼根(root, RT)。在生长健壮的当年生枝上分别在距枝顶端0~2 cm间和10~12 cm间截取2段,记为S1和S2,解剖观察发现S1木质部区域很少,可认为是未木质化茎,S2则为木质化茎。取去年生木质化茎,剥取皮层(bark, BK),另在木质部一侧刮取1~2 mm厚的未成熟木质部(xylem, XY)。用于SNP分析的无性系共40个,分别来源于广西的融水、那坡、三江,湖南会同、靖县,以及贵州锦屏等地,取无病斑、无虫咬痕迹的当年生嫩叶用于DNA提取。所有材料皆于液氮速冻后保存于-80 ℃冰箱。
1.2 基因组DNA的提取基因组DNA的提取采用CTAB法,具体参照谢一青等(2006)的报道。
1.3 总RNA的提取和cDNA第1链的合成总RNA的提取采用改良CTAB法(王钰等,2010)。cDNA合成采用TaKaRa公司的Prime ScriptTM RT reagent Kit反转录试剂盒进行合成。
1.4 ClNAC1序列的分离依据杉木根、茎、叶等混合样本转录组测序得到的数据(Accession number: SRA051493),设计特异引物ClNAC1-FF/FR,经PCR获得具完整ORF的cDNA序列,同时以基因组DNA为模板扩增对应DNA序列。将扩增得到目的片段连接到pGEM-T Easy Vector(Promega)上,转化大肠杆菌(Escherichia coli)DH5α,将筛选得到的阳性克隆送南京金斯瑞生物科技公司测序。
1.5 生物信息学分析利用NCBI数据库中的BLASTX进行序列比对,应用DNASTAR软件包确定ClNAC1的ORF,并推测编码氨基酸序列,以及蛋白质的分子量和等电点。使用DNAMAN软件对ClNAC1结构域氨基酸序列进行多序列比对。利用Spidey(http://www.ncbi.nlm.nih.gov/IEB/Research/Ostell/Spidey/)进行ClNAC1的基因结构分析。通过NCBI查找得到拟南芥、水稻(Oryza sativa)、毛果杨共42个NAC蛋白的氨基酸序列,结合ClNAC1蛋白用MEGA6.0的邻接(neighbor joining, NJ)算法构建系统进化树(Salinas et al., 2012)。
1.6 基因表达分析采用qPCR分析ClNAC1基因的表达模式,具体按照SYBR premix Extaq Ⅱ(TaKaRa)试剂盒说明书进行,以Actin基因作内参,相关引物序列见表 1。设4次重复,不同器官或组织的表达数据用Duncan法进行多重比较(Aminoroaya et al., 2016)。
|
|
依据ClNAC1基因的cDNA序列,设计特异引物ClNAC1SNP1-F和ClNAC1SNP1-R、ClNAC1SNP2-F和ClNAC1SNP2-R(序列见表 1)。以40个无性系的DNA为模板进行PCR扩增,产物回收后连接于pGEM-T Easy Vector,转化后挑取阳性克隆进行测序,将基因片段拼接成完整基因序列。在此基础上,利用MEGA6.0和DnaSP5.0软件对ClNAC1基因组序列进行比对分析,等位频率大于0.05时,对常见SNP位点、SNP频率和多样性指数(任意两序列之间核苷酸差异的平均数除以核苷酸总数)进行搜寻和计算;对同义突变、错义突变和无义突变情况进行统计;分析ClNAC1基因在自然群体当中的遗传分化和变异情况,以及基因内的LD延伸模式。
2 结果与分析 2.1 杉木ClNAC1基因序列的分离及结构特征分析利用转录组数据,经PCR和测序验证得到了ClNAC1基因的cDNA序列,相应GenBank登录号为:MG099703。该cDNA长度为1 286 bp,其中5′UTR及3′UTR分别为80 bp和114 bp,ORF为1 092 bp,编码363个氨基酸。根据该cDNA序列分离了对应的基因组DNA序列2 546 bp(GenBank登录号为:MG099704),有3个外显子和3个内含子,长度分别为172、273、647 bp和639、88、533 bp。第1个内含子位于5′UTR(图 1),与水稻蜡质基因Wx、沙冬青(Ammopiptanthus mongolicus)胁迫响应蛋白基因AmCBL1的5′UTR含有内含子的现象类似(蔡秀玲等,1997;庞涛等,2011)。
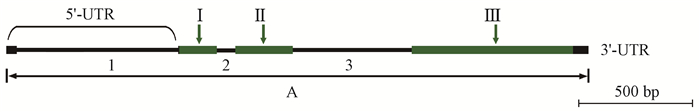
|
图 1 ClNAC1 基因结构示意 Figure 1 Schematic structure of the ClNAC1 5′-UTR: 5′未翻译区; Ⅰ:外显子1; Ⅱ:外显子2; Ⅲ:外显子3; 1: 5′-UTR内含子; 2:内含子2; 3:内含子3; 3′-UTR: 3′未翻译区; A:SNP分析区域. 5′-UTR: 5′untranslated region; Ⅰ: Exon 1; Ⅱ: Exon 2; Ⅲ: Exon 3; 1: 5′-UTR intron; 2: Intron 2; 3: Intron 3; 3′-UTR: 3′untranslated region; A: SNP analysis region. |
多序列比对发现,ClNAC1与毛果杨PtrWND2B(HQ215850)的序列相似性最高,为49%。与已报道的其他植物NAC蛋白相似,ClNAC1蛋白的N端含有128个氨基酸残基组成的NAC结构域,且具有NAC蛋白特有的保守性高的5个亚结构域A、B、C、D、E(图 2)。为了分析ClNAC1基因的系统发生关系,将ClNAC1蛋白与来自拟南芥、水稻、毛果杨的42个NAC构建进化树(图 3)。由图可知,NAC蛋白共分为6个分支(A-F),其中ClNAC1位于B分支,与拟南芥的NST1/2/3、毛果杨的PtrWND2A/B等聚在一起,属于第1大类(Class Ⅰ),该类多为与次生生长相关的NAC转录因子(李伟等,2011)。

|
图 2 ClNAC1与同源基因的编码区氨基酸序列比对 Figure 2 Alignment of the deduced amino acid sequences of ClNAC1 and homologous genes Cl:杉木;Ptr:毛果杨;At:拟南芥;黑色加粗线条:NAC结构域;A-E黑色长方形框内序列:亚结构域;方框线下方小写字母如pp:共有的氨基酸。 Cl: Cunninghamia lanceolata; Ptr: Populus trichocarpa; At: Arabidopsis thaliana; Black bold lines: NAC domain; A-E black rectangle box sequence: Subdomain; The small letters below the line like pp: Common amino acids. |

|
图 3 杉木ClNAC1蛋白与其他物种NAC的系统进化分析 Figure 3 Phylogenetic analysis of ClNAC1 and NAC proteins from related plant species At/AT:拟南芥;Ptr:毛果杨;Os:水稻。A-F:聚类分类;Class Ⅰ:次生生长相关类;Class Ⅱ:顶端分生组织形成和器官边界建立相关类;Class Ⅲ:生物胁迫响应相关类。进化树节点旁的数字为自举检验支持率,显示≥50%的分支,分支下方数字为长度。 At/AT: Arabidopsis thaliana; Ptr: Populus trichocarpa; Os: Oryza sativa. A-F: Clustering classification; Class Ⅰ: Secondary growth; Class Ⅱ: Formation of apical meristem and organ boundary; Class Ⅲ: Defense response of biological stress. The numbers beside the nodes are bootstrap values, which ≥50% are shown; The numbers below the clades are their lengths. |
利用设计的qPCR引物(表 1),对ClNAC1基因在不同组织、器官中的表达进行分析(以根为参照)。由图 4可知,ClNAC1基因在杉木的不同组织、器官中的表达量存在显著差异,其中雄球花(MC)中的表达量最大,其次为当年生嫩叶(YL),当年生成熟叶(ML)最小;ClNAC1在当年生半木质化茎(S2)中的表达量是未木质化茎(S1)的2.8倍,且在去年生茎木质部(XY)中的表达量比皮(BK)大3倍左右,表明该基因可能参与杉木球花发育和茎的木质化过程。

|
图 4 ClNAC1 在不同组织和器官的相对表达分析 Figure 4 Relative expression levels of ClNAC1 in different tissues and organs A:定量PCR所用的材料(标尺:1.0 cm)。B:ClNAC1的相对表达量。Error Bar代表 3个生物学重复的标准差SD。以RT为参照,标有相同小写字母(如a)的组织或器官表示在0.05水平差异不显著。RT:根;S1:未木质化茎;s1:未木质化茎横切片;S2:当年生半木质化茎;s2:当年生半木质化茎横切片;YL:嫩叶;ML:当年生成熟叶;BD:嫩芽;FC:雌球花;MC:雄球花;XY:去年生茎木质部;BK:去年生茎皮层。 A: Plant materials used for the qPCR analysis (Bar=1.0 cm). B: Relative expression levels of the ClNAC1. Error bars represent SD of three biological replicates. RT was used as a reference, the common small letters above columns like "a" indicate no significant difference at 0.05 levels. RT: Root; S1: Non-lignified stem; s1: The transverse section of non-lignified stem; S2: Semi-lignified stem; s2: The transverse section of semi-lignified stem; YL: Young leaf; ML: Mature leaf; BD: Bud; FC: Female cone; MC: Male cone; XY: Xylem; BK: Bark. |
以来自6个不同群体40个杉木无性系为材料,对ClNAC1的基因序列进行PCR扩增、测序,以及SNP位点的发现。结果显示在内含子区域存在不同长度的碱基缺失,其中在第1个内含子区域存在长为20 bp、24 bp的2段缺失,第3个内含子区域有长为28 bp、30 bp的2段缺失,依次命名为D1、D2、D3和D4。这些缺失在不同群体中的发生比率存在一定的差异(表 2),其中D1发生比率较高,整体达到了72.6%,D2则较低,整体发生比率仅为25.6%。另外,在3′UTR有1个具多态性的4 bp简单重复序列(simple sequence repeats,SSR)位点。后续SNP分析在包含190 bp非编码区、1 092 bp外显子、1 172 bp内含子的共有序列上进行。由表 3可见,共检测到104个常见SNP位点,平均频率为1/24 bp,其中在外显子、内含子、非编码区中分别有32、66、6个SNPs,相应的出现频率分别为1/34 bp、1/18 bp、1/32 bp。由此可知,杉木ClNAC1基因内的单核苷酸变异非常丰富,该基因序列不同区域的保守性不同,其中外显子显示出较低的变异频率,暗示该基因在进化中编码区受到了较强的选择压力;而非编码区的低变异频率可能与分析区域不完整有关。对SNP位点的变异类型进行统计(表 4),发现有53个SNP位点发生转换,分别包含23个A↔G和30个T↔C;48个SNP位点发生颠换,分别有12个T↔G、12个T↔A、8个G↔C和16个A↔C;转换与颠换比为1.10。此外,3个SNP位点发生三态变异,分别为:A↔G↔T变异、A↔G↔C变异、C↔G↔T变异。
|
|
|
|
|
|
对编码区内检测到的32个SNPs进行了突变类型分析,来确认ClNAC1编码区的SNPs是否引起编码氨基酸的改变。由表 5可知,属同义突变、错义突变、无义突变的位点分别有25、7、0个。在7个错义突变位点中,有2个处于密码子第1个核苷酸位置上,3个处于密码子第2个核苷酸位置上,2个位于密码子第3个核苷酸位置上。非同义突变氨基酸改变的类型具有多样性:1)在第2个外显子中有1个错义突变:突变位置在密码子第3位上,由GAC变为GAA,编码氨基酸由天冬氨酸(Asp)变为谷氨酸(Glu)。2)在第3个外显子中共有6个错义突变:2个第1位密码子改变,由ATC变为GTC,CTG变为ATG,编码氨基酸由异亮氨酸(Lle)变为缬氨酸(Val)、亮氨酸(Leu)变为蛋氨酸(Met);3个第2位密码子改变,由AGT变为AAT,ATC变为ACC,ACT突变为AAT,其所编码的氨基酸分别由丝氨酸(Ser)变为天冬酰胺(Asn)、异亮氨酸(Lle)变为苏氨酸(Thr)、苏氨酸(Thr)变为天冬酰胺(Asn);1个第3位密码子改变,由GAC变为GAA,氨基酸由天冬氨酸(Asp)变为谷氨酸(Glu)。
|
|
分别对湖南靖县、湖南会同、贵州锦屏、广西那坡、广西三江、广西融水6个群体内个体进行单体型、单核苷酸多样性分析和Fu and Li’s D(Fu et al., 1993)、Tajima’s D(Tajima, 1989)检验,结果见表 6。SNP多样性指数πtot、πsil、πs、πn都存在差异,但0.05<P<0.10或者P>0.10,可以看出差异不显著,说明在不同群体间ClNAC1的分化程度没有达到显著的水平。其中6个群体的非同义突变多样性πn均小于同义突变多样性πs,即比率<1,说明在杉木的进化过程中,ClNAC1基因的选择压力主要是纯化选择。在中性检验中,得出6个群体内的Fu and Li’s D和Tajima’s D均为正值,总体的Tajima’s D>0,表明杉木种群内的进化受到平衡选择的影响,具有较高的多态性且中等频率的等位基因在群体中占主导作用。
|
|
SNP间重组事件检测结果见表 7。湖南靖县和会同分别检测到了4次和3次重组事件,贵州锦屏为13次,广西三江、融水、那坡分别为8、8、15次;将其与信息位点数相比得到每一个信息位点的重组率在0.050~0.202之间变化,其中广西那坡的相应数值最高,为0.202,湖南靖县最低,仅为0.050(表 7)。在DnaSP5.0软件中对杉木不同群体内ClNAC1基因SNP位点间连锁不平衡(LD)的延伸长度和程度进行分析(图 5),结果显示ClNAC1基因内部SNPs的LD程度随核苷酸序列长度的增加迅速衰减,但6个不同群体中的衰减程度各有不同。r2>0.1时,在广西那坡、广西三江、广西融水、贵州锦屏、湖南会同和湖南靖县中LD衰退序列长度分别为1 025、1 339、1 494、1 501、1 760、2 460 bp。
|
|

|
图 5 不同群体在ClNAC1 基因内SNPs的连锁不平衡 Figure 5 Linkage disequilibrium(LD) of SNPs in ClNAC1 from different populations 横坐标代表核苷酸距离,纵坐标(r2)代表 2个位点之间的统计相关,r2=0时说明2个位点之间是完全独立的,r2=1时说明2个位点的等位基因有着相同的频率,这时2个位点组成的4种可能的单倍型中仅表现2种单倍型。 The abscissa represents the nucleotide distance, and the ordinate(r2)represents the statistical correlation between the two sites. r2=0 indicates that the two sites are completely independent, r2 =1 indicates the alleles at the two sites share the same frequency, and the four possible haplotypes of the two sites show only two haplotypes. |
首次在杉木中克隆得到了ClNAC1基因,相应编码蛋白由363个氨基酸残基组成,且在N端具特殊的NAC结构域。系统进化分析表明ClNAC1蛋白与拟南芥NST1/2/3、毛果杨PtrWND2A/B等氨基酸序列相似性较高并处于同一分支。先前的研究表明,拟南芥NAC家族成员NST1/2/3是调控花序茎次生壁发育的关键因子,主要在花序茎的束间纤维和木纤维中表达(Zhong et al., 2015)。另外,NST1/NST2在花药中也呈较高水平表达,超表达这2个基因的拟南芥表现出花药、雄蕊的木质次生壁增厚,木质素异常沉积(Mitsuda et al., 2005)。与拟南芥SND1(NST3)直系同源的毛果杨PtrWND2A/B和毛白杨PtoVNS11(VND, NST/SND, SMB-related NAC homologs11)在次生壁的发育和木质素的沉积等方面具重要调控作用,在茎的木质部优势表达(Zhong et al., 2010;Yang et al., 2015)。试验中发现ClNAC1在雄花序中表达非常高,且在木质化茎的木质部也呈较高丰度表达,因此推测该基因可能影响了杉木药室内壁和次生壁的发育。
先前的研究表明了NAC基因家族的一些成员在木材形成中的重要调控作用(Zhong et al., 2010;Yang et al., 2015),因此,本研究在木材性状变异广泛的杉木群体内开展杉木ClNAC1基因序列内SNP等位变异,可为挖掘与木材性状相关的功能SNP位点,解析木材性状变异的分子机制并以此开展分子育种提供重要理论依据。ClNAC1基因在不同无性系中存在丰富的SNP变异,多样性水平达0.012 53,平均SNP频率为1/24 bp,高于报道的毛白杨PtoCesA4(0.005 02,1/35 bp)和光皮桦(Betula luminifera) BlSPL1(0.007 12,1/26 bp)的相应数值(徐煲铧等,2009;李玉岭等,2013)。在编码区SNPs中非同义突变与同义突变的位点分别有7和25个,二者之比为0.28(<1),与毛白杨PtoSUS1、PtoCesA4的结果类似(潘炜等,2011;徐煲铧等,2009),说明ClNAC1基因编码区在进化过程中受到的选择压较大,纯化选择对其非同义位点起了非常重要的作用。虽然ClNAC1在群体间的分化程度未达到显著水平,但所有群体都显示出非同义突变单核苷酸多样性指数(πn)与同义突变单核苷酸多样性指数(πs)的比率<1,这进一步验证了该基因在进化中主要受到纯化选择作用。
连锁不平衡(LD)作图的2种策略分别是基于全基因组扫描和基于候选基因进行的,针对特定群体的作图策略选择取决于LD在该群体内的延伸长度及程度。不同物种、不同群体LD的延伸范围变化也非常大。在一些自交植物如拟南芥、小麦(Triticum aestivum)等中,LD可延伸至250 kb以上(Kraakman et al., 2004; Nordborg et al., 2002);而在异交作物玉米(Zea mays)中LD延伸在1 kb内衰减(Remington et al., 2001)。目前已开展类似研究的林木皆为异交物种,LD延伸在1 kb至几kb内消失。火炬松(Pinus taeda)的19个候选基因中,LD延伸均在2 kb之内消失(Lu et al., 2016)。欧洲水青冈(Fagus sylvatica)16个与非生物胁迫相关的候选基因在800 bp内SNPs连锁不平衡就衰退(Lalague et al., 2014)。本试验同样发现ClNAC1基因SNPs在杉木6个群体中的LD皆在较短距离内迅速消失,表明开展基于该基因的LD作图是可行的。另外,在不同杉木群体内,该基因SNPs的LD衰减程度明显不同,湖南靖县群体的LD衰减距离最长,考虑到该群体每一信息位点的重组事件数最小,推测这可能是由于该群体相对较小,且群体内个体间基因交流相对少导致突变产生的多态性较少,或群体中受到一定驯化选择,导致群体遗传多样性下降,位点间的相关性(连锁程度)加强所致。
4 结论首次在杉木中分离得到了ClNAC1基因,相应编码蛋白由363个氨基酸残基组成,且在N端具典型NAC结构域;ClNAC1基因组DNA序列包含3个外显子和3个内含子。进化树构建分析显示ClNAC1蛋白与拟南芥NST1/2/3、毛果杨PtrWND2A/B等次生生长相关的NAC聚为一类;ClNAC1在雄球花和木质化茎的木质部中优势表达,因此推测该基因在杉木茎和花药药室木质次生壁发育中具有重要调控作用。ClNAC1在不同杉木群体无性系间存在丰富的SNP变异,多样性水平达到0.012 53,且在编码区SNPs中非同义突变与同义突变的位点之比为0.28。在6个群体中ClNAC1基因SNP位点间的LD皆在基因内迅速消失,表明开展基于ClNAC1的连锁不平衡作图是可行的。
蔡秀玲, 王宗阳, 郑霏琴, 等. 1997. 水稻蜡质基因5'非翻译区一个与调控有关的内含子[J]. 植物生理学报, 23(3): 257-261. (Cai X L, Wang Z Y, Zheng F Q, et al. 1997. A regulation-related introl in 5' untranslated region of rice Waxy gene[J]. Acta Phytophysiologica Sinica, 23(3): 257-261. [in Chinese]) |
高燕. 2011. 杉木木材形成过程扩展蛋白基因的克隆与表达分析[J]. 林业科学, 47(11): 44-51. (Gao Y. 2011. Cloning and expression analysis of expansin genes during wood formation in Chinese fir[J]. Scientia Silvae Sinicae, 47(11): 44-51. DOI:10.11707/j.1001-7488.20111108 [in Chinese]) |
巩琛锐, 王璐, 杜庆章, 等. 2013. 毛白杨S-腺苷高半胱氨酸水解酶基因PtSAHHA的克隆、表达及SNP关联分析[J]. 林业科学, 49(11): 67-76. (Gong C R, Wang L, Du Q Z, et al. 2013. Isolation, expression, and association genetics of the S-adenosyl-L-homocysteine hydrolase gene A(PtSAHHA)in Populus tomentosa[J]. Scientia Silvae Sinicae, 49(11): 67-76. [in Chinese]) |
李玉岭, 周厚君, 林二培, 等. 2013. 光皮桦BlSPL1转录因子基因的克隆、表达及单核苷酸多态性分析[J]. 林业科学, 49(9): 52-61. (Li Y L, Zhou H J, Lin E P, et al. 2013. Isolation, expression and single nucleotide polymorphisms analysis of transcription factor(BlSPL1)from Betula luminifera[J]. Scientia Silvae Sinicae, 49(9): 52-61. [in Chinese]) |
李伟, 韩蕾, 钱永强, 等. 2011. 植物NAC转录因子的种类、特征及功能[J]. 应用与环境生物学报, 17(4): 596-606. (Li W, Han L, Qian Y Q, et al. 2011. Characteristics and functions of NAC transcription factors in plants[J]. Chinese Journal of Applied and Environmental Biology, 17(4): 596-606. [in Chinese]) |
潘炜, 田佳星, 杜庆章, 等. 2011. 毛白杨蔗糖合酶基因PtSUS1的克隆、表达及单核苷酸多态性分析[J]. 林业科学, 47(3): 52-58. (Pan W, Tian J X, Du Q Z, et al. 2011. Isolation, expression and single nucleotide polymorphisms analysis of sucrose synthase gene(PtSUS1)from Populus tomentosa[J]. Scientia Silvae Sinicae, 47(3): 52-58. [in Chinese]) |
庞涛, 郭丽丽, 夏新莉, 等. 2011. CBL1基因5'非翻译区内含子在旱生植物沙冬青中的作用[J]. 北京林业大学学报, 33(6): 157-165. (Pang T, Guo L L, Xia X L, et al. 2011. Effects of 5' UTR intron of AmCBL1 gene in a xerophyte Ammopiptanthus mongolicus[J]. Journal of Beijing Forestry University, 33(6): 157-165. [in Chinese]) |
王钰, 童再康, 黄华宏, 等. 2010. 光皮桦总RNA的提取及Actin基因的克隆[J]. 浙江林业科技, 30(4): 32-36. (Wang Y, Tong Z K, Huang H H, et al. 2010. Extracting of total RNA from Betula luminifera and the cloning of Actin gene[J]. Journal of Zhejiang Forestry Science and Technology, 30(4): 32-36. DOI:10.3969/j.issn.1001-3776.2010.04.008 [in Chinese]) |
谢媛, 田云, 卢向阳. 2012. 单核苷酸多态性及其在水稻中的应用(综述)[J]. 亚热带植物科学, 41(1): 68-72. (Xie Y, Tian Y, Lu X Y. 2012. Single nucleotide polymorphism and its application in rice(Review)[J]. Subtropical Plant Science, 41(1): 68-72. DOI:10.3969/j.issn.1009-7791.2012.01.018 [in Chinese]) |
谢一青, 李志真, 黄儒珠, 等. 2006. 光皮桦基因组DNA提取方法比较[J]. 浙江林学院学报, 23(6): 664-668. (Xie Y Q, Li Z Z, Huang R Z, et al. 2006. Comparison of methods of extracting genomic DNA from Betula luminifera[J]. Journal of Zhejiang Forestry College, 23(6): 664-668. DOI:10.3969/j.issn.2095-0756.2006.06.013 [in Chinese]) |
徐煲铧, 杨晓慧, 李百炼, 等. 2009. 毛白杨纤维素合酶基因PtCesA4的克隆、表达及单核苷酸多态性分析[J]. 林业科学, 45(5): 1-10. (Xu B H, Yang X H, Li B L, et al. 2009. Isolation, expression and single nucleotide polymorphisms analysis of cellulose synthase gene(PtCesA4) from Populus tomentosa[J]. Scientia Silvae Sinicae, 45(5): 1-10. DOI:10.3321/j.issn:1001-7488.2009.05.001 [in Chinese]) |
徐莉莉, 童再康, 林二培, 等. 2013. 杉木苯丙氨酸解氨酶基因ClPAL的克隆与表达分析[J]. 林业科学, 49(12): 64-72. (Xu L L, Tong Z K, Lin E P, et al. 2013. Isolation and expression of ClPAL genes in Chinese fir(Cunninghamia lanceolata)[J]. Scientia Silvae Sinicae, 49(12): 64-72. [in Chinese]) |
杨晓慧, 张有慧, 张志毅, 等. 2009. 毛白杨干细胞决定基因Wuschel的克隆及其单核苷酸多态性分析[J]. 林业科学, 45(1): 43-49. (Yang X H, Zhang Y H, Zhang Z Y, et al. 2009. Isolation and single nucleotide polymorphisms analysis of stem cell organizer gene Wuschel in Populus tomentosa[J]. Scientia Silvae Sinicae, 45(1): 43-49. DOI:10.3321/j.issn:1001-7488.2009.01.009 [in Chinese]) |
Aminoroaya K, Sadeghi A A, Ansaripirsaraei Z, et al. 2016. The effects of cyclical higher incubation temperatures on body and organs weights, thyroid hormones and HSP70 gene expression of newly hatched broiler chicks[J]. Kafkas Universitesi Veteriner Fakultesi Dergisi, 22(4): 613-618. |
Demura T, Fukuda H. 2007. Transcriptional regulation in wood formation[J]. Trends in Plant Science, 12(2): 64-70. DOI:10.1016/j.tplants.2006.12.006 |
Fu Y X, Li W H. 1993. Statistical tests of neutrality of mutations[J]. Genetics, 133(3): 693-709. |
Grant E H, Fujino T, Beers E P, et al. 2010. Characterization of NAC domain transcription factors implicated in control of vascular cell differentiation in Arabidopsis and Populus[J]. Planta, 232(2): 337-352. DOI:10.1007/s00425-010-1181-2 |
Groover A T. 2005. What genes make a tree a tree?[J]. Trends in Plant Science, 10(5): 210-214. DOI:10.1016/j.tplants.2005.03.001 |
Groover A, Robischon M. 2006. Developmental mechanisms regulating secondary growth in woody plants[J]. Current Opinion in Plant Biology, 9(1): 55-58. DOI:10.1016/j.pbi.2005.11.013 |
Huang H H, Xu L L, Tong Z K, et al. 2012. De novo characterization of the Chinese fir(Cunninghamia lanceolata)transcriptome and analysis of candidate genes involved in cellulose and lignin biosynthesis[J]. BMC Genomics, 13(1): 648-671. DOI:10.1186/1471-2164-13-648 |
Kalluri U C, Joshi C P. 2004. Differential expression patterns of two cellulose synthase genes are associated with primary and secondary cell wall development in Aspen trees[J]. Planta, 220(1): 47-55. DOI:10.1007/s00425-004-1329-z |
Kraakman A W, Niks R E, Petra M B, et al. 2004. Linkage disequilibrium mapping of yield and yield stability in modern spring barley cultivars[J]. Genetics, 168(1): 435-446. DOI:10.1534/genetics.104.026831 |
Lalague H, Csillery K, Oddou-muratorio S, et al. 2014. Nucleotide diversity and linkage disequilibrium at 58 stress response and phenology candidate genes in a European beech (Fagus sylvatica) population from southeastern France[J]. Tree Genetics & Genomes, 10(1): 15-26. |
Lu M M, Krutovsky K V, Nelson C D, et al. 2016. Exome genotyping, linkage disequilibrium and population structure in loblolly pine (Pinus taeda L.)[J]. BMC Genomics, 17(1): 730-740. DOI:10.1186/s12864-016-3081-8 |
Mitsuda N, Seki M, Shinozaki K, et al. 2005. The NAC transcription factors NST1 and NST2 of Arabidopsis regulate secondary wall thickenings and are required for anther dehiscence[J]. Plant Cell, 17(11): 2993-3006. DOI:10.1105/tpc.105.036004 |
Nordborg M, Borevitz J O, Bergelson J, et al. 2002. The extent of linkage disequilibrium in Arabidopsis thaliana[J]. Nature Genetics, 30(2): 190-193. DOI:10.1038/ng813 |
Remington D L, Thornsberry J M, Matsuoka Y, et al. 2001. Structure of linkage disequilibrium and phenotypic associations in the maize genome[J]. Proceedings of the National Academy of Sciences of the United States of America, 98(20): 11479-11484. DOI:10.1073/pnas.201394398 |
Salinas M, Xing S, Höhmann S, et al. 2012. Genomic organization, phylogenetic comparison and differential expression of the SBP-box family of transcription factors in tomato[J]. Planta, 235(6): 1171-1184. DOI:10.1007/s00425-011-1565-y |
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 123(3): 585-595. |
Wu L, Joshi C P, Chiang V L. 2000. A xylem-specific cellulose synthase gene from Aspen(Populus tremuloides)is responsive to mechanical stress[J]. Plant Journal, 22(6): 495-502. DOI:10.1046/j.1365-313x.2000.00758.x |
Yang L, Hou Y, Zhao X, et al. 2015. Identification and characterization of a wood-associated NAC domain transcription factor PtoVNS11 from Populus tomentosa Carr[J]. Trees, 29(4): 1091-1101. DOI:10.1007/s00468-015-1188-1 |
Zhong R, Ye Z H. 2010. Functional characterization of poplar wood-associated NAC domain transcription factors[J]. Plant Physiology, 152(2): 1044-1055. DOI:10.1104/pp.109.148270 |
Zhong R, Ye Z H. 2015. Secondary cell walls: biosynthesis, patterned deposition and transcriptional regulation[J]. Plant & Cell Physiology, 56(2): 195-214. |
 2018, Vol. 54
2018, Vol. 54

