文章信息
- 任健, 殷雨晴, 张慧慧, 陈典, 王可新, 王勇
- Ren Jian, Yin Yuqing, Zhang Huihui, Chen Dian, Wang Kexin, Wang Yong
- 马占相思纤维素合酶基因AmCesA1的克隆及分析
- Cloning and Analysis of AmCesA1 Gene in Acacia mangium
- 林业科学, 2018, 54(8): 79-87.
- Scientia Silvae Sinicae, 2018, 54(8): 79-87.
- DOI: 10.11707/j.1001-7488.20180809
-
文章历史
- 收稿日期:2017-10-23
- 修回日期:2017-12-01
-
作者相关文章
纤维素是植物细胞壁中必不可少的结构成分,作为一种可再生的生物材料被广泛应用于各种产品中。初生壁中的纤维素微纤丝在细胞扩增阶段调控植物的形态建成,次生壁中的纤维素也使得某些植物细胞具有特定功能(Sethapphong et al., 2016)。纤维素对植物生长的重要作用及其作为一种可再生生物材料的特质,使得对它的研究具有重要意义。
纤维素是葡萄糖分子通过无支链β-1, 4糖苷键连接形成的500~14 000个葡萄糖分子糖链,36条糖链通过氢键形成直径约为3 nm的纤维素微纤丝(Taylor, 2008; Xu et al., 2007),微纤丝相互交织形成网状,构成了细胞壁的基本框架。纤维素的葡聚糖链是由在质膜上的纤维素合酶复合体(CSC)以UDP为底物合成的,并由CSC将其分泌到胞外。
纤维素合酶基因家族中的不同成员根据功能差异被分成2类,分别负责初生壁纤维素和次生壁纤维素的合成。在拟南芥(Arabidopsis thaliana)中CESA1、CESA3、CESA6负责初生壁的形成(Arioli et al., 1998; Fagard et al., 2000; Scheible et al., 2001),CESA4、CESA7、CESA8负责次生壁的合成(Taylor et al., 2000;1999; Turner et al., 1997)。在拟南芥中无论是与初生壁合成有关的纤维素合酶基因还是与次生壁形成有关的纤维素合酶基因,其蛋白都表现出1:1:1的化学计量比(Gonneau et al., 2014; Hill et al., 2014)。
纤维素含量和品质对于以造纸为主要工业用途的速生类树木来说是一个重要的经济性状,一些树木的CesA基因已被分离、分析。林木中克隆的第1个纤维素合酶基因是美洲山杨(Populus tremuloides)的PtrCesA1(Wu et al., 2000),该基因在植物茎中表达,特别在次生壁形成期间表达量尤为显著。此后,大青杨(Populus ussuriensis)、苎麻(Boehmeria nivea)、马尾松(Pinus massoniana)等木本植物都相继克隆出CesA基因(许雷等, 2012;刘昱翔等,2014;阮维程等, 2015)。对杨树纤维素合酶的研究发现,杨树的木质部细胞膜上至少存在2种纤维素合酶复合体, 复合体Ⅰ由CesA4、CesA7、CesA8、CesA17和CesA18构成, 参与次生细胞壁合成;复合体Ⅱ由CesA3、CesA10、CesA11、CesA13、CesA15和CesA16构成,参与初生细胞壁和次生细胞壁的合成(Song et al., 2010)。
马占相思(Acacia mangium)是一种重要的速生类木材,也是我国南方广泛种植的一种造纸用树。本研究依据纤维素合酶家族的保守序列,设计了2条简并引物,利用两步RT-PCR和5′RACE技术从马占相思中得到了1个纤维素合酶基因AmCesA1,根据它的表达特征以及对激素的应答,推测该基因参与马占相思初生细胞壁的形成,为后期改善马占相思及金合欢属植物纤维素性状奠定了前期基础。
1 材料与方法 1.1 试验材料和处理方法马占相思种子购自中国林木种子公司。将马占相思种子播于体积比1:2的蛭石和草炭土的培养基质中,置于温度25~30 ℃、湿度60%~70%、光照时数为16 h的温室培养,每周定期浇水。出苗后40天取植株叶片用于总RNA的提取。取根、茎、叶作为基因表达分析试验材料。各样品取样后均用液氮速冻并移至-70 ℃保存备用。
激素处理的试验方法参照魏凯莉等(2017)的方法并加以适当修改。将40天苗龄且生长旺盛的马占相思幼苗分成3组,每组分别喷施5×10-5 mol·L-1赤霉素(gibberellin, GA3)、1×10-3 mol·L-1 6-BA(6-benzylaminopurine)、1×10-4 mol·L-1茉莉酸甲酯(methyl jasmonate, MeJA)。每隔4 h均匀喷施1次,共喷施3次,每喷施1次后立刻取其叶片并放置-80 ℃冰箱中保存。每个处理设置1个对照,喷施与激素等量的蒸馏水。每个处理设置3个重复。
1.2 两步反转录PCR和AmCesA1基因的克隆用改良CTAB法提取马占相思幼苗总RNA(李晶等, 2009)。以OligodT为引物进行反转录。以反转录产物为模板,用AmCesDe1和OligodT引物进行第1步反转录PCR扩增,PCR条件为:94 ℃预变性5 min,热启动; 94 ℃ 25 s, 55 ℃ 1 min, 72 ℃ 2 min, 33个循环;72 ℃ 10 min终止反应。PCR产物经琼脂糖凝胶电泳检测后,过柱回收,与pGEM-Teasy载体连接,转化大肠杆菌(Escherichia coli)DH5α菌株;获得的白斑以AmCesDe1和OligodT为引物对进行菌落PCR鉴定,筛选出阳性的克隆。测序后以其为模板,以Aces1.3-3、AmCesDe2进行第2步反转录PCR扩增,条件:94 ℃预变性5 min,热启动; 94 ℃ 30 s, 53 ℃ 1 min, 72 ℃ 90 s, 30个循环;72 ℃ 10 min终止反应。连接测序后利用Takara公司5′-Full RACE Kit with TAP试剂盒进行5′RACE。RACE反应按试剂盒说明操作(Code No.6106)。将第1步扩增和第2步扩增序列以及RACE所得序列进行拼接,根据拼接结果设计引物Aces5-1和Aces3-1,进行基因cDNA全长序列克隆。试验所涉及到的引物序列见表 1。
|
|
采用ExPASY(http://www.expasy.org/)对AmCesA1编码的蛋白质一级结构进行分析。利用TMHMM Sever v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构预测。使用Pfam(http://pfam.xfam.org/)对AmCesA1结构域进行分析。利用ANTHEPROT6.66软件对AmCesA1进行蛋白质二级结构预测。利用DNAMAN6.0进行氨基酸多序列比对分析。运用Clustal1.81和Mega7.0进行系统进化树分析。
1.4 基因组DNA分离和Southern杂交分析用Qiagen公司生产的DNA提取试剂盒(DNeasy Plant Mini Kits, Cat.No.69104)提取马占相思基因组DNA。采用罗氏地高辛试剂盒(Cat.No.1585606)进行Southern杂交。68 ℃洗膜,地高辛法标记,严格按照规程操作。
1.5 AmCesA1组织特异性表达和激素响应分析利用改良的CTAB法提取马占相思幼苗不同部位(根、茎、叶)RNA。使用superscript Ⅲ first-strand synthesis system (invitrogen) Cat.No.18080051合成cDNA的第1条链。在序列可变区HVR-1设计实时荧光定量PCR引物,引物序列详见表 1。利用TaKaRa公司SYBR Premix Ex Taq (TaKaRa)荧光定量试剂盒完成荧光定量试验。选择肌动蛋白基因(Actin)作为内参基因(ong et al., 2012),3次重复。实时荧光定量PCR的数据用Bio-Rad IQ5软件进行分析。
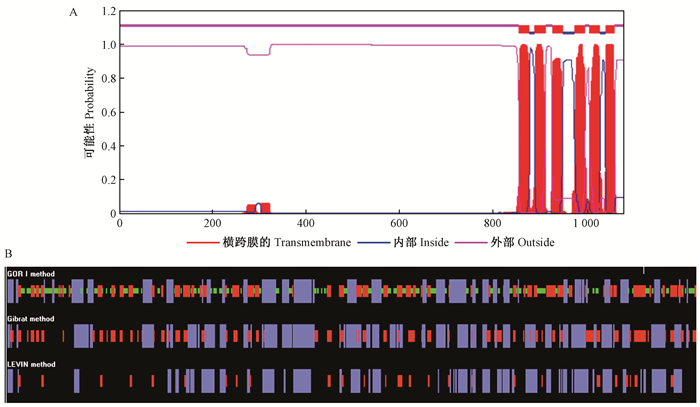
2 结果与分析 2.1 AmCesA1基因克隆与序列分析以AmcesDe1、OligodT和Aces1.3-3、AmcesDe2为引物进行扩增,分别得到了1条约1.3~1.6 kb和1.1 kb的DNA片断。之后通过5′RACE组装得到1个全长的AmCesA1。AmCesA1的cDNA全长为3 793 bp,其开放读码框(ORF)为3 249 bp,编码1 082个氨基酸,5′非翻译区(UTR)为192 bp,3′非翻译区(UTR)为352 bp, 在其3′端下游有明显的AATAA加尾信号。采用ProtParam对AmCesA1编码的蛋白质一级结构进行分析。蛋白分子式为C5495H8491N1457O1579S50,分子质量为121.83 kDa。正电荷氨基酸残基数(Arg+Lys)的数目为121,负电荷氨基酸残基数(Asp+Glu)的数目为125。等电点为6.51,为酸性蛋白质。它的不稳定系数是40.82,属于不稳定蛋白质。跨膜结构预测结果如图 1所示,N端跨膜域不明显,C端存在6个跨膜区域,对AmCesA1结构域的分析表明它存在着糖基转移酶类家族所特有的D, D, D, QxxRW结构。分别采用GOR、Gibrat、LEVIN这3种算法对AmCesA1进行蛋白质二级结构预测(表 2),结果表明AmCesA1蛋白二级结构具有较多的α-螺旋(α-helix)、β-折叠(β-sheet)、无规则卷曲(random coil)而β-转角(β-turn)很少。
|
图 1 AmCesA1氨基酸序列跨膜区分析(A)和二级结构分析(B) Figure 1 Transmembrane domain(A) and secondary structure(B) analyse of deduced amino acid sequence of AmCesA1 蓝色区域指α-螺旋;红色区域指β-折叠;绿色区指β-转角;其他区域指无规则卷曲。 B lue region indicates α-helix; Red region indicates β-sheet; Green region indicates β-turn; Other region indicates random coil. |
|
|
利用DNAman6.0软件将AmCesA1与拟南芥AtCesA1、大豆(Glycine max) GmCesA1、蔓花生(Arachis duranensis) AdCesA1、白桦(Betula platyphylla) BpCesA1进行序列比对分析,同时把相应的区域进行了标注(图 2)。在整个蛋白的结构上,于N端存在1个Zinc-finger区,其后为植物纤维素合酶家族所特有的HVR-1区,之后顺序为P-CR、HVR-2区域,其中P-CR位于保守的domain A区域内,在HVR-2后为另一个纤维素合酶家族的保守区域domain B。

|
图 2 AmCesA1氨基酸序列比对分析 Figure 2 Alignment of the deduced amino acid sequences of AmCesA1 对于CesA功能至关重要的3个Asp(D)残基和QxxRW基序(见正文)由星号标注。顶部虚线也显示了植物保守区(P-CR), 可变区(HVR-1和HVR-2)以及锌指结构。 The three Asp (D) residues and the QxxRW motif that are critical for the function of CesA (see text) are marked by asterisks. The plant conserved region (P-CR), variable regions (HVR-1 and HVR-2), and Zinc-RING finger motif are also shown by overhead dashed lines. |
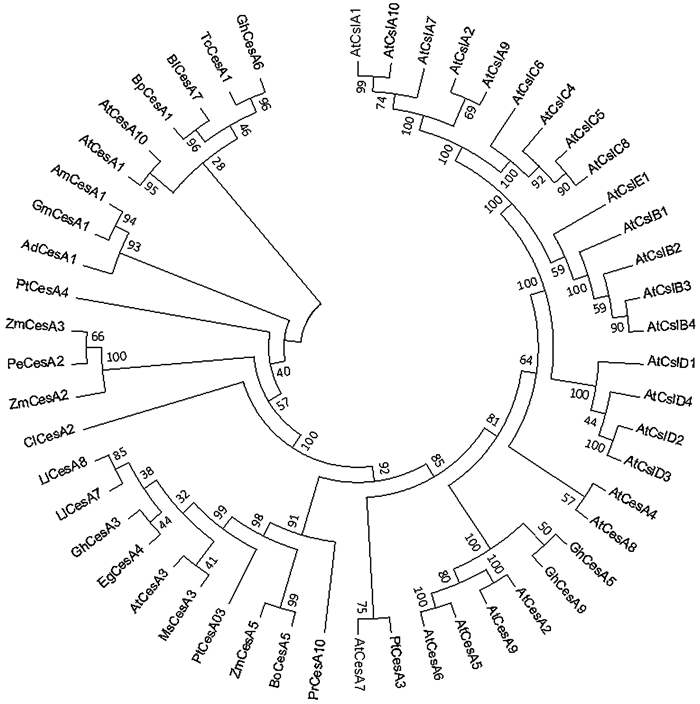
为了考察AmCesA1的进化特点,选取了拟南芥、陆地棉(Gossypium hirsutum)、光皮桦(Betula luminifera)、辐射松(Pinus radiata)、大豆等植物的纤维素合酶家族成员的氨基酸序列并选择拟南芥纤维素合酶类似蛋白(Csl)作为外类群以检验分析结果。利用Clustal1.81和Mega7的NJ算法进行进化树构建(图 3)。
|
图 3 AmCesA1的系统进化树 Figure 3 Phylogenetic tree of AmCesA1 from Acacia mangium AmCesA1:马占相思Acacia mangium (AAT66940); BpCesA1:白桦Betula platyphylla (AKN79315); GmCesA1:大豆Glycine max (XP_003522623); PrCesA10:辐射松Pinus radiata (AAQ63935); BlCesA7:光皮桦Betula luminifera (AGV22109); PtCesA3:火炬松Pinus taeda (AAX18649.1); EgCesA:巨桉Eucalyptus grandis (AAY60846); TcCesA1:可可Theobroma cacao (XP_007013842); GhCesA3, GhCesA5, GhCesA6, GhCesA9:陆地棉Gossypium hirsutum (AAD39534, AFB18634, AFB18635, AFB18639); BoCesA5:绿竹Bambusa oldhamii(AAY43222); AdCesA1:蔓花生Arachis duranensis (XP_015937454); PtCesA03:毛白杨Populus tomentosa (AIS86012); PeCesA2:毛竹Phyllostachys edulis (ACT16001); PtCesA4:美洲山杨Populus tremuloides(AAO25536); MsCesA3:苜蓿Medicago sativa (AII73575); ClCesA2:杉木Cunninghamia lanceolata (AFM37966);LlCesA7, LlCesA8:银合欢Leucaena leucocephala (ACU87559, ACU80553); ZmCesA2, ZmCesA3, ZmCesA5:玉米Zea mays (NP_001105574, NP_001292792, NP_001104955); AtCesA1, AtCesA2, AtCesA3, AtCesA4, AtCesA5, AtCesA6, AtCesA7, AtCesA8, AtCesA9, AtCesA10, AtCslA1, AtCslA2, AtCslA7, AtCslA9, AtCslA10, AtCslB1, AtCslB2, AtCslB3, AtCslB4, AtCslC4, AtCslC5, AtCslC6, AtCslC8, AtCslD1, AtCslD2, AtCslD3, AtCslD4:拟南芥Arabidopsis thaliana(NP_194967, NP_195645, NP_196136, NP_199216, NP_196549, NP_201279, NP_197244, NP_567564, NP_179768, NP_180124, NP_001319964, NP_197666, NP_565813, NP_195996, NP_173818, NP_180820, NP_180821, NP_850190, NP_180813, NP_566835, NP_194887, NP_187389, NP_180039, NP_180869, NP_001318575, NP_186955, NP_195532). |
从结果中可以看出,Csl聚为一类,CesA聚为一类,说明进化树分析的结果是可靠的。从CesA的氨基酸组成来看,,AmCesA1和大豆GmCesA1和蔓花生AdCesA1比较接近,这符合它们都是豆科(Leguminosae)植物的特性。马占相思AmCesA1并没有出现同木本植物亲缘较近的预期结果,而是与同为豆科的大豆和蔓花生分在一个组。推测CesA是一个古老的基因,它的分化早于草本植物和木本植物的分化。通过uniprot数据库得知,大豆GmCesA1和蔓花生AdCesA1均与纤维素生物合成有关并参与初生壁的合成,因此推测AmCesA1也参与初生壁的合成。在与拟南芥CESA比较中发现AmCesA1和拟南芥的AtCesA1和AtCesA10比较相似,进一步比较其相似性为86.18%和80.29%,从而说明它的功能或许与相应的拟南芥纤维素合酶异构体相近。拟南芥中,从uniprot数据库得知AtCesA1和AtCesA10都参与初级细胞壁的形成,这也进一步说明AmCesA1参与马占相思初生细胞壁的形成。
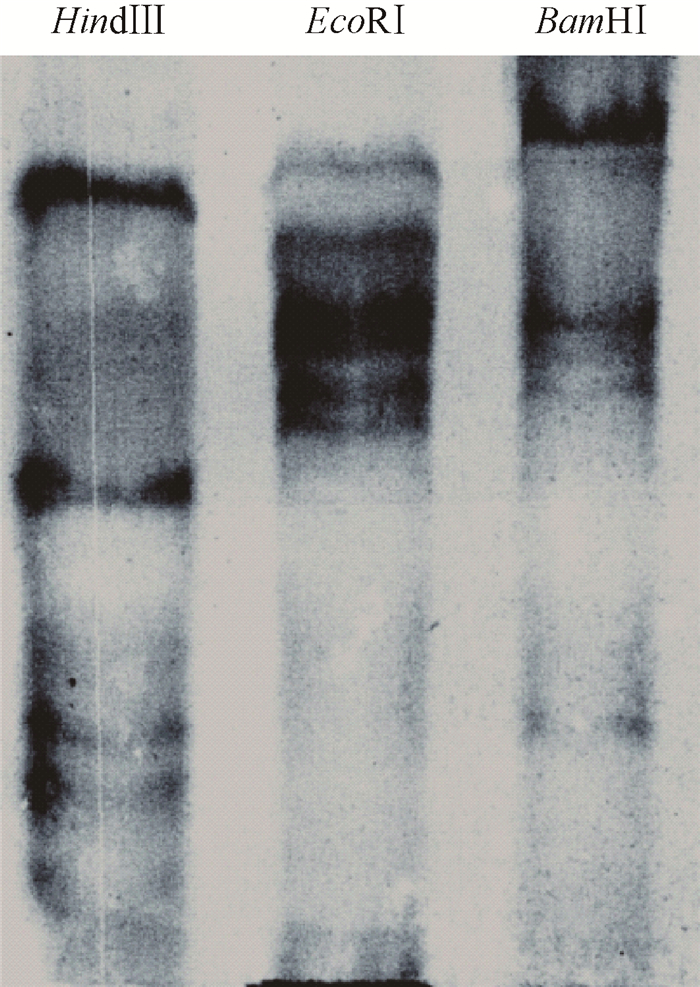
2.3 马占相思基因组中AmCesA1的Southern分析为了确定AmCesA1在马占相思基因组中的拷贝数目,在HVR-1区域设计了2条引物,以马占相思基因组作为模板进行了探针标记。AmCesA1的PCR产物大小为918 bp,测序后表明此段有2个内含子插入:以它作为探针,对马占相思基因组进行杂交。基因组消化所用的酶是BamHⅠ, EcoRⅠ, HindⅢ,杂交结果见图 4。从图中可以看到AmCesA1每个泳道都有5~6条条带且亮度比较接近,因所用探针为含有内含子的高度可变区序列,所以应该不会存在交叉杂交的现象,从而推测AmCesA1在马占相思基因组中有多个拷贝。
|
图 4 AmCesA1拷贝数分析 Figure 4 The analysis of AmCesA1 copy number |
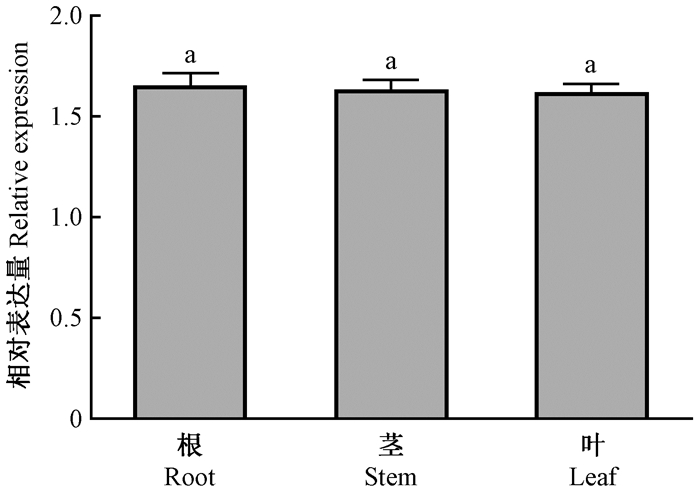
以Actin为内参,利用实时荧光定量PCR检测AmCesA1在根、茎、叶中的表达情况,结果(图 5)发现AmCesA1在根茎叶中均有表达,表达丰度之间没有显著差异。
|
图 5 AmCesA1在马占相思各器官中的相对表达量 Figure 5 Expression analysis of AmCesA1 in different organs of Acacia mangium |
为了探究AmCesA1对激素的响应情况,采用GA3、6-BA、茉莉酸甲酯(MeJA)喷施马占相思叶片并进行q-PCR分析。结果(图 6)显示,随着激素喷施次数的增加,AmCesA1的表达量也随之增加,即在喷施的第3次达到表达量的最大值。3种激素均会使AmCesA1的表达量上调,其中GA3的效果最显著,MeJA的效果相对较差,而6-BA的效果趋于这2种激素之间。在3组处理中除了MeJA在4 h喷施与对照组没有显著差异外,其余处理所造成的AmCesA1基因表达的上调均为显著水平。3种激素均促进基因表达,这也表明AmCesA1参与马占相思对激素应答的正调控。
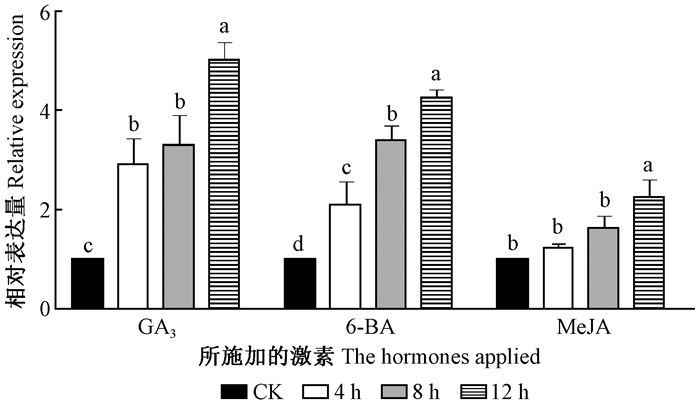
|
图 6 不同激素处理4、8、12 h后马占相思AmCesA1基因的表达情况 Figure 6 The response of AmCesA1 gene of Acacia mangium to 4, 8, 12 h hormone treatment |
纤维素是地球上最丰富的可再生资源,而且是绿色的化工原材料。对植物细胞壁碳水化合物合成途径的研究和主要成分纤维素合成途径的探索对木质纤维素的利用具有极其重要的作用(Mizrachi et al., 2012)。
植物的CesA基因通过超基因家族的形式存在,不同成员之间参与不同组织、器官或细胞壁层次纤维素的合成(庞景等,2015)。通过对CesA基因功能的研究,已陆续鉴定出与初生壁和次生壁形成相关的基因并且基因之间的冗余功能也进一步被揭示(Carroll et al., 2012)。近年来在纤维素合酶晶体结构上有所突破,包括证实球形红细菌(Rhodobacter sphaeroides)中细菌纤维素合酶亚基BcsA具有8个跨膜螺旋(TMHs),细菌纤维素合酶亚基BcsB具有1个跨膜螺旋。纤维素的合成发生在BcsA的胞质结构域,BcsB存在于周质中可能引导链的拉伸过程等,该偶联模型也得到了体外重组试验的验证(Omadjela et al., 2013)。但关于纤维素合成组装是否需要关键蛋白的参与尚不明确,对植物纤维素合成的精细调控研究还需要进一步深入。
本研究采用RT-PCR和5′RACE相结合的技术,克隆了1个马占相思纤维素合酶基因AmCesA1。 AmCesA1具有纤维素合酶家族的保守序列区D, D, D, QxxRW,含有植物高度保守区和可变区(P-CR,HVR区),而且在C末端具有纤维素合酶家族的典型的6个跨膜区域(Saxena et al., 2000)。一般情况下植物纤维素合酶共有8个跨膜结构域,2个在N端,6个在C端。跨膜结构域会在细胞质膜上形成β-1, 4葡萄糖苷链穿过细胞壁的重要通道(Harris et al., 2012)。在BcsA中,除了跨膜螺旋1和2,其他跨膜螺旋3-8均直接参与葡聚糖链的延伸,并已证实跨膜螺旋3-8形成的通道可容纳10个葡萄糖单位的转运葡聚糖(Morgan et al., 2013)。目前,N端跨膜螺旋所具有的特殊作用还不明确,本研究中AmCesA1在N端跨膜结构不明显,这种差异是否会对纤维素合酶的功能造成影响,还有待进一步研究。
从同源性分析结果来看,马占相思AmCesA1与GmCesA1、AdCesA1相近。从uniprot数据库得知,大豆GmCesA1和蔓花生AdCesA1均与纤维素生物合成有关并参与初生壁的合成,结构相似常常具有相似的生物功能(Pear et al., 1996;陈英等, 2015),因此推测AmCesA1也参与初生壁的合成。此外,AmCesA1在与拟南芥AtCesA1和AtCesA10比较中发现一致性为86.18%和80.29%,而拟南芥中,AtCesA1和AtCesA10都参与初级细胞壁的形成,从而推测 AmCesA1参与马占相思初生细胞壁的形成。
植物中许多生长发育过程均由不同植物激素介导,植物体对激素的响应最终会在细胞壁的结构和成分中表现出来。本研究荧光定量PCR结果显示AmCesA1在激素的诱导下均出现不同程度的上调表达,其中GA3上调幅度最大,另外2种激素表达量相对较低。说明GA3在AmCesA1激素通路中的主导地位。虽然激素的施加使AmCesA1表达上调从而增加纤维素的含量,但激素对AmCesA1的促进是直接过程还是间接行为,目前尚不明确。已证实合成和信号转导植物激素的关键基因突变都会造成细胞壁相关基因表达下调,例如,拟南芥中gal-3和gai突变体中果胶甲酯化程度降低和醛酸糖含量下降(Debyshire et al., 2007)。与油菜素内酯(BR)合成相关的蛋白DIM1基因功能缺失造成拟南芥植株矮化,木质素和纤维素含量分别下降38%和23%(Hossain et al., 2012)。另一方面,植物细胞壁的突变体经常表现出激素感知或合成异常。例如,拟南芥CesA3突变后造成茉莉酸和乙烯合成途径的多个基因上调表达;拟南芥CesA1突变体rsw1-1和CesA6突变体prc1-1中对茉莉酸响应基因的表达量上升(Ellis et al., 2002)。但是,这种关联被证实是直接作用的少之又少。本研究中几种激素之间是如何协调的,各个调控通路中各元件的承接关系都需要进一步研究。
4 结论本研究克隆到的马占相思AmCesA1为植物CesA家族中的一员,它在马占相思基因组中以多拷贝形式存在,且广泛表达于根、茎、叶。推测其参与初级细胞壁的形成。该基因对赤霉素、6-BA、茉莉酸甲酯处理均有响应,其中基因的表达量在不同的激素处理中都有不同程度的上调,表明该基因参与马占相思对激素应答的正调控。3种激素中,以对GA3的应答最为强烈。
陈英, 邵志龙, 王浩然, 等. 2015. 杨树PeAFB基因克隆及表达模式初步分析[J]. 林业科学, 5(8): 26-32. (Chen Y, Shao Z L, Wang H R, et al. 2015. Cloning and expression of PeAFB genes in Populus[J]. Scientia Silvae Sinicae, 5(8): 26-32. [in Chinese]) |
李晶, 王亦学, 郑德刚, 等. 2009. 一种简单高效提取棉花不同组织总RNA的方法[J]. 山西农业科学, 37(5): 17-19. (Li J, Wang Y X, Zheng D G, et al. 2009. A simply and high efficiency method of total RNA isolation in upland cotton[J]. Journal of Shanxi Agricultural Sciences, 37(5): 17-19. [in Chinese]) |
刘昱翔, 陈建荣, 彭彦, 等. 2014. 苎麻纤维素合成酶基因BnCesA4 cDNA序列的克隆与表达分析[J]. 作物研究, 28(5): 472-478. (Liu Y X, Chen J R, Peng Y, et al. 2014. The cDNA cloning and expression analysis on cellulose synthase BnCesA4 in Boehmeria nivea[J]. Crop Research, 28(5): 472-478. [in Chinese]) |
庞景, 童再康, 黄华宏, 等. 2015. 杉木纤维素合成酶基因CesA的克隆及表达分析[J]. 浙江农林大学学报, 32(1): 40-46. (Pang J, Tong Z K, Huang H H, et al. 2015. Isolation and expression analysis of cellulose synthase genes in Chinese fir (Cunninghamia lanceolata)[J]. Journal of Zhejiang A & F University, 32(1): 40-46. DOI:10.11833/j.issn.2095-0756.2015.01.006 [in Chinese]) |
阮维程, 潘婷, 季孔庶. 2015. 马尾松纤维素合成酶基因PmCesA1的克隆及其分析[J]. 分子植物育种, 13(4): 861-870. (Ruan W C, Pan T, Ji K S. 2015. Cloning and analysis on PmCesA1 gene encoding Pinus massoniana cellulose synthase[J]. Molecular Plant Breeding, 13(4): 861-870. [in Chinese]) |
魏凯莉, 周厚君, 江成, 等. 2017. 杨树次生壁纤维素合酶的表达与互作模式分析[J]. 林业科学研究, 30(2): 245-253. (Wei K L, Zhou H J, Jiang C, et al. 2017. Interaction and expression of secondary wall CESAs in Populus[J]. Forest Research, 30(2): 245-253. [in Chinese]) |
许雷, 刘一星, 方连玉. 2012. 大青杨纤维素合成酶PuCesA6基因cDNA的克隆及序列分析[J]. 西南林业大学学报, 32(5): 26-32. (Xu L, Liu Y X, Fang L Y. 2012. Cloning and sequence character analysis of full-length cDNA of cellulose synthase PuCesA6 from Populus ussuriensis[J]. Journal of Southwest Forestry University, 32(5): 26-32. [in Chinese]) |
张保才, 周奕华. 2015. 植物细胞壁形成机制的新进展[J]. 中国科学:生命科学, 45(6): 544-556. (Zhang B C, Zhou Y H. 2015. New Advances in the mechanism of plant cell wall formation[J]. Scientia Sinica Vitae, 45(6): 544-556. [in Chinese]) |
Arioli T, Peng L C, Betzner A S, et al. 1998. Molecular analysis of cellulose biosynthesis in Arabidopsis[J]. Science, 279(5351): 717-720. DOI:10.1126/science.279.5351.717 |
Carroll A, Mansoori N, Li S, et al. 2012. Complexes with mixed primary and secondary cellulose synthases are functional in Arabidopsis plants[J]. Plant Physiol, 160(2): 726-737. DOI:10.1104/pp.112.199208 |
Derbyshire P, Mccann M C, Roberts K. 2007. Restricted cell elongation in Arabidopsis hupocatyls is associated with a reduced average pectin esterification level[J]. BMC Plant Biol, 7(1): 31. DOI:10.1186/1471-2229-7-31 |
Ellis C, Karafyllidis I, Wasternack C, et al. 2002. The Arabidopsis mutant cevl links cell wall signaling to jasmonate and ethylene responses[J]. Plant Cell, 14(7): 1557-1566. DOI:10.1105/tpc.002022 |
Fagard M, Desnos T, Desprez T, et al. 2000. PROCUSTE1 encodes a cellulose synthase required for normal cell elongation specifically in roots and dark-grown hypocotyls of Arabidopsis[J]. Plant Cell, 12(12): 2409-2424. DOI:10.1105/tpc.12.12.2409 |
Gonneau M, Desprez T, Guillot A, et al. 2014. Catalytic subunit stoichiometry within the cellulose synthase complex[J]. Plant Physiol, 166(4): 1709-1712. DOI:10.1104/pp.114.250159 |
Harris D M, Corbin K, Wang T, et al. 2012. Cellulose microfibril crystallinity is reduced by mutating C-terminal transmembrane region residuces CESA1A903V and CESA3T9421 of cellulose synthase[J]. Proc Natl Acad Sci USA, 109(11): 4098-4103. DOI:10.1073/pnas.1200352109 |
Hill J L Jr, Hammudi M B, Tien M. 2014. The Arabidopsis cellulose synthase complex:a proposed hexamer of CESA trimers in an equimolar stoichipmetry[J]. Plant Cell, 26(12): 4834-4842. DOI:10.1105/tpc.114.131193 |
Hossain Z, Mcgarvey B, Amyot L, et al. 2012. DIMINUTO1 affects the lignin profile and secondary cell wall formation in Arabidopsis[J]. Planta, 235(3): 485-498. DOI:10.1007/s00425-011-1519-4 |
Mizrachi E, Mansfield S D, Myburg A A. 2012. Cellulose factories:advancing bioenergy production from forest trees[J]. New Phytol, 194(1): 54-62. DOI:10.1111/j.1469-8137.2011.03971.x |
Morgan J L, Strumillo J, Zimmer J. 2013. Crystallographic snapshot of cellulose synthesis and menmbrane translocation[J]. Nature, 493(7431): 181-186. |
Omadjela O, Narahari A, Strumillo J, et al. 2013. BcsA and BcsB form the catalytically active core of bacterial cellulose synthase sufficient for in vitro cellulose synthesis[J]. Proc Natl Acad Sci USA, 110(44): 17856-17861. DOI:10.1073/pnas.1314063110 |
Pear J R, Kawagoe Y, Schreckengost W E, et al. 1996. Higher plants contain homologs of the bacterial CelA genes encoding the catalytic subunit of cellulose synthase[J]. Proc Natl Acad Sci USA, 93(22): 12637-12642. DOI:10.1073/pnas.93.22.12637 |
Saxena I M, Brown R M Jr. 2000. Cellulose synthases and related enzymes[J]. Current Opinion in Plant Biology, 3(6): 523-531. DOI:10.1016/S1369-5266(00)00125-4 |
Scheible W R, Eshed R, Richmond T, et al. 2001. Modifications of cellulose synthase confer resistance to isoxaben and thiazolidinone herbicides in Arabidopsis Ixr1 mutants[J]. Proc Natl Acad Sci USA, 98(18): 10079-10084. DOI:10.1073/pnas.191361598 |
Ong S S, Wickneswari R. 2012. Characterization of microRNAs expressed during secondary wall biosynthesis in Acacia mangium[J]. PLoS ONE, 7(11): e49662. DOI:10.1371/journal.pone.0049662 |
Sethapphong L, Davis J K, Slabaugh E, et al. 2016. Prediction of the structures of the plant specific regions of vascular plant cellulose synthases and correlated functional analysis[J]. Cellulose, 23(1): 145-161. DOI:10.1007/s10570-015-0789-6 |
Song D, Shen J, Li L. 2010. Characterization of cellulose synthase complexes in Populus xylem differentiation[J]. New Phytologist, 187(3): 777-790. DOI:10.1111/j.1469-8137.2010.03315.x |
Taylor N G. 2008. Cellulose biosynthesis and deposition in higher plants[J]. New Phytologist, 178(2): 239-252. DOI:10.1111/j.1469-8137.2008.02385.x |
Taylor N G, Laurie S, Turner S R. 2000. Multiple cellulose synthase catalytic subunits are required for cellulose synthesis in Arabidopsis[J]. Plant Cell, 12(12): 2529-2539. DOI:10.1105/tpc.12.12.2529 |
Taylor N G, Scheible W R, Cutler S, et al. 1999. The irregular xylem3 locus of Arabidopsis encodes a cellulose synthase required for secondary cell wall synthesis[J]. Plant Cell, 11(5): 769-780. DOI:10.1105/tpc.11.5.769 |
Turner S R, Somerville C R. 1997. Collapsed xylem phenotype of Arabidopsis identifies mutants deficient in cellulose deposition in the secondary cell wall[J]. Plant Cell, 9(5): 689-701. DOI:10.1105/tpc.9.5.689 |
Wu L, Joshi C P. 2000. A xylem-specific cellulose synthase gene from aspen (Populus tremuloides) is responsive to mechanical stress[J]. The Plant Journal, 22(6): 495-502. DOI:10.1046/j.1365-313x.2000.00758.x |
Xu P, Donaldson L, Gergely Z, et al. 2007. Dual-axis electron tomography:a new approach for investigating the spatial organization of wood cellulose microfibrils[J]. Wood Sci Technol, 41(2): 101-116. DOI:10.1007/s00226-006-0088-3 |
 2018, Vol. 54
2018, Vol. 54

