文章信息
- 金玲, 刘明国, 董胜君, 吴月亮, 张欣
- Jin Ling, Liu Mingguo, Dong Shengjun, Wu Yueliang, Zhang Xin
- 97个山杏无性系的遗传多样性及SSR指纹图谱
- Genetic Diversity and Fingerprints of 97 Armeniaca sibirica Clones Based on SSR Markers
- 林业科学, 2018, 54(7): 51-61.
- Scientia Silvae Sinicae, 2018, 54(7): 51-61.
- DOI: 10.11707/j.1001-7488.20180706
-
文章历史
- 收稿日期:2017-07-26
- 修回日期:2017-12-18
-
作者相关文章
山杏(Armeniaca sibirica)是北方半干旱地区重要的生态经济型树种, 耐干旱瘠薄, 适应性强, 防风固沙、保持水土性能好, 经济价值高, 已成为产区群众脱贫致富的重要产业。杏核产量低而不稳的问题为山杏产业发展的主要瓶颈, 由于该树种长期处于野生、半野生状态, 加之自交不亲和, 种间杂交现象普遍, 种质资源异常丰富, 为丰产稳产新品种选育提供了物质基础, 但另一方面也为其种质识别增加了难度(刘明国等, 2015a)。
近年来, 山杏种质资源发掘及育种工作取得了较大进展, 已选育出一批优良无性系(刘明国等, 2015b), ‘山杏1号’、‘山杏2号’等新品种也相继通过省级审定(董胜君等, 2016)。但是这些无性系及品种间的差异往往只体现在形态性状和经济性状的某一方面, 其中的数量性状存在连续变异, 易受培育措施和环境条件的影响, 花、果方面的性状受发育阶段影响, 而抗逆性生理特性方面的指标观测并不方便。尤其是山杏种质在苗期一般不具备可用于识别的形态特征, 难以鉴别品种的真伪。所以单纯依靠形态特征已经不能满足现今山杏种质鉴定的要求, 给山杏良种选育、推广应用及知识产权保护增加了难度, 如何对山杏种质进行准确、快速鉴定成为当务之急。
DNA指纹图谱为林木种质鉴别的一种有效工具, 具有高效、准确、经济、便捷、不受季节和环境影响等优点(Rocha et al., 2002;Liu et al., 2016), 其中SSR分子标记是目前最为成熟的技术之一。当前, SSR指纹图谱的研究在果树树种上比较活跃(王立新等, 2012;高源等, 2012;李雄伟等, 2013), 在林业上集中于主要经济林树种(杨本芸等, 2008;杨阳等, 2010;周文才等, 2017)和一些用材林树种(张亚东等, 2009;沈敬理等, 2015)的研究。如, 贾会霞等(2015)利用TP-M13-SSR技术对24份杨树(Populus)种质进行指纹图谱构建和系谱关系研究, 筛选出3对可快速鉴别杨树种质的引物, 证实用SSR标记可有效地检测亲本与子代之间的系谱关系, 并准确地反映植物的倍性; 麻丽颖等(2012)采用荧光M13毛细管电泳技术, 依据12对SSR引物, 构建了36份枣(Ziziphus jujuba)品种指纹图谱; 包文泉等(2017)利用9对来自桃(Amygdalus persica)基因组DNA的SSR引物, 建立了16份仁用杏(Armeniaca vulgaris×sibirica)品种的指纹图谱, 并进行了遗传多样性评价和亲缘关系分析。但到目前为止, 有关山杏DNA指纹图谱构建及种质鉴定的研究鲜见报道。本研究以97个山杏优选无性系为试验材料, 开展SSR标记的遗传多样性和指纹图谱的构建研究, 旨在为其种质鉴别及良种选育提供科学依据。
1 材料与方法 1.1 试验材料试验材料为课题组前期初选的97个山杏无性系, 包括丰产、晚花、抗冻、抗旱、甜仁、甜肉等优特类型和其他变异类型(表 1)。其中, 辽宁喀左县、朝阳县、北票市及内蒙古敖汉旗的无性系选育始于2000年, 内蒙古扎兰屯市的无性系选育始于2010年, 基于优树选择(包括初选、复选、决选)、嫁接无性系化, 通过苗期适应性试验和资源圃中形态性状、生产性能观测试验, 筛选优良无性系。2011年通过合作研究于俄罗斯外贝加尔边疆区进行山杏优树初选, 2012年引种并开展优良无性系筛选试验。94号无性系由俄罗斯季米里亚泽夫农学院专家提供。上述97个无性系现保存于北票市林木良种繁育中心山杏种质资源圃和喀左县山杏国家林木种质资源保存库中。
|
|
6月中旬, 采集供试无性系向阳面1年生枝条上的叶片, 放入液氮罐中速冻带回实验室, 置于-80 ℃超低温冰箱中备用。
1.2 试验方法 1.2.1 DNA提取选用DNAsecure Plant Kit新型植物基因组DNA提取试剂盒进行基因组DNA的提取。
1.2.2 SSR引物筛选根据沈阳农业大学山杏课题组2014年进行的山杏简化基因组测序结果, 由北京赛百盛基因技术有限公司合成600对SSR引物, 选择4个山杏无性系(505、511、354、366)的DNA作为模板进行SSR引物筛选。
1.2.3 PCR反应体系及扩增程序PCR总反应体系为20 μL, 含DNA模板量20 ng、引物浓度0.15 μmol·L-1、Mg2+ 2.0 mmol·L-1、Taq聚合酶量1.0 U、dNTPs 0.25 mmol·L-1。PCR反应程序如下:105 ℃热盖; 94 ℃预变性5 min; 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸30 s, 共进行34个循环; 72 ℃后延伸5 min; 4 ℃保存。
1.2.4 聚丙烯酰胺凝胶电泳检测通过预试验, 本研究采用12%非变性聚丙烯酰胺凝胶电泳进行SSR-PCR产物检测, 电压设定为220 V, 电泳90 min。然后经过漂洗、银染、显影等步骤后, 置于凝胶成像系统(BIO-RAD, USA)中拍照记录。对于试验中出现的片段大小差异较小的扩增条带, 为了确认其真实存在, 采用不同胶浓度(10%和12%)进行PCR扩增验证, 如重复性好, 则认定为不同的扩增产物。
1.2.5 数据读取与分析首先通过软件Imagelab 4.0, 调取数据化模块, 依据100 bp DNA Marker(TIANGEN)计算出各位点扩增片段的大小, 再依据SSR重复基元大小进行人工矫正, 扩增片段大小差异多于1个重复基元的认定为不同等位基因(文自翔等, 2008)。最终导入软件DATAtrans2.0转换为各种分析软件所要求的格式。
采用Cervus3.0软件计算位点的多态性信息含量(PIC); 按照Nei等(1979)方法计算无性系间的遗传相似系数(GS):GS=2Nij/(Ni+Nj), 式中:Ni和Nj为i和j无性系的谱带数, Nij为i和j无性系共有的谱带数。基于遗传相似系数, 利用NTSYS 2.1软件, 采用UPGMA法对无性系进行聚类分析。
2 结果与分析 2.1 山杏无性系SSR多态性分析从合成的600对引物中筛选出155对扩增条带清晰的引物, 对97个山杏无性系的基因组DNA进行扩增, 共检测到933个等位基因, 图 1为引物Y50对20个山杏无性系的扩增图谱。由表 2可知, 每个位点的等位基因数为3~11个, 平均为6.019个。其中位点X23H、X44H的等位基因数最多(11个); 位点X99、X42H、L62、X100、L80、Y75、P57H、Y38、X61、P17、L39的等位基因数最少(3个)。155个位点的多态性信息含量(PIC)为0.476~0.885, 平均为0.681。说明此155个位点的多态性较高。
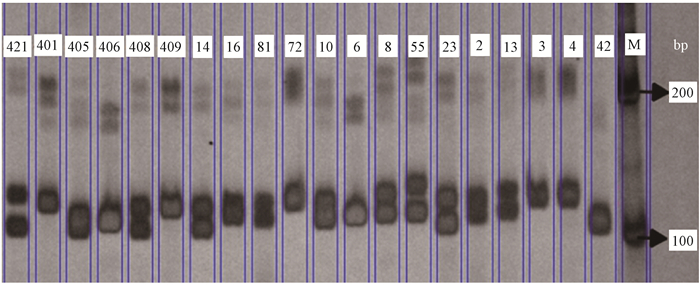
|
图 1 引物Y50对20个山杏无性系的SSR扩增 Figure 1 SSR amplification of 20 Armeniaca sibirica clones with primer Y50 M:Marker.421, 401, 405, …, 42:无性系号Clone codes. |
|
|
采用155个位点对97个山杏无性系进行指纹分析, 结果如表 3所示。由表 3可知, 50个无性系在59个位点上具有特异等位基因, 换言之, 对于50个无性系中的每一个, 仅用1个特异等位基因即可将之与其他无性系区分开。其中, 1号等34个无性系具有1个特异等位基因, 8号等11个无性系具有2个特异等位基因, 22号等5个无性系具有3个特异等位基因。
|
|
由表 3可知, 89个无性系在131个位点上具有特异基因型, 即用1个单位点特异基因型可区分某个无性系。其中, 32号等11个无性系具有1个特异基因型, 72号等11个无性系具有2个特异基因型, 10号等14个无性系具有3个特异基因型, 6号等19个无性系具有4个特异基因型, 1号等34个无性系具有5个及以上特异基因型。
上述分析表明, 特征谱带法可鉴别89个山杏无性系, 其中50个无性系既可用特异等位基因又可用特异基因型鉴别, 39个无性系只有特异基因型无特异等位基因, 尚有8个无性系不能鉴别。
2.2.2 引物组合法特征谱带法无法将所有97个山杏无性系完全区分开, 需采用不同引物扩增谱带的组合方法来进行鉴别。本着采用的引物尽量少、其扩增位点等位基因间比较容易分辨的原则, 根据本研究的实际情况, 将等位基因片段大小差异大于等于4个bp的位点按照PIC大小进行排序, 依次用前1、2、3……个位点进行遗传相似性聚类分析, 结果显示当位点数量为5时, 能够区分全部97个无性系(图 2)。所以选择5个位点(L56、X47H、L79H、P40H和X47)的SSR多态性数据构建山杏无性系指纹图谱(表 4), 5对引物的基本信息见表 5。

|
图 2 位点数与鉴别无性系数量的关系 Figure 2 The relationship between number of loci and number of identified clones |
|
|
|
|
上述研究表明, 从等位基因片段大小差异大于等于4个bp的位点中选择PIC值最大的5个位点, 其谱带组合所构建的指纹图谱可以将97个山杏无性系完全鉴别开来。之所以优先选择PIC值大的位点, 是因为PIC越大, 鉴别力越强(Smith et al., 1997)。
2.3 山杏无性系亲缘关系分析基于155个SSR位点数据, 利用NTSYS软件分析97个山杏无性系间的遗传相似性, 结果表明, 供试无性系间的遗传相似系数在0.669~0.943之间, 平均为0.757, 遗传差异较小。其中来源于内蒙古扎兰屯市的368号无性系与来源于辽宁北票市的71号无性系相似系数最小, 为0.669, 遗传差异最大; 407号与368号无性系相似系数最大, 为0.943, 遗传差异最小, 二者均来源于内蒙古扎兰屯市。
以遗传相似系数为基础的聚类结果表明, 在相似系数0.758处可以分为5大类(图 3)。

|
图 3 97个无性系的聚类结果 Figure 3 Clustering result of 97 clones |
第1大类包括25个无性系, 在相似系数0.806处又分为3个亚类。第1亚类包括辽宁朝阳市的12个无性系(1、18、19、71、21、41、46、49、53、62、11、22)和内蒙古敖汉旗的1个无性系(37);第2亚类包括内蒙古敖汉旗的9个无性系(25、31、32、27、28、33、34、35、39);第3亚类包括内蒙古扎兰屯市的3个无性系(322、328、324)。内蒙古敖汉旗与辽宁朝阳市地理位置相邻, 气候相近, 种质间相似系数较大, 所以两地的无性系聚为一大类。3个扎兰屯市的无性系位于该市大河湾镇的山杏人工林中, 其苗木种子来源于辽宁朝阳市。所以第1大类无性系来源地可概括为辽宁朝阳-内蒙古敖汉区, 处于研究区域的南部, 所属全部无性系均具丰产特性。
第2大类包括23个无性系, 在相似系数0.8处再分为3个亚类。第1亚类包括来源于内蒙古扎兰屯市的7个无性系(334、339、345、358、341、350、354)和来源于俄罗斯外贝加尔边疆区的1个无性系(516), 均具丰产特性; 第2亚类包括俄罗斯外贝加尔边疆区的13个无性系(501、502、504、503、505、506、507、508、509、510、513、517、518), 均具抗旱特性; 第3亚类包括内蒙古扎兰屯市的2个无性系(381、404), 其中1个为晚熟、丰产无性系, 1个为晚花无性系。内蒙古扎兰屯市和俄罗斯外贝加尔边疆区处于研究区域的北部, 均为低温寒冷地区, 第2大类的无性系总体看为丰产类型和抗旱类型。
第3大类包括23个无性系, 全部来源于辽宁朝阳市(2、3、4、23、55、6、8、13、14、10、81、72、16、42、43、44、48、87、89、99、BX、95、BF)。该类包括11个丰产无性系和12个变异类型无性系(甜肉、甜仁、双仁、褶皱花、香花、粉花)。
第4大类包括22个无性系, 其中20个来源于内蒙古扎兰屯市(442、443、449、455、457、459、460、462、463、464、332、367、375、366、401、405、406、421、408、409), 1个来源于俄罗斯外贝加尔边疆区(511), 1个来源于俄罗斯季米里亚泽夫农学院(94)。该类以变异类型无性系为主, 包括6个丰产无性系及16个变异类型无性系(曲枝、双雌蕊、大花、粉色花药、粉花、晚花、红果、长果、甜肉)。
第5大类包括4个无性系, 3个来源于内蒙古扎兰屯市(329、368、407), 1个来源于辽宁朝阳市(20)。该类包含3个丰产无性系及1个晚花无性系。
3 讨论科学有效的种质鉴定是育种及良种推广应用成败的关键, 山杏种质资源异常丰富, 鉴定与分类难度大。目前对山杏种质鉴定与分类研究主要限于形态标记(李明等, 2011a;马发旺等, 2013;尹健等, 2015)、蛋白质和酶标记(刘明国等, 2006;刘青华等, 2007;李明等, 2011b)以及孢粉学(刘明国等, 2015a)等方面, 但形态标记方法易受发育周期和环境影响; 蛋白质和酶标记方法能检测到的位点较少, 蛋白质种类不多, 多态性不高, 难以有效区分亲缘关系密切的种质, 同时谱带特征也受发育条件以及环境条件的影响; 利用花粉形态特征鉴别种质的缺陷是采集花粉的季节性限制, 并且种质间花粉形态特征往往无明显差异。本文基于5对SSR引物扩增位点构建了97个山杏优选无性系指纹图谱, 具有稳定、可靠、不受环境条件及发育时期限制的优点, 可实现山杏种质的快速、准确鉴定。
根据山杏简化基因组测序结果, 合成600对SSR引物, 从中筛选出155对扩增条带清晰的引物, 各位点等位基因数平均值为6.019, 这与Zhebentyayeva等(2003)对74个山杏单株扩增得到的7.64个等位基因数, 及章秋平等(2013)对67份华北生态群普通杏(Armeniaca vulgaris)扩增时得到的5.7个等位基因数相近。在155个位点中, 有149个位点PIC﹥0.5, 6个位点的PIC在0.476~0.495之间, 多态性较高; 山杏无性系间遗传相似系数平均值为0.757, 遗传差异较小, 亲缘关系较近, 与刘娟等(2015)、包文泉等(2017)研究结果相近。基于相似系数的聚类分析结果与无性系的来源区域有明显的相关性, 这与无性系来源地环境条件的差异有关。供试山杏无性系来源于辽宁朝阳-内蒙古敖汉、内蒙古扎兰屯、俄罗斯外贝加尔边疆区3个区域, 三地的年均气温分别为7~10 ℃、2.4 ℃和0 ℃以下, 降雨量分别为400~600 mm、500 mm和320 mm, 由于长期的自然选择, 来源于不同区域的无性系形成了相应的遗传特性。
本文采用特征谱带法和引物组合法开展了山杏无性系指纹图谱的构建研究。特征谱带法在鉴别种质时仅需用1对引物, 简单方便, 成本低, 实用价值大。但是, 并不是所有的种质都有特征谱带, 本研究通过155对引物的扩增, 才发现89个无性系的特异基因型。若想获取其他无性系的特征谱带, 还需要进行大量的引物筛选工作。并且特征谱带是相对的, 随着供试材料的扩大, 某一材料的特征谱带有可能出现在其他材料上。所以特征谱带法往往只有在固定的材料范围内有效, 鉴别能力相对有限(王凤格等, 2003)。引物组合法通过选择PIC高的若干位点的有限组合, 可以最少的引物来区分最多的品种(王凤格等, 2003;杨阳等, 2010)。引物组合法大大提高了种质鉴别能力, 本研究中, 采用5个等位基因片段大小差异大于等于4 bp的位点组合将97个山杏无性系完全区分开。在山杏种质资源发掘及良种选育工作中, 特征谱带法更适用数量不大的育成品种或育种亲本的真伪鉴定, 而引物组合法则适用于大规模林木种质资源的识别分类。
本研究采用引物组合法构建的山杏指纹图谱既具有相对固定性, 也具有可扩展性。随着山杏种质数量的进一步增加, 这5个位点组合的鉴别能力可能会逐渐减低, 如果出现相同的DNA指纹, 则需对指纹相同的种质从已筛选确定的155对SSR引物中再进行引物选择, 将新位点的扩增结果补充到现有DNA指纹图谱中。
4 结论本文开展了基于SSR标记的山杏优选无性系的遗传多样性及指纹图谱构建的研究, 筛选出155对SSR引物, 其扩增位点多态性较高; 研究发现89个山杏无性系具有特异基因型, 其中50个既有特异基因型也有特异等位基因, 采用5对引物扩增谱带的组合构建了97个山杏无性系指纹图谱, 为该树种种质鉴别提供了有效方法; 揭示了山杏无性系间的亲缘关系, 将97个山杏无性系分为5大类, 为杂交育种的亲本选配提供了科学依据。
包文泉, 乌云塔娜, 赵罕, 等. 2017. 基于SSR标记的仁用杏主栽品种鉴别和指纹图谱构建[J]. 西北农林科技大学学报:自然科学版, 45(6): 163-169. (Bao W Q, Wuyun T N, Zhao H, et al. 2017. Identification and fingerprinting construction of accessions of kernel-using apricot by SSR markers[J]. Journal of Northwest A & F University:Natural Science Edition, 45(6): 163-169. [in Chinese]) |
董胜君, 尹健, 刘明国, 等. 2016. 山杏新品种光合生理特性研究[J]. 经济林研究, 34(2): 67-72. (Dong S J, Yin J, Liu M G, et al. 2016. Research on photosynthetic physiological characteristics of new varieties of Armeniaca sibirica[J]. Nonwood Forest Research, 34(2): 67-72. [in Chinese]) |
高源, 田路明, 刘凤之, 等. 2012. 利用SSR荧光标记构建92个梨品种指纹图谱[J]. 园艺学报, 39(8): 1437-1446. (Gao Y, Tian L M, Liu F Z, et al. 2012. Using the SSR fluorescent labeling to establish SSR fingerprints for 92 cultivars in Pyrus[J]. Acta Horticulturae Sinica, 39(8): 1437-1446. [in Chinese]) |
贾会霞, 姬慧娟, 胡建军, 等. 2015. 杨树新品种的SSR指纹图谱构建和倍性检测[J]. 林业科学, 51(2): 69-79. (Jia H X, Ji H J, Hu J J, et al. 2015. Fingerprints of SSR markers and ploidy detection for new Populus varieties[J]. Scientia Silvae Sinicae, 51(2): 69-79. [in Chinese]) |
李明, 赵忠, 杨吉安, 等. 2011a. 黄土高原山杏种质资源分类研究[J]. 西北林学院学报, 26(1): 8-12. (Li M, Zhao Z, Yang J A, et al. 2011a. Classification on germplasm resources of Armeniaca sibirica in the Loess Plateau[J]. Journal of Northwest Forestry University, 26(1): 8-12. [in Chinese]) |
李明, 赵忠, 杨吉安, 等. 2011b. 黄土高原不同县域山杏种质遗传多样性研究[J]. 西北农林科技大学学报:自然科学版, 39(2): 143-149, 156. (Li M, Zhao Z, Yang J A, et al. 2011b. Genetic diversity analysis on germplasm of Armeniaca sibirica in different counties in Loess Plateau[J]. Journal of Northwest A & F University:Natural Science Edition, 39(2): 143-149, 156. [in Chinese]) |
李雄伟, 孟宪桥, 贾惠娟, 等. 2013. 桃品种特异性荧光SSR分子标记数据库构建[J]. 果树学报, 30(6): 924-932. (Li X W, Meng X Q, Jia H J, et al. 2013. Construction of peach genotype database with fluorescent-labeled SSR markers[J]. Journal of Fruit Science, 30(6): 924-932. [in Chinese]) |
刘娟, 廖康, 曼苏尔·那斯尔, 等. 2015. 新疆杏品种(系)遗传多样性分析及DNA指纹图谱构建. 中国农业科学, 48(4): 748-758. (Liu J, Liao K, Mansuer N, et al. 2015. Analysis of genetic diversity and construction of DNA fingerprint database of Xinjiang apricot varieties(lines). Scientia Agricultura Sinica, 48(4): 748-758. [in Chinese]) |
刘明国, 李民, 吴月亮, 等. 2015a. 山杏花粉形态特征与花粉萌发的关系[J]. 沈阳农业大学学报, 46(2): 166-172. (Liu M G, Li M, Wu Y L, et al. 2015a. Study on pollen morphological characteristics and its relationship with pollen germination in Armeniaca sibirica[J]. Journal of Shenyang Agricultural University, 46(2): 166-172. [in Chinese]) |
刘明国, 张欣, 董胜君, 等. 2015b. 西伯利亚杏优选无性系间数量性状的差异与重复力研究[J]. 沈阳农业大学学报, 46(5): 548-554. (Liu M G, Zhang X, Dong S J, et al. 2015b. Differences and repeatabilities of quantitative characters of Prunus sibirica superior clones[J]. Journal of Shenyang Agricultural University, 46(5): 548-554. [in Chinese]) |
刘明国, 赵桂玲, 董胜君. 2006. 山杏种内POD同工酶及种子可溶性蛋白分析[J]. 沈阳农业大学学报, 37(4): 582-586. (Liu M G, Zhao G L, Dong S J. 2006. Analysis of peroxidase isoenzyme and seed soluble protein in Armeniaca sibirica[J]. Journal of Shenyang Agricultural University, 37(4): 582-586. [in Chinese]) |
刘青华, 刘明国, 赵桂玲, 等. 2007. 16个山杏无性系POD及PPO同工酶的比较研究[J]. 辽宁林业科技, (4): 16-18. (Liu Q H, Liu M G, Zhao G L, et al. 2007. Comparative study on peroxidease and polyphenol for 16 clones of Prunus sibirica[J]. Journal of Liaoning Forestry Science & Technology, (4): 16-18. [in Chinese]) |
马发旺, 董胜君, 刘明国, 等. 2013. 黄土丘陵区野杏种质资源的数量分类[J]. 经济林研究, 31(4): 98-103. (Ma F W, Dong S J, Liu M G, et al. 2013. Quantitative classification of germplasm resources of Armeniaca vulgaris in loess hilly region of China[J]. Nonwood Forest Research, 31(4): 98-103. [in Chinese]) |
麻丽颖, 孔德仓, 刘华波, 等. 2012. 36份枣品种SSR指纹图谱的构建[J]. 园艺学报, 39(4): 647-654. (Ma L Y, Kong D C, Liu H B, et al. 2012. Construction of SSR fingerprint on 36 Chinese jujube cultivars[J]. Acta Horticulturae Sinica, 39(4): 647-654. [in Chinese]) |
沈敬理, 白天道, 陈亚斌, 等. 2015. 马尾松无性系种子园亲本指纹图谱构建[J]. 分子植物育种, 13(3): 646-652. (Shen J L, Bai T D, Chen Y B, et al. 2015. The fingerprints construction of clones in Pinus massoniana seed orchard[J]. Molecular Plant Breeding, 13(3): 646-652. [in Chinese]) |
王凤格, 赵久然, 郭景伦, 等. 2003. 比较三种DNA指纹分析方法在玉米品种纯度及真伪鉴定中的应用[J]. 分子植物育种, 1(5/6): 655-661. (Wang F G, Zhao J R, Guo J L, et al. 2003. Comparison of three DNA fingerprint analyzing methods for maize cultivars' identification[J]. Molecular Plant Breeding, 1(5/6): 655-661. [in Chinese]) |
王立新, 张小军, 史星雲, 等. 2012. 苹果栽培品种SSR指纹图谱的构建[J]. 果树学报, 29(6): 971-977. (Wang L X, Zhang X J, Shi X Y, et al. 2012. Establishment of SSR fingerprinting database on major apple(Malus×domestica) cultivars[J]. Journal of Fruit Science, 29(6): 971-977. [in Chinese]) |
文自翔, 赵团结, 郑永战, 等. 2008. 中国栽培和野生大豆农艺品质性状与SSR标记的关联分析I.群体结构及关联标记[J]. 作物学报, 34(7): 1169-1178. (Wen Z X, Zhao T J, Zheng Y Z, et al. 2008. Association analysis of agronomic and quality traits with SSR markers in Glycine max and Glycine soja in China:Ⅰ. Population structure and associated markers[J]. Acta Agronomica Sinica, 34(7): 1169-1178. [in Chinese]) |
杨本芸, 杨敏生, 梁海永, 等. 2008. 不同核桃品种的SSR分析[J]. 河北农业大学学报, 31(4): 51-55. (Yang B Y, Yang M S, Liang H Y, et al. 2008. SSR analysis of different walnut cultivars[J]. Journal of Agricultural University of Hebei, 31(4): 51-55. [in Chinese]) |
杨阳, 刘振, 赵洋, 等. 2010. 湖南省主要茶树品种分子指纹图谱的构建[J]. 茶叶科学, 30(5): 367-373. (Yang Y, Liu Z, Zhao Y, et al. 2010. Construction of DNA fingerprints for tea cultivars originated from Hunan province[J]. Journal of Tea Science, 30(5): 367-373. [in Chinese]) |
尹健, 董胜君, 吴智, 等. 2015. 内蒙古扎兰屯地区西伯利亚杏种质资源的数量分类[J]. 经济林研究, 33(3): 75-80. (Yin J, Dong S J, Wu Z, et al. 2015. Quantitative classification of germplasm resources of Armeniaca sibirica at Zhalantun region of Inner Mongolia[J]. Nonwood Forest Research, 33(3): 75-80. [in Chinese]) |
章秋平, 刘冬成, 刘威生, 等. 2013. 华北生态群普通杏遗传多样性与群体结构分析[J]. 中国农业科学, 46(1): 89-98. (Zhang Q P, Liu D C, Liu W S, et al. 2013. Genetic diversity and population structure of the north China populations of apricot(Prunus armeniaca L.)[J]. Scientia Agricultura Sinica, 46(1): 89-98. [in Chinese]) |
张亚东, 胡兴宜, 宋丛文. 2009. 利用新型分子标记EST-SSR鉴定湖北省内的主栽黑杨品种[J]. 分子植物育种, 7(1): 105-109. (Zhang Y D, Hu X Y, Song C W. 2009. Identification of Populus varieties from Hubei Province by EST-SSR marker[J]. Molecular Plant Breeding, 7(1): 105-109. [in Chinese]) |
周文才, 温强, 杨军, 等. 2017. 油茶栽培品种SSR指纹图谱构建及聚类分析[J]. 分子植物育种, 15(1): 238-249. (Zhou W C, Wen Q, Yang J, et al. 2017. Establishment of DNA fingerprints and cluster analysis for oil camellia cultivars based on SSR markers[J]. Molecular Plant Breeding, 15(1): 238-249. [in Chinese]) |
Liu H L, Yang W X, Hou J, et al. 2016. Genetic identification of 43 elite clonal accessions of Populus deltoides by SSR fingerprinting[J]. Canadian Journal of Plant Science, 96(3): 494-502. DOI:10.1139/cjps-2015-0272 |
Nei M, Li W H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences, 76(10): 5269-5273. DOI:10.1073/pnas.76.10.5269 |
Rocha R B, Abad J I M, Pires I E, et al. 2002. Fingerprint and genetic diversity analysis of Eucalyptus spp. genotypes using RAPD and SSR markers[J]. Scientia Forestalis, (62): 24-31. |
Smith J S C, Chin E C L, Shu H, et al. 1997. An evaluation of the utility of SSR loci as molecular markers in maize(Zea mays L.):Comparisons with data from RFLPS and pedigree[J]. Theoretical and Applied Genetics, 95(1/2): 163-173. |
Zhebentyayeva T, Reighard G, Gorina V, et al. 2003. Simple sequence repeat(SSR) analysis for assessment of genetic variability in apricot germplasm[J]. Theoretical and Applied Genetics, 106(3): 435-444. DOI:10.1007/s00122-002-1069-z |
 2018, Vol. 54
2018, Vol. 54

