文章信息
- 张恩亮, 马玲玲, 杨如同, 李林芳, 汪庆, 李亚, 王鹏
- Zhang Enliang, Ma Lingling, Yang Rutong, Li Linfang, Wang Qing, Li Ya, Wang Peng
- IBA诱导楸树嫩枝扦插不定根发育的转录组分析
- Transcriptome Profiling of IBA-Induced Adventitious Root Formation in Softwood Cuttings of Catalpa bungei 'Yu-1'
- 林业科学, 2018, 54(5): 48-61.
- Scientia Silvae Sinicae, 2018, 54(5): 48-61.
- DOI: 10.11707/j.1001-7488.20180506
-
文章历史
- 收稿日期:2017-07-13
- 修回日期:2017-12-27
-
作者相关文章
楸树(Catalpa bungei),紫葳科(Bignoniaceae)梓属(Catalpa),起源于中国的一种落叶树种,具有生长速度快、木材品质优良、适应性强等优点(石欣等,2011)。林木的扦插繁殖具有生长快、个体间整齐一致、单位面积木材产量高、造林增产效益显著等优点,利于优良品种的示范和推广。然而,这种方法依赖于不定根生根率。在课题组前期研究中发现‘豫楸1号’(C. bungei ‘Yu-1’)是易生根的楸树品种,生根率为85.6%(马玲玲等,2014)。‘豫楸1号’不定根的形成包括4个关键的发育阶段:激活期,扦插后0~5天;愈伤形成期,扦插后5~20天;不定根形成期,扦插后20~30天;根伸长期,扦插后30~40天。作为一个生根率较高的品种,‘豫楸1号’为研究楸树不定根形成的分子机制提供了重要的研究材料。
不定根发育是一个受激素调控的复杂的多基因相互作用的过程(Ahkami et al., 2009)。生长素具有明显促进不定根和侧根发生和发育的作用,乙烯通过调控生长素的代谢起到一定的作用,赤霉素对不定根和侧根形成有一定的促进作用,适当浓度的细胞分裂素促进不定根的发育,脱落酸则抑制不定根和侧根的生长。Vanneste等(2005)通过对拟南芥(Arabidopsis thaliana)slr1突变体的研究发现突变体生长素信号的传导受到抑制,阻止控制细胞周期分裂的基因的表达,进而侧根不能正常发生,推测可能是与AUX/IAA(auxin/indole-3-acetic acid)-ARF(auxin response factor)的生长素代谢途径有关。De Smet等(2007)通过对拟南芥侧根时空发生的研究,提出了侧根原基发生受生长素信号调控的模型:由生长素内向转运体(auxin transporter protein 1, AUX1)通过侧根冠转运IAA至分生组织基部,继而由生长素外向转运蛋白(auxin efflux carrier family protein, PIN)转运IAA至与原木质部接触的中柱鞘基础细胞,从而诱导停留在G1阶段的中柱鞘基础细胞重新进入S期以及G2/M期的细胞分裂过程。中柱鞘基础细胞在侧根原基发育过程中受AUX/IAA系列如IAA14/SLR基因介导的生长素信号调控(Fukaki et al., 2005)。
目前,随着基因芯片和RNA-seq测序技术的发展和应用,植物根系发育的分子机制研究取得了较大进展(Pavy et al., 2008; Felten et al., 2009; Petersson et al., 2009; Kyndt et al., 2012)。Ramírez-Carvajal等(2009)利用欧洲山杨(Populus tremula)与银白杨(P. alba)的杂交杨的全基因组基因芯片分析了在无外源激素的刺激下杂交杨插穗在切后0~6 h和6~24 h 2个时间段中基因的表达,发现蛋白的降解和磷酸化、分子转运和翻译是主要的信号通路,同时发现细胞分裂素-B类基因PtRR13通过IAA/ARF家族的基因负调控杨树的不定根发育。Kyndt等(2012)通过对水稻(Oryza sativa)根毛区和根冠的转录组分析发现3万个基因,其中1 761个基因在根毛区中优势表达,306个基因在根冠中特异表达;根毛区的基因主要是生长素、脱落酸等的应答基因,根冠区的基因主要参与线粒体电子转移、细胞器发育、次生代谢等生物学过程。Takehisa等(2012)分析水稻的38个基因表达谱数据,获得了22 297个在根不同发育时期差异表达的基因,基本阐明了植物激素如生长素、细胞分裂素等在根不同发育时期的拮抗和协同作用的机制。
楸树扦插不定根发育是一个复杂的多基因互作的过程,转录组测序在研究多基因互作机制方面较具优势。因此,本研究以楸树‘豫楸1号’为材料,对其扦插不定根发育过程中的4个阶段分别构建cDNA文库,利用高通量测序进行其转录组测序,以期获得与其扦插不定根发育相关的重要基因,并揭示其生根机制。
1 材料与方法 1.1 试验材料2013年5月,从江苏省中国科学院植物研究所梓属种质资源圃中选取楸树‘豫楸1号’当年生的嫩枝,将其处理成10~15 cm长的插穗;每个插穗留1片叶,并将其裁剪,只留下原来叶片的1/3;插穗放入浓度为3 000 mg·L-1 IBA溶液中60 s后,插入扦插床中,深度为插穗长度的1/2~2/3。扦插完之后用85%遮荫网遮盖防晒,自动喷雾装置喷雾给水。每周用500倍的多菌灵浊液喷洒浇透。采集IBA处理后0天(对照,P0)、1天(激活期,P1)、15天(愈伤形成期,P2)、25天(不定根形成期,P3)和35天(不定根伸长期,P4)的插穗基部或根的样品(图 1)。所有样品立即放入液氮进行冷冻,-80 ℃保存待用。

|
图 1 楸树嫩枝扦插不定根发育的时期 Figure 1 The stages of adventitious root formation of softwood cuttings of Catalpa bungei 'Yu-1' A-E分别是楸树嫩枝经IBA处理后0天(P0)、1天(P1)、15天(P2)、25天(P3)和35天(P4)。 A-E are 0(P0), 1(P1), 15(P2), 25(P3), 35(P4) days after cutting, respectively. |
使用TRIZOL试剂盒(Invitrogen,美国)分别提取P0-P4共5个样品的总RNA,按照试剂盒说明书进行操作。总RNA利用DNase消化DNA后,用带有Oligo(dT)的磁珠富集真核生物mRNA;加入打断试剂在Thermomixer中适温将mRNA打断成短片段,以打断后的mRNA为模板合成一链cDNA,然后配制二链合成反应体系合成二链cDNA,并使用试剂盒纯化回收、黏性末端修复、cDNA的3′末端加上碱基“A”并连接接头,然后进行片段大小选择,最后进行PCR扩增;构建好的5个cDNA文库用Agilent 2100 Bioanalyzer质检合格后,通过Illumina HiSeqTM2000进行测序。
1.2.2 原始数据的处理与拼接测序得到的所有raw reads通过去除接头、重复序列、低质量的序列,得到高质量的clean reads。所有的clean reads使用Trinity软件,在默认设置的参数下进行拼接,得到contigs。在所有的转录本中,取最长的contigs作为该转录本的unigenes。
1.2.3 Unigenes功能注释、分类和代谢通路分析Unigenes使用EMBOSS软件包中的GetORF工具进行蛋白质编码序列预测。用BLASTx将所有编码蛋白质的unigenes与NR数据库(http://www.ncbi.nlm.nih.gov/)、GO数据库(http://www.geneontology.org/)、Swiss-Prot数据库(http://www.expasy.ch/sprot)、COG数据库(http://www.ncbi.nlm.nih.gov/COG/)和KEGG数据库(http://www.genome.jp/kegg/kaas/)进行对比(E < 1e-5),得到与给定unigenes具有最高序列相似性的蛋白,作为该unigenes的蛋白功能注释信息。
1.2.4 差异基因鉴定和分析Unigenes表达量的计算使用RPKM (reads per kilobase per million mapped reads)方法(Marioni et al., 2008),即每100万条序列中,每个基因以1 000个碱基为单位比对上的reads个数。利用RSEM软件(Li et al., 2011)分析基因表达水平。以FDR(false discovery rate,错误发现率) < 0.01为标准筛选差异表达的unigenes。用MeV4.9.0软件(http://www.tm4.org/mev.html)进行聚类分析。
差异表达基因的富集分析利用Cytoscape (http://www.cytoscape.org)及其插件BINGO(http://www.psb.ugent.be/cbd/papers/BiNGO),以FDR < 0.01为标准筛选富集的GO条目和KEGG代谢通路。
1.2.5 实时荧光定量PCR分析利用M-MLV反转录酶将2 μg RNA反转成cDNA。荧光定量PCR引物利用Primer premier 5软件设计(表 1)。以楸树Actin为内参,荧光种类选择SYBR Green1,在荧光定量PCR仪上(7500型,ABI,USA)进行扩增和分析基因表达差异。以最小的样本阈值循环数(Ct值)和最高的荧光值为标准。扩增体系为cDNA稀释样1.00 μL,10×PCR Buffer 2.50 μL,dNTP (10 mmol·L-1) 0.50 μL,MgCl2(25 mmol·L-1) 1.50 μL,Primer F (10 μmol·L-1) 1.00 μL,Primer R (10 μmol·L-1) 1.00 μL,SYBR Green1 1.00 μL,rTaq polymerase(5 U·μL-1) 0.10 μL,ddH2O补充至25 μL。扩增程序为预扩增95 ℃ 30 s;95 ℃ 10 s,60 ℃ 25 s,72 ℃ 30 s,40个循环;72 ℃下延伸5 min。
|
|
楸树5个时期的文库P0、P1、P2、P3、P4产生了总共165 977 661条序列,大约33.52 Gb的转录组数据。5个cDNA文库中的鸟嘌呤(G)和胞嘧啶(C)核苷酸的GC含量变化范围为45.40%(P2)~46.48%(P3)(表 2)。序列数据已经提交到NCBI的SRA数据库,登录号SRP059272。从表 3可以发现,所有测序数据经组装产生了6 315 145个重叠群,接着组装产生平均长度为1 162.61 bp的116 941个转录本,最后获得平均长度为783.84 bp的62 955条unigenes。转录本和unigenes的N50长度分别为1 945 bp和1 417 bp(表 3)。在所有unigenes序列中,超过一半(57.89%)的unigenes的序列长度<500 bp(表 3)。利用BASTx软件进行搜索后发现有31 646条unigenes (50.27%)获得了注释,获得注释最多的数据库分别是NR(31 184, 49.53%)、GO (24 982, 39.68%)和Swiss-Prot (20 973, 33.31%)(表 4)。
|
|
|
|
|
|
从每个文库获得3 000万多个高质量的reads,然后分别比对到参考转录组数据。在每个不定根的形成阶段,发现了特异的和共同的差异表达基因。总共发现了11 100个差异表达基因,包括10 200个独特的差异表达基因和900个共同的差异表达基因(图 2A)。

|
图 2 楸树差异表达基因的表达谱分析 Figure 2 Expression profilings of differentially expressed genes(DEGs) in Catalpa bungei A:特异差异基因和共有差异基因的维恩图分析;B:文库中差异表达的基因的数量;C:差异表达基因的RPKM值分布;D:共有差异表达基因的聚类分析。 A: Venn diagrams representing the numbers of DEGs and the overlaps of sets obtained across four comparisons; B: Numbers of up- and down-regulated DEGs among four libraries (between P1 and P0, between P2 and P0, between P3 and P0, between P4 and P0, respectively); C: RPKM value distribution of DEGs; D: Hierarchical cluster heat map and cluster tree from the common DEGs(The upper indicates up-regulated genes and the lower indicates down-regulated genes). |
在P1/P0的比较中发现了4 551个差异表达基因,其中有2 383个基因上调表达,2 168个基因下调表达;在P2/P0的比较中发现了5 074个差异表达基因,其中有2 565个基因上调表达,2 509个基因下调表达;在P3/P0的比较中发现了5 182个差异表达基因,其中有3 440个基因上调表达,1 742个基因下调表达;在P4/P0的比较中发现了4 505个差异表达基因,其中2 269个基因上调表达,2 236个基因下调表达(图 2B)。上调表达的基因数量从低到高依次是P4/P0 < P1/P0 < P2/P0 < P3/P0,而下调表达的基因数量则是P3/P0 < P1/P0 < P4/P0 < P2/P0。P3/P0上调表达基因最多而P2/P0下调表达基因最多,可能是由于P3是愈伤至不定根形成的关键阶段,需要大量的基因表达从而为不定根形成提供物质基础。这些结果表明,不定根的形成与新转录本的积累有关,并且在愈伤组织形成后转录本更为丰富。
根据RPKM值将4个文库中的基因表达水平分为5类(极低、低、中、高和极高)(图 2C)。P1文库的平均RPKM值为25.61,P2文库为19.46,P3文库为29.15及P4文库为22.87。在P2和P3文库中,差异表达的基因中占比例最大的是极低表达的基因(RPKM<3),占比例最小的是极高表达的基因。然而,在P1和P4文库中,差异表达的基因中占比例最大的是极低表达的基因(RPKM<3),占比例最小的是高表达的基因(50≤RPKM<100)(图 2C)。
此外,在所有4个不定根发育阶段都检测到的差异表达基因有900个,聚类分析后被分为2个簇,簇A、B(图 2D)。有406个差异表达基因(45.11%)聚集在A类别,494个基因(54.89%)在B群中。
2.3 差异基因的GO富集分析为了进一步研究不定发育过程中的基因的生物学功能,一共有10 849个差异基因序列与拟南芥的30 467蛋白序列在E≤1e-5条件下进行比对分析。结果发现7 714(71.10%)个差异基因比对到了拟南芥的蛋白序列。差异表达基因的GO富集分析表明,在错误发现率FDR < 0.01时,57个GO条目富集在P1/P0,60个GO条目富集在P2/P0,61个GO条目富集在P3/P0,56个GO条目富集在P4/P0。基于所有GO富集分析,确定了46个共有的和7个唯一GO类别。在7个独有的GO类别中,‘高尔基体’‘溶酶体’‘核仁’‘细胞核’和‘蛋白质代谢过程’的5个GO类别在根形成期显著富集。此外,‘细胞骨架’显著富集在激活期,而‘DNA代谢过程’显著富集在愈伤组织形成期(表 5)。这些结果表明,GO类别在激素刺激下发生了动态变化,出现相应的不定根形成。在46个共有的GO条目中,‘应激反应’‘次生代谢过程’‘运输’‘生物合成过程’‘催化活性’和‘代谢过程’是富集基因最多的GO类别(表 5)。
|
|
如图 3所示,KEGG富集分析显示分别有6、4、4和8条代谢途径显著富集在P1/P0、P2/P0、P3/P0和P4/P0。苯丙氨酸代谢和苯丙素生物合成是共同显著富集的代谢途径,并且途径中上调表达的基因数量高于下调表达的基因数量(图 3)。然而,光合作用和光合天线蛋白富集在P1/P0、P2/P0和P4/P0且差异表达基因数量下调多于上调(图 3)。这些结果表明,在楸树不定根发育中,苯丙氨酸代谢和苯丙素生物合成途径具有促进作用而光合作用和光合天线蛋白途径则是阻碍作用。此外,植物激素信号转导富集在P3/P0和P4/P0,差异表达基因的上调多于下调(图 3),植物激素信号转导在楸树不定根发育中起到了促进作用。
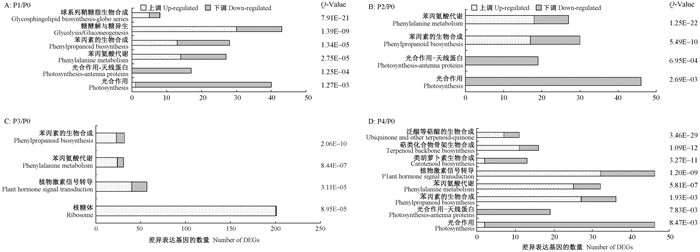
|
图 3 差异基因的KEGG分析 Figure 3 Specific significantly enriched KEGG pathways for differentially expressed genes(DEGs) Q值表示每个代谢途径的修正的错误发现率(FDR)。 Q-value was corrected false discovery rate(FDR) value of each pathway. |
KEGG富集分析表明,糖酵解和糖异生只在激活期富集且上调表达基因多于下调(图 3)。核糖体显著富集在根形成期且上调表达基因多于下调(图 3)。萜类骨架化合物生物合成和泛醌等萜醌的生物合成显著富集在根伸长期且上调表达基因多于下调(图 3)。类胡萝卜素的生物合成也显著富集在根伸长期但是上调表达基因少于下调(图 3)。这些结果表明,在IBA刺激后,代谢通路的动态变化响应了不定根的发育进程。
2.5 RNA-seq数据的验证如图 4所示,随机选取15个差异表达的RNA-Seq数据利用qPCR验证基因的表达。12个基因的qPCR数据与RNA-seq数据是一致的。qPCR分析进一步验证了PCKA(comp100533_c0)、CCoAOMT(comp110020_c1)、poxN1(comp110105_c0)和ERF110(comp114889_c1)在不定根发育过程中是上调表达的;ARF6(comp122156_c1)和S-腺苷甲硫氨酸合成酶2 (SAMS2,comp96145_c0)在不定根发育过程中是一直下调表达的。
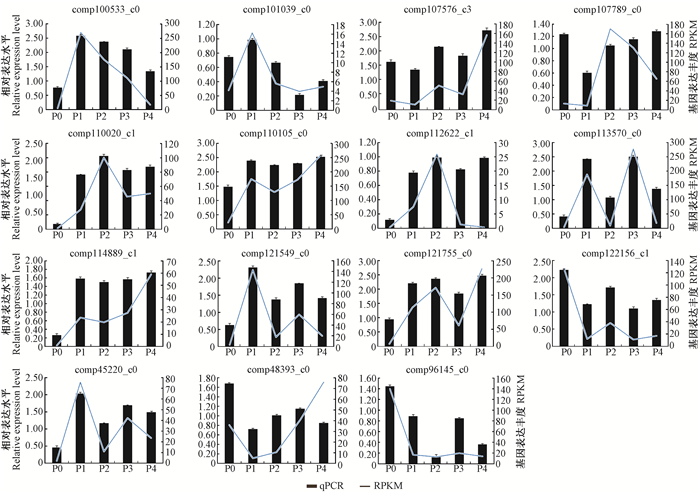
|
图 4 RNA-seq数据的荧光定量PCR验证 Figure 4 Real-time quantitative PCR validations of RNA-seq data 左纵坐标轴表示荧光定量PCR数据,右纵坐标轴为RNA-seq的数据。数据以平均值±标准误差的形式表示(n=3)。 Left axis represents expression levels determined by qPCR, and right axis represents expression levels determined by RNA-seq in RPKM units. Data are the means ± standard errors (n=3). |
生长素(auxin)是植物激素中最经典、发现最早的植物激素之一,它参与调控植物的许多生长发育过程,如不定根的形成和根的生长、顶端优势、维管组织的形成、茎叶的向光性生长、胚胎发育等(董秀春,2008)。植物生长素能诱导一些基因的快速高表达,这类基因被称为生长素原初反应基因。基于GO和KEGG注释分析发现在所有差异基因中,74个基因参与生长素代谢,17个基因参与脱落酸代谢,34个基因参与乙烯代谢,18个基因参与细胞分裂素代谢,44个基因参与油菜素内酯代谢(表 6)。随着不定根的发育,参与生长素和油菜素内酯代谢的差异基因数量基本上是逐渐增加的,而参与其他3种激素代谢的差异基因的数量变化较少(表 6)。在不定根发育的激活期,参与生长素代谢的基因表达模式是上调表达的基因数量小于下调表达,而剩余的3个时期的基因表达模式则正好相反(表 6)。参与油菜素内酯代谢的基因表达模式也具有类似的趋势。这些结果表明生长素和油菜素内酯在楸树不定根发育中起到了重要的作用,特别是在不定根伸长期,这个结果也进一步验证了KEGG富集分析的结果。从表 6中可以看出,参与细胞分裂素代谢的差异基因,愈伤期是最多的阶段同时也是唯一的下调表达基因多于上调的时期,表明细胞分裂素主要参与了楸树愈伤组织的形成。参与乙烯代谢的差异基因,愈伤期是最多的阶段同时上调表达基因多于下调,表明乙烯也在楸树的愈伤期发挥重要的作用但与细胞分裂素作用相反,可能表明二者间存在互作效应。
|
|
如表 7所示,共有30个共差异表达基因参与了激素代谢,其中17个基因上调表达而13个下调表达。随着楸树不定根的形成,参与乙烯、细胞分裂素和油菜素内酯代谢的关键基因,如1-氨基环丙烷-1-羧酸氧化酶1(1-aminocyclopropane-1-carboxylate oxidase 1, ACO1)、ACO5、细胞分裂素脱氢酶4(cytokinin dehydrogenase 4, CKX4)、CKX7和细胞色素P450 85A1(cytokrome P450 85A1, CYP85A1)是一直上调表达的(表 7)。随着楸树不定根的形成,参与激素平衡的基因如吲哚-3-乙酸酰胺合成酶(indole-3-acetic acid-amido synthetase 3.1, GH3.1)、GH3.6、生长素响应因子5(auxin response factor, ARF5)和乙烯应答转录因子3(ethylene-responsive transcription factor, ERF3)是一直上调表达,而生长素运输组分3(auxin efflux carrier component, PIN3)和ERF098却是下调表达(表 7)。
|
|
扦插繁殖是世界上木本植物的商业化生产的重要方法(Legué et al., 2014)。然而,楸树是未测序物种,基因组信息和基因表达信息缺失,阻碍了对其不定根形成的分子机制的理解。通过解剖学和生理学对不定根的分析发现楸树不定根发育是一个复杂的多步骤的过程(刘建玲等,2008)。转录组数据可以揭示基因表达谱和某个生物学过程。作为一种高通量、准确、低成本的下一代测序技术方法,RNA-seq已被广泛应用于转录组的定性和定量的分析,特别是对未测序物种。为了阐明楸树不定根发育的分子调控网络,本研究对嫩枝扦插不定根进程中的不同发育阶段进行了比较转录组分析。在本研究中,RNA-seq测序产生了116 941个unigene序列。这些序列为发掘与不定根形成相关的基因,进而为楸树的分子育种奠定了基础。
植物激素信号转导通路在植物发育控制中起着重要作用(Peat et al., 2012)。作为植物激素之一的生长素在不定根发育中的主要作用是刺激愈伤组织的形成和调节不定根发育(Lavenus et al., 2013; Legué et al., 2014)。生长素与其他的植物激素相互作用来调控植物的许多生长发育过程,如细胞分裂、伸长和分化(Muday et al., 2012)。本研究中发现IBA处理最容易影响与生长素稳态相关的基因的表达,并且大部分被诱导的基因都与游离生长素的减少有关。Staswick等(2005)研究发现,GH3基因编码的生长素酰胺合成酶能催化生长素氨基酸化使生长素失活,以维持植物体内生长素含量的动态平衡。GH3基因在大豆(Glycine max)中最先被发现,后来在水稻、拟南芥等植物中相继发现GH3类基因(孙涛等, 2008)。GH3蛋白能催化生长素与氨基酸的联接,形成生长素-氨基酸结合物致使生长素钝化失去活性,从而维持生长素的动态平衡(Zhang et al., 2009)。Nakazawa等(2001)的研究发现拟南芥中AtGH3.6的表达受生长素诱导,过表达AtGH3.6导致芽和胚轴细胞的伸长以及侧根的形成明显受到抑制。过表达水稻OsGH3.13基因的单株产生了生长素严重缺失的表型(Zhang et al., 2009)。生长素的运输是由生长素内流和外排载体蛋白完成,许多生长素内流和外排载体蛋白已经被克隆和鉴定(Muday et al., 2001; Friml et al., 2002)。AtPIN3可以通过改变生长素运输的方向,导致生长素不对称的积累,进而导致分化生长(Friml et al., 2002)。在拟南芥的pin1 pin3 pin4三突变体中产生了初生根发育缺陷的表型(Weijers et al., 2005)。本研究中,在激活期vs对照的转录组差异基因的比较中发现,分别与拟南芥的AtGH3.1和AtGH3.6同源的楸树基因comp119484_c0和comp84298_c0显著上调表达,可能导致游离态的生长素减少从而抑制不定根的发育,表明其在楸树不定根发育中起到了负调控的作用。但是,分别与拟南芥AtPIN3和AtPIN8同源的楸树基因comp122389_c0和comp115517_c0却是显著下调表达,说明在楸树不定根发育中生长素的储存和运输机制在调控生长素参与不定根发育中具有正调控的作用。同样的结果分别在愈伤期vs对照、不定根形成期vs对照及不定根伸长期vs对照的转录组差异基因的比较中被重复发现。生长素的动态平衡是不定根发育的关键,过高或过低的生长素浓度都会抑制不定根的发生和发育,本研究中发现GH3和PIN两类基因家族的基因相反的表达模式,可能起到了维持生长素浓度在合适的范围内,从而促进了楸树不定根的发生。
生长素和油菜素内酯在调节细胞扩张和增殖方面具有相似的作用(Nemhauser et al., 2004)。此外,油菜素内酯能增强生长素调控生长发育的能力,例如侧根数(Bao et al., 2004)和向地性反应(Li et al., 2005)。本研究中,随着不定根的发育,参与生长素和油菜素内酯代谢的差异基因数量基本上是逐渐增加的,而参与其他3种激素(脱落酸、乙烯和细胞分裂素)代谢的差异基因的数量变化较少(表 5), 间接表明了在不定根发育中生长素和油菜素内酯的协同作用。在不定根发育的激活期,参与生长素代谢的基因表达模式是上调表达的基因数量小于下调表达,而愈伤形成期、不定根形成期和不定根伸长期的基因表达模式则正好相反(表 5)。参与油菜素内酯代谢的基因表达模式也具有类似的趋势。参与生长素代谢和油菜素内酯代谢的差异基因数量都在不定根伸长期达到最大(表 5)。本研究的结果表明参与生长素代谢和油菜素内酯代谢的基因不仅在表达模式上具有一致的趋势而且在表达量上也具有相似的结果,为生长素和油菜素内酯在调控不定根发育中存在交互作用提供了相关的证据,然而这种相互作用的机制还不清楚,需要进一步深入的研究。本研究中发掘了许多参与生长素和油菜素内酯代谢的基因,为研究二者的交互作用奠定了基础。
细胞分裂素和乙烯对不定根的发育具有一定的作用。本研究中发现,参与细胞分裂素代谢的差异基因,愈伤期是最多的阶段同时也是唯一的下调表达基因多于上调的时期,但是参与乙烯代谢的差异基因,愈伤期则是上调表达基因多于下调(表 5),表明这2种激素在不定根发育中具有拮抗作用。Moubayidin等(2009)研究发现细胞分裂素与生长素的比值在器官分化中起到了决定性的作用。高浓度的细胞分裂素有利于芽的形成,生长素浓度高则促进根系的形成。本研究中发现外源IBA大大抑制参与细胞分裂素合成基因的表达,如细胞分裂素羟化酶,但诱导细胞分裂素降解相关基因,如细胞分裂素氧化酶(表 6)的表达。在植物的扦插中,伤害和真菌胁迫促进乙烯的合成,进而促进了不定根的发育(Kuroha et al., 2002)。乙烯生物合成始于S-腺苷甲硫氨酸(S-adenosylmethionine, SAM),包括2个关键步骤(Yang et al., 1984)。1-氨基环丙烷- 1-羧酸合酶(1-aminocyclopropane-1-carboxylate synthase, ACS, 又称为ACC合酶)催化SAM合成1-氨基环丙烷-1-羧酸(1-aminocyclopropane-1-carboxylate acid, ACC),通常被认为是乙烯合成的限速步骤。乙烯合成的最后一步是ACO催化ACC加氧转化为乙烯。在楸树不定根发育中,基因ACO1和ACO5的表达模式上调而基因ACS7和SAMS2则是下调表达,研究表明了乙烯在不定根发育调控中的复杂性。
糖酵解代谢途径是指将葡萄糖转化为丙酮酸和三磷酸腺苷(ATP)的代谢途径,它参与多种发育过程,例如发芽、开花和不定根形成。另外,蔗糖、葡萄糖和果糖之间的转化主要通过糖酵解代谢完成(Wang et al., 2007),同时发现蔗糖能显著诱导不定根的发生(Ahkami et al., 2013)。本研究中KEGG富集分析发现糖酵解只在激活期富集且上调表达基因多于下调,这个结果与糖酵解中涉及的酶的生物化学结果一致(Ahkami et al., 2014)。激活期是楸树不定根发育第1步,在这个阶段,细胞受到刺激,响应外源和內源的信号因子。因此,激活期需要能量,而糖酵解产生了能量和碳用于合成氨基酸、蛋白质和核酸的骨架,这可能是糖酵解仅在活化阶段显著富集的原因。
苯丙素生物合成是植物中最重要的次生代谢途径之一,该途径最重要的产物是木质素(Hu et al., 2010)。Cho等(2011)发现IBA处理的牛樟(Cinnamomum kanehirae)插条的木质素含量越低则不定根生根率越高。本研究通过KEGG富集分析发现苯丙素生物合成是共同显著富集的代谢途径并且途径中上调表达的基因数量高于下调表达的基因数量,表明在楸树不定根发育中,苯丙素生物合成途径具有促进作用。
4 结论通过对楸树易生根品种‘豫楸1号’的不定根发育的4个阶段的转录组分析发现,激素间的互作包括细胞分裂素和乙烯及生长素和油菜素内酯间的互作是促进楸树不定根发生的重要动力之一。虽然本研究对‘豫楸1号’不定根发育的全过程进行了表达谱比较分析,但是未能完全解析‘豫楸1号’不定根发育的分子调控机制。本研究的首次尝试为全面解析楸树不定根发育的分子机制和其他近缘物种的研究提供了基础。
董秀春. 2008. 毛白杨生长素信号转导因子基因的分离与功能的初步分析. 济南: 山东农业大学硕士学位论文. (Dong X C. 2008. Isolation and functional characterization of genes encoding auxin signaling components in Populus tomentosa. Shandong: MS thesis of Shandong Agricultural University. [in Chinese]) |
刘建玲, 何威, 苏金乐. 2008. "豫楸1号"扦插幼苗营养器官解剖构造与插穗愈伤组织形成的研究[J]. 河南科学, 26(7): 788-791. (Liu J L, He W, Su J L. 2008. The Catalpa bungei "Yu Qiu No. 1" cuttage seedling vegetative organ dissection structure with inserts the ear to injury the research which the organization forms[J]. Henan Science, 26(7): 788-791. [in Chinese]) |
马玲玲, 王鹏, 张振宇, 等. 2014. 梓属植物嫩枝扦插生根能力的评价[J]. 北方园艺, (15): 72-77. (Ma L L, Wang P, Zhang Z Y, et al. 2014. Evaluation on rooting ability of softwood cuttings on Catalpa[J]. Northern Horticulture, (15): 72-77. [in Chinese]) |
石欣, 李亚, 杨如同, 等. 2011. 中国楸树(Catalpa bungei C. A. Mey)种质资源遗传多样性的ISSR分析[J]. 江苏农业学报, 27(3): 634-639. (Shi X, Li Y, Yang R T, et al. 2011. ISSR analysis of genetic diversity of Catalpa bungei C. A. Mey germplasm resources in China[J]. Jiangsu Journal of Agricultural Science, 27(3): 634-639. [in Chinese]) |
孙涛, 柴团耀, 刘戈宇, 等. 2008. 植物GH3基因家族研究进展[J]. 生物工程学报, 24(11): 1860-1866. (Sun T, Chai T Y, Liu G Y, et al. 2008. Progress in the plant GH3 gene family[J]. Chinese Journal of Biotechnology, 24(11): 1860-1866. DOI:10.3321/j.issn:1000-3061.2008.11.002 [in Chinese]) |
Ahkami A H, Lischewski S, Haensch K T, et al. 2009. Molecular physiology of adventitious root formation in Petunia hybrida cuttings: involvement of wound response and primary metabolism[J]. New Phytologist, 181(3): 613-625. DOI:10.1111/j.1469-8137.2008.02704.x |
Ahkami A H, Melzer M, Ghaffari M R, et al. 2013. Distribution of indole-3-acetic acid in Petunia hybrida shoot tip cuttings and relationship between auxin transport, carbohydrate metabolism and adventitious root formation[J]. Planta, 238(3): 499-517. DOI:10.1007/s00425-013-1907-z |
Ahkami A, Scholz U, Steuernagel B, et al. 2014. Comprehensive transcriptome analysis unravels the existence of crucial genes regulating primary metabolism during adventitious root formation in Petunia hybrida[J]. PLoS One, 9(6): e100997. DOI:10.1371/journal.pone.0100997 |
Bao F, Shen J, Brady S R, et al. 2004. Brassinosteroids interact with auxin to promote lateral root development in Arabidopsis[J]. Plant Physiology, 134(4): 1624-1631. DOI:10.1104/pp.103.036897 |
Cho Y, Chang C Y, Huang L C, et al. 2011. Indole-3-butyric acid suppresses the activity of peroxidase while inducing adventitious roots in Cinnamomum kanehirae[J]. Botanical Studies, 52(2): 153-160. |
De Smet I, Tetsumura T, De Rybel B, et al. 2007. Auxin-dependent regulation of lateral root positioning in the basal meristem of Arabidopsis[J]. Development, 134(4): 681-690. DOI:10.1242/dev.02753 |
Felten J, Kohler A, Morin E, et al. 2009. The ectomycorrhizal fungus Laccaria bicolor stimulates lateral root formation in poplar and Arabidopsis through auxin transport and signaling[J]. Plant Physiology, 151(4): 1991-2005. DOI:10.1104/pp.109.147231 |
Friml J, Wiśniewska J, Benková E, et al. 2002. Lateral relocation of auxin efflux regulator PIN3 mediates tropism in Arabidopsis[J]. Nature, 415(6873): 806-809. DOI:10.1038/415806a |
Fukaki H, Nakao Y, Okushima Y, et al. 2005. Tissue-specific expression of stabilized SOLITARY-ROOT/IAA14 alters lateral root development in Arabidopsis[J]. The Plant Journal, 44(3): 382-395. |
Hu Y, Gai Y, Yin L, et al. 2010. Crystal structures of a Populus tomentosa 4-coumarate:CoA ligase shed light on its enzymatic mechanisms[J]. Plant Cell, 22(9): 3093-3104. DOI:10.1105/tpc.109.072652 |
Kuroha T, Kato H, Asami T, et al. 2002. A trans-zeatin riboside in root xylem sap negatively regulates adventitious root formation on cucumber hypocotyls[J]. Journal of Experiment Botany, 53(378): 2193-2200. DOI:10.1093/jxb/erf077 |
Kyndt T, Denil S, Haegeman A, et al. 2012. Transcriptome analysis of rice mature root tissue and root tips in early development by massive parallel sequencing[J]. Journal of Experimental Botany, 63(5): 2141-2157. DOI:10.1093/jxb/err435 |
Lavenus J, Goh T, Roberts I, et al. 2013. Lateral root development in Arabidopsis: fifty shades of auxin[J]. Trends in Plant Science, 18(8): 450-458. DOI:10.1016/j.tplants.2013.04.006 |
Legué V, Rigal A, Bhalerao R P. 2014. Adventitious root formation in tree species: involvement of transcription factors[J]. Physiololgia Plantarum, 151(2): 192-198. DOI:10.1111/ppl.12197 |
Li B, Dewey C N. 2011. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 12(1): 323. DOI:10.1186/1471-2105-12-323 |
Li L, Xu J, Xu Z H, et al. 2005. Brassinosteroids stimulate plant tropisms through modulation of polar auxin transport in Brassica and Arabidopsis[J]. Plant Cell, 17(10): 2738-2753. DOI:10.1105/tpc.105.034397 |
Marioni J C, Mason C E, Mane S M, et al. 2008. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays[J]. Genome Research, 18(9): 1509-1517. DOI:10.1101/gr.079558.108 |
Moubayidin L, Mambro R D, Sabatini S. 2009. Cytokinin-auxin crosstalk[J]. Trends Plant Science, 14(10): 557-562. DOI:10.1016/j.tplants.2009.06.010 |
Muday G K, Delong A. 2001. Polar auxin transport: controlling where and how much[J]. Trends in Plant Science, 6(11): 535-542. DOI:10.1016/S1360-1385(01)02101-X |
Muday G K, Rahman A, Binder B M. 2012. Auxin and ethylene: collaborators or competitors?[J]. Trends in Plant Science, 17(4): 181-195. DOI:10.1016/j.tplants.2012.02.001 |
Nakazawa M, Yabe N, Ichikawa T, et al. 2001. DFL1, an auxin-responsive GH3 gene homologue, negatively regulates shoot cell elongation and lateral root formation and positively regulates the light response of hypocotyls length[J]. The Plant Journal, 25(2): 213-221. DOI:10.1046/j.1365-313x.2001.00957.x |
Nemhauser J L, Mockler T C, Chory J. 2004. Interdependency of brassinosteroid and auxin signaling in Arabidopsis[J]. PLoS Biololgy, 2(9): e258. |
Pavy N, Boyle B, Nelson C, et al. 2008. Identification of conserved core xylem gene sets: conifer cDNA microarray development, transcript profiling and computational analyses[J]. New Phytologist, 180(4): 766-786. DOI:10.1111/nph.2008.180.issue-4 |
Peat T S, Böttcher C, Newman J, et al. 2012. Crystal structure of an indole-3-acetic acid amido synthetase from grapevine involved in auxin homeostasis[J]. Plant Cell, 24(11): 4525-4538. DOI:10.1105/tpc.112.102921 |
Petersson S V, Johansson A I, Kowalczyk M, et al. 2009. An auxin gradient and maximum in the Arabidopsis root apex shown by high-resolution cell-specific analysis of IAA distribution and synthesis[J]. The Plant Cell, 21(6): 1659-1668. DOI:10.1105/tpc.109.066480 |
Ramírez-Carvajal G A, Morse A M, Dervinis C, et al. 2009. The cytokinin type-B response regulator PtRR13 is a negative regulator of adventitious root development in Populus[J]. Plant Physiology, 150(2): 759-771. DOI:10.1104/pp.109.137505 |
Staswick P E, Serban B, Rowe M, et al. 2005. Characterization of an Arabidopsis enzyme family that conjugates amino acids to indole-3-acetic acid[J]. The Plant Cell, 17(2): 616-627. DOI:10.1105/tpc.104.026690 |
Takehisa H, Sato Y, Igarashi M, et al. 2012. Genome-wide transcriptome dissection of the rice root system: implications for developmental and physiological functions[J]. The Plant Journal, 69(1): 126-140. DOI:10.1111/tpj.2011.69.issue-1 |
Vanneste S, De Rybel B, Beemster G T S, et al. 2005. Cell cycle progression in the pericycle is not sufficient for SOLITARY ROOT/IAA14-mediated lateral root initiation in Arabidopsis thaliana[J]. The Plant Cell, 17(11): 3035-3050. DOI:10.1105/tpc.105.035493 |
Wang Y, Weathers P J. 2007. Sugars proportionately affect artemisinin production[J]. Plant Cell Report, 26(7): 1073-1081. DOI:10.1007/s00299-006-0295-2 |
Weijers D, Jürgens G. 2005. Auxin and embryo axis formation: the ends in sight?[J]. Curr Opin Plant Biol, 8(1): 32-37. DOI:10.1016/j.pbi.2004.11.001 |
Yang S F, Hoffman N E. 1984. Ethylene biosynthesis and its regulation in higher plants[J]. Annual Review Plant Physiology, 35(1): 155-189. DOI:10.1146/annurev.pp.35.060184.001103 |
Zhang S W, Li C H, Cao J, et al. 2009. Altered architecture and enhanced drought tolerance in rice via the down-regulation of indole-3-acetic acid by TLD1/OsGH3.13 activation[J]. Plant Physiology, 151(4): 1889-1901. DOI:10.1104/pp.109.146803 |
 2018, Vol. 54
2018, Vol. 54

