文章信息
- 马婧, 李政, 陈信立, 张升艳, 眭顺照, 李名扬
- Ma Jing, Li Zheng, Chen Xinli, Zhang Shengyan, Sui Shunzhao, Li Mingyang
- 蜡梅CpEXP1基因启动子的克隆及活性分析
- Cloning and Activity Analysis of CpEXP1 Gene Promoter from Chimonanthus praecox
- 林业科学, 2018, 54(3): 61-72.
- Scientia Silvae Sinicae, 2018, 54(3): 61-72.
- DOI: 10.11707/j.1001-7488.20180307
-
文章历史
- 收稿日期:2017-10-20
- 修回日期:2017-12-14
-
作者相关文章
细胞扩展蛋白(expansin)也称细胞壁松弛蛋白,它能够通过改变和修饰植物细胞壁的形态,使得细胞发生扩张和延展(高英等,2005)。细胞扩展蛋白基因家族是由α-、β-,γ-和δ-4个亚族所组成的庞大基因家族,每类亚家族由多个基因成员组成(Li et al., 2003)。目前,已经在拟南芥(Arabidopsis thaliana)(Cosgrove,2000)、玉米(Zea mays)(Wu et al., 2001)、水稻(Oryza sativa)(Choi et al., 2003)、毛白杨(Populus tomentosa)(张春玲等,2006)、白桦(Betula platyphylla)(王子剑等,2016)、桂花(Osmanthus fragrans)(罗云等,2017)等100多种植物中鉴定到细胞扩展蛋白的存在,并对其编码基因进行了克隆和功能研究。研究表明,扩展蛋白基因的表达具有器官、组织甚至细胞特异性,同时其表达调控受到发育进程、激素和环境因子的调控。目前,对扩展蛋白功能的研究主要集中在对α-expansin的研究上,它可能参与了包括植物种子萌发(Morris et al., 2011)、根系生长(Lee et al., 2003)、果实成熟(Carlos et al., 2009)、器官脱落(Cho et al., 2000)等在内的整个生长发育过程。如杉木(Cunninghamia lanceolata)中的2个α-expansin基因ClEXPA1和ClEXPA2具有较为相同的表达模式,在形成层区域表达最高,在针叶及茎尖中度表达,而在成熟木质部表达较低,推测其可能参与杉木木材形成的调控途径(高燕,2011);青杄(Picea wilsonii)PwEXP1基因在种子萌发前期被诱导表达,后期表达量急剧上升,表明其参与了种子萌发过程,可能与种子萌发后期出现幼根和幼茎的分化、细胞分裂旺盛有关(张通等,2014)。除了参与植物生长发育过程外,近几年研究还表明扩展蛋白可能通过增加细胞壁韧性来参与植物抵御高温、干旱等非生物胁迫响应过程(徐筱等,2010)。如低温、盐、重金属和渗透胁迫均强烈地抑制商陆(Phytolacca acinosa)PaEXP1基因的表达,推测该基因可能通过降低表达量来调控气孔闭合,从而降低呼吸作用和蒸腾作用,以适应外界不良环境(王辉等,2010);当低温处理高抗寒冬小麦(Triticum aestivum)东农冬麦1号时,3个expansin基因与正常冬小麦的对照组相比呈现显著性上升,推测其可能通过参与低温胁迫下冬小麦根系的形成来完成其在寒冷地区的成功越冬(李飞等,2016)。
本实验室在前期研究中从蜡梅‘磬口素心’(Chimonanthus praecox ‘Qingkousuxin’)中克隆获得了1个α-expansin家族成员CpEXP1 (Ma et al., 2012;李政等,2013),对其功能进行初步研究发现,该基因在蜡梅花器官中优势表达,并在蜡梅花发育过程中表现出不同的表达模式,可能参与蜡梅花发育及花器官脱落过程,对该基因在蜡梅营养器官生长及其他生物学过程中的具体功能还有待进一步的分析研究。本研究克隆了蜡梅CpEXP1基因上游调控序列,通过分析启动子的潜在调控元件,以及在烟草(Nicotiana tabacum)中的瞬时表达分析和在拟南芥中的稳定表达分析,探索该启动子的时空表达特性和对外界环境的响应模式,以期为进一步深入了解CpEXP1基因在蜡梅生长发育过程中的作用机制提供更多的线索。
1 材料与方法 1.1 植物材料、载体及菌株用于蜡梅基因组DNA提取的植物叶片采自西南大学苗圃中的蜡梅‘磬口素心’成年大树。转化所用的烟草组培苗品种为‘Wisconsin 38’,转化所用拟南芥的基因型为哥伦比亚系(Columbia,Col-0),以上植物材料均由西南大学园林花卉研究所扩繁保存。克隆载体pMD19-T购自TaKaRa(大连)生物公司,双元植物表达载体pBI121由本实验室保存。大肠杆菌(Escherichia coli)DH5α感受态细胞、根癌农杆菌(Agrobacterium tumefaciens)LBA4404感受态细胞均由本实验室保存。
1.2 DNA和RNA的提取与cDNA第1链的合成蜡梅和拟南芥基因组DNA均利用天根(北京)公司Plant Genomic DNA试剂盒进行提取纯化。拟南芥总RNA利用天根(北京)公司Plant RNA prep pure试剂盒进行提取后,利用TaKaRa(大连)公司的PrimeScript RT reagent Kit反转录试剂盒合成cDNA第1链,具体操作方法均按各试剂盒说明书进行。
1.3 启动子序列的克隆和生物信息学分析用Liu等(2007)改良的hiTAIL-PCR法扩增蜡梅CpEXP1基因的启动子。以前期克隆到的CpEXP1基因5′端序列为基础,分别设计3条特异的巢式引物RB0,RB1,RB2。以蜡梅基因组DNA为模板,利用文献中提供的简并引物LAD+通用引物AC0+特异引物RB0的引物对,进行预扩增;以稀释50倍的预扩增产物为模板,利用通用引物AC1+特异引物RB1的引物对,进行第1轮扩增;以稀释20倍的第1轮扩增产物为模板,利用通用引物AC1+特异引物RB2的引物对,进行第2轮扩增。简并引物、通用引物序列,PCR反应体系及程序见参考文献(Liu et al., 2007),特异引物序列见表 1。
|
|
将第1轮和第2轮产物在相邻泳道进行对比检测后,回收特异条带。目的片段进行TA克隆后,对PCR验证的阳性克隆进行测序,通过与已知序列的拼接比对,确认是否为CpEXP1基因的上游调控序列。根据获得的CpEXP1基因启动子和pBI121植物表达载体多克隆位点的序列特征,设计1对带有Hind Ⅲ和BamH Ⅰ酶切位点的特异引物,以蜡梅基因组DNA为模板扩增CpEXP1基因启动子序列。引物序列pCpEXP1-F:5′-CAAGCTTTATTCTAAT TACCATGAATGCG-3′(下划线部分为Hind Ⅲ);pCpEXP1-R:5′-CGGATCCCTCACTGTAGAAGAAGA AGAGG-3′(下划线部分为BamH Ⅰ)。
PCR反应条件:DNA模板1 μL,10 × ExTaq缓冲液2.0 μL,dNTPs 1.2 μL,上下游引物各1 μL(10 μmol·L-1),ExTaq酶0.2 μL,用ddH2O补足到20 μL。PCR反应程序:95 ℃预变性4 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸2 min,30个循环;72 ℃延伸10 min。回收特异条带进行TA克隆,将PCR检测后的阳性克隆送华大基因测序,将测序正确的CpEXP1基因启动子命名为pCpEXP1。利用在线软件Plant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和PlantPAN2.0(http://plantpan2.itps.ncku.edu.tw/promoter.php)分析该启动子序列中的潜在调控元件。
1.4 植物表达载体构建及瞬时表达分析利用Hind Ⅲ/BamH Ⅰ双酶切系统分别对含有pCpEXP1的克隆载体和pBI121植物表达载体进行酶切,回收酶切片段后连接转化大肠杆菌,即用pCpEXP1启动子代替pBI121中的35S启动子,构建pCpEXP1启动子和报告基因GUS融合的植物表达载体。将PCR和双酶切验证正确的重组质粒命名为pBI121-pCpEXP1。用电转法将该质粒转入农杆菌LBA4404,对pCpEXP1启动子进行瞬时表达分析,同时分别设置含有PBI121空载体菌株和不含表达载体的LBA4404空菌株为阳性对照和阴性对照。用含有重组质粒的工程菌(OD600=0.6)浸染烟草叶片,在黑暗条件下倒置共培养3天,具体步骤参照敬帆等(2015)的方法进行。对共培养后的烟草叶盘进行GUS染色分析(Jefferson,1987),并拍照观察,记录结果。
1.5 转CpEXP1基因启动子拟南芥的筛选鉴定利用农杆菌介导的花序侵染法将重组质粒pBI121-pCpEXP1转入拟南芥(Zhang et al., 2006),同时将含有35S启动子的pBI121植物表达载体转入拟南芥作为阳性对照。将获得的拟南芥T1代种子在含有卡那霉素(50 mg ·L-1Kan)的MS培养基上进行筛选,初步挑选出正常生长的抗性苗,移栽到营养土中。开花结实后收取T2代种子播种,继续用上述方法筛选至T3。提取T3代抗性苗的基因组DNA,用特异引物pCpEXP1-F/R和PBI121载体上的卡那霉素抗性基因Npt Ⅱ对转基因植株进行筛选检测。Npt Ⅱ基因引物序列Npt Ⅱ -F:5′-ATGATT GAACAAGATGGATTGCACG-3′;Npt Ⅱ -R:5′-TCAG AAGAACTCGTCAAGAAGGCGA-3′。PCR扩增反应体系和程序同1.4所述。
1.6 GUS组织化学染色及GUS基因表达分析对不同生长时期转pBI121空载体(35S:GUS)拟南芥(阳性对照)、野生型拟南芥(阴性对照)和转pCpEXP1启动子拟南芥的不同组织进行GUS组织化学染色分析,方法同1.4。同时采集GUS染色相应的植物材料,提取总RNA反转录后利用实时荧光定量PCR技术分析GUS基因在拟南芥不同组织和发育时期中的表达变化。对生长4周的T3代野生型和转基因拟南芥进行非生物胁迫和激素处理,每种处理选取3株长势一致的拟南芥植株作为一组,共分4组,分别进行如下处理:第1组拟南芥放置于4 ℃的人工气候箱中培养3天;第2组拟南芥放置于42 ℃的人工气候箱中培养3天;第3组拟南芥植株叶面喷施100 μmol ·L-1水杨酸;第4组拟南芥不作处理,于正常条件下培养3天。处理结束后,采集拟南芥叶片提取总RNA,反转录后利用实时荧光定量PCR检测胁迫处理后各个样本中GUS基因的表达量。GUS基因引物序列(马岩岩等,2014)GUS-F:5′-TACCGTACCTCGCATTACCC-3′;GUS-R:5′-CTGTAAGTGCGCTTGCTGAG-3′。拟南芥内参基因Actin引物如下:AtActin-F:5′-GGTAACATTGTG CTCAGTGGTGG-3′;AtActin-R:5′-AACGACCTTAAT CTTCATGCTGC-3′。
实时荧光定量PCR在BioRad(美国)公司CFX96TM荧光定量PCR仪上,利用该公司生产的SsoFastTMEvaGreen® Supermix试剂盒进行。PCR反应体系如下:10 μL Master Mix(2×),1 μL正向引物(10 μmol ·L-1),1 μL反向引物(10 μmol ·L-1),cDNA模板1 μL,用RNase Free ddH2O补足到20 μL总体积。反应条件如下:98 ℃ 30 s,95 ℃ 5 s,59 ℃ 5 s。每个循环结束后采集荧光信号,反应结束后从65~95 ℃做熔解曲线分析扩增的特异性。根据2-ΔΔCT法(Livak et al., 2001)用Bio-Rad ManagerTM Software(Version 1.1)软件对GUS基因的相对表达量进行分析。
2 结果与分析 2.1 CpEXP1基因启动子的获得、分析及植物表达载体构建用1%琼脂糖对3轮扩增产物进行检测,预扩增产物一般呈现涂抹带,不做电泳检测。将第1轮和第2轮PCR产物在相邻泳道进行比较(图 1),选择由简并引物LAD3获得的第2轮特异条带回收测序。将测序结果与已知的CpEXP1基因序列进行比对,结果显示克隆获得的片段为CpEXP1基因上游5′侧翼序列;再以蜡梅基因组DNA为模板,设计特异引物,利用PCR扩增获得了长度为2 485 bp的蜡梅CpEXP1基因启动子序列(图 2),命名为pCpEXP1(GenBank Accession:GM452931)。
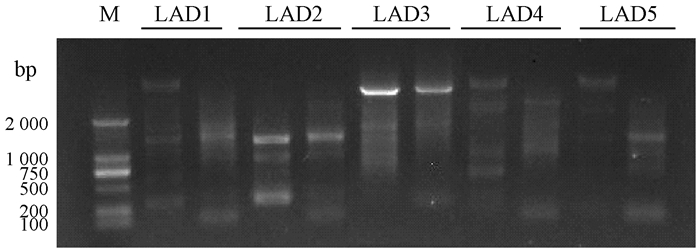
|
图 1 CpEXP1基因启动子的hiTAIL-PCR扩增 Figure 1 hiTAIL-PCR amplification of CpEXP1 gene promoter LAD1, 2, 3, 4, 5分别代表简并引物和特异引物组合的PCR产物,并列的左侧泳道为第1轮产物,右侧为第2轮产物;M:DNA分子量标记DL2000。 LAD1, 2, 3, 4, 5 stand for the PCR products of degenerate and specific primer pairs respectively; The left side is the primary and the right side is the secondary amplification product; M:DNA marker DL2000. |
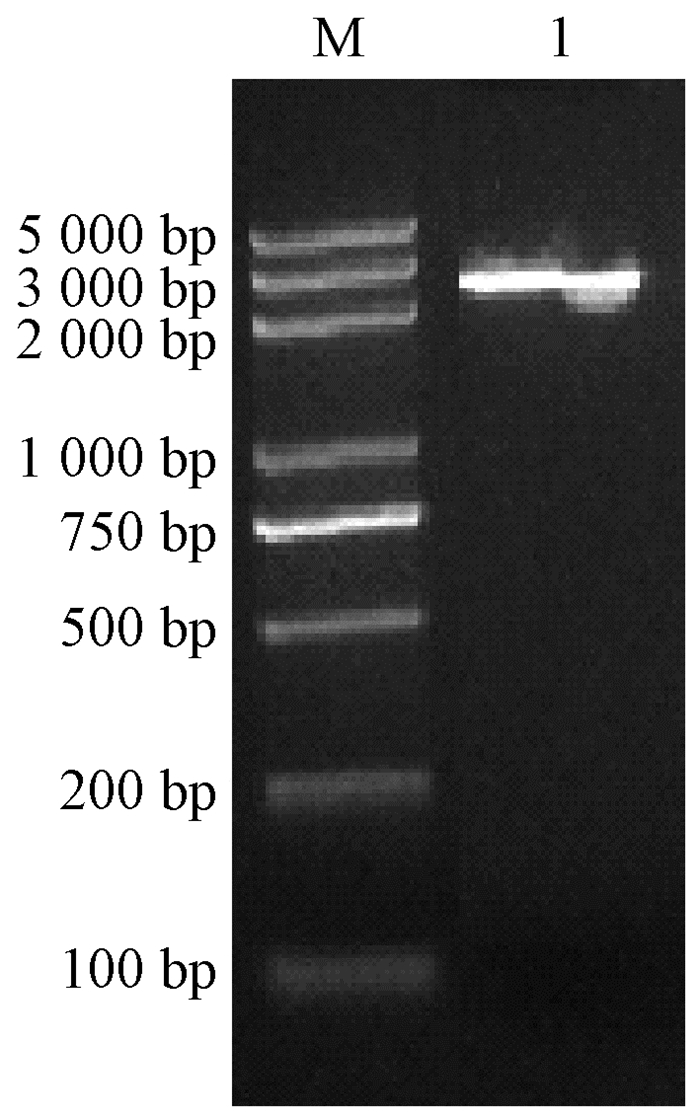
|
图 2 pCpEXP1启动子的克隆 Figure 2 Cloning of pCpEXP1 promoter by PCR 1: pCpEXP1启动子扩增产物;M:分子量标记Trans2K Plus。 1: The amplified production of pCpEXP1 promoter; M: Trans2K Plus DNA marker. |
利用PlantPan 2.0和Plant CARE软件分析pCpEXP1启动子中的顺式作用元件数量(表 2)及其分布(图 3),结果显示该启动子序列除了包含核心启动元件TATA-box 52个,CAAT-box 30个外,还包括与植物生长发育相关的元件,如发芽特异表达元件as-2-box、胚乳表达调控元件Skn-1 motif及种子特异调控元件RY-element。此外,还包含多个与植物光应答相关元件,如G-box、Ⅰ-box等,以及植物非生物胁迫响应元件,如胁迫响应元件TC-rich、水杨酸响应元件TCA-element、低温响应元件LTR、高温响应元件HSE等。
|
|

|
图 3 蜡梅CpEXP1基因启动子主要调控元件分布 Figure 3 The main regulatory elements distribution of CpEXP1 gene promoter |
对构建的植物表达载体进行PCR检测(图 4A)和酶切验证(图 4B),在电泳检测中均出现了相应大小的条带,表明已成功用pCpEXP1启动子替换了pBI121载体上的35S启动子,植物表达载体构建成功。

|
图 4 植物表达载体pBI121-pCpEXP1的PCR检测(A)和酶切验证(B) Figure 4 The identification of pBI121-pCpEXP1 by PCR (A) and digesting (B) |
农杆菌浸染烟草叶片的GUS组织化学染色结果显示,被含有pBI121空载体(35S:GUS)的农杆菌侵染的烟草叶片(图 5A)在染色后出现着色较深的蓝色斑点,符合35S启动子组成型表达的特点;以不含表达载体的农杆菌LBA4404浸染的烟草叶片作为阴性对照,结果显示无蓝色斑点,说明所用的工程菌株和烟草叶片内源均无GUS基因表达产物(图 5B),不能水解X-Gluc(5-溴-4-氯-3-吲哚-β-葡萄糖苷酸)底物,因此无法产生蓝色斑点。而被含有pBI121-pCpEXP1重组质粒农杆菌侵染后的烟草叶片出现了不均匀分布的蓝色斑点(图 5C),说明克隆获得的CpEXP1基因启动子能够驱动下游报告基因GUS的表达,具有启动活性,可以进行下一步在拟南芥中的稳定表达分析。
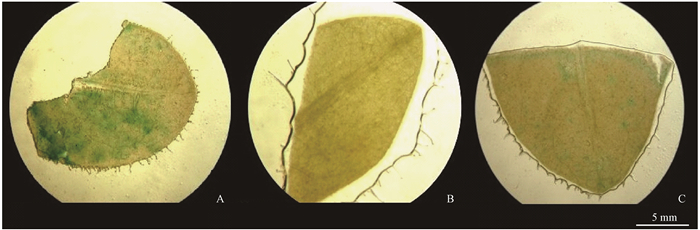
|
图 5 瞬时表达分析的GUS染色 Figure 5 GUS assay of transient expression A:pBI121空载体(35S:GUS,阳性对照);B:LBA4404空菌株(阴性对照);C:含pCpEXP1启动子的表达载体。 A: pBI121 empty vector(35S:GUS, positive control); B: LBA4404 empty Agrobacterium(negative control); C: pBI121-pCpEXP1 vector. |
通过Kan抗性筛选,共获得7株T3代抗性拟南芥转化植株。分别用特异引物pCpEXP1-F/R和PBI121载体上的Npt Ⅱ抗性基因(795 bp)引物检测所获得的T3代转化植株,在其中6株抗性植株中同时检测到Npt Ⅱ基因(图 6A)和pCpEXP1启动子(图 6B)目的片段,而在野生型拟南芥中均未扩增到目的片段,说明成功获得含有pCpEXP1启动子的转基因拟南芥植株。
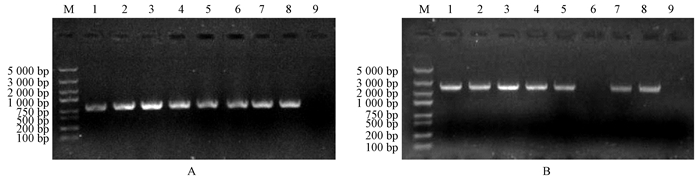
|
图 6 抗性拟南芥植株的PCR检测 Figure 6 PCR detection of resistant Arabidopsis strains A:抗性基因Npt Ⅱ的PCR检测;B:pCpEXP1启动子特异引物的PCR检测。1-7:转基因拟南芥;8:阳性对照;9:阴性对照(野生型拟南芥);M:DNA分子量标记Trans2K Plus。 A: PCR detection of Npt Ⅱ gene; B: PCR detection of pCpEXP1 promoter by specific primer.1-7: Transgenic plants; 8: Positive control; 9: Negative control(Wild type Arabidopsis); M: Trans2K Plus DNA marker. |
对转基因拟南芥进行GUS活性组织化学染色,以分析CpEXP1基因启动子的时空表达特性。结果表明在野生型拟南芥各个组织中均未检测到GUS活性。在转基因拟南芥中,CpEXP1基因启动子在种子萌芽期表现出较强的生物活性(图 7A),在子叶(图 7B)以及幼嫩叶片(图 7C)中均未检测到活性,在成熟叶片中可以检测到GUS活性,但在衰老叶叶柄处活性较强(图 7E);在根部活性较弱(图 7D);在开花结实期,在花序轴中未检测到活性,花朵中活性较弱(图 7F),在幼果和成熟果荚的果柄处也能检测到较强的GUS活性(图 7G)。

|
图 7 转pCpEXP1启动子拟南芥植株的GUS组织化学染色 Figure 7 Histochemical analysis of GUS in transgenic Arabidopsis plants with pCpEXP1 promoter A:种子;B:子叶期幼苗;C:真叶期幼苗;D:根;E:成熟(右)及衰老(左)叶片;F:花及幼果;G:成熟果荚。 A: Seeds; B: Seedling in cotyledon stage; C: Seedling in juvenile stage; D: Roots; E: Mature(right) and senescent (left) leaves; F: Flower and young fruit; G: Mature fruit pods. |
对GUS组织化学染色相应植物样本中的GUS基因表达进行实时荧光定量PCR检测(图 8),结果显示在转pCpEXP1启动子拟南芥中,GUS基因在衰老叶片的叶柄中表达量最高,在幼果和成熟果实的果柄中也具有较高表达,而在子叶、幼叶中几乎检测不到GUS基因表达,在根和花中表达量较低。这与GUS组织化学染色的结果基本一致,说明pCpEXP1启动子在拟南芥不同组织和发育阶段具有不同的启动活性。
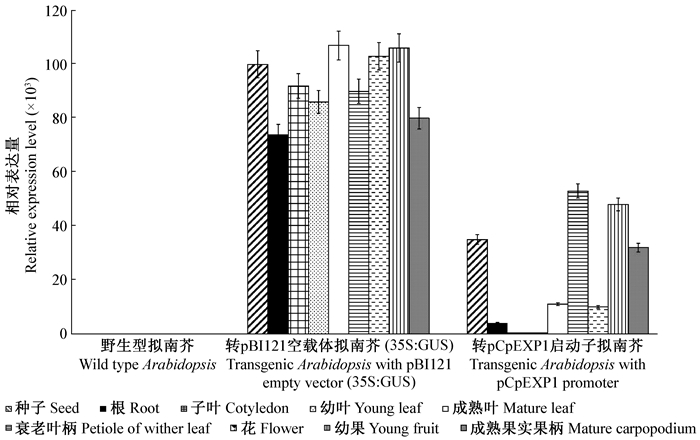
|
图 8 转基因拟南芥不同组织中GUS基因的表达分析 Figure 8 The expression of GUS gene in transgenic Arabidopsis in different tissues |
为了研究CpEXP1基因启动子对非生物胁迫的响应活性,根据生物信息学元件预测结果,对拟南芥植株分别进行高温、低温和水杨酸处理后,检测叶片中GUS基因的转录水平(图 9),结果显示:在非转基因植株中,各处理前后均未检测到GUS基因表达;转pBI121空载体阳性对照植株中的GUS基因,在各种处理前后的相对表达量无显著差异;转pCpEXP1启动子拟南芥中的GUS基因在低温、高温及水杨酸处理后,可被强烈诱导表达,其中,4 ℃低温处理后,GUS基因表达量是处理前的8.7倍,经42 ℃高温和水杨酸处理后其活性分别是处理前的5.4倍和3.3倍。由以上结果可以推测蜡梅CpEXP1基因启动子活性可以被多种非生物胁迫条件所诱导,特别对低温胁迫响应强烈。
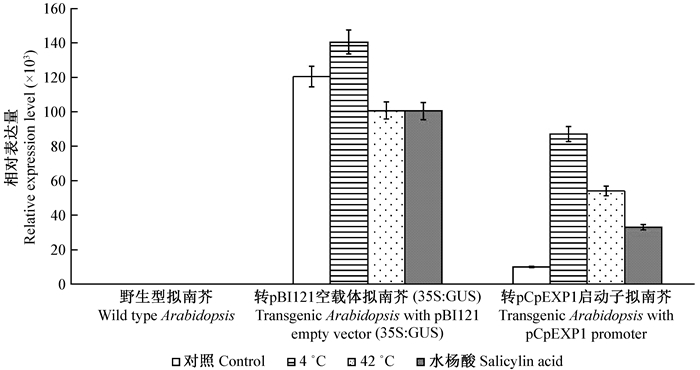
|
图 9 不同非生物胁迫处理下转基因拟南芥中GUS基因表达分析 Figure 9 The expression of GUS gene in transgenic Arabidopsis under different abiotic stress treatments |
细胞扩展蛋白是一个相对保守的多基因家族,目前的研究表明扩展蛋白几乎参与了植物发育的整个过程,在细胞生长、种子发育、根系生长、叶原基生成、果实成熟、器官脱落等过程中发挥不同功能。许多研究表明扩展蛋白基因具有明显的组织、时空甚至是细胞表达特异性(Cosgrove,2000;Chen et al., 2000;2001;欧阳昆唏等,2013),这种独特的表达模式除了受发育进程、激素、环境因子刺激外,还受到启动子的调控。许多expansin基因的启动子序列中均含有与组织表达特异性相关的调控元件,从而表现出具有组织差异的驱动活性(Cosgrove,1998;Cho et al., 2000;Sun et al., 2011)。例如,拟南芥AtEXPA7和AtEXPA18基因的启动子区含有根毛特异的顺式调控元件(RHEs)(Kim et al., 2006);酸樱桃(Prunus cerasus)PcExp2基因启动子片段包含AC-element、Myb蛋白结合位点、GAGA-box等多个与植物组织特异表达相关的顺式作用元件(Karaaslan et al., 2010)。本研究获得的蜡梅pCpEXP1启动子序列中,包含多个与种子发育相关的顺式调控元件,如发芽特异表达元件as-2-box、种子特异调控元件RY-element及胚乳表达的顺式调控元件Skn-1-motif。通过对转基因拟南芥的GUS组织化学染色和GUS基因表达分析,发现该启动子在种子萌发初期表现出较强的启动活性,而后随着种子的发育活性降低,推测该启动子可能在种子发芽早期,通过驱动下游CpEXP1基因大量积累,造成种子细胞壁松弛解体,从而进一步引起种皮的软化,促进种皮和外界物质交换,加速种子发芽过程。
前期对蜡梅CpEXP1基因表达分析的结果表明,在蜡梅的幼叶和子叶中均未检测到该基因的表达,在成熟叶片中表达量最高(Ma et al., 2012)。本研究在转基因拟南芥子叶和幼叶中也未检测到CpEXP1基因启动子驱动的下游GUS报告基因的表达,在成熟叶片中GUS活性逐渐增强,这与前人对该基因表达分析的结果一致。在转基因拟南芥植株衰老叶片的叶柄和成熟果荚果柄处均检测到较强的GUS活性,由此推测CpEXP1基因启动子可能在植物器官脱落过程中发挥一定的作用,这可能是因为该启动子驱动CpEXP1基因在叶柄、果柄等离区大量表达,从而导致该区域细胞扩张膨大,进而对脱落区域产生机械压力,促使了衰老叶片、成熟果实等的脱落。目前,关于expansin基因家族在植物器官衰老脱落间的关系已有大量文献报告,如Cho等(2000)发现拟南芥中AtEXP10的过量表达导致叶片更易脱落;Belfield等(2005)发现西洋接骨木(Sambucus nigra)中的2个expansin基因SniEXPA2和SniEXPA4都在叶柄的离区特异表达,而在非离区组织不表达。本试验中顺式调控元件预测和转基因拟南芥中启动子活性分析的结果,与前人的研究较为一致。CpEXP1基因启动子在植物不同生长阶段和组织中的活性差异,揭示了该启动子在蜡梅种子发育、器官脱落和果实成熟方面的潜在功能。
近年来的研究发现,expansin基因除了参与植物生长发育外,还可能通过改变植物细胞壁的韧性来抵御外界不良环境,参与植物抗逆反应的调控途径。如Wu等(1996)研究发现玉米根中的扩展蛋白基因在干旱条件下表达量增加,可能参与玉米抵御外界干旱胁迫,维持生长状态;剪股颖(Agrostis scabra)AsEXP1基因的表达水平与草坪草品种的耐热性呈显著正相关(Xu et al., 2007);青杄PwEXP1基因可在高温胁迫下的根和叶中被诱导表达(张通等,2014)。前人对多种植物中不同expansin基因启动子的研究表明,这些启动子序列中存在有大量与植物生物及非生物胁迫、激素诱导相关的顺式调控元件。例如,将拟南芥AtEXPA1基因启动子不同缺失片段构建成与GUS报告基因融合的转化载体,转化烟草原生质体进行瞬时表达分析,其中P12缺失体在低温处理下GUS活性降低,表明在-897~-626 bp区域间可能存在某些低温胁迫的负调控响应元件(李宏等,2008);在水稻扩展蛋白基因启动子区域也发现了与脱落酸、赤霉素、乙烯等激素相关的调控元件(Lee et al., 2005;Catalo et al., 2009)。本研究中,CpEXP1基因启动子生物信息学分析显示其含有多个逆境应答作用相关元件,如胁迫响应元件TC-rich、低温响应元件LTR、热胁迫响应元件HSE和水杨酸响应元件TCA-element,推测该启动子可能响应多种调控因子,进一步的研究也表明,低温、高温胁迫和SA的处理均可显著提升转基因拟南芥中的GUS的表达量,特别是对低温处理响应强烈。笔者前期研究(Ma et al., 2012)表明CpEXP1基因在自然栽培条件下蜡梅开放期中表达量最高,这个时期外界气温的骤降是诱导蜡梅花朵开放的主要因素,同时该时期也是蜡梅落蕾最严重的阶段(李政等,2013),结合本研究结果,推测可能是外界低温环境诱导pCpEXP1启动子驱动CpEXP1基因在花柄处大量积累导致花器官脱落。今后研究中,将继续在此基础上获得更长的上游调控序列,挖掘组织特异表达及环境诱导元件,并构建不同缺失片段长度的启动子序列,精确研究其元件的具体功能,为研究CpEXP1基因在蜡梅发育及非生物胁迫中的作用机制提供研究基础。
4 结论从蜡梅中克隆获得了1个长度为2 485 bp的扩展蛋白基因CpEXP1启动子序列,该序列中除了基本核心元件外,还含有多个与植物组织特异表达和环境因子诱导相关的顺式调控元件。CpEXP1启动子具有驱动下游报告基因GUS表达的能力,在转基因拟南芥不同发育阶段的不同组织中的启动活性具有时空特异性。同时该启动子可以被高温、低温和水杨酸诱导,特别是对低温环境响应强烈。下一步将进一步挖掘更多有意义的调控元件,研究其作用的具体机制,为全面解析蜡梅CpEXP1功能提供更多的试验依据。
高燕. 2011. 杉木木材形成过程扩展蛋白基因的克隆与表达分析[J]. 林业科学, 47(11): 44-51. (Gao Y. 2011. Cloning and expression analysis of expansin genes during wood formation in Chinese fir[J]. Scientia Silvae Sinicae, 47(11): 44-51. [in Chinese]) |
高英, 王学臣. 2005. 扩张蛋白expansin研究进展[J]. 中国农学通报, 21(7): 82-86. (Gao Y, Wang X C. 2005. The review of the development of researches on expansin[J]. Chinese Agricultural Science Bulletin, 21(7): 82-86. [in Chinese]) |
敬帆, 罗登攀, 马婧, 等. 2015. 蜡梅CpAGL6基因启动子的克隆及功能初步分析[J]. 园艺学报, 42(6): 1139-1149. (Jing F, Luo D P, Ma J, et al. 2015. Cloning and preliminary functional analysis of CpAGL6 promoter from Chimonanthus praecox[J]. Acta Horticulturae Sinica, 42(6): 1139-1149. [in Chinese]) |
李飞, 王晓磊, 徐永清, 等. 2016. 低温处理下东农冬麦1号小麦根组织EXPA基因的表达分析[J]. 麦类作物学报, 36(9): 1159-1166. (Li F, Wang X L, Xu Y Q, et al. 2016. Expression analysis of EXPA gene in root tissues of winter wheat Dongnongdongmai 1 under low temperature treatment[J]. Journal of Triticeae Crop, 36(9): 1159-1166. [in Chinese]) |
李宏, 魏鹏程, 龚喜明, 等. 2008. 拟南芥AtEXP1基因启动子的克隆及转录调控元件分析[J]. 农业生物技术学报, 16(3): 543-544. (Li H, Wei P C, Gong X M, et al. 2008. Cloning and analysis of transcriptional regulatory module of AtEXP1 promoter[J]. Journal of Agriculture Biotechnology, 16(3): 543-544. [in Chinese]) |
李政, 马婧, 眭顺照, 等. 2013. 不同脱落力下蜡梅花梗CpEXP1基因的表达与扩张蛋白活性[J]. 植物生理学报, 49(11): 1255-1260. (Li Z, Ma J, Sui S Z, et al. 2013. Expression of CpEXP1 gene and expansin activity in pedicel of wintersweet under different break strength[J]. Plant Physiology Journal, 49(11): 1255-1260. [in Chinese]) |
罗云, 张超, 付建新, 等. 2017. 桂花扩展蛋白基因家族的鉴定和表达分析[J]. 农业生物技术学报, 25(8): 1289-1299. (Luo Y, Zhang C, Fu J X, et al. 2017. Identification and expression analysis of expansin gene family in Osmanthus fragrans[J]. Journal of Agricultural Biotechnology, 25(8): 1289-1299. [in Chinese]) |
马岩岩, 陈娇, 吴天利, 等. 2014. 柑橘CsTBL1启动子的克隆及其在转基因拟南芥中的活性分析[J]. 园艺学报, 41(5): 817-824. (Ma Y Y, Chen J, Wu T L, et al. 2014. Isolation of the citrus CsTBL1 gene promoter and its activity in transgenic Arabidopsis[J]. Acta Horticulturae Sinica, 41(5): 817-824. [in Chinese]) |
欧阳昆唏, 李俊成, 黄浩, 等. 2013. 团花树α-扩展蛋白基因的克隆及表达分析[J]. 林业科学, 49(9): 62-71. (Ouyang K X, Li J C, Huang H, et al. 2013. Molecular cloning and expression analysis of α-Expansin genes in Anthocephalus chinensis[J]. Scientia Silvae Sinicae, 49(9): 62-71. [in Chinese]) |
王辉, 孙涛, 赵会君, 等. 2010. 商陆扩展蛋白基因PaEXP1在逆境胁迫下的表达[J]. 中国科学院研究生院学报, 27(4): 448-455. (Wang H, Sun T, Zhao H J, et al. 2010. Expression of PaEXP1 from Phytolacca acinosa seedling in response to adversity[J]. Journal of the Graduate School of the Chinese Academy of Sciences, 27(4): 448-455. [in Chinese]) |
王子剑, 孙丹, 于颖, 等. 2016. 白桦扩展蛋白基因的克隆分析及植物表达载体构建[J]. 西南林业大学学报, 36(1): 1-8. (Wang Z J, Sun D, Yu Y, et al. 2016. Gene cloning and construction of plant expression vector of expansin genes in Betula platyphylla[J]. Journal of Southwest Forestry University, 36(1): 1-8. [in Chinese]) |
徐筱, 徐倩, 张锴, 等. 2010. 植物扩展蛋白基因的研究进展[J]. 北京林业大学学报, 32(5): 154-162. (Xu X, Xu Q, Zhang K, et al. 2010. Advancements in expansin genes of plants[J]. Journal of Beijing Forestry University, 32(5): 154-162. [in Chinese]) |
张春玲, 张德强, 赵树堂, 等. 2006. 杨树形成层区域扩张蛋白PtEXP1基因的克隆与分析[J]. 西北农林科技大学学报:自然科学版, 34(6): 52-56. (Zhang C L, Zhang D Q, Zhao S T, et al. 2006. Cloning and analysis of a PtEXP1 gene from Populus tomentosa Carr[J]. Journal of Northwest A&F University:Natural Science Edition, 34(6): 52-56. [in Chinese]) |
张通, 李巧玲, 张凌云. 2014. PwEXP1在青杄种子萌发及逆境响应中的表达特征[J]. 林业科学, 50(12): 56-62. (Zhang T, Li Q L, Zhang L Y. 2014. Expression characteristics of PwEXP1 gene in seed germination and adversity in Picea wilsonii[J]. Scientia Silvae Sinicae, 50(12): 56-62. [in Chinese]) |
Belfield E J, Ruperti B, Robert J A, et al. 2005. Changes in expansin activity and gene expression during ethylene-promoted leaflet abscission in Sambucus nigra[J]. Journal of Experiment Botany, 56(413): 817-823. DOI:10.1093/jxb/eri076 |
Carlos G, Carlos R F, Cristian B, et al. 2009. Expression of an ethylene-related expansin gene during softening of mountain papaya fruit (Vasconcellea pubescens)[J]. Postharvest Biology and Technology, 53(1): 58-65. |
Catalo C, Rose J K C, Bennett A B. 2000. Auxin-regulated genes encoding cell wall-modifying proteins are expressed during early tomato fruits growth[J]. Plant Physiology, 122(2): 527-534. DOI:10.1104/pp.122.2.527 |
Chen F, Kent J B. 2000. Expression of an expansin is associated with endosperm weakening during tomato seed germination[J]. Plant Physiology, 124(3): 1265-1274. DOI:10.1104/pp.124.3.1265 |
Chen F, Dahal P, Bradford K J. 2001. Two tomato expansin genes show divergent expression and localization in embryos during seed development and germination[J]. Plant Physiology, 127(3): 928-936. DOI:10.1104/pp.010259 |
Cho H T, Cosgrove D J. 2000. Altered expression of expansin modulates leaf growth and pedicel abscission in Arabidopsis thaliana[J]. Proceedings of the National Academy of Sciences of the United States of America, 97(17): 9783-9788. DOI:10.1073/pnas.160276997 |
Choi D, Lee Y, Cho H, et al. 2003. Regulation of expansin gene expression affects growth and development in transgenic rice plants[J]. The Plant Cell, 15(6): 1386-1398. |
Cosgrove D J. 1998. Cell wall loosening by expansins[J]. Plant Physiology, 118(2): 333-339. DOI:10.1104/pp.118.2.333 |
Cosgrove D J. 2000. New genes and new biological roles for expansins[J]. Current Opinion in Plant Biology, 3(1): 73-78. DOI:10.1016/S1369-5266(99)00039-4 |
Jefferson R A. 1987. Assaying chimeric genes in plants:The GUS gene fusion system[J]. Plant Molecular Biology, 5(4): 387-405. DOI:10.1007/BF02667740 |
Karaaslan M, Hrazdina G. 2010. Characterization of an expansin gene and its ripening-specific promoter fragments from sour cherry (Prunus cerasus L.) cultivars[J]. Acta Physiology Plant, 32(6): 1073-1084. DOI:10.1007/s11738-010-0499-5 |
Kim D W, Lee S H, Choi S B, et al. 2006. Functional conservation of a root hair cell-specific cis-element in angiosperms with different root hair distribution patterns[J]. Plant Cell, 18(11): 2958-2970. DOI:10.1105/tpc.106.045229 |
Lee D K, Ahn J H, Song S K, et al. 2003. Expression of an expansin gene is correlated with root elongation in soybean[J]. Plant Physiology, 131(3): 985-997. DOI:10.1104/pp.009902 |
Lee Y, Choi D. 2005. Biochemical properties and localization of the β-expansin OsEXPB3 in rice (Oryza sativa L.)[J]. Molecular Cells, 20(1): 119-126. |
Li Y, Jones L, McQueen-Mason S. 2003. Expansins and cell growth[J]. Current Opinion in Plant Biology, 6(6): 603-610. DOI:10.1016/j.pbi.2003.09.003 |
Liu Y G, Chen Y L. 2007. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J]. Biotechniques, 43(5): 649-656. DOI:10.2144/000112601 |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△CT method[J]. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
Ma J, Li Z, Wang B, et al. 2012. Cloning of an expansin gene from Chimonanthus praecox flowers and its expression in flowers treated with ethephon or methylcyclopropene[J]. Hortscience, 47(10): 1472-1477. |
Morris K, Linkies A, Müller K, et al. 2011. Regulation of seed germination in the close Arabidopsis relative Lepidium sativum: A global tissue-specific transcript analysis[J]. Plant Physiology, 155(4): 1851-1870. DOI:10.1104/pp.110.169706 |
Sun T, Zhang Y, Chai T. 2011. Cloning, characterization, and expression of the BjEXPA1 gene and its promoter region from Brassica juncea L[J]. Plant Growth Regulation, 6(1): 39-51. |
Wu Y, Sharp R E, Durachko D M, et al. 1996. Growth maintenance of the maize primary root at low water potentials involves increases in cell-wall extension properties, expansin activity and wall susceptibility to expansins[J]. Plant Physiology, 111(3): 765-772. DOI:10.1104/pp.111.3.765 |
Wu Y J, Meeley R B, Cosgrove D J. 2001. Analysis and expression of the α-Expansin and β-Expansin gene families in maize[J]. Plant Physiology, 126(3): 222-232. |
Xu J C, Tian J, Belanger F C, et al. 2007. Identification and characterization of an expansin gene AsEXP1 associated with heat tolerance in C3 Agrostis grass species[J]. Journal of Experimental Botany, 58(13): 3789-3796. DOI:10.1093/jxb/erm229 |
Zhang X, Henriques R, Lin S S, et al. 2006. Agrobacterium-mediated transformation of Arabidopsis thaliana using the floral dip method[J]. Nature Protocols, 1(2): 641-646. DOI:10.1038/nprot.2006.97 |
 2018, Vol. 54
2018, Vol. 54

