文章信息
- 李洪果, 许基煌, 杜红岩, 乌云塔娜, 刘攀峰, 杜庆鑫
- Li Hongguo, Xu Jihuang, Du Hongyan, Wuyun Tana, Liu Panfeng, Du Qingxin
- 基于等位基因最大化法初步构建杜仲核心种质
- Preliminary Construction of Core Collection of Eucommia ulmoides Based on Allele Number Maximization Strategy
- 林业科学, 2018, 54(2): 42-51.
- Scientia Silvae Sinicae, 2018, 54(2): 42-51.
- DOI: 10.11707/j.1001-7488.20180205
-
文章历史
- 收稿日期:2017-03-22
- 修回日期:2017-11-06
-
作者相关文章
2. 国家林业局泡桐研究开发中心 郑州 450000
2. China Paulownia Research Center Zhengzhou 450000
杜仲(Eucommia ulmoides)是我国特有的珍贵孑遗树种,二倍体,雌雄异株,适生于年均温9~20 ℃、极端低温不低于-33 ℃、pH5.0~8.4的广大区域(杜红岩,2013)。杜仲的果、叶、皮均含有丰富的天然橡胶,可开发出橡胶弹性材料、热塑性和热弹性等用途的材料;此外,杜仲还是名贵的药用经济树种,具有强筋骨、补肝肾和轻身耐老功效(杜红岩,2013)。目前,从全国范围内收集的千余份杜仲种质资源由于引种资料匮乏等原因存在种质不清和重复收集等问题,含有一定程度的遗传冗余。另外,在杜仲栽培生产逐步向良种化发展的趋势下,一些栽培表现普通的种质面临淘汰和分布区进一步缩减的压力,存在种质灭失的风险。因此,通过构建核心种质,加强现有杜仲种质资源遗传多样性的利用和保护尤为必要。
植物种质资源是遗传改良的物质基础,随着物种多样性消失日益加剧,各国对遗传资源重要性的认知逐渐加深,相继建立了大量的种质资源库,截止2011年,全世界非原生境保存的植物收集品已达740万份(王述民等,2011)。但是种质资源的不断积累,也提高了种质资源的管理费用,增加了优异种质材料筛选和挖掘的难度。Frankel等(1984)提出核心种质的概念,用最少的资源数量和最小的遗传冗余最大限度地代表该物种最丰富的遗传多样性、结构及整个群体的地理分布。核心种质的构建为种质资源的深入评价和有效保护利用开辟了新的途径。早期构建核心种质大多采用形态及农艺性状数据,分子标记技术发明后,因其不受环境影响、精度高、稳定性好等优势,成为构建核心种质的主要手段。
目前已利用分子标记的方法在新疆野杏(Armeniaca vulgaris)(刘娟等,2015)、桂花(Osmanthus fragrans)(张维瑞等,2012)、葡萄(Vitis vinifera)(郭大龙等,2012)、柿(Diospyros kaki)(张艳芳,2010)、水稻(Oryza sativa)(王建成等,2007)等林木、花卉及农作物上建立了核心种质。本研究以887份杜仲资源为试验材料,利用9对基因组SSR引物,基于等位基因数最大化原则构建杜仲的核心种质,为杜仲种质资源的保护、利用、种质创新和建立育种群体提供依据。
1 材料与方法 1.1 试验材料试验材料为以嫁接方式保存在杜仲基因库内54个收集区的887份杜仲种质,不同材料收集区的样本数见表 1。将‘华仲1号’、‘华仲5号’-‘华仲12号’、‘华仲16号’-‘华仲18号’、‘大果1号’及‘密叶杜仲’、‘龙拐杜仲’、‘大叶杜仲’、‘小果杜仲’和‘红木杜仲’18个有特异表型或在生产中应用较广的种质作为必选材料(表 2)。于春季采集枝条顶端的幼叶装入自封塑料袋中,置于放有干冰的便携式冰箱,带回实验室超低温保存(-80 ℃)。
|
|
|
|
DNA提取按天根生化科技有限公司生产的DP320离心柱型植物基因组DNA提取试剂盒说明书进行。提取结束后,取1 μL DNA产物,用超微量紫外可见光光度计P330测量记录DNA浓度及纯度,再取2 μL DNA产物与6×Loading buffer混匀后,用1.2%的琼脂糖凝胶电泳检测。根据已报道的杜仲基因组SSR引物,筛选出扩增产物多态性高、重复性好的9对引物(表 3)用于887份杜仲种质的分子生物学分析。
|
|
引物合成及SSR检测由北京金唯智生物科技有限公司完成。SSR反应体系照参吴敏(2014)的反应体系。荧光PCR产物用ABI3730测序仪进行毛细管电泳检测,经Gene-Mapper软件分析。湖南株洲群体(23)中10003C种质基于9对引物的毛细管电泳检测结果(图 1)表明,扩增的目的片段清晰、准确、可靠。其中GEU045引物的结果呈现双峰现象,可能是由于PCR循环结束后,延伸时间不够长或延伸温度不够高,导致大部分扩增片段未能添加上额外的腺苷酸(+A),从而产生的分裂峰,而非四倍体材料或非特异扩增。该现象可以通过优化扩增条件体系(如提高终延伸温度、增加终延伸时间或选择高质量的Taq酶)加以改善,但由于毛细管电泳的信号正常,峰形也在可判读范围内,从检测结果的各项指标来看可以用于后续分析,所以未进行重新检测消除该分裂峰,而是统一选择荧光值高的片段作为毛细管电泳检测的最终结果。

|
图 1 湖南株洲群体10003C样品基于9对PCR引物扩增的毛细管电泳检测结果 Figure 1 Capillary electropherogram of 10003C from Zhuzhou, Hunan population amplified by nine primers |
1) 提出必选材料名单 根据现有资料和长期观测的结果,将曾在生产上大面积使用的、在育种中起过重要作用的、有独特利用价值的种质作为必选材料(董玉琛等,2003)。
2) 核心种质的获得 采用不分组的方法,把所有种质作为整体构建核心种质。将887份资源的毛细管电泳检测结果输入Power Core软件中,基于等位基因数最大化原则运算获得核心种质(Kim et al., 2007)。原始种质扣除核心种质后剩余的种质作为保留种质。
3) 核心种质的代表性检验 核心种质构建完成后,通过比较核心种质、保留种质、原始种质之间的遗传多样性参数及其保留比例,结合各群体相关遗传参数t检验法检验核心种质的代表性。
4) 核心种质的确认 核心种质经代表性检验后,采用主坐标分析法(PCoA)生成核心种质、原始种质样品在主坐标中的分布图,对核心种质作进一步确认。
1.3 核心种质代表性检验的评价参数及标准选择9个SSR位点检测到的全部等位基因数(n)、平均等位基因数(Na)、平均有效等位基因数(Ne)、平均Shannon指数(I)、平均Nei’s遗传多样性指数(H)、平均基因型数(Ng)、平均多态信息含量(PIC)及以上7个参数的保留比例作为验证核心种质对原始种质代表性的评价参数。PIC通过公式PIC=1-∑fij计算,式中,fij为第i个位点第j个等位基因出现的频率(Botstein et al., 1980)。其余6个遗传参数通过GenAlex6.5软件和PowerCore软件计算。评价标准:等位基因的保留比例需大于70%,其他遗传参数越大越好(Brown,2011;Franco et al., 2006;张艳芳,2010)。用SPSS16.0软件对核心种质、保留种质和原始种质的群体遗传参数进行t检验,二者差异不显著,说明构建的核心种质具有代表性。利用NTSYS-2.10e软件计算SM相似系数和进行PCoA分析,生成核心种质和原始种质在主坐标上的样品分布图,二者的分布结构越相似,构建的核心种质代表性越好。
2 结果与分析 2.1 核心种质、保留种质和原始种质的遗传多样性比较887份原始材料在Power Core软件运算得到包含18份必选材料在内的189份核心种质,取样比例为21.3%。由原始种质、核心种质、保留种质的遗传多样性结果(表 4)可知,887份种质在9个SSR位点上共扩增出107个等位基因(n),每个位点的平均等位基因数(Na)、平均有效等位基因数(Ne)、平均Shannon指数(I)、平均Nei’s遗传多样性指数(H)、平均基因型数(Ng)和平均多态信息含量(PIC)分别为11.889、5.096、1.812、0.925、46.889和0.795。核心种质包含了原始种质21.3%的样品,但保存了原始种质100%的等位基因(n)和基因型(Ng),对其他5个遗传多样性参数(Na、Ne、I、H、PIC)的保留比例分别为100%、116.5%、108.7%、101.5%、103.3%。保留种质包含了原始种质78.7%的样品,保存了原始种质86.9%的等位基因(n),对其他6个遗传多样性参数(Na、Ne、I、H、Ng、PIC)的保留比例分别为86.9%、95.7%、96.7%、99.4%、77.0%、99.0%。根据等位基因保留比例需大于70%,其他遗传参数越大越好的评价标准(Mckhann et al., 2004; Ronfort et al., 2006),本研究构建的核心种质各个遗传参数较优,符合核心种质的要求。
|
|
为进一步验证核心种质对原始种质的代表性,将核心种质和保留种质对原始种质在9个SSR位点上的平均等位基因数(Na)、平均有效等位基因数(Ne)、平均Shannon指数(I)、平均Nei’s遗传多样性指数(H)、平均基因型数(Ng)、平均多态信息含量(PIC)进行t检验(表 5),结果表明,核心种质、保留种质的6个遗传多样性参数与原始种质相比在0.01水平上差异不显著,说明构建的核心种质具有很好的代表性。
|
|
核心种质与保留种质基于6个遗传多样性参数的t检验结果表明二者在0.01水平上差异不显著(表 6),说明所构建的核心种质不仅有效反映了原始种质的遗传多样性,也没有显著影响保留种质的遗传多样性。从表 6还可以看出,核心种质的6个遗传多样性评价参数均高于保留种质,在建立杜仲育种群体时应该优先使用核心种质。
|
|
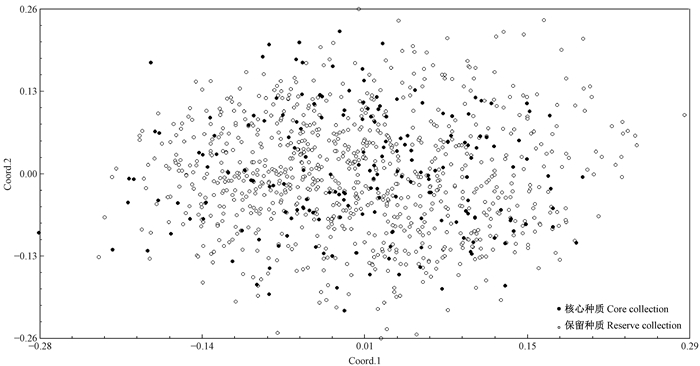
利用主坐标分析法(PCoA)对构建的核心种质进行确认。主坐标分析法是研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,找到距离矩阵中最主要的坐标,其结果是数据矩阵的一个旋转,并不改变样品点之间的相互位置关系,通过PCoA可以观察个体或群体间的差异(Jiang et al., 2013)。采用主坐标分析法(PCoA)查看核心种质样品在原始种质样品中的分布情况(图 2),结果表明:核心种质均匀分布在整个主坐标图中,较好地反映了原始样品在主坐标中的分布情况,说明所构建的核心种质具有很好的代表性。
|
图 2 核心种质和保留种质的主坐标分布 Figure 2 Principal coordinates plots of core collection and reserve collection |
数据选择。常用的构建核心种质的数据主要是形态标记数据和分子标记数据(王建成等,2008;杨汉波等,2017)。对多年生的杜仲而言,要准确测量其相关的表型数据往往需要多年的持续观测,尤其是叶、花、果和产量等经济性状数据,还需要种质保存地的立地条件、栽植年份、抚育措施等尽可能一致,以减小因环境因素引起的表型差异。因此,表型数据较难收集且易受环境等因素的影响,构建核心种质的准确性相对分子标记数据要差。分子标记以DNA多态性为基础,通常不具生物功能活性,不会或极少受植物生长期和外界环境的影响,较形态标记更适合构建核心种质和遗传多样性评价(刘新龙等,2014;杨汉波等,2017)。SSR标记是多态性最高的遗传标记之一,在基因组中分布着大量的简单重复序列,且同一个位点上重复序列的数量变化很大。因此,本研究选用9对多态性较高的SSR引物,基于等位基因数最大化策略获得189份杜仲核心材料,对杜仲资源深入研究和利用具有重要意义。
种质分组。分组的目的是为了保证取样的代表性和反映不同条件下的遗传多样性差异。在核心种质构建过程中,常将种质材料按照地理起源、植物学分类、生态区等进行分组,在分组的基础上构建核心种质(郑轶琦等,2014;李秀兰等,2013)。也有采用不分组的方法,如van Hintum等(1994)为避免具有共同亲本的种质同时选入核心种质,提出了系谱法;Hu等(2000)为避免在聚类图上选择划分切点的困难,提出了逐步聚类法;Wang等(2007)提出了最小距离逐步聚类法。杜仲是地质史上残留下来的孑遗植物,现存杜仲仅一属一种。我国历史上对杜仲的栽培利用较早,2 000多年来不同地区之间相互引种的情况一直存在。长期相互引种使杜仲地理多样性和遗传多样性的关系逐渐变得模糊,材料间地理起源的差异已不能反映其在遗传及形态上的差异。因此,常见的按植物学分类、地理起源、生态区、品种分类体系等分组的方法并不适用于杜仲分组,本研究采用了不分组的方法,将所有参试群体作为整体构建核心种质,以等位基因数最大化为标准构建杜仲的核心种质,最终用189份种质保存了887份原始材料中检测到的全部的等位基因,达到了精简杜仲基因库的目的。
构建方法。自核心种质的概念被提出以来,很多学者对其构建方法给出了不同的建议,如等位基因数目最大化法(张艳芳,2010)、基于遗传距离的聚类法(何建文等,2015)、稀有等位基因优先法(刘娟等,2015)等。迄今为止,等位基因数最大化法是其中最具优势的一种方法,已在一些模式植物和农作物的核心种质构建过程中被采用,如拟南芥(Arabidopsis thaliana)(Brown,2011)、葡萄(郭大龙等,2012)等。此策略的最大优点在于能够保存遗传多样性最为丰富的种质资源,满足分类学家和遗传学家的需要(Marita et al., 2000)。Power Core软件基于等位基因数目最大化策略构建核心种质的有效性已在葡萄、柿等作物和林木上得到验证和应用(郭大龙等,2012;张艳芳,2010)。因此,本研究采用等位基因数最大化法在Power Core软件中获得杜仲核心种质,核心种质对原始种质中7个遗传参数的保留比例均在100%及以上,保留效果较好。
评价参数及代表性检验。代表性是核心种质最重要的性质,常用的核心种质遗传多样性评价参数有:等位基因数、基因型数、Shannon多样性指数和Nei’s基因多样性指数。等位基因数被认为是最相关的指标(Mousadik et al., 1996; Petit et al., 1998),等位基因数最大化意味着保存了遗传多样性最为丰富的种质资源。王建成等(2007)在研究水稻核心种质的评价参数时认为平均Shannon多样性指数、平均多态信息含量和平均Simpson指数是评价核心种质代表性的重要参数。也有学者将多态位点百分率、观测等位基因数、有效等位基因数等参数用于核心种质的遗传多样性评价(李谋强等,2015)。综合前人的研究结果,本研究选择等位基因数、平均等位基因数、平均有效等位基因数、平均Shannon多样性指数、平均Nei’s基因多样性指数、平均基因型数和平均多态信息含量7个参数及其保留比例作为核心种质代表性的评价参数,结合不同群体相关遗传参数的t检验法和主坐标分析法(PCoA)对构建的核心种质代表性进行验证和确认,效果较好。保留比例是核心种质各个遗传参数占原始种质各个遗传参数的百分比(赵冰,2008;张艳芳,2010)。本研究中,核心种质和原始种质基于9个SSR位点的等位基因数、平均等位基因数、平均基因型数3个参数的保留比例为100%;平均有效等位基因数、平均Shannon指数、平均Nei’s遗传多样性指数、平均多态信息含量4个参数的保留比例大于100%,这主要是由于群体内样本数和等位基因频率改变引起的。这些遗传参数均基于等位基因频率按不同的方法估算获得,用于度量不同群体(原始种质、核心种质、保留种质)在多样性上的遗传冗余情况,而核心种质的构建过程是一个减少出现频率高的等位基因、增加稀有等位基因比例的过程,在去除遗传冗余的过程中,各等位基因频率的不规律增减易导致相应遗传多样性参数保留比例大于100%,这一现象在新疆野杏(刘娟等,2015)、蜡梅(Chimonanthus praecox)(赵冰,2008)、柿(张艳芳,2010)和桂花(张维瑞等,2012)等的核心种质构建中均存在。根据等位基因保留比例需大于70%,其他遗传参数越大越好的评价标准(Mckhann et al., 2004; Ronfort et al., 2006),在等位基因没有丢失的情况下,本研究构建的核心种质各个遗传参数较优,符合核心种质的要求,对原始种质的遗传多样性保留效果较好。
4 结论887份杜仲种质资源基于等位基因数目最大化原则得到189份核心种质,包含了原始种质21.3%的样品,保存了原始种质100%的等位基因和基因型。核心种质与原始种质、保留种质的6个遗传多样性参数(平均等位基因数、平均有效等位基因数、平均Shannon多样性指数、平均Nei’s基因多样性指数、平均基因型数和平均多态信息含量)经t检验,在0.01水平上差异不显著;主坐标分析也表明,核心种质与原始种质在分布图上有着相似的分布结构。以上说明构建的核心种质具有很好的代表性,可为杜仲优异基因资源发掘、育种群体建立和新品种选育提供参考,获得的189份核心种质可用于营建杜仲核心种质保存库。
董玉琛, 曹永生, 张学勇, 等. 2003. 中国普通小麦初选核心种质的产生[J]. 植物遗传资源学报, 4(1): 1-8. (Dong Y C, Cao Y S, Zhang X Y, et al. 2003. Establishment of candidate core collections in Chinese common wheat germplasm[J]. Journal of Plant Genetic Resources, 4(1): 1-8. [in Chinese]) |
杜红岩. 2013. 中国杜仲橡胶资源与产业发展报告[M]. 北京: 社会科学文献出版社: 1-25. (Du H Y. 2013. Report on development of China's Eucommia rubber resources and industry[M]. Beijing: Social Sciences Academic Press: 1-25. [in Chinese]) |
郭大龙, 刘崇怀, 张君玉, 等. 2012. 葡萄核心种质的构建[J]. 中国农业科学, 45(6): 1135-1143. (Guo D L, Liu C H, Zhang J Y, et al. 2012. Construction of grape core collections[J]. Scientia Agricultura Sinica, 45(6): 1135-1143. [in Chinese]) |
何建文, 韩世玉. 2015. 基于SSR标记不同距离聚类与抽样方法构建辣椒核心种质库[J]. 西南农业学报, 28(5): 2199-2204. (He J W, Han S Y. 2015. Construction of core capsicum germplasm bank based on different distance clustering and sampling method of SSR marker[J]. Southwest China Journal of Agricultural Sciences, 28(5): 2199-2204. [in Chinese]) |
李谋强, 师桂英, 叶树辉, 等. 2015. 基于ISSR分子标记数据的兰州百合核心种质构建方法研究[J]. 中国沙漠, 35(6): 1573-1578. (Li M Q, Shi G Y, Ye S H, et al. 2015. Methods of establishing Lanzhou lily core collection based on ISSR DNA markers[J]. Journal of Desert Research, 35(6): 1573-1578. DOI:10.7522/j.issn.1000-694X.2015.0049 [in Chinese]) |
李秀兰, 贾继文, 王军辉, 等. 2013. 灰楸形态多样性分析及核心种质初步构建[J]. 植物遗传资源学报, 14(2): 243-248. (Li X L, Jia J W, Wang J H, et al. 2013. Morphological diversity analysis and preliminary construction of core collection of Catalpa fargesii Bur[J]. Journal of Plant Genetic Resources, 14(2): 243-248. [in Chinese]) |
刘娟, 廖康, 赵世荣, 等. 2015. 利用ISSR分子标记构建新疆野杏核心种质资源[J]. 中国农业科学, 48(10): 2017-2028. (Liu J, Liao K, Zhao S R, et al. 2015. The core collection construction of Xinjiang wild apricot based on ISSR molecular markers[J]. Scientia Agricultura Sinica, 48(10): 2017-2028. DOI:10.3864/j.issn.0578-1752.2015.10.014 [in Chinese]) |
刘新龙, 刘洪博, 马丽, 等. 2014. 利用分子标记数据逐步聚类取样构建甘蔗杂交品种核心种质库[J]. 作物学报, 40(11): 1885-1894. (Liu X L, Liu H B, Ma L, et al. 2014. Construction of sugarcane hybrids core collection by using stepwise clustering sampling approach with molecular marker data[J]. Acta Agronomica Sinica, 40(11): 1885-1894. [in Chinese]) |
王建成, 胡晋, 张彩芳, 等. 2007. 建立在基因型值和分子标记信息上的水稻核心种质评价参数[J]. 中国水稻科学, 21(1): 51-58. (Wang J C, Hu J, Zhang C F, et al. 2007. Evaluating parameters of rice core collections based on genotypic values and molecular marker information[J]. Chinese Journal of Rice Science, 21(1): 51-58. [in Chinese]) |
王建成, 胡晋, 黄歆贤, 等. 2008. 植物遗传资源核心种质新概念与应用进展[J]. 种子, 27(5): 47-50. (Wang J C, Hu J, Huang X X, et al. 2008. New concept and application on core collection of plant germplasm resources[J]. Seed, 27(5): 47-50. [in Chinese]) |
王述民, 张宗文. 2011. 世界粮食和农业植物遗传资源保护与利用现状[J]. 植物遗传资源学报, 12(3): 325-338. (Wang S M, Zhang Z W. 2011. The state of the world's plant genetic resources for food and agriculture[J]. Journal of Plant Genetic Resources, 12(3): 325-338. [in Chinese]) |
吴敏. 2014. 杜仲全基因组SSR标记开发及遗传多样性评价. 北京: 中国林业科学研究院硕士学位论文. (Wu M. 2014. Development of genomics SSR markers and genetic diversity evaluation of Eucommia ulmoides Oliv. Beijing: MS thesis of Chinese Academy of Forestry. [in Chinese]) |
杨汉波, 张蕊, 王帮顺, 等. 2017. 基于SSR标记的木荷核心种质构建[J]. 林业科学, 53(6): 37-46. (Yang H B, Zhang R, Wang B S, et al. 2017. Construction of core collection of Schima superba based on SSR molecular markers[J]. Scientia Silvae Sinicae, 53(6): 37-46. DOI:10.11707/j.1001-7488.20170605 [in Chinese]) |
张维瑞, 袁王俊, 尚富德. 2012. 基于AFLP分子标记的桂花品种核心种质的构建[J]. 西北植物学报, 32(7): 1349-1354. (Zhang W R, Yuan W J, Shang F D. 2012. Development of core collection of Osmanthus fragran s Lour. cultivars based on AFLP molecular markers[J]. Acta Botanica Boreali-Occidentalia Sinica, 32(7): 1349-1354. [in Chinese]) |
张艳芳. 2010. 利用形态学和SSR标记建立柿核心种质. 武汉: 华中农业大学博士学位论文. (Zhang Y F. 2010. Development of core collection for Japanese persimmon from China based on morphological and SSR markers. Wuhan: PhD thesis of Huazhong Agricultural University. [in Chinese]) |
赵冰. 2008. 蜡梅种质资源遗传多样性与核心种质构建的研究. 北京: 北京林业大学博士学位论文. (Zhao B. 2008. Studies on genetic diversity and construction of core collection of Chimonanthus praecox. Beijing: PhD thesis of Beijing Forestry University. [in Chinese]) |
郑轶琦, 郭琰, 房淑娟, 等. 2014. 利用表型数据构建狗牙根初级核心种质[J]. 草业学报, 23(4): 49-60. (Zheng Y Q, Guo Y, Fang S J, et al. 2014. Constructing pre-core collection of Cynodon dactylon based on phenotypic data[J]. Acta Prataculturae Sinica, 23(4): 49-60. DOI:10.11686/cyxb20140406 [in Chinese]) |
Botstein D, White R L, Skolnick M, et al. 1980. Construction of genetic linkage map in man using restriction fragment length polymorphism[J]. American Journal of Human Genetics, 32(3): 314-331. |
Brown A H D. 2011. Core collections:a practical approach to genetic resources management[J]. Genome, 31(2): 818-824. |
Franco J, Crossa J, Warburton M L, et al. 2006. Sampling strategies for conserving maize diversity when forming core subsets using genetic markers[J]. Crop Science, 46(2): 854-864. DOI:10.2135/cropsci2005.07-0201 |
Frankel O, Brown A. 1984. Plant genetic resources today: a critical appraisal//Holden J H W, Williams J T. Crop genetic resources: conservation and evaluation. London: George Allen and Unwin, 249-257.
|
Hu J, Zhu J, Xu H M. 2000. Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J]. Theoretical and Applied Genetics, 101: 264-268. DOI:10.1007/s001220051478 |
Jiang X T, Peng X, Deng G H, et al. 2013. Illumina sequencing of 16S rRNA tag revealed spatial variations of bacterial communities in a mangrove wetland[J]. Microbial Ecology, 66(1): 96-104. DOI:10.1007/s00248-013-0238-8 |
Kim K W, Chung H K, Cho G T, et al. 2007. Power Core:a program applying the advanced M strategy with a heuristic search for establishing core sets[J]. Bioinformatics, 23(16): 2155-2162. DOI:10.1093/bioinformatics/btm313 |
Marita J M, Rodriguez J M, Nienhuis J. 2000. Development of an algorithm identifying maximally diverse core collections[J]. Genetic Resources and Crop Evolution, 47(5): 515-526. DOI:10.1023/A:1008784610962 |
Mckhann H I, Camilleri C, Bérard A, et al. 2004. Nested core collections maximizing genetic diversity in Arabidopsis thaliana[J]. Plant Journal for Cell & Molecular Biology, 38(1): 193-202. |
Mousadik A E, Petit R J. 1996. High level of genetic differentiation for allelic richness among populations of the argan tree (Argania spinosa (L.) Skeels) endemic to Morocco[J]. Theoretical and Applied Genetics, 92(7): 832-839. DOI:10.1007/BF00221895 |
Petit R J, Mousadik A E, Pons O. 1998. Identifying populations for conservation on the basis of genetic markers[J]. Conservation Biology, 12(4): 844-855. DOI:10.1046/j.1523-1739.1998.96489.x |
Ronfort J, Bataillon T, Santoni S, et al. 2006. Microsatellite diversity and broad scale geographic structure in a model legume:building a set of nested core collection for studying naturally occurring variation in Medicago truncatula[J]. BMC Plant Biology, 6(1): 28-41. DOI:10.1186/1471-2229-6-28 |
van Hintum T J, Haalman D. 1994. Pedigree analysis for composing a core collection of modern cultivars, with examples from barley (Hordeum vulgare s. lat.)[J]. Theor Appl Genet, 88(1): 70-74. DOI:10.1007/BF00222396 |
Wang J C, Hu J, Xu H M, et al. 2007. A strategy on constructing core collections by least distance stepwise sampling[J]. Theoretical and Applied Genetics, 115(1): 1-8. DOI:10.1007/s00122-007-0533-1 |
 2018, Vol. 54
2018, Vol. 54

