文章信息
- 包文泉, 乌云塔娜, 杜红岩, 李铁柱, 刘慧敏, 王淋, 白玉娥
- Bao Wenquan, Wuyun Tana, Du Hongyan, Li Tiezhu, Liu Huimin, Wang Lin, Bai Yu
- 基于SSR标记的西藏光核桃群体遗传多样性和遗传结构分析
- Genetic Diversity and Population Structure of Amygdalus mira in the Tibet Plateau in China Based on SSR Markers
- 林业科学, 2018, 54(2): 30-41.
- Scientia Silvae Sinicae, 2018, 54(2): 30-41.
- DOI: 10.11707/j.1001-7488.20180204
-
文章历史
- 收稿日期:2017-10-25
- 修回日期:2017-11-26
-
作者相关文章
2. 中国林业科学研究院经济林研究开发中心 郑州 450003
2. Non-Timber Forest Research and Development Center, Chinese Academy of Forestry Zhengzhou 450003
群体遗传结构的研究对物种的保护、利用和遗传改良具有重要的理论和实践意义(Millar et al., 1992;Tana et al., 2015)。其中,遗传多样性是遗传结构的核心,在揭示物种的演化历史、进化潜力和探讨物种稀有或濒危原因方面具有重要作用(Hedrick,2004;杨梅等,2014);同时,物种有效保护策略的制定,如优先保护种群的确定、有效的取样策略以及近交衰退种群的恢复策略等要依赖于遗传多样性和遗传结构的分析(Millar et al., 1992)。
光核桃(Amygdalus mira)(2n=2x=16)原产自中国西藏,属蔷薇科(Rosaceae)桃属(Amygdalus),多年生落叶乔木,是桃(A.persica)野生近缘种中唯一子叶出土的原始种(王力荣等,2012;谭江平等,2012)。光核桃寿命可达千余年,是国内外珍贵的桃种质资源“活化石群”。其树体高大,早春先叶开花,花色美,多为白色或粉红色(Guan et al., 2014;刘丽等,2013);果实营养丰富,含糖量高达11.6%,Vc、钙、铁等营养成分含量也很高;核仁富含油酸、亚油酸、硬脂酸和棕榈酸,具有极高的食用价值,还可用于治疗月经不调、跌打损伤、瘀血作痛诸症等,是食品、化妆品和制药工业等的重要原料(张姗姗等,2016;邢春等,2013)。另外光核桃具有抗逆性强、结果力高、适应性强等优良特性,可为桃种质的抗旱、抗病育种及改良提供重要的遗传基础(Yan et al., 2015)。
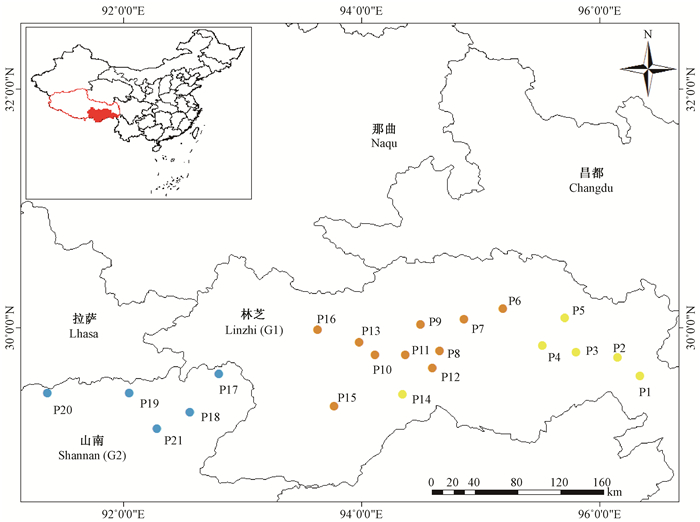
西藏光核桃主要分布于雅鲁藏布江河谷及其支流帕隆藏布和尼洋河流域的20个县境内,其中,雅鲁藏布江中下游的林芝和山南地区最为集中(Guan et al., 2014;刘丽等,2013)。山南地区平均海拔3 700 m,属于典型的藏南谷地,具有独特的高原温带干旱性气候(白淑英等,2015),该地区光核桃资源年龄结构稳定,开花期3月15日至3月27日;而林芝地区平均海拔3 000 m,地貌复杂,东南部以高山峡谷为主,北部存留有较完整的高原宽谷地貌和高原丘陵地貌,在气候类型上,东南部属于热带、亚热带山地季风湿润气候,西部为高原温带季风半湿润气候,东北部为高原温带季风湿润气候(葛立雯等,2013),该区光核桃资源年龄结构处于衰退阶段,树龄均高于10年,开花期3月7日至3月20日。
近年来,西藏地区过度开垦、基础设施修建以及乱砍滥伐,造成光核桃生境严重破坏,使其分布范围和数量逐年减少。同时,光核桃果实遭人为过度采摘和野生动物抢食,造成种群无法更新,面临濒危状况(张姗姗等,2016;邢春等,2013)。因此,研究西藏光核桃遗传多样性和遗传结构对光核桃资源合理保护与利用具有重要意义。传统的遗传多样性分析主要利用形态指标,但获得形态特征的周期较长,且易受环境及基因显隐性的影响(曾秀丽,2016),而随着分子生物学技术的快速发展和不断完善,分子标记已被普遍应用于群体遗传多样性和遗传结构等研究中(陈新民等,2003;Bertini et al., 2006),如SSR(simple sequence repeat)、RFLP(restriction fragment length polymorphism)、RAPD(random amplified polymorphism DNA)、AFLP(amplified fragment length polymorphism)和ISSR(inter simple sequence repeat)等(Bovine et al., 2009;Hagen et al., 2002;Sánchez-Pérez et al., 2006;Vicente et al., 1998),其中SSR分子标记具有操作简单、多态性高、覆盖面广、共显性、重复性高等优点,并且在近缘物种间具有很高的通用性,能够很好地满足遗传多样性等研究要求(Kijas et al., 1995;Du et al., 2012)。目前,SSR标记已用于光核桃遗传多样性等研究中(Mu et al., 2015;Chun et al., 2015)。但以往的研究所涉及的SSR标记和种群数量太少,缺乏代表性。因此,本文选用25个SSR分子标记对西藏地区21个光核桃天然群体进行研究,拟通过大规模样本间遗传多样性及遗传结构的详细比较和分析,揭示西藏光核桃的遗传多样性地理分布特点和群体间的遗传结构,为光核桃资源可持续利用提供科学依据。
1 材料与方法 1.1 试验材料2013年7—8月间,对西藏光核桃自然分布区进行全面的野外调查和采集,收集到21个天然群体共420个单株,具体样品分布及信息见图 1和表 1。采集新鲜、健康的叶片,立即放入预装变色硅胶的自封袋内干燥保存,用于DNA的提取。每个样品进行GPS定位,相邻株间距至少50 m。基于行政区地理分布可将调查的21个群体分为2组,分别为林芝地区(G1)和山南地区(G2),G1组包括P1-P16群体,其中P1-P5、P14分布于低海拔区(2 921~2 979 m),而P6-P13、P15、P16分布于中海拔区(3 111~3 394 m);G2组包括剩余的5个群体(P17-P21),分布于高海拔区(3 661~3 844 m)(表 1)。供试21个群体均处于野生状态,但由于西藏地区的快速开发和修建,有的群体位于农户周围或公路边,其中P7、P9和P12在农户周围,而P6、P10、P13和P15处于公路边;另外群体P1-P5位于雅鲁藏布江大峡谷自然保护区内。
|
|
采用改良的CTAB法(冯晨静等,2005)提取光核桃基因组DNA,1%的琼脂糖凝胶电泳及微量分光光度计检测其纯度后,将浓度调至20 ng·μL-1,-20 ℃保存备用。
1.3 SSR扩增采用25对多态性高、特异性强、重复性高的SSR引物(表 2),对光核桃种质进行遗传多样性分析。PCR扩增反应体系为20 μL的混合体系,由0.2 μL的2×Taq Plus Master Mix,1 μL的DNA模板(10~20 ng·μL-1),2 μL的10×buffer,0.5 μL的正向引物和反向引物(0.8 μmol·L-1),0.5 μL的dNTP(0.25 mol·L-1),0.5 μL的DMSO(10%)和14.8 μL的无菌去离子水(ddH2O)组成;PCR反应程序为:95 ℃预变性5 min;95 ℃变性30 s,按各引物退火温度(50 ~ 55 ℃)退火30 s,72 ℃延伸30 s,30次循环;72 ℃终延伸5 min。PCR扩增产物经由ABI 3500XL(Applied Biosystems,USA)DNA分析仪进行检测,利用Gene-Marker软件(Soft Genetics LLC,USA)读取每个位点的片段大小。
|
|
将检测到的SSR片段大小,录入Excel表格中,采用Cervus 3.0.7软件(Kalinowski et al., 2007)计算各SSR位点的无效等位基因(null alleles)频率。采用GenAlEx 6.501 (Peakall et al., 2006)软件计算等位基因数(Na)、有效等位基因数(Ne)、期望杂合度(He)、观察杂合度(Ho)、Shannon’s多样性指数(I)、群体内部近交系数(F)、等位基因频率、特有等位基因数、Nei’s遗传距离(D)和基因流(Nm)。为了衡量群体遗传分化程度,利用GenAlEx 6.501统计各位点的近交系数(FIS)和遗传分化系数(FST)等F-统计量(Nei,1973)。
基于Arlequin v3.1(Raffaele et al., 2000)软件检测群体的Hardy-Weinberg(HWE)平衡,并计算SSR引物的多态性信息含量(PIC)值,评价所用25对SSR引物在光核桃遗传多样性分析中的多态性信息指数,其值越大,表明多态性越大,越适合遗传多样性研究。
利用STRUCTURE(Pritchard et al., 2000)软件的贝叶斯聚类法对西藏光核桃群体遗传结构进行分析;将类群数K设置为2 ~ 21,开始时不作数迭代(length of burn-in period)和MCMC(Markov Chain Monie Carfo)值均设为100 000次,重复运行20次。采用ΔK值最大原则选取最佳K值(Evanno et al., 2005),结果由STRUCTURE Harvester(Earl et al., 2012)在线(http://taylor0.biology.ucla.edu/struct_harvest/)计算,并通过CLUMPP(Mattias et al., 2007)软件重复抽样分析,由Distruct软件(Noah et al., 2002)绘制群体遗传结构图。
为了估计群体间及群体内部的遗传差异,使用Arlequin v3.1(Raffaele et al., 2000)软件对物种水平的遗传变异进行分子方差分析(AMOVA)。为了检验西藏光核桃天然群体间是否存在地理隔离效应及群体间遗传变异是否受海拔梯度的影响,采用GenAlEx 6.501 (Peakall et al., 2006)软件进行Mantel检测(Zhe et al., 2014),分析其遗传距离与地理距离、海拔梯度的相关性。采用NTSYS pc version 2.10(Rohlf,1994)软件,以Nei’s遗传距离(D)(Nei,1973)作为参数,完成非加权成组配对算术平均法(UPGMA)的树状聚类分析。主坐标分析(PCoA)以Nei’s遗传距离为参数,用GenAlEx软件(Peakall et al., 2006)完成。
2 结果与分析 2.1 SSR位点的多样性25对SSR引物中,有6对引物的无效等位基因(null alleles)频率为正值,但其频率均小于0.2,表明供试25对引物在后续遗传多样性分析中的结果准确可靠(表 2)。25对SSR引物在420份光核桃样品中,共检测到214个等位基因,平均每位点8.6个等位基因(Na)和3.6个有效等位基因(Ne)(表 2)。Shannon’s信息指数(I)变异范围为0.18 ~ 2.07,平均为1.19。物种水平上的观察杂合度(Ho)和期望杂合度(He)分别为0.46和0.52。25个SSR位点的F统计量检测显示,西藏光核桃的FST为0.15,FIS为0.13,说明西藏光核桃遗传分化程度适中。各位点多态信息含量指数(PIC)在0.32 ~ 0.82之间,平均为0.62,表明所选SSR引物均能有效揭示光核桃遗传多样性。
2.2 光核桃群体遗传多样性21个光核桃天然群体遗传多样性分析结果(表 3)显示,各群体遗传多样性水平存在差异。其中,群体P17遗传多样性最高,而群体P18和P19遗传多样性最低。在21个群体中,除P4、P17和P20群体外其余的18个群体均含有特有等位基因,特有等位基因数为1 ~ 4,其中P18和P19群体较特殊,具有的特有等位基因数最多(4和3),然而遗传多样性却最低。
|
|
西藏光核桃各群体内近交系数(F)均大于零(0.06 ~ 0.26),平均为0.17。Hardy-Weinberg平衡(HWE)检测显示,有10个群体(P1、P6、P7、P9、P10、P12、P13、P15、P18和P19)显著偏离于HWE。
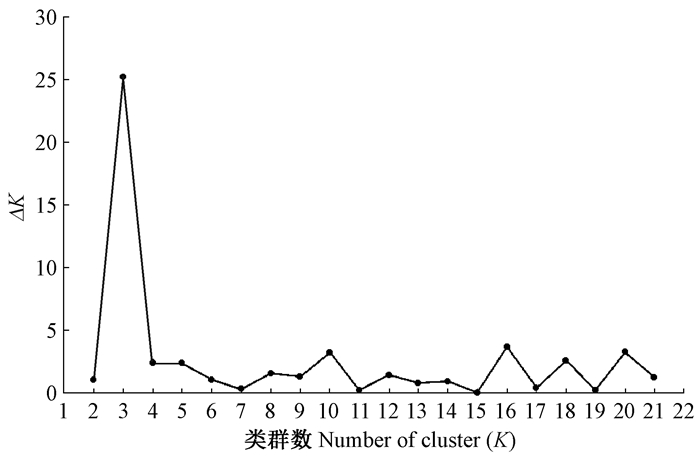
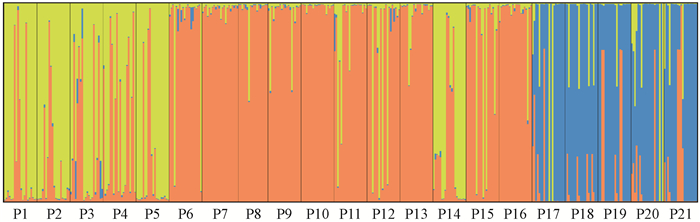
2.3 光核桃群体遗传关系和遗传结构特征利用STRUCTURE分析西藏光核桃群体遗传结构,当K=3时ΔK值为最大(图 2),表明420份光核桃材料具有3个不同类群(图 3),虽然类群间存在一定的遗传混杂(基因交流现象),但仍存在较明显的分化。对420个个体的Q(即第i材料基因组变异源于第K群体的概率)值进行统计,有394个(占93.8%)个体遗传组分相对单一(Q>0.6),并被划分到相应的3个类群中。第1类群(黄色基因池)包括6个低海拔区群体(P1、P2、P3、P4、P5和P14),其中有7个个体(5.8%)Q值小于0.6;有15个个体来自其他类群被划分至该组,包括P6、P11、P17和P20的各2份,P12的4份以及P15的3份。第2类群(橙色基因池)包括10个中海拔区群体(P6、P7、P8、P9、P10、P11、P12、P13、P15和P16),有11个个体(3.4%)Q值小于0.6,有33个个体来自其他类群被划分至该组,包括P1和P21的各3份,P2和P5的各2份,P3和P4的各6份,P14的5份,P17和P20的各1份,P19的4份。第3类群(蓝色基因池)包括5个高海拔区群体(P17、P18、P19、P20和P21),有12个个体(12%)的遗传组分较混杂,Q值小于0.6。
|
图 2 最佳类群数K与推断值ΔK的关系b Figure 2 Relations between the rational groups number K and estimated value ΔK |
|
图 3 基于STRUCTURE分析的西藏光核桃21个群体的遗传结构 Figure 3 Genetic structural map of 21 wild populations of Amygdalus mira in Tibet based on STRUCTURE analysis 图中相同的颜色代表同一个类群;黄、橙和蓝色分别代表类群Ⅰ、Ⅱ和Ⅲ。 The same color indicates the same group; Yellow, orange and blue in figure represent the groupⅠ, Ⅱ and Ⅲ, respectively. |
为了进一步探讨西藏光核桃群体间的遗传关系,分别利用21个群体和420份个体的Nei’s遗传距离进行UPGMA聚类分析(图 4A)和主坐标分析(PCoA)(图 4B),其结果与STRUCTURE划分结果基本一致,均将供试群体分为3组,大部分地理位置相近、海拔高度相近的群体依次划分至同一类群中。这与Mantel检测结果相吻合,即西藏光核桃群体遗传距离与地理距离(r=0.50,P<0.01)(图 5A)、海拔梯度(r=0.61,P<0.01)(图 5B)呈显著正相关。表明西藏光核桃群体间存在地理距离和海拔梯度的遗传变异,具有明显的地理区域特性,群体间存在地理隔离效应。

|
图 4 基于遗传距离的光核桃21个群体的UPGMA聚类分析(A)及420个个体的主坐标分析(B) Figure 4 UPGMA dendrogram (A) based on genetic distance of the 21 populations of Amygdalus mira and Principal coordinates analysis (B) based on genetic distance of 420 individuals of A. mira |

|
图 5 供试光核桃群体的遗传距离与地理距离(A)、海拔梯度(B)的相关性 Figure 5 Correlation between genetic distance and geographic distance (A), geographic altitudes (B) of all sampled populations of Amygdalus mira |
通过分子方差分析(AMOVA)(表 4)可知,西藏光核桃遗传变异主要来自群体内个体间(83.7%),群体间的遗传变异占16.3%。检测各群体间的遗传分化(FST)和基因流(Nm)(表 5)可知,21个光核桃群体间的遗传分化系数为0.01 ~ 0.36,基因流水平为0.44 ~ 20.33,其中相同遗传组群体间遗传分化较小,基因交流较频繁,而来自不同遗传组群体间的分化较大,基因交流较少,这与划分结果相吻合;21个群体中有4对群体间的FST值较小,分别为P10-P11(0.02)、P10-P13(0.02)、P12-P15(0.01)和P18-P21(0.01)。3个遗传组中C1-C3(0.14)和C2-C3(0.13)的遗传分化程度较高。
|
|
|
|
遗传多样性是物种长期生存和保持进化的基础(Nei,1978;Botstein et al., 1980)。一般而言,特有种、濒危种的遗传多样性水平较低(Hamrick et al., 1996),但也有些保持较高水平的遗传多样性(Richter et al., 1994)。本研究中,25个SSR位点的平均He和Ho分别为0.52和0.46;21个群体平均He和Ho分别为0.52和0.44,高于此前利用SSR标记分析西藏光核桃的遗传多样性[He=0.340 (Mu et al., 2015); He=0.352 (Chun et al., 2015)]以及同属的桃(He=0.46,Ho=0.34)(Maria et al., 2010)和扁桃(Amygdalus communis)(Ho=0.339,He=0.219)(Tahan et al., 2009),而低于同一生活史的一些物种,如西藏核桃(Juglans regia)(He=0.605)和西藏泡核桃(Juglans sigillata)(He=0.678)(王滑,2010),表明西藏光核桃属于中度杂合物种,遗传多样性处于中等水平。
光核桃属于多年生、虫媒花、自交不亲和植物,广泛分布于青藏高原,能够适应各种恶劣环境(王力荣等,2012;Guan et al., 2014),其遗传多样性水平应与多数学者认为的分布广、适应性强、多年生、异交物种的遗传多样性水平较高的特点(Hamrick,1990;刘华波等,2012)相契合;然而,本研究中,西藏光核桃遗传多样性处于中等水平,究其原因,可能是光核桃寿命较长,分布范围较为广泛,长期的演化使其沉积了丰富的遗传变异,只是后期人为乱砍滥伐造成光核桃生境的破坏,分布逐渐呈现片段化,但片段化时间较短,遗传多样性还未丧失。
生境片段化是影响群体遗传结构和遗传多样性的重要因素,生境片段化会降低有效种群大小,使物种的适应能力下降,引起有些等位基因的消失,而降低群体的多样性(Templeton et al., 1990;Saunders et al., 1991;Young et al., 1996)。作者在调查时发现,西藏光核桃各群体的生境差异较大,部分群体在其分布区内形成优势种(如P11和P17),分布连续,数量较多;有些群体则生境退化或遭人为破坏严重,导致个体数量减少,种群分布呈现片段化(如P7、P8、P13、P15、P18和P19),后者必然会限制群体间和群体内部的基因流,降低其遗传多样性水平,并会加剧群体遗传分化。通过Hardy-Weinberg平衡(HWE)检验显示,有10个群体显著偏离于HWE,其中有7个群体是位于人类活动较频繁的农户周围或公路边,表明西藏光核桃受人为活动的干扰比较明显。另一方面,种群规模的下降也会使种群的近交频率增加,从而降低该物种遗传多样性及生存能力(Emerson,2002;Yao et al., 2011)。通常异花授粉植物不接受自花授粉或近亲交配,但因生境片段化或地理因素,花粉传播被限制,随机交配系统被打破,群体中容易产生近亲繁殖(Murawski et al., 1991;1992)。本研究结果也表明21个群体近交系数均大于0(0.06 ~ 0.26),群体内均存在近亲繁殖,使物种缺乏足够的杂合子(Ho<He)来维持高水平的遗传多样性。类似结果同样发现于其他树种中,如中国野生山杏(Armeniaca sibirica)由于群体内存在较高的近亲繁殖,表现杂合子不足(Zhe et al., 2014);山茱萸(Cornus officinalis)群体内存在较多的半同胞间的交配,导致山茱萸群体杂合子不足(Hadziabdic et al., 2012)。综上可知,西藏光核桃群体遗传多样性处于中等水平,这主要与西藏光核桃生境片段化、自身生物学特性及悠久的演化历史相关。
3.2 西藏光核桃群体遗传结构遗传分化系数(FST)是反映群体遗传结构的重要指标(Hamrick et al., 1996)。本研究中,AMOVA分析(FST=0.16)显示,西藏光核桃遗传分化水平适中,遗传变异主要来自于群体内个体间。这与谭江平等(2012)研究结果相一致,也与多年生木本植物的遗传变异规律相符(Yeh et al., 1995;Hamrick et al., 1996;Gillies et al., 1999),即群体内遗传变异水平较高,而群体间遗传变异水平较低。相比于其他一些近缘种,山杏遗传分化系数(FST)为0.06(Zhe et al., 2014)、豆梨(Pyrus calleryana)的FST为0.088 (Mitsui et al., 2010)、秋子梨(Pyrus ussuriensis)的FST为0.072 (Tana et al., 2015)等,西藏光核桃表现较高的遗传分化,不仅山南地区和林芝地区间存在较为明显的差异,林芝区域群体间也表现明显的遗传分化。造成这一结果的可能原因是西藏地区高山环绕,峡谷深切,山川骈列,地形十分复杂,使物种区域分布的山脉阻隔特点突出,导致各居群间的隔离(万雪琴等,2009);因此,山南、林芝地区之间较大的遗传差异可能源自中部地区山川及河流地势所产生的地理隔离。另外,光核桃群体间的海拔差异较大(2 921~3 844 m)。一般情况下,尤其是高山地区,海拔差异构成不同的水、光、热、土壤梯度,使植物生长的自然地理环境多样化,也使植物对环境产生复杂的适应性变化而加剧群体间的遗传分化(Hirao et al., 2004;Ortego et al., 2012)。UPGMA聚类分析和STRUCTURE群体遗传结构分析均表明,绝大部分群体均按海拔高度相近而聚为一类。如本研究中,群体P14没有与地理位置相近的P11、P12和P13等群体划分在一起,而与地理位置较远但海拔高度相近的5个群体(P1-P5)划分至一起。推测群体P1-P5和P14在类似的海拔高度,受到类似的自然选择,发生了相似的等位变异,从而表现相似的遗传结构。
3.3 西藏光核桃资源保护策略遗传多样性和遗传结构的研究是制定物种保护措施的前提条件(Newton et al., 1999)。西藏光核桃群体遗传多样性处于中等水平,但目前受人类活动的影响较严重,应及时采取保护措施,保证西藏光核桃资源的合理利用及有效保护。
本研究结果表明西藏光核桃可划分为3个保护单位(C1、C2和C3)。C1类群的5个群体位于国家自然保护区,保护有保障;今后的重点应放在C2和C3类群的保护。其中,应对C2和C3类群中少数地区遗传多样性高的群体进行优先就地保护,如C2中的P11群体、C3中的P17群体。另外,西藏光核桃受人为影响较严重,尤其人类集中的农户周围和公路边的群体遭破坏较严重,如P6、P7、P9、P10、P12、P13和P15,这些群体在保护中也应重视种群基因流,加强抚育管理。对于一些树龄较大的种质,应挂牌进行保护。同时也要迁地保护一些特异群体,避免一些特殊基因的消失,如P18和P19群体遗传多样性低却含有较多的特异等位基因。
4 结论本文利用25对SSR引物对西藏地区21个光核桃群体的420份个体进行遗传多样性及遗传结构分析,共得到214个等位基因,多态信息含量(PIC)为0.62。西藏光核桃遗传多样性适中,群体间存在地理隔离效应和海拔梯度的遗传变异。西藏光核桃受人为干扰较严重,遗传分化程度较高,且个体间的近亲繁殖较频繁。STRUCTURE分析、UPGMA分析和主坐标分析(PCoA)均将21个群体划分为3个类群,可将西藏光核桃分为3个保护单元;应限制人为活动对西藏光核桃的破坏,实施就地保护的同时,建立光核桃种质资源库,促进不同居群间的基因交流,保护西藏光核桃的遗传多样性。
白淑英, 吴奇, 史建桥, 等. 2015. 基于时间序列遥感数据的西藏山南地区植被覆盖变化特征分析[J]. 中国沙漠, 35(5): 1396-1402. (Bai S Y, Wu Q, Shi J Q, et al. 2015. Analysis on vegetation coverage change in Shannan, Tibet, China based on remotely sensed data[J]. Journal of Desert Research, 35(5): 1396-1402. DOI:10.7522/j.issn.1000-694X.2015.00032 [in Chinese]) |
陈新民, 何中虎, 史建荣, 等. 2003. 利用SSR标记进行优质冬小麦品种(系)的遗传多样性研究[J]. 作物学报, 29(1): 13-19. (Chen X M, He Z H, Shi J R, et al. 2003. Genetic diversity of high quality winter wheat varieties (lines) based on SSR markers[J]. Acta Agronomica Sinica, 29(1): 13-19. [in Chinese]) |
冯晨静, 张元慧, 徐秀英, 等. 2005. 14份杏种质的ISSR分析[J]. 河北农业大学学报, 28(5): 52-62. (Feng C J, Zhang Y H, Xu X Y, et al. 2005. Genetic diversity revealed by ISSR marker in apricot[J]. Journal of Agricultural University of Hebei, 28(5): 52-62. [in Chinese]) |
葛立雯, 潘刚, 任德智, 等. 2013. 西藏林芝地区森林碳储量、碳密度及其分布[J]. 应用生态学报, 24(2): 319-325. (Ge L W, Pan G, Ren D Z, et al. 2013. Forest carbon storage, carbon density, and their distribution characteristics in Linzhi area of Tibet, China[J]. Chinese Journal of Applied Ecology, 24(2): 319-325. [in Chinese]) |
刘华波, 王哲, 刘君, 等. 2012. 燕山山脉西伯利亚杏的遗传多样性和遗传结构[J]. 林业科学, 48(8): 68-74. (Liu H B, Wang Z, Liu J, et al. 2012. Genetic diversity and genetic structure of Siberian apricot populations in the Yan mountains[J]. Scientia Silvae Sinicae, 48(8): 68-74. DOI:10.11707/j.1001-7488.20120811 [in Chinese]) |
刘丽, 孟凡娟. 2013. 西藏光核桃果实的品质分析[J]. 安徽农业科学, 41(35): 13740-13743. (Liu L, Meng F J. 2013. Fruit quality analysis of Amygalus mira in Tibet[J]. Journal of Anhui Agricultural Sciences, 41(35): 13740-13743. DOI:10.3969/j.issn.0517-6611.2013.35.114 [in Chinese]) |
谭江平, 曾秀丽, 廖明安. 2012. 西藏光核桃自然居群遗传多样性的SRAP分析[J]. 草业学报, 21(6): 213-220. (Tan J P, Zeng X L, Liao M A. 2012. Using SRAP markers to study genetic diversity of Prunus mira Koehne in Tibet[J]. Acta Prataculturae Sinica, 21(6): 213-220. DOI:10.11686/cyxb20120628 [in Chinese]) |
万雪琴, 张帆, 钟宇, 等. 2009. 中国西南地区乡土杨树基因资源的保护与利用[J]. 林业科学, 45(4): 139-144. (Wan X Q, Zhang F, Zhong Y, et al. 2009. Conservation and application of the genetic resource of native poplars in southwest China[J]. Scientia Silvae Sinicae, 45(4): 139-144. DOI:10.11707/j.1001-7488.20090423 [in Chinese]) |
王滑. 2010. 西藏核桃种质资源遗传多样性研究[M]. .
|
王力荣, 朱更瑞, 方伟超. 2012. 中国桃种质资源[M]. 北京: 中国农业出版社: 12-15. (Wang L R, Zhu G R, Fang W C. 2012. Peach genetic resource in China[M]. Beijing: China Agriculture Press: 12-15. [in Chinese]) |
邢春, 李荣钦, 王超, 等. 2013. 光核桃栽培与生理研究进展及展望[J]. 内蒙古农业科技, 2(6): 1-2. (Xing C, Li R Q, Wang C, et al. 2013. The current situation and prospect of research on Amygdalus mira Koehne cultivation and physiology[J]. Inner Mongolia Agricultural Science and Technology, 2(6): 1-2. [in Chinese]) |
杨梅, 张敏, 师守国, 等. 2014. 武当木兰种群遗传结构的ISSR分析[J]. 林业科学, 50(1): 76-81. (Yang M, Zhang M, Shi S G, et al. 2014. Analysis of genetic structure of Magnolia sprengeri populations based on ISSR markers[J]. Scientia Silvae Sinicae, 50(1): 76-81. [in Chinese]) |
曾秀丽. 2016. 西藏野生光核桃花的多样性研究[J]. 安徽农学通报, 22(1): 12-15. (Zeng X L. 2016. Genetic diversity of flower characteristics of wild Amygdalus mira Koehne in Tibet[J]. Anhui Agricutural Science Bulletin, 22(1): 12-15. [in Chinese]) |
张姗姗, 邓岚, 曾秀丽. 2016. 西藏光核桃研究进展及展望[J]. 农学学报, 6(2): 88-91. (Zhang S S, Deng L, Zeng X L. 2016. Research progress and prospects of Amygdalus mira (Koehne)[J]. Journal of Agriculture, 6(2): 88-91. DOI:10.11923/j.issn.2095-4050.cjas15040018 [in Chinese]) |
Bertini C H C, Schuster I, Sediyama T, et al. 2006. Characterization and genetic diversity analysis of cotton cultivars using microsatellites[J]. Genetics and Molecular Biology, 29(2): 321-329. DOI:10.1590/S1415-47572006000200021 |
Botstein D, White R L, Skolnick M, et al. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 32(3): 314-331. |
Bovine H M C, Gibbs R A, Taylor J F, et al. 2009. Genome-wide survey of SNP variation uncovers the genetic structure of cattle breeds[J]. Science, 324(5926): 528-532. DOI:10.1126/science.1167936 |
Cantini C, Iezzoni A F, Lamboy W F, et al. 2001. DNA fingerprinting of tetraploid cherry germplasm using simple sequence repeats[J]. Journal of the American Society for Horticulturalence, 126(2): 205-209. |
Chun X, Yan T, Guan F C, et al. 2015. Evaluation of genetic diversity in Amygdalus mira (Koehne) Ricker using SSR and ISSR markers[J]. Plant Syst Evol, 301(3): 1-10. |
Cipriani G, Lot G, Huang W G, et al. 1999. AC/GT and AG/CT microsatellite repeats in peach[Prunus persica (L) Batsch]:isolation, characterisation and cross-species amplification in Prunus[J]. Theor Appl Genet, 99(1/2): 65-72. |
Dirlewanger E, Cosson P, Tavaud M, et al. 2002. Development of microsatellite markers in peach[Prunus persica (L.) Batsch] and their use in genetic diversity analysis in peach and sweet cherry (Prunus avium L.)[J]. Theor Appl Genet, 105(6): 127-138. |
Du Q, Wang B, Wei Z, et al. 2012. Genetic diversity and population structure of Chinese white poplar (Populus tomentosa) revealed by SSR markers[J]. J Hered, 103(6): 853-862. DOI:10.1093/jhered/ess061 |
Earl D A, Vonholdt B M. 2012. STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conservation Genetics Resources, 4(2): 359-361. DOI:10.1007/s12686-011-9548-7 |
Emerson B C. 2002. Evolution on oceanic islands:molecular phylogenetic approaches to understanding pattern and process[J]. Molecular Ecology, 11(6): 951-966. DOI:10.1046/j.1365-294X.2002.01507.x |
Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Molecular Ecology, 14(2): 2611-2620. |
Gillies A C M, Navarro C, Lowe A J, et al. 1999. Genetic diversity in Mesoamerican populations of mahogany (Swietenia macrophylla), assessed using RAPDs[J]. Heredity, 83(6): 722-732. DOI:10.1046/j.1365-2540.1999.00626.x |
Guan F C, Wang S P, Li R Q, et al. 2014. Genetic diversity of wild peach (Prunus mira Koehne kov et. Kpst) from different altitudes in the Tibetan plateau by pollen morphous and RAPD markers[J]. Hortscience, 49(8): 1017-1022. |
Hadziabdic D, Wang X, Wadl P A, et al. 2012. Genetic diversity of flowering dogwood in the Great Smoky Mountains National Park[J]. Tree Genetics & Genomes, 8(4): 855-871. |
Hagen S, Khadari B, Lambert P, et al. 2002. Genetic diversity in apricot revealed by AFLP markers:species and cultivar comparisons[J]. Theor Appl Genet, 105(2/3): 298-305. |
Hamrick J L. 1990. Isozymes and the analysis of genetic structure in plant populations//Douglas E S, Pamela S S, Theodore R D. Isozymes in Plant Biology. London: Chapman and Hall, 87-105.
|
Hamrick J L, Godt M J W. 1996. Effects of life history traits on genetic diversity in plant species[J]. Philosophical Transactions of the Royal Society, 351(1345): 1291-1298. DOI:10.1098/rstb.1996.0112 |
Hedrick P W. 2004. Recent developments in conservation genetics[J]. Forest Ecology and Management, 197(1): 3-19. |
Hirao A, Kudo G. 2004. Landscape genetics of alpine-snowbed plants:comparisons along geographic and snowmelt gradients[J]. Heredity, 93(3): 290-298. DOI:10.1038/sj.hdy.6800503 |
Kalinowski S T, Taper M L, Marshall T C. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J]. Molecular Ecology, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x |
Kijas J, Fowler J, Thomas M. 1995. An evaluation of sequence tagged microsatellite site markers for genetic analysis within Citrus and related species[J]. Genome, 38(2): 349-355. DOI:10.1139/g95-045 |
Maria J A, El-Kadri A, Werner H, et al. 2010. Genetic variation, population structure and linkage disequilibrium in peach commercial varieties[J]. BMC Genetics, 69(11): 1-13. |
Mattias J, Noah A R. 2007. CLUMPP:a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J]. Bioinformatics, 23(14): 1801-1806. DOI:10.1093/bioinformatics/btm233 |
Millar C I, Westfall R D. 1992. Allozyme markers in forest genetic conservation[J]. New Forest, 6(1-4): 347-371. DOI:10.1007/BF00120652 |
Mitsui Y, Isagi Y, Setoguchi H. 2010. Multiple spatial scale patterns of genetic diversity in riparian populations of Ainsliaea faurieana (Asteraceae) on Yakushima island, Japan[J]. Amer J Bot, 97(1): 101-110. DOI:10.3732/ajb.0800220 |
Mu P, Guan F C, Tao L, et al. 2015. Analysis of genetic relationship on Amygdalus mira (Koehne) Ricker with other peach species using simple sequence repeat (SSR)[J]. Biochemical Systematics and Ecology, 62: 98-105. DOI:10.1016/j.bse.2015.06.026 |
Murawski D A, Hamrick J L. 1991. The effect of the density of flowering individuals on the mating systems of nine tropical tree species[J]. Heredity, 67(2): 167-174. DOI:10.1038/hdy.1991.76 |
Murawski D A, Hamrick J L. 1992. The mating system of Cavanillesia platanifolia under extremes of flowering-tree density:A test of predictions[J]. Biotropica, 24(1): 99-101. DOI:10.2307/2388478 |
Nei M. 1973. Analysis of gene diversity in subdivided populations[J]. Proceedings of the National Academy of Sciences, 70(12): 3321-3323. DOI:10.1073/pnas.70.12.3321 |
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 89(3): 583-590. |
Newton A C, Allunutt T R, Gillies A C M, et al. 1999. Molecular phylogeography, intraspecific variation and the conservation of tree species[J]. Trends Ecol Evol, 14(4): 140-145. DOI:10.1016/S0169-5347(98)01555-9 |
Noah A R, Jonathan K P, James L W, et al. 2002. Genetic structure of human populations[J]. Science, 298(5602): 2381-2385. DOI:10.1126/science.1078311 |
Ortego J, Riordan E C, Gugger P F, et al. 2012. Influence of environmental heterogeneity on genetic diversity and structure in an endemic southern Californian oak[J]. Molecular Ecology, 21(13): 3210-3223. DOI:10.1111/j.1365-294X.2012.05591.x |
Peakall R, Smouse P E. 2006. GENALEX 6:genetic analysis in Excel. Population genetic software for teaching and research[J]. Molecular Ecology Notes, 6(1): 288-295. |
Pritchard J K, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data[J]. Genetics, 155(2): 945-959. |
Raffaele T, Teresa M, Guido C, et al. 2000. Microsatellite DNA in peach (Prunus persica L. Batsch) and its use in fingerprinting and testing the genetic origin of cultivars[J]. Genome, 43(3): 512-520. DOI:10.1139/g00-010 |
Richter T S, Soltis P S, Soltis D E, et al. 1994. Genetic variation within and among populations of the narrow endemic, Delphinium viridescens (Ranunculaceae)[J]. American Journal of Botany, 8(81): 1070-1076. |
Rohlf F J. 1994. NTSYS-PC: Numerical taxonomy and multivariate analysis system version 1. 80. Setauket New York: distribution by Exeter software.
|
Sánchez-Pérez R, Martínez-Gómez P, Dicenta F, et al. 2006. Level and transmission of genetic heterozygosity in apricot (Prunus armeniaca L.) explored using simple sequence repeat markers[J]. Genetic Resources and Crop Evolution, 53(4): 763-770. DOI:10.1007/s10722-004-4636-0 |
Saunders D A, Hobbs R J, Margules C R. 1991. Biological consequences of ecosystem fragmentation:A review[J]. Conservation Biology, 5(1): 18-32. DOI:10.1111/cbi.1991.5.issue-1 |
Sosinski B, Gannavarapu M, Hager L D, et al. 2000. Characterization of microsatellite markers in peach[Prunus persica (L.) Batsch][J]. Theor Appl Genet, 101(3): 421-428. DOI:10.1007/s001220051499 |
Tahan O, Geng Y, Zeng L, et al. 2009. Assessment of genetic diversity and population structure of Chinese wild almond, Amygdalus nana, using EST-and genomic SSRs[J]. Biochemical Systematics and Ecology, 37(3): 146-153. DOI:10.1016/j.bse.2009.02.006 |
Tana W Y, Hitomi A, Jingshi X, et al. 2015. Population structure of and conservation strategies for wild Pyrus ussuriensis Maxim. in China[J]. PLoS One, 10(8): e0133686. DOI:10.1371/journal.pone.0133686 |
Templeton A R, Shaw K, Routman E. 1990. The genetic consequences of habitat fragmentation[J]. Annals of the Missouri Botanical Garden, 77(1): 13-27. DOI:10.2307/2399621 |
Testolin R, Marrazzo T, Cipriani G, et al. 2000. Microsatellite DNA in peach (Prunus persica L. Batsch) and its use in fingerprinting and testing the genetic origin of cultivars[J]. Genome, 43(3): 512-520. DOI:10.1139/g00-010 |
Vicente M, Truco M, Egea J, et al. 1998. RFLP variability in apricot (Prunus armeniaca L.)[J]. Plant Breeding, 117(2): 153-158. DOI:10.1111/j.1439-0523.1998.tb01470.x |
Wang Y, Georgi L L, Zhebentyayeva T N, et al. 2002. High-throughput targeted SSR marker development in peach (Prunus persica)[J]. Genome, 45(2): 319-328. DOI:10.1139/g01-153 |
Xie H, Sui Y, Chang F Q, et al. 2006. SSR allelic variation in almond (Prunus dulcis Mill.)[J]. Theor Appl Genet, 112(2): 366-372. DOI:10.1007/s00122-005-0138-5 |
Yamamoto T, Mochida K, Imai T, et al. 2002. Microsatellite markers in peach[Prunus persica (L.) Batsch] derived from an enriched genomic and cDNA libraries[J]. Mol Ecol, 2(6): 298-301. |
Yan T, Chun X, Yuan C, et al. 2015. Evaluation of genetic diversity on Prunus mira Koehne by using ISSR and RAPD markers[J]. Biotechnology & Biotechnological Equipment, 29(6): 1053-1061. |
Yao X, Zhang J, Ye Q, et al. 2011. Fine-scale spatial genetic structure and gene flow in a small, fragmented population of Sinojackia rehderiana (Styracaceae), an endangered tree species endemic to China[J]. Plant Biology, 13(2): 401-410. DOI:10.1111/plb.2011.13.issue-2 |
Yeh F C, Chong D K X, Yang R C. 1995. RAPD variation within and among natural populations of trembling aspen (Populus tremuloides Michx.) from Alberta[J]. Journal of Heredity, 86(6): 454-460. DOI:10.1093/oxfordjournals.jhered.a111620 |
Young A, Boyle T, Brown T. 1996. The population genetic consequences of habitat fragmentation for plants[J]. Trends in Ecology & Evolution, 11(10): 413-418. |
Zhe W, Ming K, Huabo L, et al. 2014. High-level genetic diversity and complex population structure of Siberian apricot (Prunus sibirica L.) in China as revealed by nuclear SSR markers[J]. PLoS One, 9(2): e87381. DOI:10.1371/journal.pone.0087381 |
 2018, Vol. 54
2018, Vol. 54