文章信息
- 倪飞, 励文豪, 林二培, 童再康, 黄华宏.
- Ni Fei, Li Wenhao, Lin Erpei, Tong Zaikang, Huang Huahong.
- 光皮桦MYB基因的克隆及表达和调控分析
- Cloning, Expression and Regulation of MYB Genes in Betula luminifera
- 林业科学, 2018, 54(12): 70-81.
- Scientia Silvae Sinicae, 2018, 54(12): 70-81.
- DOI: 10.11707/j.1001-7488.20181208
-
文章历史
- 收稿日期:2017-07-11
- 修回日期:2018-08-21
-
作者相关文章
木材的形成主要包括5个阶段:维管形成层细胞的分裂、细胞的增大、次生壁的形成、细胞程序性凋亡和心材的形成,是一个复杂且被有序调控的生物学过程(Larson,2012)。其中,木材品质的优劣与次生壁的形成息息相关,次生壁的形成与众多代谢过程有关,比如纤维素、木质素以及半纤维素等的生物合成,已成为近几年最为活跃的研究领域之一。借助于分子遗传学、生物化学等手段,参与纤维素、木质素合成的纤维素合成酶(CesA)、咖啡酸辅酶A-3-O-甲基转移酶(CCoAOMT)、4-香豆酰辅酶A连接酶(4CL)等的编码基因(Wu et al., 2000; Kalluri et al., 2004; Nairn et al., 2005; Krauskopf et al., 2005),以及多数调控次生壁形成的MYB、NAC等转录因子(刘雪梅,2005;Groover et al., 2005; 2006; Demura et al., 2006; Karpinska et al., 2004)都相继被分离和鉴定。其中,MYB转录因子日益受到研究者的关注。该类转录因子具有非常保守的MYB结构域(R),可根据MYB结构域的个数不同分为1R-MYB、2R-MYB、3R-MYB和4R-MYB 4个亚类,其中包含2个结构域的R2R3-MYB数量最多。草本植物中的研究认为R2R3-MYB转录因子广泛参与发育和代谢的调节,特别是参与了苯丙烷类次生代谢途径和末端细胞分化的调控。近年来此亚类的一些成员也逐渐被证实通过调控木质素、纤维素合成,从而参与林木细胞次生壁的形成。毛果杨(Populus trichocarpa)PtrMYB3和PtrMYB20在功能上与拟南芥(Arabidopsis thaliana)AtMYB46和AtMYB83相似,在拟南芥中超表达这2个基因均能促进纤维素、木质素和木聚糖的生物合成,被认为是毛果杨次生壁形成转录调控的第2层次总开关(McCarthy et al., 2010)。火炬松(Pinus taeda)PtMYB4和巨桉(Eucalyptus grandis)EgMYB2在功能上也属AtMYB46和AtMYB83一类,它们分别在烟草(Nicotiana tabacum)和拟南芥中超表达后,均能导致木质素含量增加和细胞壁结构异常(Patzlaff et al., 2003;Goicoechea et al., 2005)。另有一些MYB转录因子则仅调控木质素合成相关酶基因的转录。Bomal等(2008)研究认为火炬松PtMYB1和PtMYB8参与木质素合成途径上肉桂酸4-羟基化酶(C4H)、4CL、肉桂醇脱氢酶(CAD)等酶基因的转录调控。毛果杨PtrMYB26、PtrMYB28、PtrMYB90和PtrMYB152,以及毛白杨(Populus tomentosa)PtoMYB92也被认为是这一类的转录因子(Zhong et al., 2011; Li et al., 2014; 2015)。可见,MYB转录因子在木材形成中具有重要作用。
光皮桦(Betula luminifera)属桦木科(Betulaceae)桦木属(Betula)落叶乔木,材质优良,用途广泛。目前,光皮桦材质性状的改良是桦木育种的重要内容之一,围绕该树种木材形成的分子生物学研究也日益受到重视。程龙军等(2010)采用同源克隆策略分离到光皮桦Bl4CL基因全长序列,该基因主要在根和茎中表达,而在花和叶中表达较弱。Huang等(2014)在转录组测序的基础上,分离到8个BlCesA基因的全长cDNA序列,并结合序列分析基因的器官和组织表达特异性、激素响应以及应拉木诱导处理的表达变化,多层次分析了基因表达与基因功能之间的关系,明确BlCesA1、BlCesA3、BlCesA4主要参与光皮桦次生壁的形成。然而,迄今为止尚未有涉及光皮桦MYB转录因子的研究报道。为此,本研究克隆得到光皮桦MYB基因,在序列特征和进化树构建分析的基础上,分析这些基因在不同组织、器官,以及应拉木诱导形成中的表达变化,挖掘参与木材形成的MYB基因,以期为后续解析基因功能和分子辅助育种提供理论依据和基因资源。
1 材料与方法 1.1 试验材料栽种在浙江农林大学平山实验基地的2年生光皮桦嫁接无性系1-Ⅴ-25-2,共16种器官或组织被采集,取样时间为3月初至7月。花序取自树冠上部枝条,其中雌花序包括长1~1.5 cm的雌花序(3月初采集)和长5~6 cm的雌花序(4月初采集),分别记为FF1和FF2;雄花序包括长1~1.5 cm的雄花序(7月初采集)和即将散粉的雄花序(长6~8 cm,4月初采集),分别记为MF1和MF2。根(Root)来自同一无性系的水培植株,记为R。即将萌发的叶芽、幼叶、成熟叶分别记为L1、L2、L3;茎取自树冠上部的当年生枝条,5月下旬采集,包括第1、2、3、4和5节的茎段(Stem),分别标记为Stem1、Stem2、Stem3、Stem4和Stem5。经解剖观测第1、2节茎段的木质化区域很少,第3、4和5节为正在木质化的茎,木质部比例较高。在植株胸径部位截取10 cm左右主干,剥皮后在木质部一侧刮取厚1~2 mm的外层木质部(outer xylem, OX)和主要包含髓心厚约2 mm的内层木质部(inner xylem, Ⅸ);在树皮一侧分别刮取韧皮部(phloem, PH)、形成层(cambium, CA)(江成,2014)。所有样品采集后立即放入液氮冻存备用。
1.2 应拉木诱导处理采用拉弯处理诱导应拉木,选取生长相对一致的2年生光皮桦(1-Ⅴ-25-2)无性系植株,将其拉弯与垂直方向成45°,具体参照何辉等(2016)的报道。在处理6 h、48 h、7天时,采集对照组(CK)、应拉木(tension wood, TW)和对应木(opposite wood, OW)的外层木质部。
1.3 总RNA的提取采用PureLinkTM Plant RNA Reagent(上海英骏生物技术有限公司)提取样品的总RNA,RNA浓度和质量分别用分光光度计和琼脂糖凝胶电泳检测。
1.4 MYB基因cDNA的克隆根据已有的光皮桦转录组序列,设计合成MYB基因的RACE扩增引物(表 1)。采用SMARTerTM RACE cDNA Amplification Kit(ClonTech)获得MYB基因的5'和3'序列,具体操作步骤参照该试剂盒说明书。琼脂糖凝胶电泳后,使用AxyPrepTM DNA Gel Extraction Kit(AXYGEN)对目的片段进行回收,回收片段与pEASY-T1 Cloning Kit(TransGen)连接后转化大肠杆菌(Escherichia coli)DH5α,挑取阳性克隆送往南京金斯瑞生物科技有限公司测序。在基因的5'和3'非翻译区设计引物(表 2),利用PCR与测序技术验证克隆片段的正确性。
|
|
|
|
使用PrimeScriptTM RT reagent Kit (TaKaRa)进行反转录合成cDNA。采用BIO-RAD CFX96 Real-Time System(BIO-RAD)进行荧光定量PCR,分析4个BlMYB基因、7个预测的下游基因在不同器官、组织,以及应拉木中的表达。荧光定量PCR按照TaKaRa的SYBR® Premix Ex TaqTM(Tli RNaseH Plus)试剂盒说明书进行,每个样品均做3个重复,均以EF1a为内参基因。所有计算在CFX96 ManagerTM V 1.6(Bio-Rad, USA)、Excel 2007和Graphed Prim Mac软件上完成。荧光定量PCR引物序列见表 3。不同基因表达间的皮尔逊相关系数用SPSS 11.0计算。
|
|
利用DNASTAR软件中的EditSeq工具查找光皮桦MYB基因的开放阅读框(open reading frame,ORF),并预测氨基酸。使用MatGAT软件(Campanella et al., 2003)计算序列间的一致性和相似性。通过NCBI(http://www.ncbi.nlm.nih.gov/pubmed/)和phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)平台下载不同物种的MYB氨基酸序列,运用Clustal X2软件比对氨基酸多序列。利用InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)数据库推测蛋白质结构域,利用在线Weblogo平台(http://weblogo.berkeley.edu/logo.cgi)构建结构域的隐马尔可夫模型(Hidden Markov Model,HMM)。使用MEGA7软件构建不同物种的MYB蛋白进化树。通过在线基因结构显示系统(GSDS,http://gsds.cbi.pku.edu.cn/)绘制4个BlMYB基因的结构图。
1.7 MYB家族基因调控途径分析基于课题组已有的光皮桦基因表达谱数据(黄华宏,2012),基因共表达网络运用相互排名法(mutual rank, MR)进行构建(Obayashi et al., 2007)。依据(http://atted.jp/help/coex_cal.shtml)在线帮助,以RPKM值进行样品冗余度计算。再以MYB为指导基因分别计算与其他基因间的皮尔逊相关系数(Pearson's correlation coefficients, PCC),并转换为MR值(http://atted.jp/help/mr.shtml)。针对每个指导基因,选取排名前50位的基因,利用Cytoscape软件(V2.8.2)构建基因共表达网。根据基因注释信息选择纤维素、木质素合成相关酶基因,在其转录起始位点上游1 499 bp的启动子序列,搜寻与MYB家族基因特异性结合的AC元件(AC-Ⅰ:ACCTACC;AC-Ⅱ:ACCAACC),筛选可能与MYB基因协同表达且受MYB基因调控的基因。其中,启动子区AC元件利用Pant CARE在线数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)查找。
2 结果与分析 2.1 BlMYB基因和蛋白序列分析利用已有光皮桦转录组数据和RACE技术克隆到4个BlMYB基因的全长序列,分别命名为BlMYB1、BlMYB2、BlMYB3、BlMYB4,它们都含有2个MYB结构域,属于R2R3亚类MYB转录因子家族成员。这些cDNA序列的全长、ORF、3'UTR、5'UTR、预测编码氨基酸数目及分子量见表 4和图 1。cDNA序列的长度在888~1 628 bp之间,3'UTR和5'UTR序列长度差异明显,在36~505 bp间变化,可能部分基因的非翻译区仍不完整。BlMYB1的ORF序列长度最长,为1 188 bp,BlMYB2的ORF序列长度最短,为759 bp。预测的编码氨基酸数目介于252~395个之间,分子量变幅为28.8~40.1 kDa。
|
|
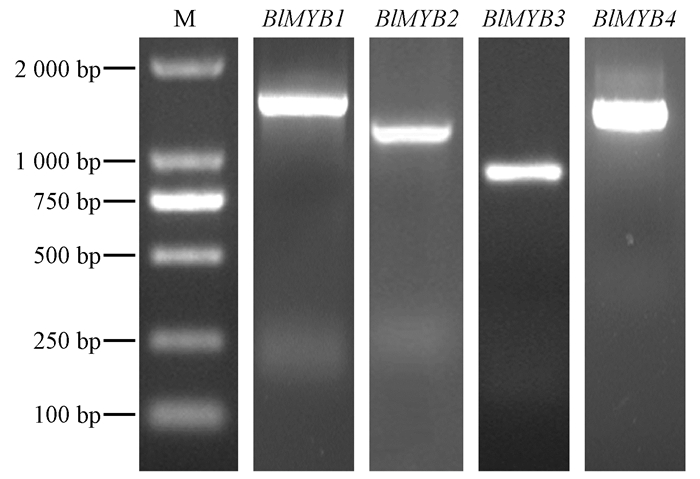
|
图 1 光皮桦MYB基因全长电泳 Fig. 1 The agarose gel electrophoresis of full-length MYB genes in B. luminifera |
由光皮桦cDNA序列推测的氨基酸序列之间的比对结果见表 5。从表中可以看出,4个BlMYB一致性在27%~37%之间,而与它们相应的具最高相似度的拟南芥AtMYB氨基酸序列间的一致性为39%~55%,说明克隆到的4个BlMYB基因是MYB家族的不同成员,而非等位变异或同一基因的不同拷贝。
|
|
通过对光皮桦BlMYB和拟南芥AtMYB氨基酸序列的多重比对分析,发现BlMYB和AtMYB氨基酸序列的N端都有R2、R3结构域(图 2中箭头所示部分),序列相似性较高,而在远离R2R3结构域的地方,序列的相似性明显降低。对R2R3结构域作进一步分析,确定了R2和R3结构域在每个位置上最大可能氨基酸的频率(图 3),证实2个结构域均有典型的色氨酸残基(W)。这些结果进一步说明了分离到的4个cDNA序列确为光皮桦R2R3-MYB类的转录因子。
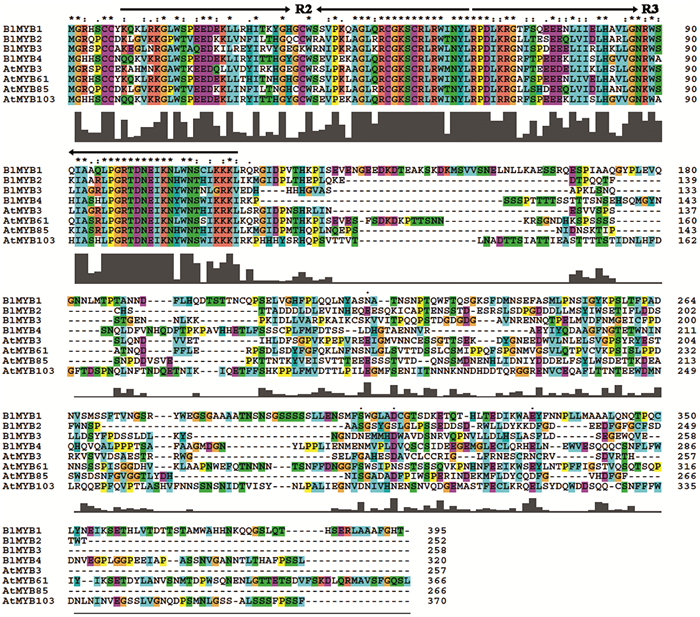
|
图 2 光皮桦BlMYB及拟南芥AtMYB氨基酸序列比对分析 Fig. 2 Amino acid residue alignment and analysis of the predicted BlMYB and AtMYB protein sequence |
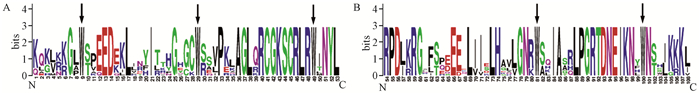
|
图 3 光皮桦和拟南芥MYB蛋白结构域的隐马尔可夫模型 Fig. 3 HMM logos of conserved domains in BlMYB and AtMYB proteins A、B分别为光皮桦和拟南芥MYB蛋白结构域的隐马尔可夫模型。隐马尔可夫模型使用字母表示氨基酸,字母高度表示保守性水平,很清楚地显示了BlMYB和AtMYB蛋白保守区各位置氨基酸的保守性。图中箭头所指的是R2R3结构域中所特有的氨基酸。 A and B show HMM logos of conserved MYB domains of B. luminifera and A. thaliana, respectively. In HMM Figure, letters represent different amino acid, the height of letter indicates conserved level. Obviously, it shows each position of conservative amino acid from BlMYB and AtMYB protein conserved region. What the arrow points is specific amino acid in R2R3 domain. |
本研究根据4个BlMYB蛋白的氨基酸序列与拟南芥、毛果杨、水稻(Oryza sativa)、巨桉、杉木(Cunninghamia lanceolata)、玉米(Zea mays)等的MYB蛋白的氨基酸序列比对结果构建系统进化树(图 4)。从进化树上看,MYB蛋白家族主要分为5个分支。BlMYB1与来自拟南芥的AtMYB55/50/61/86和来自火炬松的PtMYB8等聚在一起,属第13亚组。BlMYB3与拟南芥的AtMYB123具有较高的相似性,属第5亚组。BlMYB2与来自毛果杨的PtrMYB75最为相似,并与拟南芥的AtMYB42/85等聚为一支,属参与细胞次生壁形成的一类。BlMYB4先与毛果杨的PtrMYB10/128蛋白聚合,再与拟南芥AtMYB103蛋白聚为一簇,也属于细胞次生壁形成有关的类别。进化树分析结果为选择参与光皮桦木材形成有关的BlMYB基因提供了初步基础。
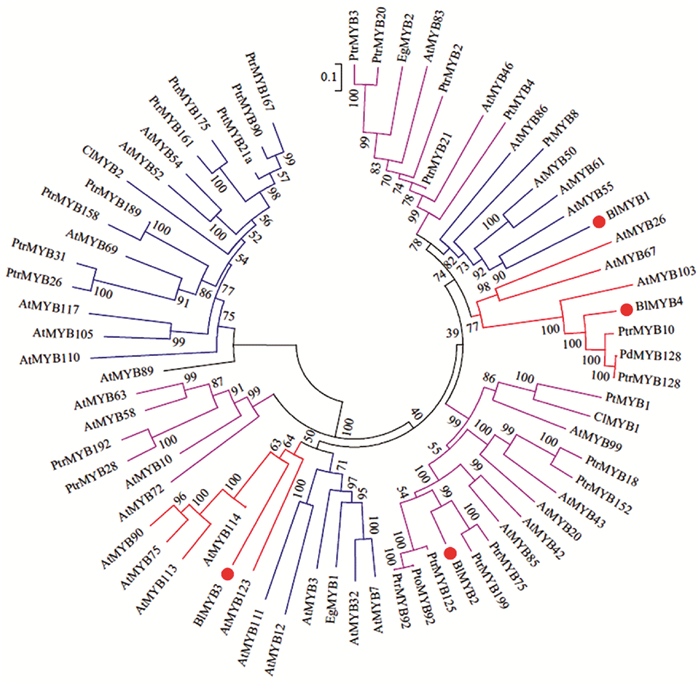
|
图 4 植物MYB蛋白的系统进化树 Fig. 4 Phylogenetic tree of MYB protein sequences in plants Ptr:毛果杨Populus trichocarpa;At:拟南芥Arabidopsis thaliana;Cl:杉木Cunninghamia lanceolata;Zm:玉米Zea mays;Os:水稻Oryza sativa;Eg:巨桉Eucalyptus grandis;Bl:光皮桦Betula luminifera. |
通过将4个光皮桦BlMYB的基因组序列(GenBank登录号分别为:FJ410443.1、KC238465.1、MF797016、MF797017)与转录组序列进行比对分析,得到4个BlMYB的基因结构(图 5)。4个BlMYB基因都含有1~2个内含子,除BlMYB4序列外,其余BlMYB基因的内含子的片段长度差别不大,长度在169~190 bp之间;外显子的数目为2~3个,长度介于759~1 186 bp之间。BlMYB2只有1个内含子(190 bp),BlMYB4第2个内含子长度最长(821 bp),这些特征可能对其基因功能的分化有重要作用。

|
图 5 BlMYB基因的结构 Fig. 5 The structure of BlMYB genes |
首先对荧光定量PCR引物进行验证,电泳结果显示扩增产物条带特异性良好,测序结果显示产物序列正确,可进行荧光定量PCR实验。基于定量PCR数据,采用2-ΔΔCt法(Livak et al., 2001)分析4个BlMYB基因在各器官中的表达量(图 6)。BlMYB1、BlMYB3在叶芽(L1)、嫩叶(L2)、成熟叶(L3)中的表达量呈现递增趋势,且成熟叶片的数值在所有检测器官中为最高。BlMYB2在木质化茎(S5)中表达量最高,是低木质化茎(S1)的20多倍;根(R)、成熟叶片(L3)和成熟雄花序(MF2)中的表达量居第2。BlMYB4在成熟雄花序(MF2)中表达量最高,在根(R)、叶芽(L1)、嫩叶(L2)中的表达量较低;在木质化茎(S5)中的表达量居其次,且是低木质化茎(S1)相应数值的19倍。

|
图 6 BlMYB基因在光皮桦不同器官中的表达 Fig. 6 Expression of BlMYB genes in different organs of B. luminifera FF1:1~1.5 cm的雌花序;FF2:5~6 cm的雌花序;MF1:1~1.5 cm的雄花序;MF2:即将散粉的雄花序;L1:芽;L2:嫩叶;L3:成熟叶;S1:第1节茎;S5:第5节茎;R:根。以R为参照。 FF1:1~1.5 cm female inflorescence; FF2:5~6 cm female inflorescence; MF1:1~1.5 cm male inflorescence; MF2: Male inflorescence that is about to powder; L1: Bud; L2: Young leaf; L3: Mature leaf; S1: The first stem; S5: The fifth stem; R: Root.R is used as a reference. |
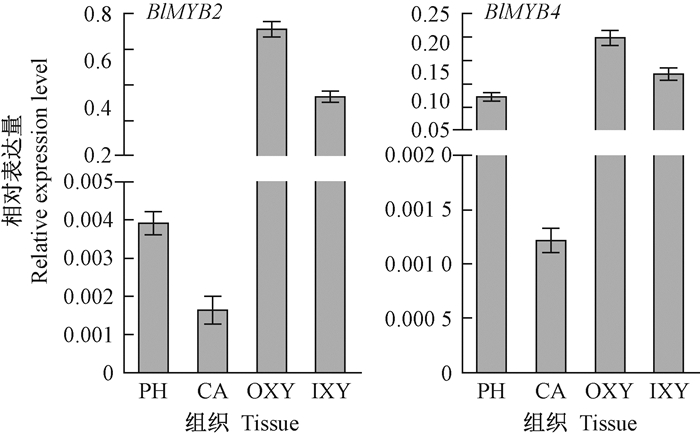
依据4个BlMYB基因在10种器官中表达模式,结合生物信息学分析结果,选择可能参与木质化过程的BlMYB2和BlMYB4作进一步基于标准曲线的定量PCR分析。图 7所示为这2个基因在不同木质化茎段中的表达结果。BlMYB2和BlMYB4在茎中的表达模式相似,即随茎段木质化程度的提高表达逐渐增强,在木质化茎(S4和S5)中的表达水平是低木质化茎(S1)的18~25倍。进一步以来自木质化茎的韧皮部、形成层、木质部为材料分析BlMYB2和BlMYB4的表达差异,结果(图 8)显示这2个基因均在外层木质部(OXY)中表达最强,而在形成层(CA)中表达最弱,在内层木质部(IXY)和韧皮部(PH)中的表达水平居中。值得注意的是,BlMYB2在茎中的总体表达量明显高于BlMYB4,暗示BlMYB2在光皮桦木质部发育中可能发挥更重要的作用。

|
图 7 BlMYB2和BlMYB4基因在光皮桦不同木质化茎中的表达分析 Fig. 7 Analysis of BlMYB2 and BlMYB4 genes expression in different lignified stems of B. luminifera S1:第1节茎;S2:第2节茎;S3:第3节茎;S4:第4节茎;S5:第5节茎。 S1: The first stem; S2: The second stem; S3: The third stem; S4: The forth stem; S5: The fifth stem. |
|
图 8 BlMYB2和BlMYB4基因在光皮桦不同组织中的表达分析 Fig. 8 Analysis of BlMYB2 and BlMYB4 genes expression in different tissues of B. luminifera PH:韧皮部;CA:形成层;OXY:外层木质部;IXY:内层木质部。 PH: Phloem; CA: Cambium; OXY: Outer xylem; IXY: Inner xylem. |
应拉木是指在阔叶材倾斜或弯曲树干和枝条的上方,即受拉部位的木质部,在其横切面上一部分年轮呈现明显偏宽的现象。人为拉弯处理后,光皮桦应拉木的细胞壁显著增厚,形成典型的胶质纤维层;纤维素含量相对增加,而木质素含量则下降(何辉等,2016)。为了探讨BlMYB2和BlMYB4在应拉木形成初期的诱导情况,采用荧光定量PCR分析它们在不同拉弯处理时间应拉木和对应木中的表达差异(图 9)。BlMYB2在应拉木中的表达量呈下降趋势,拉弯处理6 h、48 h和7天时的相应数值分别为对照的0.62、0.37和0.37;而BlMYB4整体呈上升趋势,拉弯处理48 h和7天时表达水平分别是对照组的2.2和1.9倍。相同处理下,这2个基因在对应木中的表达模式也不一致,BlMYB2先下调表达,到拉弯处理48 h后恢复到对照水平;BlMYB4在3个处理时间点的表达量都显著低于对照组。由此可见,BlMYB2和BlMYB4在应拉木形成早期阶段表现出明显的表达差异,表明它们可能参与不同的调控途径。

|
图 9 光皮桦应拉木形成中BlMYB2和BlMYB4基因的表达分析 Fig. 9 Analysis of BlMYB2 and BlMYB4 genes expression during the formation of tension wood of B. luminifera TW:应拉木;OW:应压木;CK:对照组。以CK为参照。*表示处理与对照间的数值差异2倍以上。 TW: Tension wood; OW: Opposite wood; CK: The control. The CK was used as a reference. * shows more than 2.0-fold difference between the treatment and control. |
利用多样本RNA-Seq数据,分别以BlMYB2和BlMYB4作为指导基因,构建了基因共表达网(图 10),发现共有98个Unigenes与相应指导基因间具有很高的表达相关性。BlMYB2、BlMYB4分别与毛果杨的PtrMYB92和PtrMYB128具较近的亲缘关系,后二者被证实调控纤维素、木质素合成途径中相关酶基因的表达(Chai et al., 2014; Li et al., 2015)。因此,依据注释信息从共表达的基因中筛选出10个编码纤维素、木质素合成相关酶的Unigenes,作进一步启动子区顺式作用元件搜寻。其中有7个基因(表 6)的启动子区皆富含该类MYB蛋白结合的AC元件(AC-Ⅰ:ACCTACC;AC-Ⅱ:ACCAACC)。其中Unigene4426.2和Unigene4901.4分别编码羟基-肉桂酰辅酶A莽草/奎宁酸酯转移酶(HCT)、CesA3,它们的表达与BlMYB2、BlMYB4间都呈正相关,皮尔逊相关系数(Pearson's correlation coefficients, PCC)大于0.91。Unigene5760.3和Unigene11074分别注释为4CL、咖啡酸-甲基转移酶(COMT),仅与BlMYB2的表达显著相关,PCC均在0.93以上。Unigene1899.2、Unigene4901.7、Unigene572分别注释为CCoAOMT、CesA4和果糖激酶(FRK),其表达水平与BlMYB4的表达显著正相关(PCC > 0.91)。为验证这7个预测的下游基因与BlMYB2和BlMYB4表达的相关性,利用荧光定量PCR分析它们在不同组织、器官中的表达(图 11)。结果显示:随着茎段木质化程度(S1-S5)的提高,7个下游基因的表达水平也越来越高,在S3、S4和S5中的表达量明显高于S1、S2的相应数值;且在木质部(OXY,IXY)的表达水平明显高于韧皮部(PH)和形成层(CA),这与BlMYB2、BlMYB4的表达模式十分相似,对应的表达相关系数在0.72~0.97间(n=9,P < 0.05)。这也进一步说明BlMYB2和BlMYB4可能在木质素、纤维素合成相关基因的调控中发挥重要作用。

|
图 10 BlMYB2和BlMYB4基因的共表达网 Fig. 10 Co-expression network of BlMYB2 and BlMYB4 矩形表示MYB基因,圆圈表示与MYB共表达的Unigenes,圈内数字为Unigene编号。 Rectangles represent the MYB genes, and circles represent the co-expression Unigenes. Unigene numbers are indicated inside the circles. |
|
|

|
图 11 光皮桦不同组织部位中下游基因的表达分析 Fig. 11 Analysis of downstream genes expression in different tissue positions of B. luminifera 以S1和CA为参照。 The S1 and CA are used as controls. |
在转录组测序基础上分离到4个BlMYB基因序列,相应编码蛋白的N端都有保守的R2R3结构域。荧光定量PCR发现,BlMYB2在木质化茎的木质部高表达,在叶片、韧皮部等组织器官中低表达。系统进化分析发现,BlMYB2与来自毛果杨的PtrMYB92、PtrMYB125、PtrMYB152等序列相似性较高,聚在同一分支。PtoMYB92与PtrMYB92为直系同源基因,同属R2R3-MYB类的转录激活因子,PtoMYB92在功能上主要是通过控制木质素单体的合成参与调控毛白杨(Populus tomentosa)次生壁的形成,它在木质部优势表达,在幼叶中较弱表达(Li et al., 2015)。来自毛果杨的PtrMYB152也属R2R3-MYB转录因子,是一个参与木质素生物合成的特异转录激活因子,主要在茎的木质部、根和叶柄中表达(Li et al., 2014)。同时,在应拉木诱导形成早期阶段,应拉木中BlMYB2的表达量呈下降趋势,尤其是拉弯处理48 h和7天时的相应数值显著低于CK。结合应拉木中木质素含量的相对下降,暗示BlMYB2可能参与光皮桦次生壁中木质素的生物合成。BlMYB4与来自拟南芥的AtMYB103及毛果杨的PtrMYB10、PtrMYB128等亲缘关系较近,在茎的木质部、成熟雄花序中优势表达,在根、嫩叶中的表达水平较低。拟南芥AtMYB103被证实不仅在纤维素生物合成中发挥作用,而且调控阿魏酸-5-羟化酶(F5H)基因表达和S木质素单体合成,主要在束间纤维和木质部表达(Zhong et al., 2008; Öhman et al., 2013)。美洲黑杨(Populus deltoides)的PdMYB10和PdMYB128分别与PtrMYB10和PtrMYB128互为直系同源基因,均为转录激活因子,前二者在美洲黑杨木质部中高水平表达;这2个基因异源转化拟南芥均导致纤维细胞壁明显增厚,木质素、纤维素含量显著增加(Chai et al., 2014)。本研究也发现,在拉弯处理48 h时,BlMYB4在应拉木中表达水平明显高于对照。因此,考虑先前研究发现2个木质部优势表达的F5H基因在光皮桦应拉木形成早期阶段也呈上调表达(黄华宏,2012),以及应拉木中典型胶质纤维层的出现和纤维素含量相对增加,推测BlMYB4可能在光皮桦木质部木质素、纤维素的生物合成中发挥重要作用。
参与木质素、纤维素生物合成的MYB转录因子多通过结合下游靶基因启动子区的AC元件实现调控。玉米ZmMYB11是一个木质素合成的负向调控因子,能结合到来自ZmCOMT基因启动子的AC顺式作用元件上(Vélez-Bermúdez et al., 2015)。电泳迁移率变动和转录激活分析证实,调控木质素、纤维素合成的PtrMYB2/3/20/21、EgMYB2和PtMYB4蛋白都能与AC元件结合(Patzlaff et al., 2003; Zhong et al., 2013)。其次,该类MYB与所调控靶基因的表达高度相关。在烟草中超表达EgMYB2可导致木质素合成酶基因HCT、COMT、F5H、CCoAOMT、CCR和CAD等表达的显著增加,说明EgMYB2与这些酶基因表达间呈正相关(Goicoechea et al., 2005)。同样,相比野生型,在超表达PtoMYB92的毛白杨中,CCoAOMT1、CCR2、COMT2、C3H、PAL4、4CL5、HCT1、C4H2和CAD1等木质素单体合成酶基因的表达显著上调(Li et al., 2015)。因此,本研究基于以上2个特征,利用转录组测序获得的基因表达数据,筛选到FRK、COMT、HCT、CesA3、CesA4、4CL和CCoAOMT 7个可能受BlMYB2和BlMYB4所调控的纤维素、木质素合成酶基因。荧光定量PCR也显示这7个下游基因在不同木质化组织、器官中的表达模式与BlMYB2、BlMYB4的基本一致。根据本研究结果,可以推测BlMYB2可能调控4个假定下游靶基因的表达(其中3个为参与木质素单体合成途径的酶基因);BlMYB4可能调控3个纤维素合成酶基因和2个木质素单体合成相关酶基因的表达。尽管以上假定的调控关系还需要进一步的试验验证,但这些基于生物信息学的结果可为后续调控机制解析提供重要线索。
4 结论利用转录组序列克隆到4个光皮桦R2R3-MYB基因(BlMYB1-4)。BlMYB2与毛果杨PtrMYB92、PtrMYB125和PtrMYB152等的序列高度相似,BlMYB4与AtMYB103、PtrMYB10、PtrMYB128等亲缘关系较近。荧光定量PCR结果显示BlMYB2在木质化茎的木质部高表达,在叶片、韧皮部等组织器官中低表达;BlMYB4在茎的木质部、成熟雄花序中优势表达,在根、嫩叶中的表达水平较低。根据共表达网构建、荧光定量PCR和特异性AC元件分析结果,推测在光皮桦中,FRK、COMT、HCT、CesA3、CesA4、4CL和CCoAOMT 7个纤维素、木质素合成酶基因的转录可能受BlMYB2、BlMYB4基因调控。因此,BlMYB2、BlMYB4极有可能参与调控光皮桦木材的形成。
程龙军, 童再康, 黄华宏, 等. 2010. 光皮桦中4-香豆酸辅酶A连接酶基因Bl4CL的克隆和表达分析. 植物生理学通讯, (1): 5-10. (Cheng L J, Tong Z K, Huang H H, et al. 2010. Cloning and expression analysis of 4-coumarate: CoA ligase gene Bl4CL in Betula luminifera H. Winkl. Plant Physiology Communications, (1): 5-10. [in Chinese]) |
何辉, 楼雄珍, 林二培, 等. 2016. 光皮桦应拉木的显微特征及其形成早期内源激素分布. 林业科学, 52(10): 38-44. (He H, Lou X Z, Lin E P, et al. 2016. Xylem characteristics of tension wood and endogenous hormones distributions during its early formation period in Betula luminifera. Scientia Silvae Sinicae, 52(10): 38-44. DOI:10.11707/j.1001-7488.20161005 [in Chinese]) |
黄华宏. 2012. 基于转录组测序的光皮桦应拉木形成分子机制研究. 杭州: 浙江大学博士学位论文. (Huang H H. 2012. Studies on molecular mechanism of tension wood formation in Betula luminifera using RNA-Seq. Hangzhou: PhD thesis of Zhejiang University. [in Chinese]) |
江成. 2014. 光皮桦BlOFPs基因的克隆及其功能研究. 杭州: 浙江农林大学硕士学位论文. (Jiang C. 2014. Isolation and functional analysis of BlOFPs genes in Betula luminifera. Hangzhou: MS thesis of Zhejiang Agricultural and Forestry University. [in Chinese]) |
刘雪梅. 2005. 白桦木质素生物合成酶基因分离及遗传转化的研究. 哈尔滨: 东北林业大学博士学位论文. (Liu X M. 2005. Genes isolation and genetic transformation of biosynthetic enzymes in Betula platyphylla. Harbin: PhD thesis of Northeast Forestry University. [in Chinese]) |
Bomal C, Bedon F, Caron S, et al. 2008. Involvement of Pinus taeda MYB1 and MYB8 in phenylpropanoid metabolism and secondary cell wall biogenesis: a comparative in planta analysis. Journal of Experimental Botany, 59(14): 3925-3939. DOI:10.1093/jxb/ern234 |
Campanella J J, Bitincka L, Smalley J. 2003. MatGAT: an application that generates similarity/identity matrices using protein or DNA sequences. BMC Bioinformatics, 4(1): 29. DOI:10.1186/1471-2105-4-29 |
Chai G, Wang Z, Tang X, et al. 2014. R2R3-MYB gene pairs in Populus: evolution and contribution to secondary wall formation and flowering time. Journal of Experimental Botany, 65(15): 4255-4269. DOI:10.1093/jxb/eru196 |
Demura T, Fukuda H. 2006. Transcriptional regulation in wood formation. Trends in Plant Science, 12(2): 64-70. |
Goicoechea M, Lacombe E, Legay S, et al. 2005. EgMYB2, a new transcriptional activator from Eucalyptus xylem, regulates secondary cell wall formation and lignin biosynthesis. Plant Journal, 43(4): 553-567. DOI:10.1111/tpj.2005.43.issue-4 |
Groover A T. 2005. What genes make a tree a tree?. Trends in Plant Science, 10(5): 210-214. DOI:10.1016/j.tplants.2005.03.001 |
Groover A, Robischon M. 2006. Developmental mechanisms regulating secondary growth in woody plants. Current Opinion in Plant Biology, 9(1): 55-58. DOI:10.1016/j.pbi.2005.11.013 |
Huang H, Jiang C, Tong Z, et al. 2014. Eight distinct cellulose synthase catalytic subunit genes from Betula luminifera are associated with primary and secondary cell wall biosynthesis. Cellulose, 21(4): 2183-2198. DOI:10.1007/s10570-014-0261-z |
Kalluri U C, Joshi C P. 2004. Differential expression patterns of two cellulose synthase genes are associated with primary and secondary cell wall development in aspen trees. Planta, 220(1): 47-55. DOI:10.1007/s00425-004-1329-z |
Karpinska B, Karlesson M, Srivastava M, et al. 2004. MYB transcription factors are differentially expressed and regulated during secondary vascular tissue development in hybrid aspen. Plant Molecular Biology, 56(2): 255-270. DOI:10.1007/s11103-004-3354-5 |
Krauskopf E, Harris P J, Putterill J. 2005. The cellulose synthase gene PrCESA10 is involved in cellulose biosynthesis in developing tracheids of the gymnosperm Pinus radiata. Gene, 350(2): 107-116. |
Larson P R. 2012. The vascular cambium: development and structure. Springer Science & Business Media.
|
Li C, Wang X, Lu W, et al. 2014. A poplar R2R3-MYB transcription factor, PtrMYB152, is involved in regulation of lignin biosynthesis during secondary cell wall formation. Plant Cell, Tissue and Organ Culture, 119(3): 553-563. DOI:10.1007/s11240-014-0555-8 |
Li C, Wang X, Ran L, et al. 2015. PtoMYB92 is a transcriptional activator of the lignin biosynthetic pathway during secondary cell wall formation in Populus tomentosa. Plant and Cell Physiology, 56(12): 2436-2446. DOI:10.1093/pcp/pcv157 |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
McCarthy R L, Zhong R, Fowler S, et al. 2010. The poplar MYB transcription factors, PtrMYB3 and PtrMYB20, are involved in the regulation of secondary wall biosynthesis. Plant and Cell Physiology, 51(6): 1084-1090. DOI:10.1093/pcp/pcq064 |
Nairn C J, Haselkorn T. 2005. Three loblolly pine CesA genes expressed in developing xylem are orthologous to secondary cell wall CesA genes of angiosperms. New Phytologist, 166(3): 907-915. DOI:10.1111/nph.2005.166.issue-3 |
Obayashi T, Kinoshita K, Nakai K, et al. 2007. ATTED-Ⅱ: a database of co-expressed genes and cis elements for identifying co-regulated gene groups in Arabidopsis. Nucleic Acids Research, 35(suppl 1): D863-D869. |
Öhman D, Demedts B, Kumar M, 等. 2013. MYB103 is required for FERULATE-5-HYDROXYLASE expression and syringyl lignin biosynthesis in Arabidopsis stems. The Plant Journal, 73(1): 63-76. |
Patzlaff A, Mclnnis S, Courtenay A, et al. 2003. Characterisation of a pine MYB that regulates lignification. The Plant Journal, 36(6): 743-754. DOI:10.1046/j.1365-313X.2003.01916.x |
Vélez-Bermúdez I C, Salazar-Henao J E, Fornalé S, et al. 2015. A MYB/ZML complex regulates wound-induced lignin genes in maize. The Plant Cell, 27(11): 3245-3259. DOI:10.1105/tpc.15.00545 |
Wu L, Joshi C P, Chiang V L. 2000. A xylem-specific cellulose synthase gene from aspen (Popolus tremuloides) is responsive to mechanical stress. The Plant Journal, 22(6): 495-502. DOI:10.1046/j.1365-313x.2000.00758.x |
Zhong R, Lee C, Zhou J, et al. 2008. A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis. The Plant Cell, 20(10): 2763-2782. DOI:10.1105/tpc.108.061325 |
Zhong R, McCarthy R L, Lee C, et al. 2011. Dissection of the transcriptional program regulating secondary wall biosynthesis during wood formation in poplar. Plant Physiology, 157(3): 1452-1468. DOI:10.1104/pp.111.181354 |
Zhong R, Mccarthy R L, Haghighat M, et al. 2013. The poplar MYB master switches bind to the SMRE site and activate the secondary wall biosynthetic program during wood formation. PLoS One, 8(7): e69219. DOI:10.1371/journal.pone.0069219 |
 2018, Vol. 54
2018, Vol. 54

