文章信息
- 王源杰, 郭雪峰, 赵蕾, 郭成, 王煜炜.
- Wang Yuanjie, Guo Xuefeng, Zhao Lei, Guo Cheng, Wang Yuwei.
- 毛竹叶黄酮碳苷C-糖基化途径及C-糖基转移酶的分析
- Characterization of the C-glycosylation Pathway and C-glucosyltransferase of C-glycosyl Flavonoids from Phyllostachys edulis Leaves
- 林业科学, 2018, 54(12): 60-69.
- Scientia Silvae Sinicae, 2018, 54(12): 60-69.
- DOI: 10.11707/j.1001-7488.20181207
-
文章历史
- 收稿日期:2018-02-01
- 修回日期:2018-06-22
-
作者相关文章
黄酮糖苷类化合物是植物中广泛存在的天然产物,具有抗炎、抗氧化、抗过敏、镇静催眠和抗癌等作用(Chen et al., 2013; Zhang et al., 2005; Guo et al., 2013)。根据黄酮糖苷类化合物中糖基与黄酮母环连接方式,黄酮糖苷类化合物可以分为黄酮氧苷和黄酮碳苷2大类,黄酮氧苷是指糖基部分通过1个氧原子(-O-)与黄酮母环连接的化合物,黄酮碳苷是指糖基部分与黄酮母环的1个碳原子(-C-)直接连接的化合物。在黄酮糖苷类化合物的糖基化过程中,糖基转移酶是一个关键酶,起着非常重要的作用,催化合成黄酮氧苷的是O-糖基转移酶(O-glycosyltransferase,OGT),催化合成黄酮碳苷的是C-糖基转移酶(C-glycosyltransferase,CGT)。到目前为止,碳水化合物活性酶数据库(Carbohydrate-Active enZyme,CAZy)(http://www.cazy.org/GlycosylTransferases.html)内有超过359 000个糖基转移酶(glycosyltransferase,GT)的序列,基于序列相似性,把这些糖基转移酶分为104个家族(Egorova et al., 2016)。
糖基供体主要是核苷二磷酸化的含糖基化合物,包括UDP-葡萄糖、UDP-半乳糖、UDP-鼠李糖、UDP-木糖和UDP-葡萄糖醛酸等,其中UDP-葡萄糖最为常见(Hofer, 2016;Sawada et al., 2005)。并且大多数已知的CGT显示出相对较窄的底物选择性(Chen et al., 2016)。关于黄酮碳苷的C-糖基化合成途径主要有2类报道:第1类是糖基供体和黄酮苷元底物受体在CGT催化下直接一步合成黄酮碳苷,如三花龙胆(Gentiana triflora)中重组的CGT(GtUF6CGT1),以木犀草素为底物受体和UDP-葡萄糖为糖基供体,直接C-糖基化合成异荭草苷(Sasaki et al., 2015);第2类是底物经过多种酶共同作用合成黄酮碳苷,如Brazier-Hicks等(2009)从水稻(Oryza sativa)中提取、纯化和表征了一种黄酮C-葡萄糖基转移酶(OsCGT),OsCGT以2-羟基环烷酮开链结构底物受体和UDP-葡萄糖为糖基供体,催化合成相应的糖苷,然后该糖苷在黄酮脱水酶(dehydration)的作用下合成异牡荆苷。而且多数报道是关于第2类C-糖基化合成途径(Brazier-Hicks et al., 2009; Nagatomo et al., 2014; Falcone Ferreyra et al., 2013; Hirade et al., 2015; Wang et al., 2017; Hamilton et al., 2012),第1类C-糖基化合成途径的报道较少。
植物中糖基转移酶所含氨基酸大致在442~507之间,不同糖基转移酶氨基酸序列相似性在95%~24%之间(Fukuchi-Mizutani et al., 2003; Costa et al., 2017)。而且毛竹(Phyllostachys edulis)基因组测序工作以及毛竹基因组数据库已经完成(Peng et al., 2013;Zhao et al., 2014),这项成果为寻找毛竹黄酮碳苷合成代谢途径中相关酶的基因提供了丰富的信息。由于毛竹叶中含有大量的黄酮碳苷,CGT是黄酮碳苷生物合成代谢途径中C-糖基化的关键酶,但毛竹叶CGT的具体基因序列是什么,CGT的酶学性质和结构特征是什么,以及CGT催化完成的C-糖基化途径是什么,现在还不清楚,也没有相关报道。本研究,主要目的是从毛竹叶中分离纯化CGT,通过建立CGT催化合成黄酮碳苷(异荭草苷)的反应体系,运用数据库搜索比对和Q-TOF检测分析等方法手段,研究毛竹叶黄酮碳苷的C-糖基化途径以及CGT的酶学性质和一级结构氨基酸序列特征,以期为后续深入研究毛竹叶中的CGT打下良好的基础。
1 材料与方法 1.1 材料来源毛竹叶采自江西省林业科学院,采后迅速冷藏处理运输,储存于实验室-80 ℃超低温冰箱。
1.2 HPLC检测方法色谱柱:Water XTerra® MS C18 5 μm (250 mm×4.6 mm);流动相A:乙腈;流动相B:5‰乙酸水溶液。梯度洗脱条件:0~3 min, 15%的流动相A;3~5 min,15%~20%流动相A;5~10 min,20%~27%流动相A;10~31 min,27%流动相A;32~37 min,95%流动相A;38~42 min,15%流动相A。流速1 mL·min-1;柱温30 ℃;进样量10 μL。圣草素查尔酮、圣草素的检测波长是289 nm,木犀草素、荭草苷以及异荭草苷的检测波长设为350 nm。
用上述HPLC条件分别建立底物(圣草素查尔酮、圣草素、木犀草素)和产物(异荭草苷和荭草苷)的标准曲线,得到标准曲线方程。圣草素查尔酮:y=6 838.4x-14 902,R2=0.999 8;圣草素:y=11 176x-34 790,R2=0.999 6;木犀草素:y=11 254 x-3 088.8,R2=0.999 8;异荭草苷:y=4 474.9x-2 898.1,R2=0.999 8;荭草苷:y=4 434.6x-26.394,R2=0.999 8。式中:x为样品浓度,y为HPLC检测峰面积。
1.3 毛竹叶黄酮碳苷C-糖基转移酶(CGT)催化C-糖基化反应体系的建立CGT催化的C-糖基化反应体系溶液总体积为880 μL,其中0.1 μmol·L-1的Tris-HCl缓冲液为480 μL,粗酶溶液160 μL,31.76 μmol·L-1底物(糖基受体)溶液和448 μmol·L-1UDP-葡萄糖(糖基供体)溶液共240 μL。CGT催化反应体系温度为28 ℃,反应时间为1 h,加入40 μL纯乙酸终止反应。
分别选择圣草素查尔酮、圣草素、木犀草素3种可能的底物(糖基受体),UDP-葡萄糖作为糖基供体,用HPLC法检测反应体系中合成产物异荭草苷或荭草苷的增加量。CGT催化活性的表示方法:以反应体系中合成产物(异荭草苷或荭草苷)的浓度值增加量来表示CGT的活性大小。
1.4 CGT的提取分离与纯化酶的提取和纯化过程均在4 ℃以下环境中进行。将冷冻的毛竹叶样品(200 g鲜质量)加入到600 mL预冷的缓冲液A(0.1 mol·L-1 Tris-HCl,5 mmol·L-1 DTT,pH8.0)中,提取4 h,然后用4层纱布过滤除去细胞碎片,将滤液以10 000 r·min-1离心30 min,上清液即是得到的粗酶液。将粗酶液通过硫酸铵分级沉淀、透析、葡聚样凝胶过滤、超滤浓缩、阴离子交换层析逐步进行纯化。上述每一步分离纯化后,均用已经建立的CGT活性检测方法检测各馏分中CGT活性,合并具有CGT活性的各馏分溶液,然后再进行下一步纯化。
离心后的粗酶液中加入硫酸铵,取30%~80%饱和度沉淀溶解于约10 mL缓冲液A中,透析脱盐。将透析浓缩后的样品上样到用二乙醇胺(pH8.9)平衡的Sephadex G-25 (GE, USA)柱(1.6 cm × 40 cm)上。用二乙醇胺(pH8.9)洗脱,收集2 mL为1个馏分,分别检测其CGT活性,合并具有CGT活性的各馏分,用超滤管Amicon Ultra-15 Ultracel-10k(Merck Millipore)浓缩。将超滤管浓缩后具有CGT活性的酶液上样到已用120 mL缓冲液Buffer B(0.1 mol·L-1 Tris-HCl,5 mmol·L-1 DTT,pH7.5)平衡的DEAE-纤维素柱(Waterman, UK)(1.5 cm × 20 cm)上,然后依次用120 mL含有0.06,0.10,0.15,0.25,0.35,0.5 mol·L-1 NaCl的Buffer B(0.1 mol·L-1 Tris-HCl,5 mmol·L-1 DTT,pH7.5)进行洗脱,收集并合并具有酶活性的馏分,并用超滤管Amicon Ultra-15 Ultracel-10k浓缩脱盐。最后用SDS-PAGE (Bio-Rad, USA)法检测分析得到的CGT样品的纯度和大致分子量,以备下一步分析检测用。
1.5 CGT催化的C-糖基化反应体系参数优化用葡聚糖凝胶过滤后,具有CGT活性的粗酶液作为样品。按照CGT酶活性测定方法,分别研究不同反应时间(20, 30, 40, 50, 60, 80 min)、不同反应温度(22, 25, 28, 31, 34 ℃)、缓冲盐pH(7.1, 7.6, 8.1, 8.6, 9.1),以及不同底物(木犀草素)浓度(3.18, 15.88, 31.76, 47.64, 63.52 μmol·L-1)对酶的影响,以反应体系中产物异荭草苷浓度增加量为标准,确定最佳反应时间、反应温度、缓冲盐pH和底物浓度。
1.6 毛竹叶黄酮碳苷C-糖基化途径分析分别选择圣草素查尔酮、圣草素、木犀草素3种可能的底物(糖基受体),UDP-葡萄糖作为糖基供体,建立3个反应体系(圣草素查尔酮为底物的是反应体系1,圣草素为底物的是反应体系2,木犀草素为底物的是反应体系3),用HPLC法检测3个反应体系中底物的减少量,以及合成产物异荭草苷或荭草苷的增加量。根据试验结果,参考已报道的黄酮碳苷C-糖基化途径,分析毛竹叶黄酮碳苷生物合成过程中可能的C-糖基化途径。
1.7 HPLC-Q-TOF-MS/MS对CGT的检测分析样品回收:用手术刀片切下电泳胶上目标条带置于1 mL离心管中,先用100 μL 100 mmol·L-1 NH4HCO3进行清洗,清洗后除去清洗液,然后再加入100 μL 25 mmol·L-1 NH4HCO3/50%乙腈脱色液,直至将目的蛋白条带清洗至透明,弃去脱色液得到脱色后CGT样品。
胰蛋白酶酶解处理:按照Devouge等(2007)报道的方法进行。将脱色后的CGT样品用乙腈吸水干燥,保持10 min。在57 ℃条件下,加入50 μL 10 mmol·L-1二硫苏糖醇(DTT)进行还原45 min,冷却至室温后加入50 μL 55 mol·L-1碘代乙酰胺(IAA)在黑暗条件下进行烷基化30 min。将得到的样品进行离心并冷冻干燥,溶解于1 mL 100 mmol·L-1 NH4HCO3,并按照质量比1: 20(胰蛋白酶:蛋白)的比例加入质谱级的胰蛋白酶,过夜消化16 h。反应结束后快速升温至100 ℃终止反应。最后将酶解后CGT样品过0.22 μm微孔滤膜后,进行HPLC-Q-TOF-MS/MS (1290-6540, Agilent, USA)检测分析。
HPLC-Q-TOF-MS/MS检测分析条件:1)HPLC条件:ZORBAX Eclipse Plus C18色谱柱,流动相A和B分别为水和乙腈,流动相梯度洗脱条件如下:0~1 min,5%的流动相B;1~36 min,5%~25%的流动相B,流速为0.2 mL·min-1,上样量为5 μL。2)质谱条件:正离子模式,干燥器温度350 ℃, 干燥器流速10 L·min-1, 雾化器压力30 psi(207 kPa), 鞘气温度350 ℃, 鞘气流速11 L·min-1, 毛细管电压3 500 V, 喷嘴电压500 V。
1.8 毛竹CGT基因序列和一级结构氨基酸序列分析根据肽段裂解规律,分析HPLC-Q-TOF-MS/MS测定的CGT样品质谱图,得到CGT样品肽段序列信息,并与毛竹基因组数据库(http://www.bamboogdb.org)可能的毛竹CGT一级结构氨基酸序列进行比对分析,之后利用数据库(https://www.ebi.ac.uk/Tools/sss/ncbiblast/)搜索已报道的黄酮碳苷CGT的基因序列,进行BLAST(Basic Local Alignment Search Tool)比对。最终得到可能的毛竹CGT基因序列和一级结构氨基酸序列。
2 结果与分析 2.1 毛竹叶黄酮碳苷C-糖基转移酶(CGT)分离纯化毛竹叶样品经硫酸铵分层沉淀、透析、葡聚糖凝胶过滤、阴离子交换层析、超滤脱盐等方法纯化后,得到毛竹叶CGT样品,将纯化后的CGT样品进行SDS-PAGE凝胶电泳。SDS-PAGE分析纯化后CGT样品如图 1所示,在50 kDa处有1条明显的蛋白带,且纯化后条带单一,说明CGT纯度较高,分子量初步判定约为50 kDa。同时该结果证明以上分离纯化步骤可行,可以达到较好的纯化效果。

|
图 1 SDS-PAGE分析纯化后C-糖基转移酶(CGT)样品 Fig. 1 SDS-PAGE analysis of the purified C-glycosyltransferase(CGT) samples A:蛋白质标准品; B, C, D:纯化后CGT样品。 A: Protein marker; B, C, D: Purified CGT samples. |
采用纯化后CGT样品,以木犀草素为底物研究毛竹叶中黄酮碳苷CGT酶学性质,对CGT催化反应体系中缓冲盐pH值、温度、反应时间和底物浓度4个参数进行优化(图 2)。由图 2可知,毛竹叶CGT的最佳反应时间是40 min,最适反应温度是28 ℃,最佳缓冲盐pH为8.1,最适底物(木犀草素)浓度为31.76 μmol·L-1。
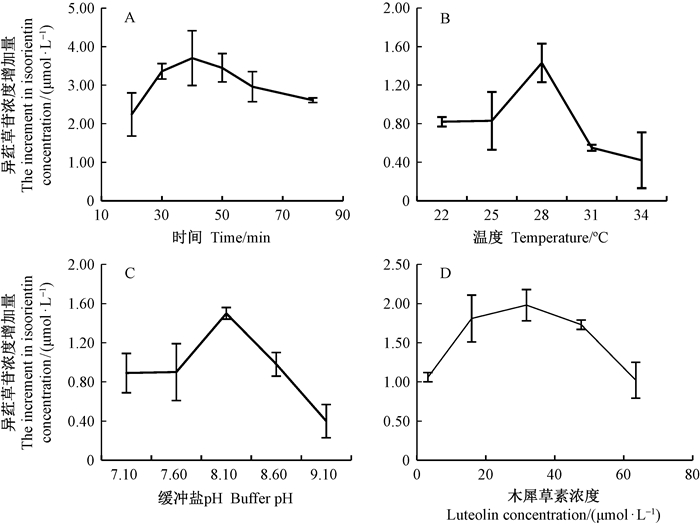
|
图 2 CGT催化反应体系中反应时间、反应温度、缓冲盐pH和底物浓度对CGT活性的影响 Fig. 2 Effects of time, temperature, buffer pH and luteolin concentration on CGT activity in the CGT reaction system |
毛竹叶含有大量的黄酮碳苷,主要是异荭草苷和荭草苷。参考已有报道,毛竹叶黄酮碳苷可能的C-糖基化途径见图 3。根据图 3,选择了圣草素查尔酮、圣草素和木犀草素3种可能的底物,根据已建立的CGT催化的C-糖基化反应体系,进行可能的C-糖基化途径验证。不同样品(不同底物反应体系在CGT催化反应后样品、粗酶液样品、3个底物和2个产物标准品)的HPLC检测见图 4。不同底物反应体系在CGT催化反应前后底物和产物浓度变化见表 1。在底物分别为圣草素查尔酮、圣草素、木犀草素的反应体系1、2、3中,CGT催化反应后底物浓度的减少量分别为20.42,16.09,8.00 μmol·L-1,3个反应体系底物减少量都很大,且3个体系间均差异显著(P<0.05),说明3个反应体系中底物均大量消耗生成了别的产物;产物异荭草苷浓度的增加量分别为0.75,0.81,3.62 μmol·L-1,反应体系3增加量最大,1和2增加量不明显,且3与1、2差异显著,1与2无显著差异,而产物荭草苷浓度在3个反应体系里基本没有增加,也无显著差异,且反应体系1和2有大量未知产物(图 4 A中峰6,图 4 B中峰7)生成,反应体系3则无该未知产物生成,说明反应体系3中底物木犀草素在CGT催化下大量转化生成了异荭草苷(图 3路线3),反应体系1和2中的底物圣草素查尔酮和圣草素在CGT催化下大量转化生成了未知产物,只有少量转化生成异荭草苷。根据3个反应体系异荭草苷生成量来判断,毛竹叶中异荭草苷主要生成途径是以CGT催化木犀草素和UDP-葡萄糖直接生成异荭草苷,而CGT催化圣草素查尔酮、圣草素和UDP-葡萄糖间接生成异荭草苷则是次要途径。
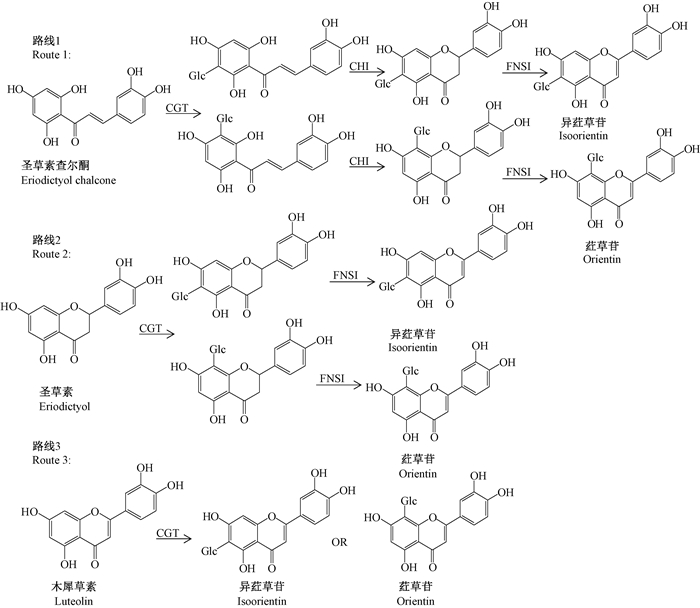
|
图 3 毛竹叶黄酮碳苷生物合成过程中可能的C-糖基化途径 Fig. 3 The possible C-glycosylation pathways for C-glycosyl flavonoid biosynthesis in leaves of Phyllostachys edulis CHI:查尔酮异构酶; FNSI:黄酮合成酶。 CHI: Chalcone isomerase; FNSI: Flavone synthase. |

|
图 4 不同底物的反应体系中CGT催化反应后样品、粗酶液、底物和产物标准品HPLC检测 Fig. 4 HPLC analysis of the different reaction system solution samples, including the crude enzyme solution, substrate and product standards, and CGT catalytic reaction samples A:反应体系1(底物为圣草素查尔酮)反应后HPLC检测;B:反应体系2(底物为圣草素)反应后HPLC检测;C:反应体系3(底物为木犀草素)反应后HPLC检测;D:粗酶液HPLC检测;E:标准品圣草素查尔酮(Peak 3)HPLC检测;F:标准品圣草素(Peak 4)HPLC检测;G:标准品木犀草素(Peak 5)HPLC检测;H:标准品异荭草苷(Peak 1)HPLC检测;I:标准品荭草苷(Peak 2)HPLC检测。HPLC检测图中色谱峰分别是:1:异荭草苷;2:荭草苷;3:圣草素查尔酮;4:圣草素;5:木犀草素;6:未知产物1;7:未知产物2. A: Reaction system 1 solution sample (eriodictyol chalcone was used as the substrate); B: Reaction system 2 solution sample (eriodictyol was used as the substrate); C: Reaction system 3 solution sample (luteolin was used as the substrate); D: Crude enzyme solution sample; E: Eriodictyol chalcone standard sample (Peak 3); F: Eriodictyol standard sample (Peak 4); G: Luteolin standard sample (Peak 5); H: Isoorientin standard sample (Peak 1); I: Orientin standard sample (Peak 2). The peaks in the chromatograms of HPLC: 1: Isoorientin; 2: Orientin; 3: Eriodictyol chalcone; 4: Eriodictyol; 5: Luteolin; 6: Unknown product 1; 7: Unknown product 2. |
|
|
将SDS-PAGE纯化后的CGT片段割胶回收,并经胰蛋白酶酶解处理后,进行HPLC-Q-TOF-MS/MS检测分析。根据蛋白质质谱裂解规律,解析出4个CGT肽段,CGT 4个肽段的提取离子流图(EIC)及其MS/MS图见图 5。由图 5可知,4个肽段的氨基酸序列分别是:PSSSD,DQLSELAAGLEASGCR,GTVVDR,DDDR。将结果在毛竹基因数据库(http://www.bamboogdb.org)中进行搜索比对。将BLAST结果与毛竹基因组数据库(http://www.bamboogdb.org)的结果进行比较找到可能的毛竹CGT基因。由图 6可知,4个毛竹CGT肽段的氨基酸序列均可与毛竹基因(PH01000603G0510)编码的蛋白质氨基酸序列匹配。

|
图 5 纯化后CGT 4个肽段的提取离子流(EIC)及其MS/MS分析 Fig. 5 Extracted ion chromatogram (EIC) of the four peptide ions of the purified CGT samples and MS/MS analysis A:蛋白质肽离子的命名和碎片离子; B:纯化的CGT样品的4个肽段离子色谱图(EIC); C:肽段1的质谱分析; D:肽段2的质谱分析; E:肽段3的质谱分析; F:肽段4的质谱分析。 A: The nomenclature and diagnostic fragmentation of the protein peptide ions; B: Extracted ion chromatogram (EIC) of the four peptide ions of the purified CGT samples; C: MS/MS analysis of peptide ion 1; D: MS/MS analysis of peptide ion 2; E: MS/MS analysis of peptide ion 3; F: MS/MS analysis of peptide ion 4. |

|
图 6 4个毛竹CGT肽段的氨基酸序列(1: GTVVDR;2: PSSSD;3: DQLSELAAGLEASGCR;4: DDDR)与毛竹基因(PH01000603G0510)编码的蛋白质氨基酸序列比对结果 Fig. 6 The result from aligning the amino acid sequence of the four peptide ions (1: GTVVDR; 2: PSSSD; 3: DQLSELAAGLEASGCR; 4: DDDR) with the amino acid sequence encoded by the gene (PH01000603G0510) from Phyllostachys edulis |
由于水稻和毛竹同属于禾本科(Gramineae),具有较近的亲缘关系,水稻CGT的基因序列已经报道,且水稻CGT催化生物合成最终产物也是异荭草苷,所以选择已报道的水稻(Oryza sativa Japonica Group)的CGT基因序列(FM179712),将毛竹基因(PH01000603G0510)的碱基序列与水稻的CGT基因的碱基序列进行比对(图 7)。2个基因的匹配率为81%,可以初步认定该毛竹基因序列(PH01000603G510)可能就是毛竹CGT基因序列。毛竹基因(PH01000603G0510)碱基数为1 416,其编码的蛋白质及其氨基酸序列氨基酸数为471。

|
图 7 毛竹基因序列(Ⅱ: PH01000603G0510)与水稻基因序列(Ⅰ: FM179712)比对结果 Fig. 7 The alignment result of the base sequence of a gene (Ⅱ: PH01000603G0510) from Phyllostachys edulis with the base sequence of the CGT gene (Ⅰ: FM179712) from Oryza sativa |
目前,黄酮碳苷的获得途径主要是从植物中提取分离、化学合成或结构改造以及植物细胞或组织培养物中获得,但是以上几种方法获得的黄酮碳苷产量小且成本大。而代谢工程为规模化生产黄酮碳苷等次生代谢物提供了新的前景。研究植物黄酮碳苷合成代谢中的C-糖基化途径及其C-糖基转移酶(CGT),然后找到相关基因,是通过代谢工程大量获得黄酮碳苷的必要前提条件。在本研究中,反应体系中反应时间、反应温度、缓冲盐pH值和底物浓度等参数均对CGT活性有较大的影响。在CGT反应体系最佳缓冲液pH值为8.1,这与Karim等(2002)的研究结果最适pH范围7.5~8.5基本一致, 过酸和过碱不利于酶反应。随着温度升高,酶促反应速率加快,当温度过高时,酶变性并失活。最佳反应温度为28 ℃,这也与很多GT的最佳反应温度一致(Yabuya et al., 2002)。最适的木犀草素浓度为31.76 μmol·L-1。低浓度木犀草素可能反应不充分,而木犀草素浓度过高(≥47.64 μmol·L-1)可能对酶活性有抑制作用。
本研究首次证实了毛竹叶黄酮碳苷C-糖基化主要途径,即CGT催化木犀草素和UDP-葡萄糖直接生成异荭草苷,该结果与三花龙胆中的C-糖基化途径是一致的(Sasaki et al., 2015)。在底物为圣草素查尔酮和圣草素时,反应中产生的2个未知产物可能是底物圣草素查尔酮或圣草素连接1个UDP-葡萄糖生成的,而少量的异荭草苷极有可能就是由未知产物再次转化而生成的,这也是只有少量异荭草苷生成的原因。以上是毛竹叶异荭草苷C-糖基化次要途径,该途径与Brazier-Hicks等(2009)研究的水稻中黄酮碳苷C-糖基化途径相类似。另外,虽然毛竹叶中的异荭草苷和荭草苷是同分异构体,但毛竹叶CGT只催化木犀草素生成异荭草苷,没有生成荭草苷,这表明毛竹叶CGT极易催化生成C-6黄酮苷(异荭草苷),而极少催化生成C-8黄酮苷(荭草苷),这个结果也与Brazier-Hicks等(2009)和Sasaki等(2015)研究结果一致。该结果说明底物受体的化学空间结构对于CGT催化其接受糖基的位置起着至关重要的作用。
根据文献记载,大多数植物糖基转移酶的分子量在45~60 kDa之间(Vogt et al., 2000)。本研究首次分离纯化并鉴定了来自毛竹叶的CGT。该CGT的分子量约为50 kDa,与文献报道的分子量范围一致。再根据可能的毛竹CGT基因(PH01000603G0510)编码的蛋白质氨基酸序列,计算出其分子量为50.5 kDa,与SDS-PAGE分析毛竹CGT分子量的结果基本一致。
4 结论通过对毛竹叶黄酮碳苷C-糖基转移酶(CGT)提取、分离纯化及质谱鉴定得到可能的毛竹叶黄酮碳苷CGT基因序列(PH01000603G0510)。并确定毛竹叶黄酮碳苷C-糖基化途径,主要途径是CGT催化木犀草素和UDP-葡萄糖直接合成,次要途径是CGT催化圣草素查尔酮和UDP-葡萄糖或圣草素和UDP-葡萄糖间接合成。毛竹叶CGT极易催化生成C-6黄酮苷(异荭草苷),而极少催化生成C-8黄酮苷(荭草苷)。CGT催化生物合成异荭草苷的反应体系中,CGT最佳催化反应时间为40 min,反应温度28 ℃,缓冲盐pH8.1,木犀草素浓度31.76 μmol·L-1。
Brazier-Hicks M, Evans K M, Gershater M C, et al. 2009. The C-glycosylation of flavonoids in cereals. Journal of Biological Chemistry, 284(27): 17926-17934. DOI:10.1074/jbc.M109.009258 |
Chen A Y, Chen Y C. 2013. A review of the dietary flavonoid, kaempferol on human health and cancer chemoprevention. Food Chemistry, 138(4): 2099-2107. DOI:10.1016/j.foodchem.2012.11.139 |
Chen D W, Sun L L, Chen R D, et al. 2016. Enzymatic synthesis of acylphloroglucinol 3-C-glucosides from 2-O-glucosides using a C-glycosyltransferase from Mangifera indica. Chemistry-A European Journal, 22(17): 5873-5877. DOI:10.1002/chem.201600411 |
Costa F D, Barber C J S, Kim Y B, et al. 2017. Molecular cloning of an ester-forming triterpenoid: UDP-glucose 28-O-glucosyltransferase involved in saponin biosynthesis from the medicinal plant Centella asiatica. Plant Science, 262: 9-17. DOI:10.1016/j.plantsci.2017.05.009 |
Devouge V, Rogniaux H, Nési N, et al. 2007. Differential proteomic analysis of four near-isogenic Brassica napus varieties bred for their erucic acid and glucosinolate contents. Journal of Proteome Research, 6(4): 1342-1353. DOI:10.1021/pr060450b |
Egorova K S, Toukach P V. 2016. CSDB_GT: a new curated database on glycosyltransferases. Glycobiology, 27(4): 285-290. |
Falcone Ferreyra M L, Rodriguez E, Casas M L, et al. 2013. Identification of a bifunctional maize C- and O-glucosyltransferase. Journal of Biological Chemistry, 288(44): 31678-31688. DOI:10.1074/jbc.M113.510040 |
Fukuchi-Mizutani M, Okuhara H, Fukui Y, et al. 2003. Biochemical and molecular characterization of a novel UDP-glucose: Anthocyanin 3'-O-glucosyltransferase, a key enzyme for blue anthocyanin biosynthesis, from gentian. Plant Physiology, 132(3): 1652-1663. DOI:10.1104/pp.102.018242 |
Guo X F, Yue Y D, Tang F, et al. 2013. A comparison of C-glycosidic flavonoid isomers by electrospray ionization quadrupole time-of-flight tandem mass spectrometry in negative and positive ion mode. International Journal of Mass Spectrometry, 333(1): 59-66. |
Hamilton M L, Kuate S P, Brazier-Hicks M, et al. 2012. Elucidation of the biosynthesis of the di-C-glycosylflavone isoschaftoside, an allelopathic component from Desmodium spp. that inhibits Striga spp. development. Phytochemistr, 84(6): 169-176. |
Hirade Y, Kotoku N, Terasaka K, et al. 2015. Identification and functional analysis of 2-hydroxyflavanone C-glucosyltransferase in soybean (Glycine max). Febs Letters, 589(15): 1778-1786. DOI:10.1016/j.febslet.2015.05.010 |
Hofer B. 2016. Recent developments in the enzymatic O-glycosylation of flavonoids. Applied Microbiology and Biotechnology, 100(10): 4269-4281. DOI:10.1007/s00253-016-7465-0 |
Karim M R, Hashinaga F. 2002. Isolation and characterization of limonoid glucosyltransferase from pummelo albedo tissue. Food Chemistry, 76(4): 431-436. DOI:10.1016/S0308-8146(01)00300-4 |
Nagatomo Y, Usui S, Ito T, et al. 2014. Purification, molecular cloning and functional characterization of flavonoid C-glucosyltransferases from Fagopyrum esculentum M. (buckwheat) cotyledon. The Plant Journal, 80(3): 437-448. DOI:10.1111/tpj.2014.80.issue-3 |
Peng Z H, Lu Y, Li L B, et al. 2013. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla). Nature Genetics, 45(4): 456-461. DOI:10.1038/ng.2569 |
Sasaki N, Nishizaki Y, Yamada E, et al. 2015. Identification of the glucosyltransferase that mediates direct flavone C-glucosylation in Gentiana triflora. Febs Letters, 589(1): 182-187. DOI:10.1016/j.febslet.2014.11.045 |
Sawada S, Suzuki H, Ichimaida F, et al. 2005. UDP-glucoselucuronic acid: anthocyanin glucuronosyltransferase from red daisy (Bellis perennis) flowers. Journal of Biological Chemistry, 280(2): 899-906. DOI:10.1074/jbc.M410537200 |
Vogt T, Jones P. 2000. Glycosyltransferases in plant natural product synthesis: characterization of a supergene family. Trends in Plant Science, 5(9): 380-386. DOI:10.1016/S1360-1385(00)01720-9 |
Wang T T, Wang H Y, Cai D W, et al. 2017. Comprehensive profiling of rhizome-associated alternative splicing and alternative polyadenylation in moso bamboo (Phyllostachys edulis). The Plant Journal, 91(4): 684-699. DOI:10.1111/tpj.2017.91.issue-4 |
Yabuya T, Yamaguchi M A, Imayama T, et al. 2002. Anthocyanin 5-O-glucosyltransferase in flowers of Iris ensata. Plant Science, 162(5): 779-784. DOI:10.1016/S0168-9452(02)00021-3 |
Zhang Y, Bao B L, Lu B Y, et al. 2005. Determination of flavone C-glucosides in antioxidant of bamboo leaves (AOB) fortified foods by reversed-phase high-performance liquid chromatography with ultraviolet diode array detection. Journal of Chromatography A, 1065(2): 177-185. DOI:10.1016/j.chroma.2004.12.086 |
Zhao H S, Peng Z H, Fei B H, et al. 2014. BambooGDB: a bamboo genome database with functional annotation and an analysis platform. Database-OXFORD, 2014(10): bau006. |
 2018, Vol. 54
2018, Vol. 54

