文章信息
- 邱帅, 沈柏春, 李婷婷, 郭娟, 王霁, 孙丽娜, 陈徐平, 胡绍庆
- Qiu Shuai, Shen Baichun, Li Tingting, Guo Juan, Wang Ji, Sun Lina, Chen Xuping, Hu Shaoqing
- 基于随机森林算法和SRAP分子标记的桂花品种鉴定方法
- A Method of Osmanthus fragrans Cultivars Identification Based on Random Forest Algorithm and SRAP Molecular Markers
- 林业科学, 2018, 54(1): 32-45.
- Scientia Silvae Sinicae, 2018, 54(1): 32-45.
- DOI: 10.11707/j.1001-7488.20180104
-
文章历史
- 收稿日期:2017-04-11
- 修回日期:2017-08-16
-
作者相关文章
2. 浙江省林业科学研究院 杭州 310023;
3. 浙江理工大学 杭州 310018
2. Zhejiang Forestry Academy Hangzhou 310023;
3. Zhejiang Sci-Tech University Hangzhou 310018
桂花(Osmanthus fragrans)系木犀科(Oleaceae)木犀属(Osmanthus)木犀组(Sect.Osmanthus)植物, 是我国传统十大名花之一, 至今已有2 500多年栽培历史, 广泛应用于园林绿化(向其柏等, 2008)。桂花品种繁多, 仅2008年版《中国桂花品种图志》就详细记录了122个品种(向其柏等, 2008), 且每年仍有不少的新品种被选育出来。不同桂花品种的营养性状相似, 而生殖性状差异较大, 品种鉴别主要依据花色、花序类型、花形态和花期等生殖器官特质(杨康民, 2013), 因此未到开花年龄的桂花幼苗很难进行品种鉴定。此外, 一些品种的性状差异极小, 且同一品种的桂花在不同立地条件下, 花、叶等器官易出现变异, 也大大增加了桂花品种鉴定的难度。目前, 桂花苗木生产和园林应用中常出现品种混杂、以次充好的现象, 急需一种快速、简便、准确的桂花品种鉴定方法。
DNA分子标记反映植物遗传差异, 不受环境因素影响和生长年龄限制, 理论上可以提供无限多的标记, 以此为基础的DNA指纹图谱广泛应用于品种鉴定、品种权益保护和种质资源保护中(李婷婷等, 2016)。国内外已使用SSR、AFLP、RAPD、DArT(多样性序列芯片技术, diversity arrays technology)、SRAP和ISSR等多种分子标记构建了DNA指纹图谱, 用于鉴定玫瑰(Rosaspp.)(Akond et al., 2012)、欧洲榛(Corylus avellana)(Ferrari et al., 2005)、芒果(Mangifera indica)(Faleiro et al., 2010)、六倍体小麦(Triticum aestivum)(Akbari et al., 2006)、大叶榉树(Zelkova schneideriana)(李婷婷等, 2016)、非洲菊(Gerbera jamesonii)(Bhatia et al., 2009)、橡胶树(Hevea brasiliensis)(钟淦彬等, 2013)和新疆野苹果(Malus sieversii)(马衣努尔姑·吐地等, 2016)等多种植物的栽培品种、种质资源和杂交系。桂花的DNA指纹图谱研究较少, 刘龙昌等(2004)使用2条RAPD引物构建了34个桂花品种、6个野生桂花个体、6个木犀属其他种的DNA指纹图谱; 段一凡等(2014)使用11对SSR引物建立了64个桂花品种的荧光SSR指纹图谱; 乔中全等(2016)使用10条ISSR引物建立了45个桂花品种的DNA指纹图谱。
无论是使用表型标记, 还是DNA分子标记, 植物品种鉴定的要求都是确定未知样品的类别, 其实质是一个分类算法(classification), 即通过对已知类别训练集的学习, 得到一个模型, 将每个属性映射到目标属性, 以此预测新数据的类别, 是一种有监督的学习(罗可等, 2005)。因此, 植物品种分子鉴定必须包括以下2个主要步骤:1)根据已知品种的样品DNA标记信息构建每个品种的DNA指纹图谱; 2)将待鉴定样品的DNA标记信息与DNA指纹图谱进行比对, 确定未知样品为哪个品种。而传统DNA指纹图谱研究通常使用分子标记信息计算遗传距离, 从而进行聚类分析(陈鹭真等, 2002), 检验能否有效区分已知样品, 但无法鉴定未知样品。可见, 错误的统计分析方法是导致DNA指纹图谱无法广泛应用于品种鉴定的主要原因。
目前, 已有多种分类算法在众多领域得到广泛应用, 包括决策树、贝叶斯分类、支持向量机和神经网络等, 不同分类算法都有各自的优缺点(罗可等, 2005)。由Breiman(2001a)和Cutler等(2007)提出的随机森林(random forest, RF)算法是一种基于分类树的算法, 在变量和数据的使用上进行随机化, 生产很多分类树, 再汇总分类树的结果(李欣海, 2013)。在自助法(Bootstrap)重采样过程中, 原始数据中约2/3的样本作为训练集, 约1/3未被选取的样本作为袋外数据(out of bag, OOB), 用于预测分类的准确率。Breiman(2001b)证明, 袋外数据误差估计(OOB error)是泛化误差估计的组成, 可以取代测试集的误差估计。随机森林算法具有诸多优点: 1)预测精度高; 2)能够处理大量数据集和变量, 运算速度快; 3)能够给出变量的重要性估计; 4)可以产生泛化误差的内部无偏估计(OOB error), 无需交叉验证或者采用单独的测试集进行误差估计; 5)能够有效估计缺失值; 6)不会产生过度拟合(Breiman, 2001b)。基于多种优点, 随机森林算法被誉为目前最好的算法之一, 在多个领域得到了广泛应用(李欣海, 2013)。
本研究采用随机森林算法和SRAP分子标记, 建立了45个桂花品种的分类模型, 实现桂花品种鉴定的自动化, 为桂花种苗生产、推广应用和种质资源保护提供了一种快速、准确的品种鉴定方法, 并可以应用于其他植物品种的鉴定。
1 材料与方法 1.1 试验材料以45个桂花品种或变异类型为材料(表 1), 所有材料保存于浙江省杭州市余杭区杭州市园林绿化股份有限公司国家桂花种质资源库。采集新鲜的叶片, 装入冰袋中, 迅速保存于-20 ℃备用。每个品种采集5株叶片, 作为重复。
|
|
桂花栽培品种的鉴定步骤包括: 1)取样和DNA提取; 2) SRAP-PCR扩增; 3)毛细管电泳检测; 4)单对引物鉴定能力的评价; 5)引物的选择、分类模型建模及评价; 6)未知样品鉴定。为了实现品种高效、准确的鉴定, 须按照以下描述的方法严格执行。
1.2.1 DNA提取取0.5 ~1 g健康、新鲜的叶片, 用75%酒精擦拭干净, 液氮速冻后快速研磨至粉末, 使用植物基因组快速提取试剂盒(购自北京百泰克生物技术有限公司)提取DNA。使用1.0%琼脂糖凝胶电泳检测DNA质量, 并使用NanaDrop 2000/2000c分光光度计(购自Thermo公司)测定浓度, 稀释至20~50 ng·μL-1, -20 ℃储存备用。
1.2.2 SRAP-PCR扩增和毛细管电泳检测SRAP-PCR采用10 μL反应体系, 含有DNA模板1 μL, 10 μmol ·L-1正反引物各1 μL, 2×Power Taq PCR MasterMix 5 μL(购自北京百泰克生物技术有限公司, 内含Tag酶、dNTPs、Mg2+、缓冲液和反应稳定剂), 双蒸水2 μL。SRAP引物采用李婷婷等(2016)收集整理的10个正向引物和9个反向引物组成90对引物组合(表 2), 由上海英骏生物技术有限公司合成。使用ABI VeritiTM基因扩增仪(购自ABI公司)进行PCR扩增, 反应程序参考李婷婷等(2016)并稍做改进:94 ℃预变性8 min; 94 ℃变性1 min, 35 ℃退火1 min, 72 ℃延伸1 min 30 s, 5个循环; 94 ℃变性1 min, 59.3 ℃退火1 min, 72 ℃延伸1 min 30 s, 35个循环; 最后72 ℃延伸8 min, 4 ℃保存。
|
|
PCR产物使用2.0%琼脂糖凝胶电泳检测筛选引物, 根据扩增稳定性、条带的清晰度和多态性条带数筛选出10对引物组合, 将扩增产物稀释10倍, 取1 μL上样至Qsep100TM全自动核酸分析仪(购自Bioptic公司)进行毛细管电泳检测, 以5 000 bp DNA Ladder标定扩增条带大小, 使用Q-Editor(Bioptic公司)软件收集记录扩增条带的峰值信号, 采用李婷婷等(2016)方法对毛细管电泳图进行处理, 输出0/1数据。
1.2.3 单对引物对桂花品种鉴别能力评价采用PowerMarker软件计算每对引物多态信息含量(PIC)。使用R语言对引物的带型分布进行χ2检测, 计算χ2值量化每对引物的带型分布情况。
采用Tessier等(1999)构建的参数鉴别能力(the discriminating power, D)作为评价单对引物对桂花品种鉴定能力的指标, 该参数描述的是从所用样品中随机抽取2个样品具有不同带型的概率, 即引物能够区分这2个样品的概率, 第j对引物的Dj按照以下公式计算:
| $ {D_j} = 1 - \sum\limits_{i = 1}^n {{p_i}} \frac{{\left( {N{p_i} - 1} \right)}}{{N - 1}}。$ |
式中:N为样品数; n为第j个引物的带型数; pi为第i个带型的频率。当N趋于无穷大, 即样品数无穷多时, D极限值DL按照以下公式估算:
| $ {D_{\rm{L}}} = {\rm{lim}}{D_j} = 1 - \sum\limits_{i = 1}^n {{p_i}^2}。$ |
随着测试样品数增加, 引物的分辨能力D逐渐减小至极限值DL, 因此当样品数需要增加时, 使用DL作为引物分辨能力的指标。
可以用于鉴定某个样品的带型称为有效带型, 其数量P按照Belaj等(2003)的公式计算:
| $ P = \frac{1}{{1 - {D_{\rm{L}}}}}。$ |
使用R语言根据上述公式编程, 计算各指标值。使用R语言对D、多态性条带、带型数、有效带型数、PIC和χ2进行相关性分析, 并采用scatterplot.matrix函数绘制散点矩阵图(Templ et al., 2012), 确定各参数与引物鉴别能力的关系。
1.2.4 引物对组合筛选第j对引物无法区分的样品对数xj= [N(N-1)/2](1-Dj)。当单对引物的xj不为0时, 即无法完全区分所有样品时, 需要通过多对引物组合增加鉴别能力。假设用于分类的引物相互独立, 则k对引物组合无法区分的样品对数期望值为:
| $ {X_{{\rm{E}},k}} = \frac{{N\left( {N - 1} \right)}}{2}\prod\limits_{j = 1}^k {\left( {1 - {D_j}} \right)}。$ |
当不相互独立, k对引物组合无法区分的样品形成m个亚群时, 无法区分的样品对数观测值XO, k由以下公式计算:
| $ {X_{{\rm{O}},k}} = \sum\limits_{i = 1}^m {{{\rm{C}}_{{n_i}}}^2} 。$ |
式中:ni为每个亚群的样品数。由于连锁现象常见, 引物组合的XE, k常常无法完全为0, 但只要观测值XO, k为0, 即可区分所有的样品。
使用R语言编程实现引物组合的优化:采用穷举法计算出k(k为0~引物数之间的正整数, 初始值设为1)对引物所有可能组合的XO,k, 当所有XO, k均大于0时, 使用k=k+1重复上述步骤, 直到XO,k=0时终止计算, 并输出引物组合。
1.2.5 随机森林分类模型构建与评估以筛选出的引物组合的条带数据作为分类特征(自变量), 每个品种5个重复, 总共225个样本, 确保通过Bootstrap重采样获得的训练集中含有每个品种的数据。采用随机森林算法, 使用R语言程序包randomForest构建分类模型(Breiman, 2002)。参数设置为:树的数量(ntree)为1 000, 每个分支所选择的变量数(mtry)为4, 并计算临近矩阵, 其他参数为默认。使用平均OOB误判率评估模型泛化能力。使用MDSplot函数输出经过标准化临近矩阵产生的3个维度坐标数据, 并使用rgl包绘制样品在三维空间的分布图(Murdoch, 2017), 图形化展示分类效果。采用predict函数鉴定品种, 设置参数type="prob", 输出每个鉴定结果的准确性估计。
2 结果与分析 2.1 单对引物分辨能力评价从90对SRAP引物中筛选出10对多态性较高的引物组合, 对225个样本(45个桂花品种, 每个品种5个重复)进行扩增, 共检测出151个重复性较好的多态性位点, 平均每对引物15.1个, 多态信息指数(PIC)为0.20~0.34, 平均0.26(表 3)。10对引物的多态性位点共形成339个带型组合, 平均每对引物33.9个带型。以χ2表示各引物带型的分布情况, 其值越小, 则带型分布越均匀, 结果(表 3)显示, 引物对me5/em5的带型接近均匀分布, 而me9/em6和me5/em8的带型表现为严重的偏分布。
|
|
10对引物的平均分辨能力(D)为0.97, 不同引物对的分辨能力差异较大(表 4)。引物对me5/em5和me4/em5的分辨能力最强, 都为0.99, DL为0.97, 有效带型数都为38.21, 无法区分的样品对数x都为4, 但品种不同:前者为ZYH, CHDG; ZY, LGZY; MHZY, FDZ; CYQ, YYSJ; 后者为BYJ, HLJDTZ; ZYH, ZSDG, CHDG。引物对me5/em8的D最小, 仅为0.89, DL为0.87, 有效带型数仅为7.82, 无法区分品种:ZY, JQZ; ZYH, ZSDG; BYJ, CHDG; YLL, BJ; KYZY, MBX; ZYG, DZ; WY, LZH; XHG, FDZ, RXG; QJ, XYS, CYQ, DHJG; YLYS, MHZY, JQG, HZH, ZZH, ZJH, HLJD, HLJDTZ, LZDG, CYG, YYSJ, CHSJ, TNSH, GZ, x为107。DL为样品无穷多时估计的分辨能力, 所以当样品数增加时, 根据DL评价引物的分辨能力较为合适。
|
|
图 1显示, 引物的分辨能力与引物多个指标存在相关性。D为PIC的拓展(Teesier et al., 1999), 前者依据引物的带型频率, 后者依据引物的等位基因频率, 因此D与PIC为较弱正相关关系, 但与带型数和有效带型数为强正相关关系(图 1)。部分样品共同具有的带型为无效带型, 因此有效带型数与D的相关性强于带型数。D与χ2为强负相关关系(图 1), 表明带型分布显著影响引物的鉴别能力, 带型分布均匀的引物鉴别能力强于偏分布的引物。图 1显示, D与多态性条带数无显著相关关系, 这可能是因为本研究中样品数较少且亲缘关系较近, 含有n个多态性条带的引物产生的实际带型数远小于理论最大值2n。
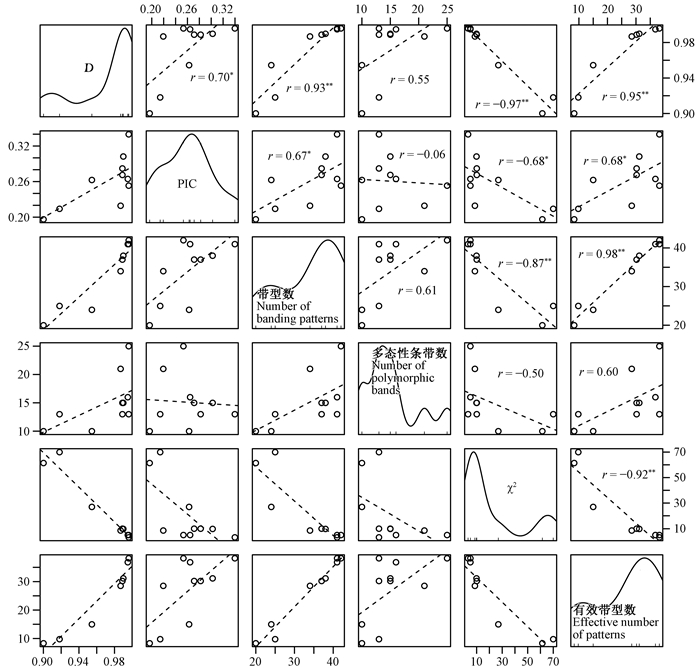
|
图 1 SRAP引物指标与分辨能力(D)的相关性分析 Figure 1 Correlation analysis of index and the discriminating power(D) of SRAP primer pairs *:在0.05水平显著; **:在0.01水平显著; r为Person相关系数; 对角为指标的概率密度。 *:Significant at 0.05 level; **:Significant at 0.01 level; r is Person's correlation coefficient; Diagonal is probability density. |
由于所有10对SRAP引物都无法完全区分45个桂花品种, 因此需要考虑多个引物对的组合。结果(表 5)显示, 2个引物组合就能完全区分所有桂花品种, 共有8对引物组合, D都为1, 都能完全区分所有45个桂花品种, 条带数为20~27。
|
|
以8对引物组合的条带数据作为分类特征, 构建8个分类模型rf1-rf8(表 5)。图 2为8个分类模型的OOB估计和对每个品种的OOB误判率, 值越低则泛化能力越强, 即该模型对预测样本的适应能力越强, 鉴定能力越强。8个分类模型都有极小的平均OOB估计, 在0.004 4~0.013 9之间, 且差异不大, 表明8个分类模型总体上都有较高的泛化能力。其中, 模型rf5和rf3的OOB估计最小, 而rf8最高。不同分类模型对不同品种的OOB误判率存在差异, 表明不同分类模型对不同品种的鉴定能力存在一定差异。其中rf1、rf3、rf4、rf5和rf7对各桂花品种的OOB误判率差异不大, 表明这些模型对不同品种的鉴定能力差异较小。rf2和rf8对桂花品种LZDG的OOB误判率达到0.104 8和0.111 0, 远高于其他品种, 因此对该品种的鉴定能力较差。rf6对品种ZYH的OOB误判率达到0.101 4, 表明该模型对其鉴定的能力较差。

|
图 2 不同分类模型的OOB估计 Figure 2 The OOB estimation of different classification models OOB为使用袋外数据估算的误差。OOB is the estimated error using out of bag data. |
图 3的坐标为模型中的条带数据(特征)经过缩放后得到, 用于展示不同分类模型中所有样品在三维空间的分布情况, 图中相同颜色的点为同一品种。由图 3可知, 8个分类模型都有较高的分类能力, 但存在一定差异。根据样品的分布情况, 8个模型可以大致分为3类:模型rf1中, 45个桂花品种分布较散, 可以明显区分, 具有极强的分类能力; 模型rf3、rf4、rf5和rf7中, 部分桂花品种距离较近, 但无交叉现象, 仍然能够较为明显地区分45个桂花品种; 模型rf2、rf6和rf8中, 少量桂花品种出现聚集交叉现象, 尤其rf2中的交叉现象最为明显, 对分类效果会产生不利影响。
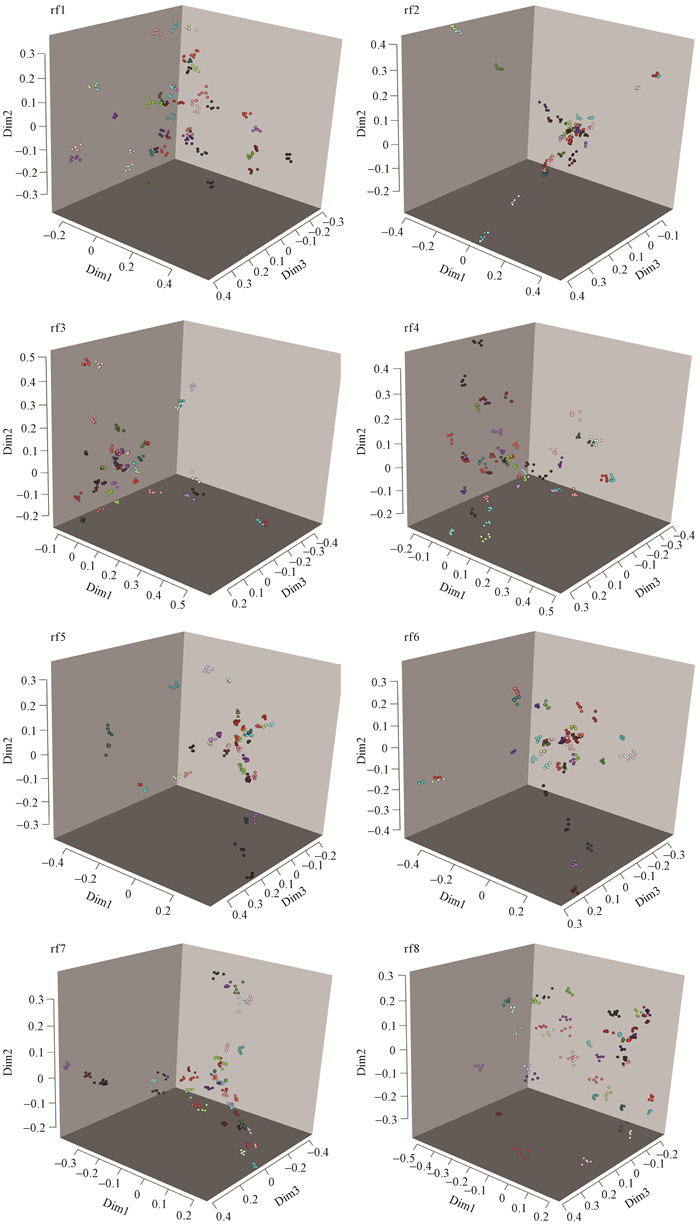
|
图 3 不同桂花品种的三维分布 Figure 3 The 3-D distribution of different Osmanthus fragrans cultivars rf1-rf8为8个分类模型; 3个坐标为原有特征经过缩放后得到, 只有数学意义, 并非实际存在的坐标; 相同颜色的点表示相同品种, 总共225个样品, 每个品种5个重复。rf1-rf8 are 8 classification models; 3 coordinates is scaled attributes, only have mathematical meaning, not real coordinates; The dots with same color mean the same varieties, total of 225 samples, 5 samples per cultivars. |
综上所述, 8个分类模型中, rf1、rf3、rf4、rf5和rf7具有较强的泛化能力和分类能力, 其对应的引物组合分别为me1/em3+me9/em6、me4/em5+me9/em6、me4/em8+me9/em6、me6/em9+me9/em6和me5/em5+me9/em6, DNA指纹图谱见表 6。
|
|
Tessier等(1999)研究认为, 使用分辨能力较高的引物进行组合能获得高分辨能力。本研究中, 8个分类模型所选的单对引物分辨能力并不是都很强, 只有4个模型采用D值最高的引物me5/em5和me4/em5, 而6个模型采用D值只为0.918 2的引物me9/em6, 可见低分辨能力的引物进行组合也能获得较高的分辨能力。考虑到不同引物的带型在样品中共分布的程度会影响引物组合的分类效果, 共分布的样品常常无法区分, 而引物的相关性可以反映它们之间共分布的程度, 因此使用卡方检验检测10对引物的相关性。图 4显示, 8个分类模型所选择的引物都不相关。引物me5/em5和me4/em5的分辨能力最强, 仅无法区分4对样品, 但有一定的相关性, 都无法区分ZYH和CHDG这2个品种(表 4)。引物对me9/em6虽然分辨能力差, 但与me5/em5和me4/em5的相关性极弱, 无法区分的样品对没有重合, 所以me9/em6分别与me5/em5和me4/em5组合的模型rf3和rf7具有极强的分类能力。而其他模型也基本是由1对分辨能力较强的引物加1对分辨能力较弱且不显著相关的引物组合而成。8个模型的分类能力也与模型中2对引物之间的相关性有一定相关性, 即rf1、rf3、rf4、rf5和rf7的分类能力最强, 其引物之间的相关性也最弱。因此, 在进行引物筛选时, 除了引物自身的鉴别能力外, 还需要考虑引物之间的相关性。

|
图 4 10对引物的相关性 Figure 4 The correlation of 10 primer pairs 点的颜色代表卡方检验的显著性, 大于0.05表明2对引物不相关, 反之则显著相关。The colours of dots mean the significance of Chi square test.The significance greater than 0.05 indicates 2 primer pairs are uncorrelated, otherwise, significantly correlated. |
作为分类属性, DNA分子标记是决定桂花品种分类模型分类能力的主要因素之一, 需要满足操作简单、易开发、结果稳定等要求。根据单对或单个引物扩增位点的数量, DNA分子标记可分为单位点标记和多位点标记。以SSR和SNP标记为代表的单位点标记具有多态性高、共显性、结果稳定等优点, 在遗传研究中得到了广泛应用(黄秦军等, 2002; 曹廷杰等, 2015); 而以AFLP、ISSR和RAPD为代表的多位点标记则都有一系列设计好的通用引物, 无需单独开发引物, 少量引物就可以产生大量多态性位点。SSR和SNP标记开发需要基因组或转录组信息, 随着高通量测序技术的普及, 很多物种都已开发了大量的SSR和SNP标记, 但桂花或木犀属植物仍然无相关报道, 单独开发的成本较高。SNP标记一般通过重测序技术或生物芯片技术实现高通量检测, 但成本高、技术复杂, 需要专业的仪器, 而针对每个位点设计引物进行PCR扩增, 则需要进行大量的引物筛选工作, 且需要高精度的检测技术检测单个碱基的多态性, 如Sanger测序技术或高分辨率溶解曲线技术(Han et al., 2011)。相似地, SSR标记的开发也需要设计大量的引物并进行筛选, 虽然已经开发了少量的桂花SSR标记, 但桂花品种繁多, 且亲缘关系较近, 不同品种出现相同带型的概率较大, 仍然需要开发更多的SSR标记才能满足品种鉴定需求(段一凡等, 2014)。Belaj等(2003)研究表明, 多位点标记与单位点标记具有相似的品种鉴别能力。AFLP技术虽然结果稳定, 但操作复杂(Vos et al., 1995), 而ISSR(Reddy et al., 2002)和RAPD技术(Sharma et al., 1996)由于采用单引物设计, 虽然通用性强, 但是扩增结果不稳。相关序列扩增多态性(sequence-related amplified polymorphic, SRAP)是Li等(2001)开发的一种新型分子标记, 该标记根据外显子富含GC而启动子、内含子富含AT设计引物进行扩增, 具有AFLP技术优点, 但操作简单, 是用于品种鉴别的理想分子标记(邱帅等, 2013)。本研究采用毛细管电泳SRAP分子标记技术, 实现了稳定快速的桂花DNA检测, 为桂花的分类模型提供了大量多态性位点。综上所述, SRAP标记通用性强、操作简单、稳定性高、成本低, 能够提供较为丰富的多态性位点, 是苗木生产和园林应用中品种鉴定的理想分子标记。
桂花品种繁多, 且不断有新的品种被选育出来, 很难一次性收集所有品种的DNA标记信息, 当新增加品种DNA信息与已有品种一致时, 会出现错误鉴定情况, 这也是DNA指纹图谱应用于植物品种鉴定的一大难题。本研究从以下几个方面尝试解决该难题。首先, 桂花品种虽多, 但广泛应用的较少。本研究所选材料中, 40个品种获得木犀属国际登录权, 为目前应用较为广泛或具有较高推广价值的品种(向其柏等, 2008), 使用这些品种构建的分类模型能基本满足目前的需求; 其他材料为原有品种中筛选出的变异株, 与原品种极为相似, 用于测定鉴定方法的精度, 如‘彩叶桂’为‘天女散花’变异, ‘火炼金丹(跳枝)’为‘火炼金丹’芽变枝条, ‘四季桂黄’为‘四季桂’变异, 本研究的鉴定方法可以有效地分辨这些材料, 具有极高的鉴定精度。其次, SRAP标记具有较高的多态性, 对于鉴定45个桂花品种, 本研究中检测到的SRAP标记多态性信息是充分冗余的:分类模型只使用了部分多态性位点, 如模型rf2只使用了所选引物60%的位点, 即可分辨所有45个桂花品种, 剩下的位点可以用于鉴定新增加的品种; 10对SRAP引物中, 8个分类模型总共只选择了6对引物, 而每个模型只选择了2对引物, 当模型内冗余位点信息无法分辨新增加的品种时, 通过增加引物数量, 可以大大增加模型的鉴定容量。因此, 本研究所描述的方法不仅确保了常用桂花品种的准确鉴定, 也为其他品种或新品种预留足够的冗余SRAP标记信息, 只需将新增加品种的SRAP标记信息增加到训练集, 重新训练, 而无需单独开发引物, 即可用于该品种的鉴定。
Tessier等(1999)描述了一种逐步最小化无法区分的样品对数期望值的引物组合优化方法, 该方法假设所有备选引物带型分布相互独立; 但由于长期的人工选育, 栽培品种往往有较高的连锁不平衡性, 导致不同引物的带型分布很难相互独立, 使得无法区分的样品数期望值与实际值存在较大差异, 只能得出局部最优解, 无法得出全局最优解。本研究10对SRAP引物中, 有较多引物的带型在45个桂花品种中出现了不同程度的共分布现象, 采用此种方法得出需要3对引物能够完全区分所有品种, 即me5/em5+ me4/em5+ me9/em6, 而实际上只需2对引物就能完全区分所有品种。考虑到本研究所用引物较少, 因此采用穷举法, 计算出k对引物所有组合的无法区分样品对数实际值X, k从1开始增加, 直至X=0, 能够得到精确的全局最优解。但当引物数较多时, 此种方法需要消耗较长运算时间并占用较大内存, 此时Tessier等(1999)的方法是一种快速得到近似最优解的方法。此外, 随机森林模型也能够给出位点或引物对品种鉴定的重要性估计(李欣海, 2013), 作为引物组合筛选的依据, 并且计算速度和内存占用率优于穷举法, 但是很难给出一定的阈值作为选择标准, 并且剔除一部分引物或位点后, 模型的分类能力和剩下引物或位点对分类的重要性会发生较大改变, 因此每轮剔除都需要回溯。值得注意的是, 本研究中引物的分辨能力与其对分类的重要性并不是线性正相关的, 因此, 随机森林模型重要性估计能否作为引物组合优化的指标需要进一步研究。
4 结论采用SRAP分子标记技术, 构建了8个随机森林分类模型, 能够鉴别所有45个桂花品种, 根据模型的泛化能力和分类效果, 模型rf1、rf3、rf4、rf5和rf7的分类能力最佳, 引物对组合分别为me1/em3+me9/em6、me4/em5+me9/em6、me4/em8+me9/em6、me6/em9+me9/em6和me5/em5+me9/em6。引物自身的分辨能力越强, 引物之间的相关性越小, 引物组合的分类效果越好。SRAP标记的高多态性使得分类模型有较高的冗余信息, 不但能确保常用桂花品种的准确鉴定, 也能满足不断出现新品种的鉴定。采用毛细管电泳技术实现了SRAP标记信息自动检测, 而采用随机森林算法实现了学习、建模和鉴定的自动化和标准化, 该方法不仅可以应用于桂花品种的鉴定, 也可以应用于其他植物品种的鉴定。
曹廷杰, 谢菁忠, 吴秋红, 等. 2015. 河南省近年审定小麦品种基于系谱和SNP标记的遗传多样性分析[J]. 作物学报, 41(2): 197-206. (Cao T J, Xie J Z, Wu Q H, et al. 2015. Genetic diversity of registered wheat varieties in Henan Province based on pedigree and single-nucleotide polymorphism[J]. Acta Agronomica Sinica, 41(2): 197-206. [in Chinese]) |
陈鹭真, 李振基, 周涵韬. 2002. 福建省桫椤科植物的分子分类学研究[J]. 厦门大学学报:自然科学版, 41(4): 481-486. (Chen L Z, Li Z J, Zhou H T. 2002. Study on molecular taxonomy of 5 species of Cyatheaceae in Fujian[J]. Journal of Xiamen University:Natural Science Edition, 41(4): 481-486. [in Chinese]) |
段一凡, 王贤荣, 梁丽丽, 等. 2014. 桂花品种SSR荧光指纹图谱的构建[J]. 南京林业大学学报:自然科学版, 38(s1): 1-6. (Duan Y F, Wang X R, Liang L L, et al. 2014. Fingerprinting and identification of Osmanthus fragrans cultivars using fluorescence-labeled SSR markers[J]. Journal of Nanjing Forestry University:Natural Science Edition, 38(s1): 1-6. [in Chinese]) |
黄秦军, 苏晓华, 张香华. 2002. SSR分子标记与林木遗传育种[J]. 世界林业研究, 15(3): 14-21. (Huang Q J, Su X H, Zhang X H. 2002. Microsatellite marker and its application in tree genetics and breeding[J]. World Forestry Research, 15(3): 14-21. [in Chinese]) |
李欣海. 2013. 随机森林模型在分类与回归分析中的应用[J]. 应用昆虫学报, 50(4): 1190-1197. (Li X H. 2013. Using "random forest" for classification and regression[J]. Chinese Bulletin of Entomology, 50(4): 1190-1197. DOI:10.7679/j.issn.2095-1353.2013.163 [in Chinese]) |
李婷婷, 朱锦茹, 邱帅, 等. 2016. 基于CE-SRAP标记的榉树优良单株指纹图谱构建[J]. 植物遗传资源学报, 17(1): 169-176. (Li T T, Zhu J R, Qiu S, et al. 2016. Fingerprinting construction for superior individuals of Zelkova schneideriana based on CE-SRAP[J]. Journal of Plant Genetic Resources, 17(1): 169-176. [in Chinese]) |
刘龙昌, 向其柏, 刘玉莲. 2004. RAPD标记在桂花遗传多样性检测和品种鉴定中的应用(英文)[J]. 南京林业大学学报:自然科学版, 28(s1): 76-82. (Liu L C, Xiang Q B, Liu Y L. 2004. The application of RAPD markers in diversity detection and cultivars identification of Osmanthus fragrans[J]. Journal of Nanjing Forestry University:Natural Sciences Edition, 28(s1): 76-82. [in Chinese]) |
罗可, 林睦纲, 郗东妹. 2005. 数据挖掘中分类算法综述[J]. 计算机工程, 31(1): 3-5. (Luo K, Lin M G, Xi D M. 2005. Review of classification algorithms in data mining[J]. Computer Engineering, 31(1): 3-5. [in Chinese]) |
马衣努尔姑·吐地, 张延辉, 秦伟, 等. 2016. 基于SSR分子标记技术的新疆苹果资源指纹图谱的构建. 新疆农业大学学报, 39(1): 26-34. (Maynur T, Zhang Y H, Qing W, et al. 2016. Construction of fingerprint of apple resources in Xinjiang based on SSR molecular markers. Journal of Xinjiang Agricultural University, 39(1): 26-34. [in Chinese]) |
乔中全, 王晓明, 李永欣, 等. 2016. 桂花优良品种'珍珠彩桂'遗传多样性的ISSR分析及指纹图谱构建[J]. 湖南林业科技, 43(3): 1-5. (Qiao Z Q, Wang X M, Li Y X, et al. 2016. Genetic diversity and fingerprint construction of varieties of Osmanthus fragrans 'Zhenzhu Caigui' by ISSR markers[J]. Hunan Forestry Science Technology, 43(3): 1-5. [in Chinese]) |
邱帅, 丁信誉, 席梦利, 等. 2013. 东方百合SRAP体系优化及引物筛选[J]. 南京林业大学学报:自然科学版, 37(3): 6-10. (Qiu S, Ding X Y, Xi M L, et al. 2013. Optimization of SRAP PCR system and primers screening of oriental lily hybrids[J]. Journal of Nanjing Forestry University:Natural Science Edition, 37(3): 6-10. [in Chinese]) |
向其柏, 刘玉莲. 2008. 中国桂花品种图志[M]. 杭州: 浙江科学技术出版社: 2, 8, 80-81, 86. (Xiang Q B, Liu Y L. 2008. An illustrated monograph of the sweet osmanthus cultivars in China[M]. Hangzhou: Zhejiang Science & Technology Press: 2, 8, 80-81, 86. [in Chinese]) |
杨康民. 2013. 中国桂花[M]. 北京: 中国林业出版社: 11-24. (Yang K M. 2013. Chinese osmanthus[M]. Beijing: China Forestry Publishing House: 11-24. [in Chinese]) |
钟淦彬, 李维国, 吴春太, 等. 2013. 188份巴西橡胶树种质材料AFLP指纹图谱分析[J]. 热带作物学报, 34(1): 1-9. (Zhong G B, Li W G, Wu C T, et al. 2013. AFLP analysis of 188 germplasm material for rubber tree(Hevea brasiliensis)[J]. Chinese Journal of Tropical Crops, 34(1): 1-9. [in Chinese]) |
Akbari M, Wenzl P, Caig V, et al. 2006. Diversity arrays technology(DArT) for high-throughput profiling of the hexaploid wheat genome[J]. Theoretical and Applied Genetics, 113(8): 1409-1420. DOI:10.1007/s00122-006-0365-4 |
Akond M, Jin S, Wang X. 2012. Molecular characterization of selected wild species and miniature roses based on SSR markers[J]. Scientia Horticulturae, 147: 89-97. DOI:10.1016/j.scienta.2012.08.028 |
Belaj A, Satovic Z, Cipriani G, et al. 2003. Comparative study of the discriminating capacity of RAPD, AFLP and SSR markers and of their effectiveness in establishing genetic relationships in olive[J]. Theoretical and Applied Genetics, 107(4): 736-744. DOI:10.1007/s00122-003-1301-5 |
Bhatia R, Singh K P, Jhang T, et al. 2009. Assessment of clonal fidelity of micropropagated gerbera plants by ISSR markers[J]. Scientia Horticulturae, 119: 208-211. DOI:10.1016/j.scienta.2008.07.024 |
Breiman L. 2001a. Random forests[J]. Machine Learning, 45(1): 5-32. DOI:10.1023/A:1010933404324 |
Breiman L. 2001b. Statistical modeling:the two cultures[J]. Statistical Science, 16(3): 199-215. |
Breiman L. 2002. Manual on setting up, using, and understanding Random Forests V3. 1.https://www.stat.berkeley.edu/~breiman/Using_random_forests_V3.1.pdf.
|
Cutler D R, Edwards T C, Beard K H, et al. 2007. Random forests for classification in ecology[J]. Ecology, 88(1): 2783-2792. |
Faleiro F G, Cordeiro M C R, Pinto A, et al. 2010. Fingerprinting analysis of Mango(Mangifera indica L.) cultivars introduced in Brazil using RAPD markers[J]. Acta Horticulturae, 864: 1127-1132. |
Ferrari M, Gori M, Monnanni R, et al. 2005. DNA fingerprinting of Corylus avellana L[J]. accessions revealed by AFLP molecular markers.Acta Horticulturae, 686: 125-134. |
Han Y H, Kang Y, Torres-Jerez I, et al. 2011. Genome-wide SNP discovery in tetraploid alfalfa using 454 sequencing and high resolution melting analysis[J]. BMC Genomics, 12(1): 350. DOI:10.1186/1471-2164-12-350 |
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 103(2): 455-461. |
Murdoch D. 2017. rgl Overview. https://cran.r-project.org/web/packages/rgl/vignettes/rgl.html.
|
Reddy M P, Sarla N, Siddiq E A. 2002. Inter simple sequence repeat(ISSR) polymorphism and its application in plant breeding[J]. Euphytica, 128(1): 9-17. DOI:10.1023/A:1020691618797 |
Sharma S K, Knox M R, Ellis T H. 1996. AFLP analysis of the diversity and phylogeny of Lens and its comparison with RAPD analysis[J]. Theoretical and Applied Genetics, 93(5): 751-758. |
Templ M, Alfons A, Filzmoser P. 2012. Exploring incomplete data using visualization techniques[J]. Advances in Data Analysis and Classification, 6(1): 29-47. DOI:10.1007/s11634-011-0102-y |
Tessier C, David J, This P, et al. 1999. Optimization of the choice of molecular markers for varietal identification in Vitis vinifera L[J]. Theoretical and Applied Genetics, 98(1): 171-177. DOI:10.1007/s001220051054 |
Vos P, Hogers R, Bleeker M, et al. 1995. AFLP:a new technique for DNA fingerprinting[J]. Nucleic Acids Research, 23(21): 4407-4414. DOI:10.1093/nar/23.21.4407 |
 2018, Vol. 54
2018, Vol. 54

