文章信息
- 闫文凯, 林文武, 杨文婷, 杜雅馨, 吴祖建, 杨靓
- Yan Wenkai, Lin Wenwu, Yang Wenting, Du Yaxin, Wu Zujian, Yang Liang
- 竹花叶病毒福州分离物基因组序列分析及其侵染性克隆的构建
- Complete Genome Sequence Analysis and Infectious Clone Construction of Bamboo Mosaic Virus Isolated from Fuzhou
- 林业科学, 2017, 53(8): 35-42.
- Scientia Silvae Sinicae, 2017, 53(8): 35-42.
- DOI: 10.11707/j.1001-7488.20170805
-
文章历史
- 收稿日期:2016-06-12
- 修回日期:2016-07-30
-
作者相关文章
竹亚科(Bambusoideae)属单子叶禾本科(Gramineae)植物,全世界有70多属1 200多种,主要分布于低纬度的热带或亚热带季风气候区。我国是其世界分布中心之一,是世界上竹类资源最丰富、竹林面积最大、产量最高的国家,全国竹类植物有500多种。然而在竹类栽培过程中,竹子花叶病毒(bamboo mosaic virus, BaMV)的发生和流行威胁着竹类的生存且可能带来严重的经济损失(Lin et al., 2016)。BaMV可使叶片花叶、竹笋和竹杆内部呈褐色条纹病变,造成竹笋木质化、发笋率降低、竹杆节间变短,进而影响竹材的经济价值(Lin et al., 1979;Yeh et al. , 1992)。BaMV最早在巴西泰山竹(Bambusa vulgaris)和孝顺竹(Bambusa multiples)上被发现,后来相继在中国台湾、美国加利福尼亚和佛罗里达以及中国福建等地的不同竹种中被发现,分布范围以及寄主相当广泛(Elliott et al., 1996; Lin et al., 2016)。
在电子显微镜下BaMV为长490~510 nm的弯曲线形(Lin et al., 1977),其基因组为正单链RNA分子(Lin et al., 1992),全长6 366 bp左右,不计poly(A),5′端带有帽子结构,3′端带有poly(A)尾巴,基因组结构包含5个开放阅读框(open reading frame, ORF)。ORF1可编码1个分子质量约为155 KDa的复制酶(replicase)。ORF2、3和4组成重叠三基因功能区(triple gene block, TGB),其基因产物分别为TGBp1(28 KDa)、TGBp2(13 KDa)、TGBp3(6 KDa),与病毒在寄主细胞间的运动有关,故又称运动蛋白(Lin et al., 2006; Wung et al., 1999;Ju et al., 2007)。ORF5主要编码25 KDa的外壳蛋白(coat protein,CP),起包被核酸的作用(Lin et al., 1992;1994)。BaMV作为Potexvirus模式研究对象之一,其基因组结构和功能、与寄主植物互作途径和机制以及作为表达载体的应用上都有了较为深入研究;然而,由于目前报道的BaMV全长序列较少,该病毒的遗传多样性研究至今还没有较大进展。
以病毒侵染性克隆为基础的反向遗传学技术体系是现代分子病毒学研究中最基本、最重要的技术手段。对于BaMV侵染性克隆研究方面,Lin等(2004)构建了BaMV侵染性克隆;Yang等(2007)将BaMV发展成为表达猪口蹄疫病毒抗原表位的疫苗载体;Liou等(2014)成功将BaMV以及satBaMV构建成为病毒诱导的基因沉默(virus induced gene silencing, VIGS)载体用于本氏烟(Nicotiana benthamiana)的基因功能验证。然而,这些侵染性克隆的构建方法过于繁琐,且易受到酶切位点的限制。无缝克隆(scarless assembly)也称为吉布森重组(Gibson assembly)的构建方法可以克服这些缺点,快速高效地将片段连接在载体上,目前还未见运用该方法构建BaMV侵染性克隆的研究报道。
本研究对麻竹(Dendrocalamus latiflorus)中的BaMV-TMS1以及satBaMV-TMS1分离物进行全基因组序列测定,并将BaMV-TMS1构建成侵染性克隆,为进一步研究BaMV和satBaMV的遗传多样性、基因功能、致病机制以及和寄主植物互作的途径和机制提供基础。
1 材料与方法 1.1 试验材料福州分离物BaMV-TMS1和satBaMV-TMS1于2013年11月采自福州永泰天门山景区,呈典型花叶症状的麻竹上,前期经电镜观察及RT-PCR检测等确定分别为BaMV和satBaMV分离物,新鲜样品置于密封袋中,保存于-80 ℃超低温冰箱。侵染性克隆载体pCAMBIATuMV:6K2-GFP、对照载体pCAMGFP、农杆菌(Agrobacterium)菌株GV3101由本实验室保存。侵染性克隆载体pKB(Liou et al., 2014) 由台湾中兴大学生物科技学研究所徐尧辉老师惠赠。
1.2 主要试剂RNeasy Plant Mini Kit购自Qiagen公司;GoScriptTMReverse Transcription System购自Promega公司;TransStart KD Plus DNA Polymerase、EasyPure Quick Gel Extraction Kit、EasyPure Plasmid MiniPrep Kit、pEASY-Blunt Zero Cloning Kit、DNA Marker、Trans1-T1感受态细胞均购自北京全式金公司(TransGen);ClonExpressTM MultiS One Step Cloning Kit购自南京诺唯赞生物科技有限公司(Vazyme)。
1.3 试验方法 1.3.1 RNA提取及基因克隆根据RNeasy Plant Mini Kit提供的使用说明从感病的麻竹心叶中提取总RNA。由于BaMV和satBaMV在3’UTR末尾都带有polyA,因此反转录过程中直接选用Oligo(dT)作为cDNA第一链合成的引物。本反应体系参照Promega公司GoScriptTM Reverse Transcription System说明进行,获得BaMV和satBaMV全长的cDNA。根据GenBank已报道的BaMV和satBaMV的全长序列分别设计2对和1对扩增引物(表 1),由北京六合华大基因科技有限公司合成。
|
|
PCR扩增采用25 μL反应体系:5×KD Plus buffer 5 μL,dNTPs(2.5mM)2 μL,正向引物和反向引物(10 μmol ·L-1)各0.5 μL,ddH2O 15.5 μL,KD Plus DNA Polymerase 0.5 μL,cDNA 1 μL。PCR反应条件为:94 ℃预变性3 min;94 ℃变性30 s,复性30 s(退火温度视基因片段而定),68 ℃延伸若干分钟(延伸时间按1 min ·kb-1计),共34个循环;最后一轮循环后68 ℃延伸10 min。PCR反应结束后,取产物5 μL用1%琼脂糖凝胶电泳进行检测。PCR产物经胶回收试剂盒纯化后与pEASY-Blunt Zero克隆载体连接,并转化到Trans1-T1感受态细胞中。经菌落PCR鉴定筛选得到的阳性克隆子,每个片段各挑取5个委托南京金斯瑞生物技术有限公司测序,并通过测序峰图及序列比对分析。
1.3.2 序列比对及组装测序获得的多个克隆子序列使用DNAMAN 7.0软件进行处理和拼接。序列一致性使用BLAST数据库(http://blast.ncbi.nlm.nih.gov/)在线比对。
1.3.3 系统发育分析为分析BaMV以及satBaMV的系统发育关系,从GenBank中下载BaMV和satBaMV其他分离物的核苷酸序列作为参考,使用最大似然法(maximum likelihood,ML)基于BaMV和satBaMV全基因组的核苷酸序列构建系统发育树。建树前,使用MEGA5(Tamura et al., 2011)的Muscle比对方法对建树序列进行多重序列比对,利用Mrmodeltest选择最优化的核苷酸替换模型,并按照AIC(akaike information criterion)标准设置相应参数,最后应用自举法(bootstrap)评估各分支节点的自举置信度(bootstrap confidence level)。
1.3.4 侵染性克隆载体构建根据侵染性克隆载体pCAMBIATuMV:6K2-GFP的序列设计特异性反向扩增引物(表 1),对载体基体进行扩增(去除TuMV的序列),可制备7 469 bp的线性化载体。由于载体片段较大,因此为减少由于PCR带来的随机突变,将PCR反应循环数降低到30左右。另外,为减少转化过程中由于模板质粒而引进的假阳性率,使用高质量的试剂盒对线性化克隆载体进行胶回收纯化,以提高DNA纯度并去除一部分未线性化的环状质粒。
为实现下一步重组反应,分别设计2对特异性引物pCAM1F/1R和pCAM2F/2R对阳性克隆菌液进行扩增(表 1)。PCR反应体系应进行适当调整:94 ℃预变性3 min;94 ℃变性30 s,55 ℃复性30 s,72 ℃延伸若干分钟,共10个循环;94 ℃变性30 s,65 ℃复性30 s,68 ℃延伸若干分钟,共24个循环;最后一轮循环后68 ℃延伸10 min。PCR反应预计分别扩增约3 527 bp和2 838 bp的目的片段。最后,根据ClonExpressTM MultiS One Step Cloning Kit的使用说明将载体和片段进行重组连接。
1.3.5 农杆菌侵染pCAMBaMV-TMS1载体通过电击转化农杆菌感受态GV3101,含有载体的农杆菌培养后,经注射悬浮液(10 mmol ·L-1 MgCl2, 10 mmol ·L-1 MES, 200 μmol ·L-1 acetosyringone)室温放置3 h后,用1 mL注射器将农杆菌注射到4周龄的本氏烟叶片,共注射3次,每次3株。注射后在温室中培养(昼27 ℃/16 h;夜22 ℃/8 h),14天后观察烟草发病情况。
1.3.6 RT-PCR检测BaMV的侵染对病毒侵染的本氏烟叶片提取总RNA,反转录获得cDNA第一链,然过用特异性引物CpC-F/R进行PCR扩增,PCR产物经1%琼脂糖凝胶电泳检测。
1.3.7 电镜负染观察BaMV病毒粒子参照韦石泉等(1985)和Hitchborn等(1965)的方法对感病烟草中的病毒粒子进行电镜观察。
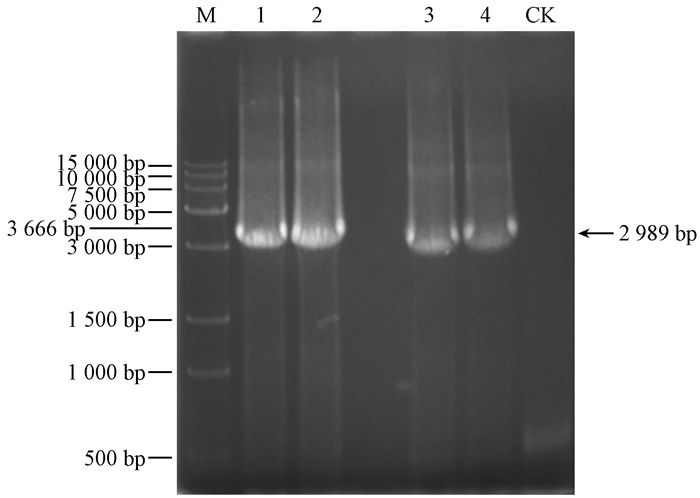
2 结果与分析 2.1 BaMV-TMS1和satBaMV-TMS1分离物全基因组的扩增与序列分析利用特异性引物,经PCR扩增获得覆盖BaMV和satBaMV基因组全长的3条特异性片段,电泳结果如图 1、2所示。克隆、测序、拼接后获得BaMV-TMS1与satBaMV-MAZBZ基因组序列。BaMV-TMS1分离物全长6 366 nt(NCBI登录号为KU936346),satBaMV-TMS1分离物全长834 nt(NCBI登录号为KU936347),都分别具有典型的BaMV和satBaMV基因组结构特征,其基因的核苷酸和氨基酸序列与其他分离物的序列一致性比较如表 2、3所示。
|
图 1 BaMV-TMS1基因组全长的扩增产物 Fig.1 The amplified products of full length gene of BaMV-TMS1 1、2和3、4分别为BaMV-TMS1基因组全长的2个片段;M.Trans15K DNA Marker; CK:阴性对照。 1, 2 and 3, 4 were the two separate amplified proolucts of full length gene of BaMV-TMS1;M:Trans15K DNA Marker; CK:negative control. |

|
图 2 satBaMV-TMS1全长的扩增产物 Fig.2 The amplified products of satBaMV-TMS1 5和6为satBaMV-TMS1全长的扩增产物;M:D2000 plus DNA ladder; CK:阴性对照。 5 and 6 were amplified products of full length; M:D2000 plus DNA ladder; CK:negative control. |
|
|
|
|
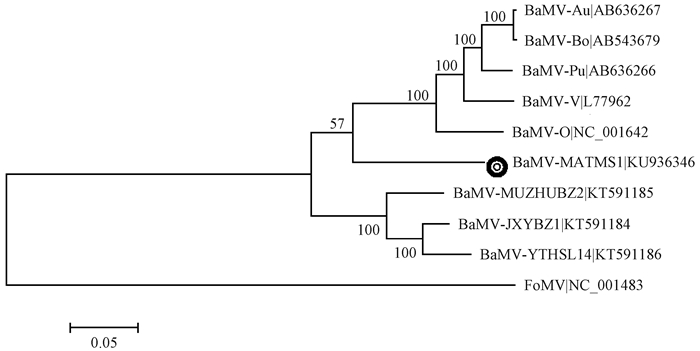
根据Mrmodeltest软件的AIC标准,建树序列最适合的核苷酸替换模型为GTR+I+G。ML法重建的系统发育树如图 3、4所示,BaMV-TMS1分离物可形成一个新的分支;同样,satBaMV-TMS1分离物也单独形成一个新的分支,且与台湾分离物具有明显的地域性。
|
图 3 BaMV-TMS1的系统发育树 Fig.3 The phylogenetic tree of BaMV-TMS1 |

|
图 4 satBaMV-TMS1的系统发育树 Fig.4 The phylogenetic tree of satBaMV-TMS1 |
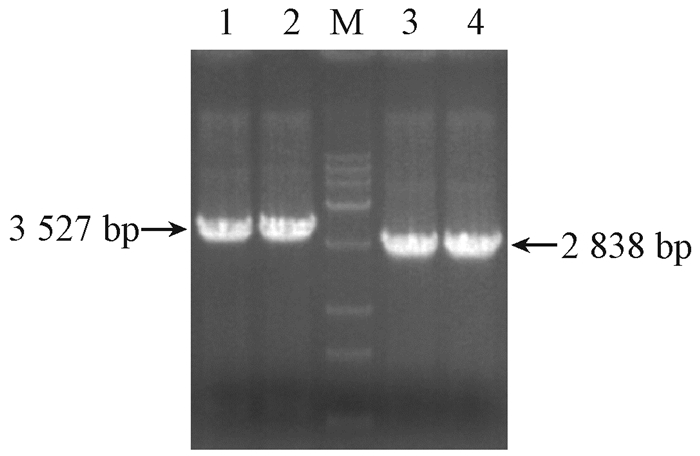
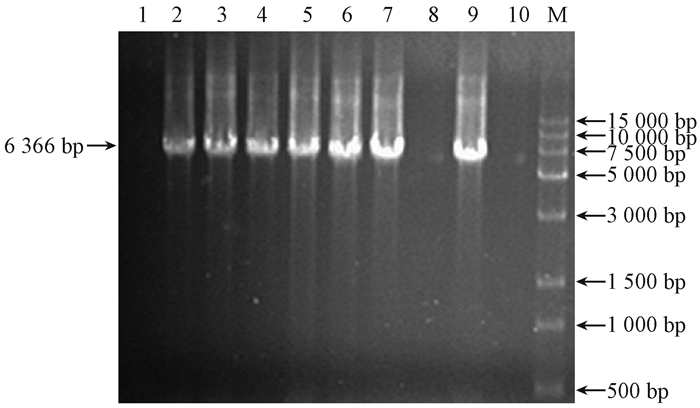
利用设计的特异性引物,分别对载体和病毒片段进行PCR扩增,所获得的片段均符合预期大小(图 5、6)。将上述PCR产物回收后,分别加入150 ng载体片段、70 ng病毒片段1和57 ng病毒片段2于重组反应体系中进行反应。经挑取单菌落检测全长片段(6 366 nt),阳性率高达70%(图 7)。

|
图 5 载体pCAMBIA的扩增 Fig.5 The amplification of vector pCAMBIA 1:阴性对照; 2~11:载体扩增; M:Trans15K DNA Marker。 1: Negative control; 2-11: The amplification of vector; M:Trans15K DNA Marker. |
|
图 6 BaMV基因的扩增 Fig.6 The amplification of BaMV gene 1和2、3和4分别为2个基因片段; M:Trans15K DNA Marker。 1 and 2、3 and 4: The two separate amplified target fragments; M:Trans15K DNA Marker. |
|
图 7 BaMV-TMS1基因全长的检测 Fig.7 The detection of full genome sequences of BaMV-TMS1 1~10:单菌落;M:Trans15K DNA Marker。 1-10: Single colony; M:Trans15K DNA Marker. |
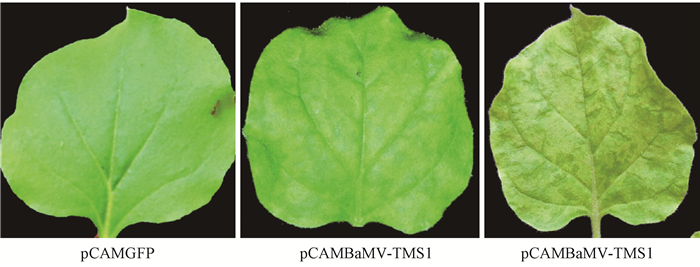
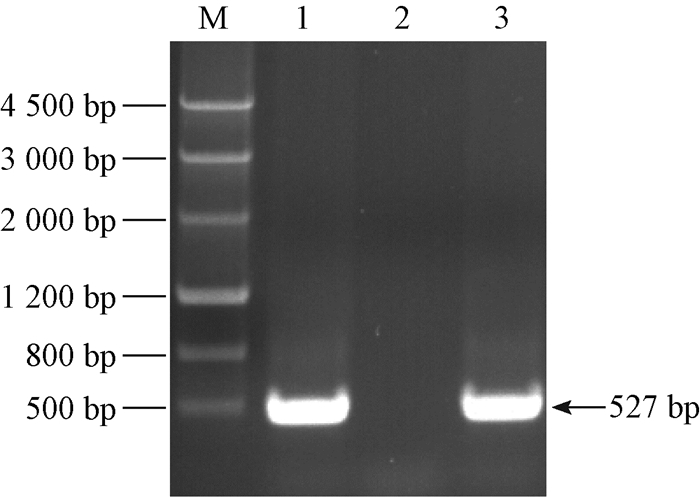
将构建的pCAMBaMV-TMS1侵染性克隆导入农杆菌GV3101后,注射4周龄的本氏烟;同时,分别以导入pKB和pCAMGFP载体的农杆菌侵染烟草植株作为阳性和阴性对照。结果表明:含有pKB载体的农杆菌接种本氏烟10天以后,上部系统叶片开始出现花叶症状,随后病状加重;含有载体pCAMBaMV-TMS1的农杆菌侵染本氏烟20天后出现轻微的花叶症状;而含有载体pCAMGFP的农杆菌侵染本氏烟后没有出现任何症状(图 8)。分别提取3组本氏烟的总RNA,以BaMV外壳蛋白保守区设计的特异性检测引物CpC-F/R(Lin et al., 2016)(表 1)进行RT-PCR扩增,结果表明,经含有侵染性克隆pCAMBaMV-TMS1和pKB的农杆菌侵染本氏烟后,PCR扩增都得到527 bp左右的特异性条带,而阴性对照组则没有扩增到相应条带(图 9)。采用病叶榨汁登网并负染的方法,以pKB为对照,对病叶中的病毒进行电镜负染观察,结果显示,在电镜下观察到许多杆状、长度约为490~510 nm弯曲线形的病毒粒子(图 10),是BaMV病毒粒子的形态特征。
|
图 8 BamV-TMS1侵染的本氏烟症状 Fig.8 The Nicotiana benthamiana symptom of BaMV inoculation |
|
图 9 RT-PCR检测 Fig.9 The detection of RT-PCR 1:pCAMBaMV-TMS1; 2:阴性对照; 3:阳性对照; M:TIANGEN MarkerⅢ。 1:pCAMBaMV-TMS1; 2: negative control; 3:positive control; M: MarkerⅢ of TIANGEN. |

|
图 10 电镜观察病毒粒子 Fig.10 The virus particle in electron microscope |
本研究从福州永泰天门山景区采得带有重型花叶症状的麻竹样品,根据GenBank中已知的BaMV和satBaMV不同分离物序列设计特异性的扩增引物,利用RT-PCR扩增和测序成功获得BaMV-TMS1和satBaMV-TMS1分离物的全基因组序列。经NCBI BLAST比对显示,BaMV-TMS1分离物与已报道的序列一致性为82% ~83%,而satBaMV-TMS1与其他分离物的序列一致性为92% ~93%。系统进化树分析表明,BaMV-TMS1分离物并没有与已知的福州分离物聚在一起,而是与台湾分离物亲缘关系较为接近,且同时形成一个新的分支。这可能与台湾与大陆地区竹类品种往来运输有关,但该病毒具体的起源地在何处,还需要进行更多的研究。同样,satBaMV-TMS1分离物在系统发育分析中也形成一个单独的分支,该结果表明BaMV和satBaMV在自然界中受到较大的选择压力影响,序列变异度较大,存在较高的遗传多样性,但其基因组结构及其编码的功能蛋白没有受到影响。
随着RNA病毒的cDNA侵染性克隆技术的发展,可在DNA水平上对RNA病毒进行有目的性的改造,这为研究RNA病毒与寄主的相互作用机制提供了很好的手段。植物病毒侵染性克隆首先在雀麦花叶病毒中获得成功(Ahlquist et al., 1984), 至今已有多种植物病毒成功地构建了全长cDNA侵染性克隆。然而,传统的cDNA侵染性克隆的构建方法步骤相对繁琐、操作不易且成功率低,为避免由于长片段扩增而带来的随机突变,常通过短片段的扩增,经过限制性酶切位点的切割和连接将片段和载体重新连接而成。但是,这种方法往往会受到酶切位点的限制,且会引入一些非病毒序列的片段影响病毒的转录起始和终止,从而导致丧失病毒侵染性(Chiang et al., 1997)。无缝克隆技术是由Gibson等(2009)首次提出的,可实现多个DNA片段同时无缝接合到1个载体上。目前,该技术已成功运用于多种RNA病毒的基因组重构和侵染性克隆构建,如流感病毒A(influenza A virus)(Zhou et al., 2009)、登革热病毒(dengue virus)(Siridechadilok et al., 2013)、西尼罗病毒(West Nile virus)(Vandergaast et al., 2014)、猪繁殖和呼吸综合征病毒(porcine reproductive and respiratory syndrome virus)(Suhardiman et al., 2015)、莴苣花叶病毒(Lettuce mosaic virus)(Bordat et al., 2015)等。目前还没有报道使用该方法对BaMV进行侵染性克隆的构建。本研究中,根据已知的病毒和载体序列,分别设计引物进行PCR扩增;回收产物经纯化后按照一定比例加入连接体系,PCR检测阳性率高达70%。该方法采用农杆菌瞬时表达法转录病毒基因组侵染植物,无需体外转录,操作方便快速,不需要昂贵的试剂,并且接种效率高达100%,适合大规模的遗传操作。因此,BaMV侵染性克隆快速构建方法的成功建立,将为下一步研究不同BaMV分离物的侵染性、发展运用于验证竹类植物内源基因功能的VIGS载体以及研究抗病毒策略等科学问题提供有力的技术手段。
4 结论通过分析大陆和台湾BaMV及satBaMV分离物的全基因组序列,发现BaMV及satBaMV具有较高的遗传多样性,系统进化树分析也得到相似的结果。另外,本试验成功快速构建了大陆BaMV-TMS1分离物的侵染性克隆,接种本氏烟后有明显的花叶症状,并通过RT-PCR检测和电镜负染的方法验证了其侵染性。
| [] |
韦石泉, 徐慧民. 1985. 应用负染色技术电镜观察快速检测植物病毒粒子的研究. 微生物学杂志, 5(4): 7–10.
( Wei S Q, Xu H M. 1985. Study of rapid detection of plant virus particles by applying of negative staining electron microscope. Journal of Microbiology, 5(4): 7–10. [in Chinese] ) |
| [] | Ahlquist P, French R, Janda M, et al. 1984. Multicomponent RNA plant virus infection derived from cloned viral cDNA. Proceedings of the National Academy of Sciences, 81(22): 7066–7070. DOI:10.1073/pnas.81.22.7066 |
| [] | Bordat A, Houvenaghel M C, German-Retana S. 2015. Gibson assembly:an easy way to clone potyviral full-length infectious cDNA clones expressing an ectopic VPg. Virology Journal, 12(1): 89. DOI:10.1186/s12985-015-0315-3 |
| [] | Chiang C H, Yeh S D. 1997. Infectivity assays of in vitro and in vivo transcripts of papaya ringspot potyvirus. Botanical Bulletin of Academia Sinica, 38: 153–163. |
| [] | Elliott M S, Zettler F W. 1996. Bamboo mosaic virus detected in ornamental bamboo species in Florida. Proc Fla State Hortic Soc, Florida:Florida State Horticultural Society. |
| [] | Gibson D G, Young L, Chuang R Y, et al. 2009. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 6(5): 343–345. DOI:10.1038/nmeth.1318 |
| [] | Hitchborn J H, Hills G J. 1965. The use of negative staining in the electron microscopic examination of plant viruses in crude extracts. Virology, 27(4): 528–540. DOI:10.1016/0042-6822(65)90178-9 |
| [] | Ju H J, Brown J E, Ye C M, et al. 2007. Mutations in the central domain of potato virus X TGBp2 eliminate granular vesicles and virus cell-to-cell trafficking. Journal of Virology, 81(4): 1899–1911. DOI:10.1128/JVI.02009-06 |
| [] | Lin M K, Chang B Y, Liao J T, et al. 2004. Arg-16 and Arg-21 in the N-terminal region of the triple-gene-block protein 1 of bamboo mosaic virus are essential for virus movement. Journal of General Virology, 85(1): 251–259. DOI:10.1099/vir.0.19442-0 |
| [] | Lin M K, Hu C C, Lin N S, et al. 2006. Movement of potexviruses requires species-specific interactions among the cognate triple gene block proteins, as revealed by a trans-complementation assay based on the bamboo mosaic virus satellite RNA-mediated expression system. Journal of General Virology, 87(5): 1357–1367. DOI:10.1099/vir.0.81625-0 |
| [] | Lin M T, Kitajima E W, Cupertino F P, et al. 1977. Partial purification and some properties of bamboo mosaic virus. Phytopathology, 67(12): 1439–1443. |
| [] | Lin N S, Chen M C, Chiang T, et al. 1979. Preliminary studies on bamboo mosaic disease in Taiwan. Taiwan, Science and Education Press. |
| [] | Lin N S, Lin B Y, Lo N W, et al. 1994. Nucleotide sequence of the genomic RNA of bamboo mosaic potexvirus. Journal of General Virology, 75(9): 2513–2518. DOI:10.1099/0022-1317-75-9-2513 |
| [] | Lin N S, Lin F Z, Huang T Y, et al. 1992. Genome properties of bamboo mosaic virus. Phytopathology, 82(7): 731–734. DOI:10.1094/Phyto-82-731 |
| [] | Lin W W, Gao F L, Yang W T, et al. 2016. Molecular characterization and detection of a recombinant isolate of bamboo mosaic virus from China. Archives of Virology, 161(4): 1091–1094. DOI:10.1007/s00705-016-2773-3 |
| [] | Liou M R, Huang Y W, Hu C C, et al. 2014. A dual gene-silencing vector system for monocot and dicot plants. Plant biotechnology Journal, 12(3): 330–343. DOI:10.1111/pbi.2014.12.issue-3 |
| [] | Siridechadilok B, Gomutsukhavadee M, Sawaengpol T, et al. 2013. A simplified positive-sense-RNA virus construction approach that enhances analysis throughput. Journal of Virology, 87(23): 12667–12674. DOI:10.1128/JVI.02261-13 |
| [] | Suhardiman M, Kramyu J, Narkpuk J, et al. 2015. Generation of porcine reproductive and respiratory syndrome virus by in vitro assembly of viral genomic cDNA fragments. Virus Research, 195: 1–8. DOI:10.1016/j.virusres.2014.09.008 |
| [] | Tamura K, Peterson D, Peterson N, et al. 2011. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 28(10): 2731–2739. DOI:10.1093/molbev/msr121 |
| [] | Vandergaast R, Hoover L I, Zheng K, et al. 2014. Generation of west nile virus infectious clones containing amino acid insertions between capsid and capsid anchor. Viruses, 6(4): 1637–1653. DOI:10.3390/v6041637 |
| [] | Wung C H, Hsu Y H, Liou D Y, et al. 1999. Identification of the RNA-binding sites of the triple gene block protein 1 of bamboo mosaic potexvirus. Journal of General Virology, 80(5): 1119–1126. DOI:10.1099/0022-1317-80-5-1119 |
| [] | Yang C D, Liao J T, Lai C Y, et al. 2007. Induction of protective immunity in swine by recombinant bamboo mosaic virus expressing foot-and-mouth disease virus epitopes. BMC Biotechnology, 7(1): 62. DOI:10.1186/1472-6750-7-62 |
| [] | Yeh C C, Cheng A H, Hwang H Y. 1992. Indexing of bamboo mosaic virus and extension of virus-free clone/plant. Research Journal of Agricultural Improvement Field, Tainan District, 29: 82–88. |
| [] | Zhou B, Donnelly M E, Scholes D T, et al. 2009. Single-reaction genomic amplification accelerates sequencing and vaccine production for classical and Swine origin human influenza a viruses. Journal of Virology, 83(19): 10309–10313. DOI:10.1128/JVI.01109-09 |
 2017, Vol. 53
2017, Vol. 53

