文章信息
- 杨汉波, 张蕊, 王帮顺, 徐肇友, 陈焕伟, 周志春
- Yang Hanbo, Zhang Rui, Wang Bangshun, Xu Zhaoyou, Chen Huanwei, Zhou Zhichun
- 木荷优树无性系种质SSR标记的遗传多样性分析
- Analysis of Genetic Diversity in Schima superba Plus Tree Germplasms by SSR Markers
- 林业科学, 2017, 53(5): 43-53.
- Scientia Silvae Sinicae, 2017, 53(5): 43-53.
- DOI: 10.11707/j.1001-7488.20170506
-
文章历史
- 收稿日期:2016-04-25
- 修回日期:2016-06-15
-
作者相关文章
2. 浙江省龙泉市林业科学研究院 龙泉 323700
2. Longquan Forestry Research Institute, Zhejiang Province Longquan 323700
种质资源的收集是林木育种的关键环节。截止2013年底,我国已收集保存各类林木种质资源16万份。多样性的评价和遗传背景的研究是种质资源利用的前提(Chuanfu et al., 2008)。许多学者借助表型性状变异开展植物种质资源遗传多样性研究(王永康等,2014;Zeka et al., 2015)。然而,由于某些性状受环境因素和生长期的影响,基于形态和生长性状对种质资源遗传多样性的研究具有一定的局限性,不能准确反映种质资源个体间的遗传差异和亲缘关系(Terzopoulos et al., 2008)。自20世纪90年代以来,具有共显性、高分辨率和重复性好等特点的SSR(Simple Sequence Repeat)标记技术已广泛应用于作物及多年生木本植物的遗传多样性和遗传结构分析中,并取得了一系列的成果(Ferrão et al., 2015;赵爽等,2016;Lassois et al., 2016)。另外,在种质亲缘关系研究中,主坐标分析(PCoA)与STRUCTURE分析方法的结合已得到许多应用。对于遗传标记结果,主坐标分析(PCoA)通过相似性进行个体区分,可以清晰地显示个体间的相互关系;STRUCTURE分析采用混合模型,可以对所有个体进行划分,反映群体的遗传结构本质,还可以根据个体等位基因的组分数,推断群体中具有复杂遗传背景的个体或者发生迁移的个体,对群体中的个体进行归属判断(Sun et al., 2003;宗绪晓等,2010)。如在林木亲缘关系研究中,采用主坐标(PCoA)与STRUCTURE 2种分析方法相结合处理鹅掌楸(Liriodendron chinense)(杨爱红等,2014)、红椿(Toona ciliata)(李培等,2016)等分子标记数据,得到了更为准确、丰富的遗传多样性与遗传结构分析结果。
木荷(Schima superba)为山茶科(Theaceae)木荷属(Schima)常绿大乔木,自然分布于31°N以南105°E以东的广大地区,是该区域常绿阔叶林的主要建群种。木荷早期速生,材性优良,抗逆性强,亦是我国南方各省区的珍贵优质阔叶用材和高效生物防火树种,在商品用材林和生态防火林建设中占有重要地位(张蕊等,2013;楚秀丽等,2014),以珍贵优质用材和高效生物防火为目标的木荷育种研究已成为林木育种学家研究的重点。作者所在研究组自2001年开展木荷育种工作以来,经过不断搜集、补充,目前已保存国内5个省份24个地区的木荷优树无性系种质734份,并借助表型标记开展了木荷地理种源遗传变异研究(张萍等,2004;周志春等,2006;王秀花等,2011)。本研究在辛娜娜等(2015)对部分木荷育种亲本进行遗传多样性分析的基础上,利用SSR标记进一步对搜集保存的全部木荷优树无性系种质进行遗传多样性分析,拟通过大规模样本间的遗传多样性及遗传结构的详细系统比较和分析,全面揭示我国木荷优树无性系种质遗传多样性地理分布特点和种质群体(个体)间的遗传关系,为我国木荷优树无性系种质的深入研究和开发利用提供科学依据,同时也为我国木荷的长期多目标育种奠定基础。
1 材料与方法 1.1 试验材料研究材料来源于浙江省龙泉市林业科学研究院上圩基地木荷优树无性系种质基因库(28°03′N, 119°06′E)。基地面积为6.7 hm2,海拔200~300 m,相对湿度79%,年均降雨量1 664.8~1 706.2 mm。2010年至今,在浙江、江西、福建等地选择优树1 000余株,嫁接保存734份。选优林分要求林龄20年、面积1.0 hm2以上,以木荷为主的优良天然林或起源明确的人工林;优树选择条件为树型高大,干形通直圆满,生长量明显高于附近3~5株同龄优势木等。以本地1~2年生木荷容器苗为砧木,在3—4月份选用带有休眠芽的穗条,采用切接的方法嫁接优树无性系,在苗圃地集中培育2年生大容器嫁接苗,然后根据木荷优树无性系种质基因库配置图(株行距为4 m×4 m)将嫁接苗移栽定植至基因库相应的位置上。将所有木荷优树无性系种质按照产地来源分为24个种质群体(表 1),2015年6月,对全部734份木荷优树无性系种质采集其顶端新发枝条上的新鲜嫩叶,将其放入液氮中带回实验室,置于-80 ℃冰箱中保存备用。
|
|
采用试剂盒提取木荷基因组DNA,1.0%琼脂糖凝胶电泳检测DNA纯度和完整性,NanoDrop-2000超微量分光光度计(Thermo,美国)检测其浓度,最后稀释成20 ng·μL-1,-20 ℃保存备用。本研究选用13对条带清晰、多态性强的SSR引物进行PCR扩增(辛娜娜等,2015)。PCR扩增反应在TaKaRa PCR Thermal Cycler Dice Touch上进行。PCR扩增反应体系25 μL:含12.5 μL 2×Taq Plus Master Mix, 10 μmol·L-1上下游引物和50 ng基因组DNA。PCR扩增程序为:95 ℃预变性5 min;94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸45 s(35个循环);72 ℃延伸10 min (辛娜娜等,2015)。扩增产物在Qsep100TM全自动核酸蛋白自动分析系统上进行电泳分离检测和片段大小的测定,Qsep100TM能以1~4 bp的分辨率高效区分20~20 000 bp DNA片段(http://www.sciencemag.org/content/348/6241/1383.1.full)。
1.3 遗传参数分析对SSR检测结果进行峰图分析及等位基因的读取,然后采用Cervus 3.0.7软件(Kalinowski et al., 2007)计算多态信息含量(PIC),并检测各SSR位点的无效等位基因(null alleles)频率,去除无效等位基因频率超过0.2的引物组合,以确保后续遗传多样性分析结果的准确性(文亚峰等,2013)。利用GenAlEx6.502软件(Peakall et al., 2012)对结果进行遗传多样性分析。分别计算:1) 等位基因数(Na);2) 有效等位基因数(Ne);3) Shannon’s信息多样性指数(I);4) 观测杂合度(Ho)与期望杂合度(He);5) 群体间Nei’s遗传相似系数和遗传距离,并利用NTSYS pc 2.1软件(Rohlf, 2000)绘制基于UPGMA法的树状聚类图。利用GenAlEx6.502软件,计算各木荷优树无性系种质个体间的遗传距离,并进行主坐标(PCoA)分析。用Arlequin软件对群体间和种群内分子遗传变异进行AMOVA分子方差分析(Excoffier et al., 2010)。
1.4 群体遗传结构和模拟聚类分析利用Genepop 4.5软件(Rousset, 2008),采用马尔科夫链方法(Markov chain method,MC)对SSR位点之间的关联性进行显著性检验,得到无偏估计P值,当P < 0.05表明连锁不平衡具有显著性。采用软件STRUCTURE 2.3(Pritchard et al., 2000)进行木荷优树无性系种质遗传结构分析。这一软件分析目的在于找到个体合适的分组数,K值。首先确定K值取值范围为1~20,然后运行软件,对每个K值进行个体分组分析,每个K值重复运行100次,参数iterations和burn-in period均设为10 000。再根据Evanno等(2005)提供的ΔK方法确定合适的分组K值。
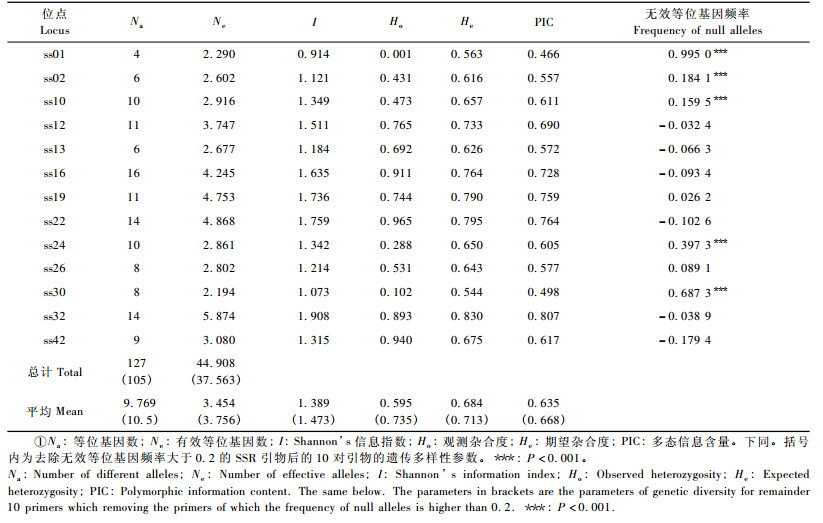
2 结果与分析 2.1 SSR位点多态性分析13对SSR引物中,有7对引物的无效等位基因(null alleles)频率为正值,其中ss01、ss24和ss30的无效等位基因频率均大于0.2,并且显著偏离哈迪-温伯格平衡(表 2),因此,在后续遗传多样性分析中去除这3个位点以确保结果的准确性。筛选出的10对引物组合能准确获得不同材料在不同位点的等位基因片段大小及相应的电泳峰图(图 1)。10对SSR引物在734份木荷优树无性系种质中扩增出105个等位基因(Na),平均每对引物扩增出10.5个等位基因,有效等位基因数(Ne)为3.756(表 2)。10对木荷SSR引物间存在较大的差异,其中,等位基因数(Na)最多的引物是ss16,为16个,其有效等位基因数(Ne)为4.245;其次是ss32、ss22,Na均为14个,Ne分别为5.874个和4.868个;Na最少的是引物ss02和ss13,均为6个,Ne分别为2.602个和2.677个。10对SSR引物的多态信息含量(PIC)范围为0.557~0.807,平均值为0.668,表明所选的SSR引物在木荷优树无性系种质上多态性丰富。期望杂合度(He)和观测杂合度(Ho)的变化范围分别为0.616(ss02)~0.830(ss32) 和0.431(ss02)~0.965(ss22),平均分别为0.713和0.735。Shannon’s信息多样性指数(I)变化范围为1.121~1.908,平均为1.473,这表明木荷优树无性系种质具有较丰富的遗传多样性。
|
|

|
图 1 引物ss12,ss16,ss22和ss24在部分材料中的电泳图谱 Fig.1 The part of electrophoretogram of primer ss12, ss16, ss22 and ss24 |
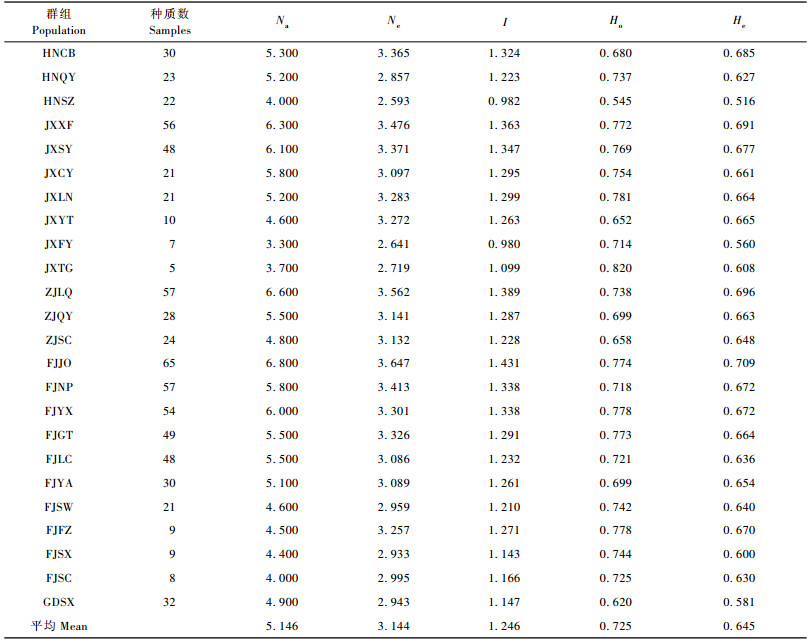
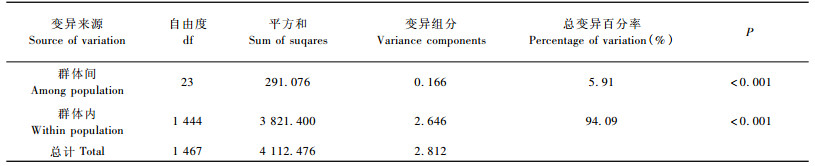
24个种质群体的有效等位基因数(Ne)变化范围较大,在2.593(HNSZ)~3.647(FJJO)之间(表 3)。各参试群体的Shannon’s信息指数(I)变化在0.980(JXFY)~1.431(FJJO)之间,平均为1.246。观测杂合度(Ho)最小的是HNSZ(0.545),最大的是JXTG(0.820),JXLN次之(0.781),平均为0.725。期望杂合度(He)最小的是HNSZ(0.516),最大的是FJJO(0.709),平均为0.645。部分群体的有效等位基因数(Ne)和Shannon’s信息指数(I)的变化趋势不一致,如FJNP的Ne高于FJYX, 而二者的I值均为1.338,HNCB的Ne高于FJYX,但I却低于FJYX。同样,部分群体的观察杂合度(Ho)和期望杂合度(He)也表现出这种不一致的变化趋势。AMOVA分析结果(表 4)显示,木荷优树无性系种质群体间的遗传变异为5.91%,而群体内遗传变异显著,为94.09%。
|
|
|
|
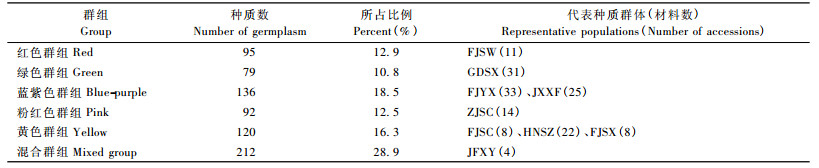
连锁不平衡(LD)检测结果显示各SSR位点间不存在连锁,处于完全独立状态(表 5)。STRUCTURE分析中,ΔK在K=5时有明显的峰(图 2),因此将734份木荷优树无性系种质分为5个群组,并绘制出遗传结构图(图 3)。分析各木荷优树无性系种质在不同群组的Q值,发现522份种质(71.1%)在某一群组中的Q值大于0.5,推测其遗传组分相对比较单一,被划分到5个群组中的1个,而剩余的212份(28.9%)在5个群组中Q值均小于0.5,没有明确的群组归属特性,形成了1个混合群(mixed group)(表 6)。GDSX中的96.9%(31份)归属于1个群组(绿色群组),Q平均值为0.88,仅1份种质划分到混合群,表明该群体遗传组分相对单一。来自FJSC(8份,100%)、FJSX(8份,88.9%)和HNSZ(22份,100%)的大部分种质被归为1个群组(黄色群组),表明它们的遗传结构相似。参试木荷优树无性系种质遗传结构与地理分布不完全相关,如JXSY、FJNP和JXXF等群体的种质材料在5个群组中均有分布。
|
|

|
图 2 待定群体数K与估计值ΔK的关系 Fig.2 Relations between the number of determined group K and estimated value ΔK |

|
图 3 参试木荷优树无性系种质遗传结构分组 Fig.3 Estimated population STRUCTURE for Schima superba resources populations 图中5种颜色表示5个不同的群组,每条彩色竖线代表一份种质,不同颜色所占比例越大,则该种质被划分到相应群组的可能性就越大。 Each germplasm is represented by a single color line; there are five population groups; the more proportion of the color, the more possibility of the represented germplasm by the color divided into the corresponding population. |
|
|
主坐标(PCoA)分析结果显示,734份木荷优树无性系种质可分为A、B和C 3个类群(图 4)。A类群所包含的种质数量最多,占全部种质的41%,B、C 2个类群所包含的种质数量差异不大,分别占材料总数的29.7%和29.3%。5个省(市)的种质在3个类群中均有分布,3个类群间有少量相互渗透的现象。A类群的典型代表是HNSZ(20份)、ZJSC(14份)和ZJQY(16份)等,B类群的典型代表是FJSW(13份)、HNCB(16份)和FJFZ(4份)等,C类群的典型代表有FJYX(37份)、JXFY(4份)和GDSX(27份)等。总体上看,福建、浙江和江西的资源在大范围内呈分散分布,说明其遗传背景宽广。基于两两个体间遗传距离的主坐标(PCoA)分析与遗传结构分析聚类结果基本可以保持一致:A类群与粉红色和黄色群组对应,B类群与红色群组和蓝紫色群组相对应,C类群与绿色群组对应;群体结构分析中划分到混合群体的材料在PCoA分析中也被划分到与Q值最大的类群所对应的区域,或与该区域较近的位置。
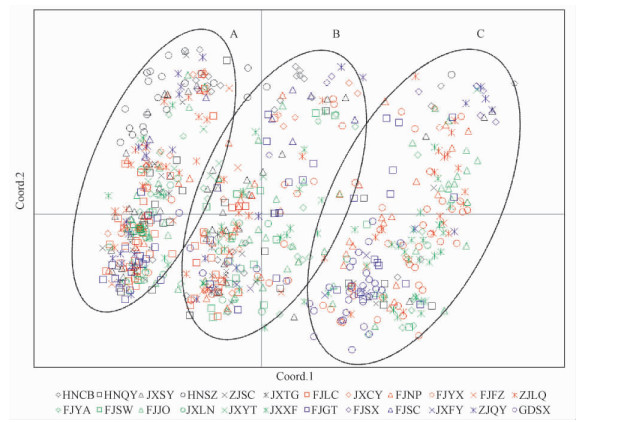
|
图 4 基于10对SSR引物的734份木荷种质二维PCoA分析 Fig.4 Two-dimension principal coordinate analysis of the 734 Schima superba accessions based on 10 SSR primer pairs |
为进一步探明种质遗传多样性与地理来源的相关性,对24个种质群体绘制基于UPGMA法的树状聚类图(图 5)。24个木荷种质群体间遗传距离差异很大,介于0.030~0.804之间,平均为0.230。从聚类图看出,GDSX、HNSZ和FJSX 3个群体与其他群体间遗传距离均较大,分别单独聚为一组,其中HNSZ与GDSX间遗传距离最大,为0.804。HNCB和FJSC聚为一组,它们之间的遗传距离为0.149。其余19个种质群体全部聚为一组,遗传距离介于0.030~0.323,其中FJYX与JXXF间的遗传距离最小,FJSW与JXFY间的遗传距离最大。
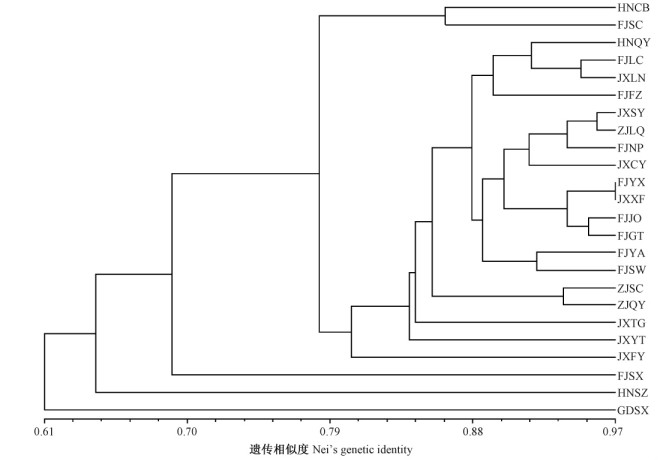
|
图 5 基于遗传距离的木荷种质群体聚类 Fig.5 Phylogenetic dendrogram based on the genetic distance in the Schima superba germplasm populations |
无效等位基因(null alleles)会对遗传学相关研究结果造成显著影响,如降低群体遗传多样性,加大群体间遗传分化和遗传距离等,是SSR标记最大的缺陷之一(Paetkau et al., 1997;文亚峰等,2013)。本研究中,3对高无效等位基因频率引物的存在,明显降低了观测杂合度(Ho)、期望杂合度(He)和群体总体遗传多样性(表 2),相应地,使得群体遗传分化和遗传距离增大。在进行引物能力评估时,PIC>0.5时,引物贡献率较高,多态性较好,选取的引物可以最大程度地反映遗传多样性(Wang et al., 2014)。本研究中,筛选后的10对SSR引物组合的PIC平均值为0.668,说明选择的10对SSR引物可以很好反映引物的区分能力,能准确有效地揭示木荷优树无性系种质的遗传多样性。保持林木遗传多样性是对林木进一步选择和改良的前提(李悦等,2000)。本研究结果显示,参试木荷优树无性系种质具有较高的遗传多样性,10对多态性SSR引物共检测出105个等位基因(Na),平均每对引物为10.5个。检测到等位基因(Na)最多的引物是ss16(16个等位基因),而辛娜娜等(2015)利用ss16引物仅检测到6个等位基因,Niu等(2012)也只检测到7个等位基因。本研究中的SSR引物之所以能检测到更为丰富的变异类型,可能与参试种质丰富、参试种质来源广泛、群体间的地理位置相距较远有关。Shannon’s信息指数(I)可以有效反映种质各群体的遗传多样性,也是评价遗传多样性水平的重要指标(Nybom, 2004)。本研究中木荷优树无性系种质Shannon’s信息指数(I)为1.473,高于辛娜娜等(2015)利用SSR对木荷一代育种群体遗传多样性研究结果(I=1.225)。原因可能有以下2个方面:一是研究材料来源于木荷主产区的5个省市具有广泛代表性,这本身就代表着相对较高的遗传多样性;二是与木荷本身的交配系统有关(雌雄同花、虫媒传粉且自交不亲和)。与其他阔叶树种相比较,如大花序桉(Eucalyptus cloeziana) (I=1.250)、灰叶胡杨(Populus pruinosa)(I=1.185) 等(邓紫宇,2012;张玲等,2012),本研究中木荷优树无性系种质表现出较高的遗传多样性(I=1.473),这与其本身的生物学特性有关。木荷是一种适应性很强的物种,在群落演替的各个阶段都出现,分布广泛,种群较大,使得木荷在总体上表现出很高的遗传多样性。
通过对比分析不同地理来源木荷优树无性系种质群体间的遗传多样性参数,发现FJJO、ZJLQ和JXXF的遗传多样性最为丰富,其可能原因为这些地区处于南岭山脉-武夷山脉附近,与张萍等(2006)划分的南部种源区和王秀花等(2011)划分的中心种源区位置大致相同,是木荷的中心产区,其种质不仅最为速生,而且遗传多样性也最高。JXFY的遗传多样性最低,遗传变异最小;这可能与该群体的种质数量较少有关(N=7),如JXTG (N=5) 和FJSX(N=9) 的遗传多样性水平均较低。因此,在今后的木荷种质保存和育种策略制定工作中,应重点考虑遗传多样性相对较为丰富的群体(如FJJO、ZJLQ和JXXF等种质群体),优先对这些地区的木荷种质做进一步系统研究和开发利用,同时还应加强对优树种质的搜集和保存,以拓宽遗传背景,提高育种成效。育种亲本间的遗传基础差异越大,产生的杂交后代中越容易选出性状超越亲本和适应性较强的新品种,因此,科学的育种亲本选配是杂交育种工作的关键,选配亲本重要原则之一就是选择不同生态型、不同地理来源和不同亲缘关系的材料作为杂交育种亲本(余亚莹等,2015)。本研究结果显示,JXXF与FJYX、JXSY与ZJLQ等种质群体间的遗传距离较小、亲缘关系较近,因此,在木荷杂种育种工作中应避免在这些群体间进行杂交制种;应在遗传距离大、亲缘关系远的群体间(如HNCB与FJSX、GDSX与HNSZ)进行杂交制种,以期获得更多的遗传变异和杂种优势。主坐标分析(PCoA)与STRUCTURE分析属于2种不同的运算模式,2种分析方法的结合,有助于结果的相互验证,使结果更为准确,更全面了解不同材料间的遗传关系(陈斐等,2013)。在本研究中,主坐标分析(PCoA)及STRUCTURE分析结果均显示,不同地区种质聚集交错,与地理来源并无直接关系,这与国内外相关研究结果一致(Laurentin et al., 2006; 辛娜娜等,2015)。因此,在木荷杂交育种亲本选配时不仅要考虑地理远缘,还应考虑亲本群体(个体)间的亲缘关系。同时还要根据其他参考性状,如木材质量、抗逆性等性状来考虑是否作为杂交育种的亲本材料。
分子方差分析(AMOVA)结果表明,木荷优树无性系种质的遗传变异主要来源于群体内。因此,在木荷育种工作中,需要侧重于优树个体的选择和改良,同时兼顾地理群体的选择。群体遗传分化的动力主要来源于自然选择(李康琴,2013)。木荷优树无性系种质群体间遗传分化程度低,原因可能是木荷分布范围较广,木荷优树采集地保留种群一般比较大,呈集群分布,群体间基因流较大而未发生严重的遗传分化。
近年来,基于DNA水平的分子标记在作物关联作图分析上得到广泛应用,而在多年生木本植物中应用相对较少(Zhu et al., 2015; Xu et al., 2015)。对种质遗传多样性的研究及遗传结构的分析,是关联作图的前提,当研究所用材料遗传结构简单时,关联分析的功效就会达到最大,结果出现假阳性的可能性也最低(Cardon et al., 2003;Aulchenko, 2011)。在群体遗传结构分析中,Wang等(2008)在玉米(Zea mays)研究中将Q>0.8的材料认为结构相对单一,在小麦(Triticum aestivum)研究中刘丽华等(2009)将Q>0.6视为结构单一。本文根据种质资源遗传复杂程度的不同,将Q>0.5作为判断遗传结构是否单一的标准,发现71.1%木荷优树无性系种质遗传组分相对单一,说明不同地区的木荷优树无性系种质间存在一定的混杂,同时也表明这些木荷优树无性系是较好的关联作图材料。然而进行表型与基因型的关联分析时,应具体分析遗传结构与表型的关系,对关联分析的结果做出正确的判断。
4 结论利用筛选的10对条带清晰、多态性SSR引物对734份木荷优树无性系种质进行遗传多样性分析,共获得105个等位基因(Na),多态信息含量(PIC)为0.668。根据地理来源将其划分为24个种质群体,各种质群体均具有较高的遗传多样性。遗传变异主要存在于群体内,而群体间的交流有限。在选择高遗传多样性群体的基础上,木荷选育应侧重于群体内个体的选择。主坐标分析(PCoA)及STRUCTURE分析均显示群体划分与种质来源不完全相关,在木荷杂交育种亲本选配时须同时考虑地理远缘和亲本群体(个体)间的亲缘关系。木荷优树无性系种质的遗传多样性丰富,可以为种质创新、优良基因挖掘以及关联分析提供基础。
| [] |
陈斐, 魏臻武, 李伟民, 等. 2013. 基于SSR标记的苜蓿种质资源遗传多样性与群体结构分析. 草地学报, 21(4): 759–768.
(Chen F, Wei Z W, Li W M, et al. 2013. Analysis of genetic diversity and population structure in Medicago germplasm by SSR markers. Aata Agrestia Sinica, 21(4): 759–768. DOI:10.11733/j.issn.1007-0435.2013.04.020[in Chinese]) |
| [] |
楚秀丽, 王艺, 金国庆, 等. 2014. 不同生境、初植密度及林龄木荷人工林生长、材性变异及林分分化. 林业科学, 50(6): 152–159.
(Chu X L, Wang Y, Jin G Q, et al. 2014. Variation in growth and wood property and the structure differentiation of Schima superba plantation with different sites, stand densities and ages. Scientia Silvae Sinicae, 50(6): 152–159. [in Chinese]) |
| [] |
邓紫宇. 2012. 利用SSR分子标记研究大花序桉遗传结构. 南宁: 广西大学硕士学位论文. ( Deng Z Y. 2012. Genetic structure of Eucalyptus cloeziana by SSR markers. Nanning:MS thesis of Guangxi University. [in Chinese]) |
| [] |
李康琴. 2013. 鹅掌楸属群体遗传结构及分子系统地理学分析. 南京: 南京林业大学博士学位论文. ( Li K Q. 2013. Studies on populations genetic and molecular phylogeography of Liriodendron. Nanjing:PhD thesis of Nanjing Forestry University. [in Chinese]) |
| [] |
李悦, 张春晓. 2000. 油松育种系统遗传多样性研究. 北京林业大学学报, 22(1): 12–19.
(Li Y, Zhang C X. 2000. Genetic diversity within a breeding system of Pinus tabulaeformis. Journal of Beijing Forestry University, 22(1): 12–19. [in Chinese]) |
| [] |
李培, 阙青敏, 欧阳昆唏, 等. 2016. 不同种源红椿SRAP标记的遗传多样性分析. 林业科学, 52(1): 62–70.
(Li P, Que Q M, Ouyang K X, et al. 2016. Genetic diversity of Toona ciliata from different provenances based on sequence-related amplified polymorphism (SRAP) makers. Scientia Silvae Sinicae, 52(1): 62–70. [in Chinese]) |
| [] |
刘丽华, 王丽新, 赵昌平, 等. 2009. 光温敏二系杂交小麦恢复系遗传多样性和群体结构分析. 中国生物化学与分子生物学报, 25(9): 867–875.
(Liu L H, Wang L X, Zhao C P, et al. 2009. Genetic diversity and alterations of population structure in restorers of dual cross-line hybrid wheat with thermo-photoperiod sensitive male sterile. Chinese Journal of Biochemistry and Molecular Biology, 25(9): 867–875. [in Chinese]) |
| [] |
王秀花, 陈柳英, 马丽珍, 等. 2011. 7年生木荷生长和木材基本密度地理遗传变异及种源选择. 林业科学研究, 24(3): 307–313.
(Wang X H, Chen L Y, Ma L Z, et al. 2011. Geographical provenance variation of growth and wood basic density of 7-year-old Schima superba and its provenance selection. Forest Research, 24(3): 307–313. [in Chinese]) |
| [] |
王永康, 吴国良, 赵爱玲, 等. 2014. 枣种质资源的表型遗传多样性. 林业科学, 50(10): 33–41.
(Wang Y K, Wu G L, Zhao A L, et al. 2014. Phenotypic genetic diversity of jujube germplasm resources. Scientia Silvae Sinicae, 50(10): 33–41. [in Chinese]) |
| [] |
文亚峰, KentaroUchiyama, 韩文军, 等. 2013. 微卫星标记中的无效等位基因. 生物多样性, 21(1): 117–126.
(Wen Y F, Kentaro Uchiyama, Han W J, et al. 2013. Null alleles in microsatellite markers. Biodiversity Science, 21(1): 117–126. [in Chinese]) |
| [] |
辛娜娜, 张蕊, 徐肇友, 等. 2015. 木荷1代育种群体遗传多样性分析. 林业科学研究, 28(3): 332–338.
(Xin N N, Zhang R, Xu Z Y, et al. 2015. Genetic diversity among breeding parents of Schima superba revealed by SSR. Forest Research, 28(3): 332–338. [in Chinese]) |
| [] |
杨爱红, 张金菊, 田华, 等. 2014. 鹅掌楸贵州烂木山居群的微卫星遗传多样性及空间遗传结构. 生物多样性, 22(3): 375–384.
(Yang A H, Zhang J J, Tian H, et al. 2014. Microsatellite genetic diversity and fine-scale spatial genetic structure within a natural stand of Liriodendron chinense (Magnoliaceae) in Lanmushan, Duyun city, Guizhou province. Biodiversity Science, 22(3): 375–384. [in Chinese]) |
| [] |
余亚莹, 邵高能, 圣忠华, 等. 2015. 国内外香稻资源遗传多样性研究. 植物分类与资源学报, 37(6): 871–880.
(Yu Y Y, Shao G N, Sheng Z H, et al. 2015. Genetic diversity of global aromatic rice varieties. Plant Diversity and Resources, 37(6): 871–880. [in Chinese]) |
| [] |
张萍, 金国庆, 周志春, 等. 2004. 木荷苗木性状的种源变异和地理模式. 林业科学研究, 17(2): 192–198.
(Zhang P, Jin G Q, Zhou Z C, et al. 2004. Provenance difference and geographic variation pattern for seedling trait of Schima superba. Forest Research, 17(2): 192–198. [in Chinese]) |
| [] |
张萍, 周志春, 金国庆, 等. 2006. 木荷种源遗传多样性和种源区初步划分. 林业科学, 42(2): 38–42.
(Zhang P, Zhou Z C, Jin G Q, et al. 2006. Genetic diversity analysis and provenance zone allocation of Schima superba in China using RAPD markers. Scientia Silvae Sinicae, 42(2): 38–42. [in Chinese]) |
| [] |
张玲, 焦培培, 李志军. 2012. 中国新疆灰叶胡杨群体遗传多样性的SSR分析. 生态学杂志, 31(11): 2755–2761.
(Zhang L, Jiao P P, Li Z J. 2012. Genetic diversity of Populus pruinosa populations in Xinjiang of China based on SSR analysis. Chinese Journal of Ecology, 31(11): 2755–2761. [in Chinese]) |
| [] |
张蕊, 王艺, 金国庆, 等. 2013. 施氮对木荷3个种源幼苗根系发育和氮磷效率的影响. 生态学报, 33(12): 3611–3621.
(Zhang R, Wang Y, Jin G Q, et al. 2013. Nitrogen addition affects root growth, phosphorus and nitrogen efficiency of three provenances of Schima superba in barren soil. Acta Ecologica Sinica, 33(12): 3611–3621. [in Chinese]) |
| [] |
赵爽, 苏淑钗, 陈志钢, 等. 2016. 基于SSR的辽宁铁岭地区平榛遗传多样性与居群遗传结构分析. 果树学报, 33(1): 24–33.
(Zhao S, Su S C, Chen Z G, et al. 2016. An assessment of the genetic diversity and population genetic structure concerning the Corylus heterophylla Fisch.grown in the Tieling district of Liaoning province, using SSR markers. Journal of Fruit Science, 33(1): 24–33. [in Chinese]) |
| [] |
周志春, 范辉华, 金国庆, 等. 2006. 木荷地理遗传变异和优良种源初选. 林业科学, 19(6): 718–724.
(Zhou Z C, Fan H H, Jin G Q, et al. 2006. Geographic genetic variation and preliminary selection of superior provenance in Schima superba. Scientia Silvae Sincae, 19(6): 718–724. [in Chinese]) |
| [] |
宗绪晓, 关建平, 王海飞, 等. 2010. 世界栽培豌豆(Pisum sativum L.)资源群体结构与遗传多样性分析. 中国农业科学, 43(2): 240–251.
(Zong X X, Guan J P, Wang H F, et al. 2010. Population structure and genetic diversity of global pea (Pisum sativum L.) germplasm resources. Scientia Agricultura Sinica, 43(2): 240–251. [in Chinese]) |
| [] | Aulchenko Y S. 2011. Effects of population structure in genome-wide association studies//Marchini J. Analysis of Complex Disease Association Studies. San Diego:Academic Press. |
| [] | Cardon L R, Palmer L J. 2003. Population stratification and spurious allelic association. The Lancet, 361(9357): 598–604. DOI:10.1016/S0140-6736(03)12520-2 |
| [] | Chuanfu A, Sukumar Saha, Jenkins Johnie N, et al. 2008. Cotton (Gossypium spp.) R2R3-MYB transcription factors SNP identification, phylogenomic characterization, chromosome location, and linkage mapping. Theoretical and Applied Genetics, 116(7): 1015–1026. DOI:10.1007/s00122-008-0732-4 |
| [] | Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software structure:a simulation study. Molecular Ecology, 14(8): 2611–2620. DOI:10.1111/mec.2005.14.issue-8 |
| [] | Excoffier L, Lischer H. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| [] | Ferrão L F V, Caixeta E T, Pena G, et al. 2015. New EST-SSR markers of Coffea arabica:transferability and application to studies of molecular characterization and genetic mapping. Molecular Breeding, 35(1): 1–5. DOI:10.1007/s11032-015-0202-z |
| [] | Kalinowski S T, Taper M L, Marshall T C. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16(5): 1099–1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [] | Lassois L, Denancé C, Ravon E, et al. 2016. Genetic diversity, population structure, parentage analysis, and construction of core collection in the French apple germplasm based on SSR markers. Plant Molecular Biology Reporter, 34(4): 827–844. DOI:10.1007/s11105-015-0966-7 |
| [] | Laurentin H E, Karlovsky P. 2006. Genetic relationship and diversity in a sesame (Sesamum indicum L.) germplasm collection using amplified fragment length polymorphism (AFLP). BMC Genetics, 7(1): 10. DOI:10.1186/1471-2156-7-10 |
| [] | Niu H Y, Li X Y, Ye W H, et al. Isolation and characterization of 36 polymorphic microsatellite markers in Schima superba (Theaceae). Journal of Cell Biology, 18(1): 153–166. DOI:10.1083/jcb.18.1.153 |
| [] | Nybom H. 2004. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants. Molecular Ecology, 13(5): 1143–1155. DOI:10.1111/mec.2004.13.issue-5 |
| [] | Paetkau D, Waits L P, Clarkson P L. 1997. An empirical evaluation of genetic distance statistics using microsatellite data from bear (Ursidae) populations. Genetics, 147(4): 1943–1957. |
| [] | Peakall R, Smouse P E. 2012. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 28(19): 2537–2539. |
| [] | Pritchard J K, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data. Genetics, 7(4): 574–578. |
| [] | Rohlf F J. 2000. NTSYS pc, numerical taxonomy and multivariate analysis system, version 2.1. New York, USA:Exeter Publication Ltd Setauket. |
| [] | Rousset F. 2008. Genepop' 007:a complete re-implementation of the Genepop, software for windows and Linux. Molecular Ecology Resources, 8(1): 103–106. DOI:10.1111/j.1471-8286.2007.01931.x |
| [] | Sun G, Bond M, Nass H, et al. 2003. RAPD polymorphisms in spring wheat cultivars and lines with different level of Fusarium resistance. Theoretical and Applied Genetics, 106(6): 1059–1067. DOI:10.1007/s00122-002-1163-2 |
| [] | Terzopoulos P J, Bebeli P J. 2008. Genetic diversity analysis of Mediterranean faba bean (Vicia faba L.) with ISSR markers. Field Crops Research, 108(1): 39–44. DOI:10.1016/j.fcr.2008.02.015 |
| [] | Wang R, Yu Y, Zhao J, et al. 2008. Population structure and linkage disequilibrium of a mini core set of maize inbred lines in China. Theoretical and Applied Genetics, 117(7): 1141–1153. DOI:10.1007/s00122-008-0852-x |
| [] | Wang S, Liu Y, Ma L, et al. 2014. Isolation and characterization of microsatellite markers and analysis of genetic diversity in Chinese jujube (Ziziphus jujuba Mill.).. PloS One, 9(6): e99842. DOI:10.1371/journal.pone.0099842 |
| [] | Xu Y, Huang L, Ji D, et al. 2015. Construction of a dense genetic linkage map and mapping quantitative trait loci for economic traits of a doubled haploid population of Pyropia haitanensis (Bangiales, Rhodophyta). BMC Plant Biology, 15(1): 228. DOI:10.1186/s12870-015-0604-4 |
| [] | Zeka D, Sedlák P, Sedláková V, et al. 2015. Phenotype and molecular diversity evaluation of some wild 2n Solanum species (super series Rotata). Chilean Journal of Agricultural Research, 75(2): 147–151. DOI:10.4067/S0718-58392015000200002 |
| [] | Zhu Y, Yin Y, Yang K, et al. 2015. Construction of a high-density genetic map using specific length amplified fragment markers and identification of a quantitative trait locus for anthracnose resistance in walnut (Juglans regia L.). BMC Genomics, 16(1): 614. DOI:10.1186/s12864-015-1822-8 |
 2017, Vol. 53
2017, Vol. 53