文章信息
- 王凯利, 傅鹰, 周明兵
- Wang Kaili, Fu Ying, Zhou Mingbing
- 毛竹SQUAMOSA启动子结合蛋白SBP家族基因的鉴定及表达模式分析
- Identification and Expression Pattern of SQUAMOSA Promoter Binding Protein (SBP) Family Genes in Phyllostachys edulis
- 林业科学, 2017, 53(12): 50-61.
- Scientia Silvae Sinicae, 2017, 53(12): 50-61.
- DOI: 10.11707/j.1001-7488.20171206
-
文章历史
- 收稿日期:2017-03-27
- 修回日期:2017-05-19
-
作者相关文章
2. 浙江省竹资源与高效利用协同创新中心 杭州 311300
2. Zhejiang Provincial Collaborative Innovation Center for Bamboo Resources and High-Efficiency Utilization Hangzhou 311300
SBP(SQUAMOSA promoter-binding-protein,SQUA启动子结合蛋白)-box基因家族编码一类植物特有的转录因子——SBP转录因子(辛婧,2006),由于SBP转录因子能够识别并结合MADS-box基因SQUAMOSA(SQUA)启动子,所以又被称为SQUA启动子结合蛋白(辛婧,2006;Chen et al., 2010)。SBP基因首先在金鱼草(Antirhinum majus)中发现(Klein et al., 1996),近年来,在越来越多的植物中发现了SBP基因,如,拟南芥(Arabidopsis thaliana)(Cardon et al., 1997)、水稻(Oryza sativa)(Shao et al., 1999)、番茄(Lycopersicon esculentum)(Salinas et al., 2012)、葡萄(Vitis vinifera)(Hou et al., 2013)、大白菜(Brassica rapa subsp. pekinensis)(Tan et al., 2015)、梅(Prunus mume)(Xu et al., 2015)、甜瓜(Cucumis melo)(Ma et al., 2015)、菊花(Chrysanthemum morifolium)(Song et al., 2016)、玉米(Zea mays)(Zhang et al., 2016)等。但是关于毛竹(Phyllostachys edulis)中SBP家族基因的分析还未见报道。
SBP基因编码的植物SBP转录因子参与了植物营养生长和生殖生长的调控。Martin等(2010)发现AtSBP13会影响拟南芥第1片真叶的发生,Wu等(2006)发现AtSBP3与拟南芥芽的发育有关。在生殖生长阶段,SBP家族基因还与果实的成熟有关(Preston et al., 2013)。Jung等(2011)研究发现AtSBP3,AtSBP4与AtSBP5参与调控拟南芥营养生长与生殖生长的转换。AtSBP8在植物中可以影响花粉囊与雌蕊群的发育(Unte et al., 2003;Xing et al., 2013)。Yu等(2010)研究发现AtSBP3,AtSBP9,AtSBP10和AtSBP13可以影响拟南芥花中毛状体的分布状况。Teotia等(2015)发现SBP家族基因是植物开花过程中的年龄基因,并且它们的反馈作用可以调节植物开花的时期。此外,SBP家族基因还广泛参与植物对非生物与生物胁迫的应答(Hou et al., 2013)。Hou等(2013)发现葡萄的VvSBP6基因对外界非生物胁迫应答明显,在高盐处理与干旱处理时均出现明显增高状态。SBP基因还参与了对抗铜(Eriksson et al., 2004)与真菌毒素胁迫(Stone et al., 2005)等应答反应。SBP家族基因在进化上高度保守,具有保守的典型SBP结构域(Tan et al., 2015)。SBP转录因子的DNA结合结构域有大约76~79个氨基酸残基(Cardon et al., 1999;Klein et al., 1996),可结合顺式作用元件TNCGTACAA(N表示任意碱基)(Cardon et al., 1997;1999;Kropat et al., 2005),它包括2个锌指结构域。2个锌-结合位点蛋白序列为C-C-C-H和C-C-H-C,均具有3个反平行β折叠片。第2个锌指结构域C端具有1个NLS结构域(nuclear localization signal)(Yamasaki et al., 2004;Xu et al., 2015)。2个锌指结构域各司其职,Yamasaki等(2006)发现,第1个锌-结合位点的功能为维持SBP转录因子三级结构的稳定,若缺失位于C端结合区的Cys64则SBP转录因子不能结合在启动子上,第2个锌-结合位点则引导C端loop环正确进入DNA大沟,进行DNA结合。
SBP基因是miR156的靶基因(Song et al., 2016)。水稻有19条SBP基因,其中有11条具有miR156位点(Xie et al., 2006)。miR156不仅可以结合在基因的编码区也可以结合在3′UTR区(3′非翻译区)(Hou et al., 2013; Li et al., 2013)。例如,梅有15条SBP基因,有8条SBP基因的编码区为miR156的靶点,3条SBP基因的3′UTR为miR156靶点,余下的4条未检测到miR156结合位点。大白菜的miR156可以调节SBP家族基因编码区或者3′UTR区(Tan et al., 2015)。在葡萄(Hou et al., 2013)、拟南芥(Gandikota et al., 2007)等其他物种中也有类似的报道。
miR156是SBP家族基因表达调控的重要成员,高盐胁迫可以引起miR156的表达量上调,进而抑制SBP基因的表达(Sun et al., 2012)。Teotia等(2015)发现,miR156是拟南芥成花诱导5大途径(年龄途径、赤霉素途径、光周期途径、春化途径、自主途径)中年龄途径中的一个关键microRNA。在植物开花过程中,SBP家族基因表达受到miR156在RNA水平上的调节,同样,某些SBP转录因子会反馈调节miR156的表达水平,例如,Wei等(2012)发现,AtSBP15以结合miR156B启动子的方式抑制其表达,而Wu等(2009)发现AtSBP9与AtSBP10促进miR156表达。
毛竹在我国分布广泛,依据第八次全国森林资源清查报告,我国毛竹林面积为443万hm2,占竹林总面积的74%(郭起荣等,2015)。Peng等(2013)公布了毛竹基因组草图与数据,这为毛竹的研究提供了可靠的基因组数据支持。自然状态下毛竹开花周期长、结籽率低(Ge et al., 2017;张莹等,2016),因此研究毛竹开花相关基因对于毛竹开花分子机制的解析具有重要意义。
本文主要对与毛竹开花密切相关的SBP家族基因进行全基因组鉴定,并通过生物信息学工具对其结构和进化模式开展系统的调查,利用实时荧光定量PCR分析毛竹SBP基因在毛竹的不同组织部位及在盐胁迫过程中的表达模式,为毛竹SBP家族基因功能分析奠定基础。
1 材料与方法 1.1 毛竹的SBP家族基因序列的获得、筛选及进化树的构建为了鉴定毛竹的SBP家族基因序列,首先在PlanTFDB数据库(http://plntfdb.bio.uni-potsdam.de/v3.0/)下载拟南芥和水稻SBP家族基因蛋白序列,然后在毛竹基因组数据库(http://www.bamboogdb.org/index.jsp)下载毛竹基因组蛋白序列。以拟南芥和水稻的SBP家族基因序列为query,通过blastp检索毛竹SBP家族基因蛋白序列(E < -10)。为了剔除假阳性序列,利用MEME软件(http://meme-suite.org/tools/meme)检索毛竹SBP家族基因蛋白序列保守结构域。
将毛竹SBP家族基因序列命名为PeSBP-N (N为序号)。
鉴定出的阳性毛竹SBP家族基因蛋白序列,利用MEGA6.0软件,通过最大似然法(maximum likelihood,ML)分析构建系统发育树的最佳模型。根据分析出的最佳进化模型构建毛竹SBP家族基因蛋白进化树,重复次数为1 000次。
1.2 毛竹、拟南芥和水稻SBP家族基因进化树的构建将拟南芥、禾本科(Gramineae)植物水稻与毛竹SBP基因家族蛋白序列通过MEGA 6.0软件比对,通过最大似然法(maximum likelihood,ML)分析构建其系统发育树的最佳模型。根据分析出的最佳进化模型构建3个物种间的系统发育树,重复次数为1 000次。
1.3 毛竹SBP家族基因结构分析在毛竹基因组数据库(http://www.bamboogdb.org/index.jsp)下载SBP家族基因对应的DNA序列,并在scaffold的位置,利用Gene Structure Display Server 2.0(http://gsds.cbi.pku.edu.cn/index.php)软件分析毛竹SBP家族基因结构。
1.4 毛竹SBP家族基因miR156靶位点预测在毛竹基因组数据库(http://www.bamboogdb.org/index.jsp)下载毛竹miR156基因序列,利用psRNATarget(http://plantgrn.noble.org/psRNATarget/?function=3)软件检索毛竹SBP家族基因中miR156靶位点。
1.5 毛竹SBP家族基因表达量分析毛竹种子种植于泥炭:蛭石:珍珠岩体积比为1:1:1的人工培养基上,放置于温室,16 h/8 h昼夜时数,23 ℃/18 ℃昼夜温度,相对湿度控制在55%~60%(Wu et al., 2015)。待株高达20~25 cm时,分别选取根、茎、叶(图 1A)立即放于液氮中速冻。

|
图 1 实时荧光定量PCR材料采集 Figure 1 Samples used in the quantitative real-time fluorescence PCR A.毛竹根、茎、叶取样;B.毛竹花序取样。 A. Sampled tissues including root, stem and leaf of Phyllostachys edulis;B. The inflorescences of P. edulis. |
2015年6—7月间在广西灵川县收集毛竹花序样品,将毛竹花序发育分为4个阶段:花序形成初期(P1)、小穗的分化(P2)、颖花的分化(P3)和小穗的生长(P4)(图 1B)(郭起荣等,2015)。收集4个阶段的花器官,迅速将样品放入液氮速冻。
以上述相同的方法种植毛竹实生苗,待株高到达20~25 cm时,对照组浇灌无菌水,试验组浇灌等量200 mmol·L-1的NaCl溶液(张智俊等,2011),分别在第1天、第3天、第5天取毛竹叶片,对照组分别命名为CK1,CK2与CK3,试验组分别命名为T1,T2与T3,迅速将样品放入液氮速冻。
所有样品通过Trizol试剂(TaKaRa,日本)提取总RNA,采用反转录试剂盒(TaKaRa,日本)将RNA反转录为cDNA。以毛竹NTB基因(Nucleotide Tract-Binding Protein)为内参基因(Fan et al., 2013),通过Primer premier 5.0软件设计SBP家族基因引物(表 1)。选用SYBR® Premix Ex TaqTM Ⅱ定量试剂(TaKaRa日本),在CFX96荧光定量PCR仪(Bio-Rad,美国)上进行扩增:95 ℃ 30 s预变性;95 ℃ 5 s变性,55 ℃ 30 s复性延伸,40个循环;最后得到熔解曲线。
|
|
在PlanTFDB数据库分别获得15条拟南芥和19条水稻SBP家族基因序列,通过blastp比对获得37条疑似毛竹SBP家族基因序列。通过MEME软件获得具有完整SBP结构域的基因18条(表 2,图 2A,B)。毛竹中SBP结构域自C-Q-V到R-R-R,共2个锌指结构域和1个核定位信号结构域(NLS),共有74个氨基酸残基(图 2C)。
|
|
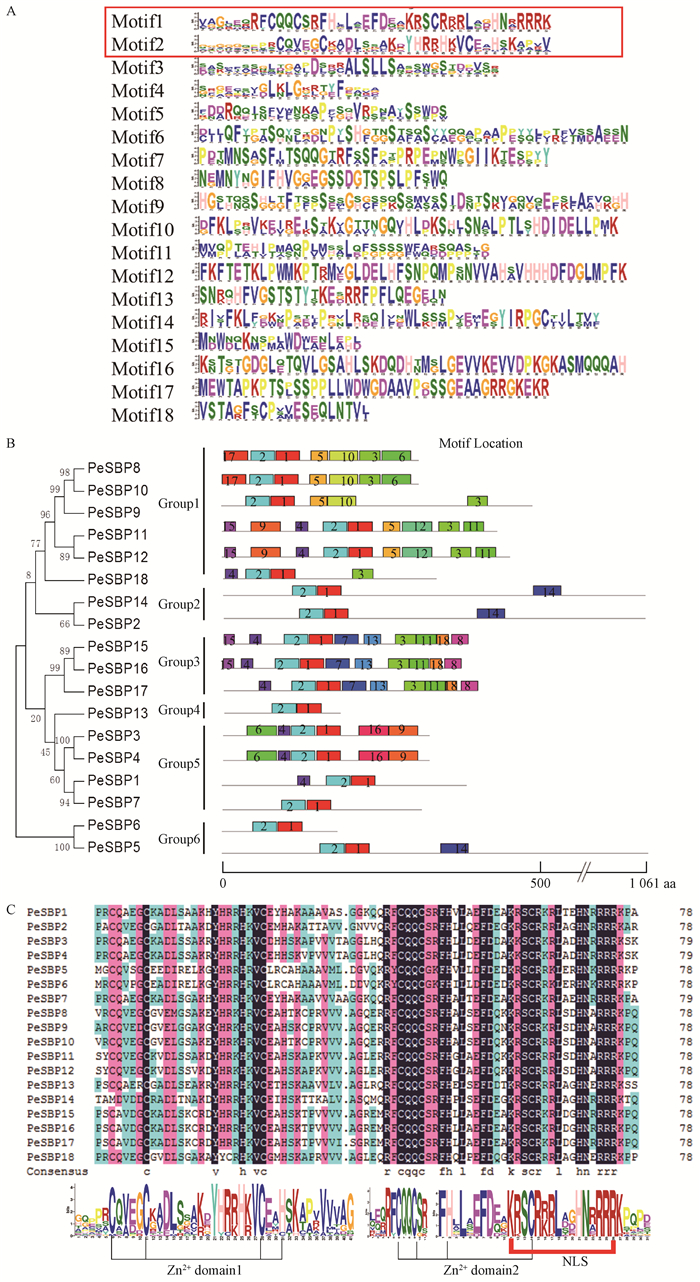
|
图 2 毛竹SBP家族基因蛋白序列保守结构鉴定 Figure 2 Characterization of conserved motifs of amino acid sequences of Phyllostachys edulis SBP family genes A.毛竹SBP家族基因蛋白序列18个保守结构域;B.毛竹SBP家族基因保守基序分布图,每一个Motif都分别以不同的彩色背景与数字标注,左侧为进化树;C.毛竹SBP蛋白序列SBP结构域,利用DNAMAN获得多序列比对结果,2个锌离子结合结构域与NLS已经标注。氨基酸的高度表示此处氨基酸的保守性。A. Eighteen motifs of SBP transcription factor. B. Motif distribution of P. edulis SBP family genes. Each motif is represented by a colored block with a number. The left is phylogenetic tree of P. edulis SBP family genes. C. Multiple sequence alignment and sequence logo of the P. edulis SBP domain. Multiple sequence alignment was performed using DNAMAN. The two conserved zinc finger and NLS are indicated. The overall height of the stack indicates the sequence conservation at that position. |
通过MEME软件鉴定出的18个结构域中,Motif 2和Motif 1组成完整的SBP结构域(图 2A)。Motif 2是第1个锌指结构域(C-C-C-H),Motif 1是第2个锌指结构域(C-C-H-C)和NLS结构域(核定位信号)(图 2C)。18条SBP蛋白中Motif超过5个的有10条(图 2B)。
毛竹18条SBP家族基因蛋白序列最佳进化树分析模型为JTT+G+I,重复1 000次,根据进化关系远近可分为6组(图 2B)。
2.2 毛竹、拟南芥和水稻SBP基因亲缘关系分析拟南芥、水稻与毛竹3个物种SBP家族基因蛋白序列最佳进化模型为JTT+G,重复1 000次。54条SBP蛋白序列可以分为12组(Clade1-Clade12)(图 3)。毛竹SBP家族基因与水稻SBP家族基因亲缘关系最近,每一条毛竹中的SBP蛋白序列均能找到进化关系相近的水稻SBP转录因子。
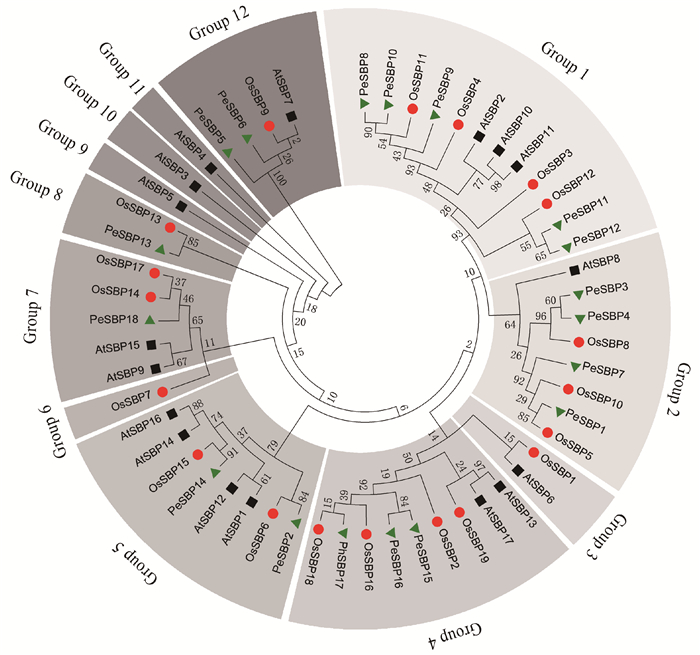
|
图 3 拟南芥、水稻与毛竹中SBP 家族基因进化关系 Figure 3 Unrooted phylogenetic tree of SBP family genes from Phyllostachys edulis,Arabidopsis thaliana and Oryza sativa |
通过Gene Structure Display Server 2.0软件分析,得到毛竹SBP家族基因DNA序列中内含子、外显子、5′UTR和3′UTR的分布情况(图 4A)。18条SBP家族基因的平均长度为5 464 bp。Group1、Group4与Group5相比于Group2、Group3和Group6 SBP家族基因DNA序列较长。PeSBP2、PeSBP5和PeSBP14的内含子与外显子最多,均有10个内含子和11个外显子;PeSBP13仅有1个内含子与外显子。
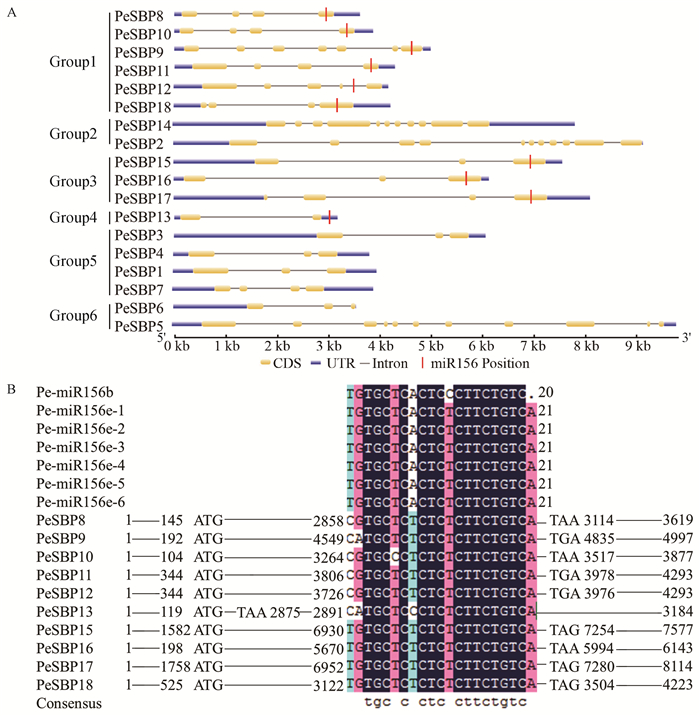
|
图 4 毛竹SBP家族基因结构及miRNA靶位点 Figure 4 The structure of Phyllostachys edulis SBP family genes and target sites of miRNA A.毛竹SBP家族基因内含子、外显子、5′与3′UTR分布图。黑色直线表示内含子,左侧蓝色圆柱形表示5′UTR,右侧蓝色圆柱形表示3′UTR,黄色圆柱形表示外显子。红色标记为miR156位置。标尺标注了基因的长度。B.毛竹miR156序列与毛竹SBP家族基因DNA序列比对图,数字表示碱基位置。 A. Exon-intron structures of P. edulis SBP family genes. Phylogenetic tree was constructed based on P. edulis SBP protein sequences. UTR, exons and introns are indicated by blue rounded rectangles, yellow rounded rectangles, and black lines, respectively. Red line indicated the miR156 position. The scale represents the lengths of the genes. B. Multiple sequence alignment of Ph-miR156 complementary sequences with P. edulis SBP family genes. Numbers indicate the position of the nucleotide sequences. |
在毛竹基因组数据库里共找到miR156序列7条,分别为Pe-miR156b以及Pe-miR156e-1-Pe-miR156e-6。经过DNAMAN比对分析后发现,Pe-miR156e-1-Pe-miR156e-6序列完全一致,序列为UGACAGAAGAGAGUGAGCACA,共21 nt;而Pe-miR156b为GACAGAAGGGAGUGAGCACA,共有20 nt,相比较Pe-miR156e,缺失第1个碱基U并且第10位碱基为G,而Pe-miR156e第10位碱基为A(图 4B)。
将7条miR156序列与毛竹SBP基因序列比对之后发现,有10条SBP家族基因序列有miR156靶位点,9条基因的miR156靶位点位于基因的编码区,1条位于3′UTR区(图 4B)。PeSBP8,PeSBP10,PeSBP11,PeSBP17与PeSBP18的miR156靶位点位于各自第4外显子处,PeSBP15,PeSBP16 miR156靶位点位于各自第3外显子处,PeSBP9 miR156靶位点位于其第7外显子处,PeSBP12 miR156靶位点位于其第4内含子处,仅PeSBP13的miR156靶位点位于3′UTR区。并且miR156靶位点均位于基因接近尾部的位置(图 4A)。
2.5 毛竹SBP家族基因表达量分析通过实时荧光定量PCR检测了毛竹SBP家族基因在毛竹实生苗根、茎、叶中的表达情况。在根中,PeSBP16相对表达量最高,PeSBP9相对表达量较高,其他PeSBP基因在根中的表达量较低。在茎中,PeSBP11相对表达量最高,PeSBP5,PeSBP9和PeSBP10有较高的表达量,而其他PeSBP基因在茎中相对表达量较低。在叶中,PeSBP5相对表达量最高,PeSBP6在叶中的相对表达量高于其在根和茎中的相对表达量(图 5)。
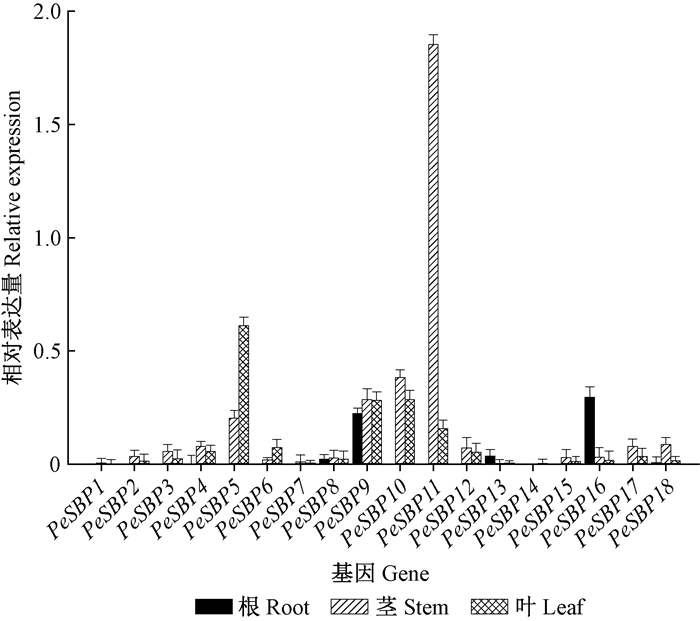
|
图 5 毛竹不同组织SBP家族基因相对表达量 Figure 5 Relative expression of Phyllostachys edulis SBP family genes in different organs |
本研究分析了毛竹SBP家族基因自花序形成初期(P1)经过小穗的分化(P2)和颖花的分化(P3)到小穗的生长(P4)4个阶段表达模式的变化(图 1B;图 6)。在毛竹花序发育的4个时期,毛竹SBP家族基因相对表达量发生了不同程度的变化,根据基因相对表达量趋势,可以将SBP家族基因分成5类。第1类:无明显表达量,且表达趋势变化不明显,包括有PeSBP1,PeSBP2,PeSBP3,PeSBP7,PeSBP15与PeSBP17共6个基因。第2类:先降后升,P1-P3期PeSBP4与PeSBP13相对表达量逐渐下降,在P3期到达最低点,P4期又重新上升。第3类:先升后降,PeSBP10与PeSBP14相对表达量逐渐上升在P3期到达最高点,PeSBP12与PeSBP18相对表达量逐渐上升在P2期到达最高点,之后相对表达量均下降。第4类:先升后降再升,P1-P2期PeSBP5,PeSBP9与PeSBP16相对表达量逐渐升高,P3期下降,到P4期相对表达量又上升。第5类:先降后升再降,P1-P2期PeSBP6,PeSBP8与PeSBP11相对表达量逐渐下降,P3期相对表达量上升,到P4期相对表达量又下降。
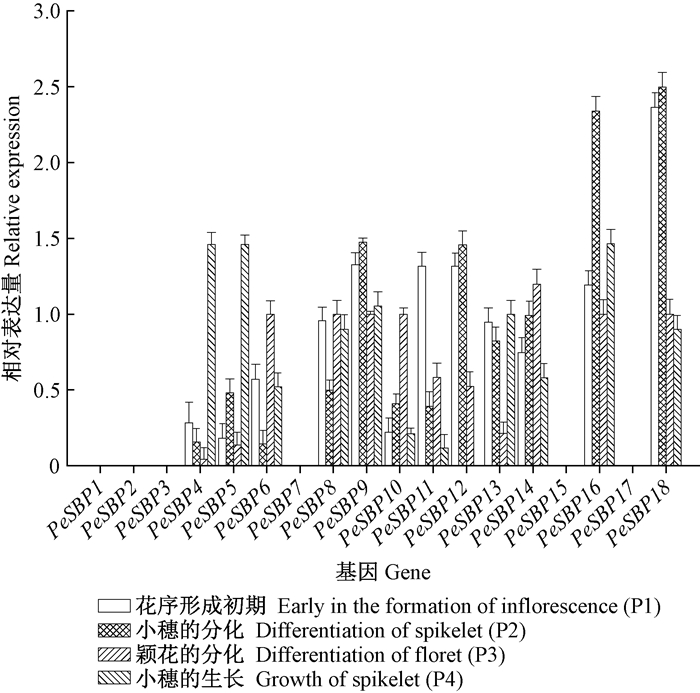
|
图 6 毛竹花序发育过程SBP家族基因相对表达量 Figure 6 Relative expression levels of Phyllostachys edulis SBP genes in the development of inflorescences |
本研究也调查了毛竹SBP基因受盐胁迫过程表达模式的变化,当毛竹实生苗受高盐胁迫后,毛竹所有SBP家族基因均发生了不同程度的表达量变化(图 7)。根据基因表达量变化趋势可以将毛竹SBP家族基因分为3类。第1类:基因相对表达量逐渐下降,包括PeSBP1,PeSBP5,PeSBP6,PeSBP7,PeSBP13,PeSBP15,PeSBP16,PeSBP17与PeSBP18。第2类:基因相对表达量逐渐上升,包括PeSBP2,PeSBP3,PeSBP4,PeSBP8,PeSBP10,PeSBP11,PeSBP12与PeSBP14。第3类:基因相对表达量先升后降,PeSBP9在高盐胁迫的第1~3天(T1-T2期)相对表达量逐渐上升,在第3天(T2期)到达最高点,在第5天(T3期)相对表达量下降并低于第1天(T1期)相对表达量。

|
图 7 盐胁迫下毛竹SBP家族基因相对表达量 Figure 7 Relative expression levels of Phyllostachys edulis SBP family genes under salinity treatment |
SBP家族基因编码的SBP转录因子为植物所特有,此类家族基因具有高度保守性,参与植物生长发育的多个阶段,在花发育中的作用尤为重要。SBP基因在多种植物中均有发现,尤其在模式植物拟南芥中研究较为透彻,但在竹亚科(Bambusoideae)植物毛竹中却未有报道。
3.1 毛竹SBP家族基因特性本研究最初在毛竹中共发现37条疑似SBP家族基因序列,经MEME检索结构域确认,18条SBP转录因子具有完整的74个氨基酸残基的SBP结构域。毛竹SBP家族基因蛋白序列中NLS序列为K-R-S-C-R-R-R-L-A-G-H-N-R-R-R-R,仅1个氨基酸残基与梅(Yu et al., 2010)中的NLS序列(K-R-S-C-R-R-R-L-A-G-H-N-E-R-R-R)不同。这18条基因被认为是真正的SBP家族基因。构建进化树发现,进化关系相近的毛竹SBP转录因子的Motif和基因结构均具相似性,例如,PeSBP11与PeSBP12亲缘关系比较近,2条基因的Motif完全一致。组内Motif数目也大致具有相似性,例如,Group3中PeSBP15与PeSBP16的Motif最多达到了10个,而PeSBP6、PeSBP7与PeSBP13仅有1个SBP结构域。PeSBP2、PeSBP5和PeSBP14均有10个内含子和11个外显子,PeSBP13仅1个内含子与外显子。拟南芥、水稻和毛竹SBP家族基因进化树分析得出,毛竹SBP家族基因与拟南芥SBP家族基因亲缘关系较远,毛竹SBP家族基因与水稻SBP家族基因进化关系近,并且在水稻中都可以找到相应的毛竹SBP同源基因,推测在进化过程中毛竹基因组并未出现特异的SBP家族基因。也从侧面证实了SBP家族基因在进化上的保守性。10条毛竹SBP家族基因具有miR156靶位点,并且均靠近基因尾端,miR156既可以结合于基因编码区也可以结合于基因UTR区(Hou et al., 2013; Li et al., 2013)。
3.2 毛竹SBP家族基因具有多样的表达模式调查了毛竹SBP家族基因在毛竹实生苗根、茎、叶中的相对表达量。PeSBP11和PeSBP16相对表达量分别在茎和根中最高,暗示它们分别在茎和根发育中可能发挥着重要作用。PeSBP9在根中相对表达量较高(图 5),可能参与了毛竹根的发育。PeSBP5,PeSBP9,PeSBP10与PeSBP11在茎和叶中均呈现出高相对表达量,表明这些基因可能参与了毛竹茎和叶的发育(Wang et al., 2008;Martin et al., 2010)。
在花序发育过程的P1-P4期,有6个基因(PeSBP1,PeSBP2,PeSBP3,PeSBP7,PeSBP15与PeSBP17)表达量较低,且无明显的表达量变化,暗示它们可能与花序发育关系不大。有7个毛竹SBP家族基因(PeSBP5,PeSBP9,PeSBP10,PeSBP12,PeSBP14,PeSBP16和PeSBP18)在P1-P2表达量上升,表明可能参与了早期的花序发育(王翊等,2011),其中PeSBP10与PeSBP14高表达延续到P3期。在P3期毛竹花序继续伸长,生长锥形成,雄雌蕊发育成花器官,第1朵小花形成。7个基因(PeSBP6,PeSBP8,PeSBP9,PeSBP10,PeSBP14,PeSBP16和PeSBP18)在P3期表达值较高,表明这7个基因可能参与了雄雌蕊发育(郭起荣等,2015)。P4期侧生与顶端小穗迅速生长(郭起荣等,2015),毛竹SBP家族基因中有5个基因(PeSBP4,PeSBP5,PeSBP9,PeSBP13与PeSBP16)在P4期表达值较高,表明这5个基因可能参与了小穗的生长(郭起荣等,2015)。
AtSBP8可以影响花粉囊与雌蕊群的发育(Unte et al., 2003;Xing et al., 2013)。AtSBP8缺失突变体导致植物生育能力降低,它的敲除可能也会影响萼片上的毛状体形成和雄蕊丝伸长(Unte et al., 2003)。拟南芥的花器官具有典型的花萼但是毛竹却无明显的花萼(田大刚等,2011;孙立方等,2012),与AtSBP8同源的PeSBP3在花发育4个时期相对表达量均比较低,可能与毛竹特有花序结构有关。AtSBP9和AtSBP15在拟南芥由营养生长到生殖生长的转换中起到了促进作用(Schwarz et al., 2008;Wang et al., 2008),与AtSBP9同源的PeSBP5,PeSBP6及与AtSBP15同源的PeSBP14在毛竹花发育的4个时期均有较为明显的表达,暗示着这3个基因可能参与毛竹营养生长向生殖生长转变的调控。
葡萄SBP家族基因在高盐环境下表现出了不同的表达模式,参与了葡萄对于盐胁迫的反应(Hou et al., 2013)。在菊花的盐胁迫中,CmSBP4,CmSBP9与CmSBP11对盐胁迫的反应趋势比其他9个菊花SBP家族基因反应更为明显(Song et al., 2016)。高盐胁迫环境下毛竹SBP家族基因表现出了3类不同的表达趋势。第1类,PeSBP1,PeSBP5,PeSBP6,PeSBP7,PeSBP13,PeSBP15,PeSBP16,PeSBP17与PeSBP18随着高盐处理时间从第1~5天(T1-T3期)相对表达量逐渐下降,可能是它们的表达受盐胁迫反馈抑制(Hou et al., 2013;Song et al., 2016);第2类,自高盐胁迫第1天(T1期)开始随着高盐胁迫处理时间的延长,PeSBP2,PeSBP3,PeSBP4,PeSBP8,PeSBP10,PeSBP11,PeSBP12与PeSBP14相对表达量逐渐上升,暗示着这些基因可能在毛竹高盐胁迫响应的过程中起到了主要作用(Hou et al., 2013;Song et al., 2016);第3类,相对表达量先升后降(PeSBP9),表明PeSBP9在高盐胁迫第1~3天(T1-T2期)参与了盐胁迫的响应,随着处理时间延长又被抑制。
4 结论本研究利用生物信息学技术鉴定了毛竹18条SBP家族基因,证明了毛竹18条SBP家族基因具有完整的SBP结构域并且非常保守。毛竹与水稻SBP家族基因进化关系较近。在18条毛竹SBP家族基因中有10条基因在靠近基因尾端有miR156识别的靶位点。SBP家族基因参与了毛竹营养生长与生殖生长的调控,PeSBP5,PeSBP9,PeSBP10与PeSBP11对叶的发育具有重要的影响,PeSBP8,PeSBP9,PeSBP10,PeSBP11,PeSBP12,PeSBP13,PeSBP16与PeSBP18可能在毛竹花序发育过程中发挥了重要的作用。在毛竹高盐胁迫处理中,SBP家族基因均表现出了相对表达量的变化,大部分基因参与了毛竹对高盐胁迫的应答反应。
郭起荣, 周建梅, 孙立方, 等. 2015. 毛竹的花序发育研究[J]. 植物科学学报, 33(1): 19-24. (Guo Q R, Zhou J M, Sun L F, et al. 2015. Development of Phyllostachys edulis inflorescences[J]. Plant Science Journal, 33(1): 19-24. DOI:10.11913/PSJ.2095-0837.2015.10019 [in Chinese]) |
孙立方, 郭起荣, 王青, 等. 2012. 毛竹花器官的形态与结构[J]. 林业科学, 48(11): 124-129. (Sun L F, Guo Q R, Wang Q, et al. 2012. Flower organs morphology and structure of Phyllostachys edulis[J]. Scientia Silvae Sinicae, 48(11): 124-129. DOI:10.11707/j.1001-7488.20121120 [in Chinese]) |
田大刚, 刘华清, 苏军, 等. 2011. 水稻与拟南芥中控制花器官发育MADS-box基因的比较研究进展[J]. 福建农业学报, 26(2): 309-320. (Tian D G, Liu H Q, Su J, et al. 2011. Flower-development-controlling MADS-box genes in rice and Arabidopsis thaliana[J]. Fujian Journal of Agricultural Sciences, 26(2): 309-320. [in Chinese]) |
王翊, 胡宗利, 杨妤欣, 等. 2011. 水稻SBP基因家族的生物信息学分析[J]. 生物信息学, 9(1): 82-87. (Wang Y, Hu Z L, Yang Y X, et al. 2011. Bioinformatics analysis of SBP-box gene family in rice[J]. China Journal of Bioinformatics, 9(1): 82-87. [in Chinese]) |
辛婧. 2006. SBP转录因子的结构和功能[J]. 现代生物医学进展, 6(12): 140-142. (Xin J. 2006. The structure and function of SBP transcription factors[J]. Progress in Modern Biomedicine, 6(12): 140-142. [in Chinese]) |
张智俊, 杨洋, 罗淑萍, 等. 2011. 毛竹液泡膜Na+/H+逆向运输蛋白基因克隆及表达分析[J]. 农业生物技术学报, 19(1): 69-76. (Zhang Z J, Yang Y, Luo S P, et al. 2011. Molecular cloning and expression analysis of a vacuolar Na+/H+ antiporter gene in Moso bamboo[J]. Journal of Agricultural Biotechnology, 19(1): 69-76. [in Chinese]) |
张莹, 孙立方, 冉洪, 等. 2016. 毛竹花粉的形态及双受精过程研究[J]. 广西植物, 36(11): 1325-1329. (Zhang Y, Sun L F, Ran H, et al. 2016. Pollen morphology and double fertilization of Phyllostachys edulis[J]. Guihaia, 36(11): 1325-1329. [in Chinese]) |
Cardon G H, Höhmann S, Nettesheim K, et al. 1997. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3: a novel gene involved in the floral transition[J]. Plant Journal for Cell & Molecular Biology, 12(2): 367-377. |
Cardon G, Höhmann S, Klein J, et al. 1999. Molecular characterisation of the Arabidopsis, SBP-box genes[J]. Gene, 237(1): 91-104. DOI:10.1016/S0378-1119(99)00308-X |
Chen X, Zhang Z, Liu D, et al. 2010. SQUAMOSA promoter-binding protein-like transcription factors:star players for plant growth and development[J]. Journal of Integrative Plant Biology, 52(11): 946-951. DOI:10.1111/jipb.2010.52.issue-11 |
Eriksson M, Moseley J L, Tottey S, et al. 2004. Genetic dissection of nutritional copper signaling in Chlamydomonas distinguishes regulatory and target genes[J]. Genetics, 168(2): 795-807. DOI:10.1534/genetics.104.030460 |
Fan C, Ma J, Guo Q, et al. 2013. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis)[J]. PLoS One, 8(2): e56573. DOI:10.1371/journal.pone.0056573 |
Gandikota M, Birkenbihl R P, Höhmann S, et al. 2007. The miRNA156/157 recognition element in the 3'UTR of the Arabidopsis SBP box gene SPL3 prevents early flowering by translational inhibition in seedlings[J]. Plant Journal for Cell & Molecular Biology, 49(4): 683. |
Ge W, Zhang Y, Cheng Z, et al. 2017. Main regulatory pathways, key genes and microRNAs involved in flower formation and development of Moso bamboo (Phyllostachys edulis)[J]. Plant Biotechnology Journal, 15(1): 82-96. DOI:10.1111/pbi.2017.15.issue-1 |
Hou H, Li J, Gao M, et al. 2013. Genomic organization, phylogenetic comparison and differential expression of the SBP-box family genes in grape[J]. PLoS One, 8(3): e59358. DOI:10.1371/journal.pone.0059358 |
Jung J H, Seo P J, Kang S K, et al. 2011. miR172 signals are incorporated into the miR156 signaling pathway at the SPL3/4/5, genes in Arabidopsis, developmental transitions[J]. Plant Molecular Biology, 76(1): 35-45. |
Klein J, Saedler H, Huijser P. 1996. A new family of DNA binding proteins includes putative transcriptional regulators of the Antirrhinum majus ffloral meristem identity gene SQUAMOSA[J]. Mol Gen Genet, 250(1): 7-16. |
Kropat J, Tottey S, Birkenbihl R P, et al. 2005. A regulator of nutritional copper signaling in Chlamydomonas is an SBP domain protein that recognizes the GTAC core of copper response element[J]. Proceedings of the National Academy of Sciences of the United States of America, 102(51): 18730-18735. DOI:10.1073/pnas.0507693102 |
Li J, Hou H M, Li X Q, et al. 2013. Genome-wide identification and analysis of the SBP-box family genes in apple (Malus×domestica Borkh.)[J]. Plant Physiology and Biochemistry, 70(1): 100-114. |
Ma Y, Guo J W, Bade R, et al. 2015. Genome-wide identification and phylogenetic analysis of the SBP-box gene family in melons[J]. Journal of Plant Growth Regulation, 13(1): 8794-8806. |
Martin R C, Asahina M, Liu P P, et al. 2010. The regulation of post-germinative transition from the cotyledon-to vegetative-leaf stages by microRNA-targeted SQUAMOSA PROMOTER-BINDING PROTEIN LIKE13 in Arabidopsis[J]. Seed Science Research, 20(2): 89-96. DOI:10.1017/S0960258510000073 |
Peng Z H, Lu Y, Li L B, et al. 2013. The draft genome of the fast-growing non-timber forest species Moso bamboo (Phyllostachys heterocycla)[J]. Nature Genetics, 45(4): 1-2. |
Preston J C, Hileman L C. 2013. Functional evolution in the plant SQUAMOSA-PROMOTER BINDING PROTEIN-LIKE (SPL) gene family[J]. Frontiers in Plant Science, 4(80): 1-13. |
Salinas M, Xing S, Höhmann S, et al. 2012. Genomic organization, phylogenetic comparison and differential expression of the SBP-box family of transcription factors in tomato[J]. Planta, 235(6): 1171-1184. DOI:10.1007/s00425-011-1565-y |
Schwarz S, Grande A V, Bujdoso N, et al. 2008. The microRNA regulated SBP-box genes SPL9 and SPL15 control shoot maturation in Arabidopsis[J]. Plant Molecular Biology, 67(1): 183-195. |
Shao C X, Takenda Y, Hatano S, et al. 1999. Rice gene encoding the SBP domain protein, which is a new type of transcription factor controlling plant development[J]. Rice Genet Newsl, 16: 114. |
Song A P, Wu D, Fan Q Q, et al. 2016. Transcriptome-wide identification and expression profiling analysis of Chrysanthemum trihelix transcription factors[J]. Plant Physiology and Biochemistry, 102(2): 10-16. |
Stone J M, Liang X W, Nekl E R, et al. 2005. Arabidopsis AtSPL14, a plant-specific SBP-domain transcription factor, participates in plant development and sensitivity to fumonisin B1[J]. The Plant Journal, 41(5): 744-754. DOI:10.1111/tpj.2005.41.issue-5 |
Sun G L, Stewart C N, Xiao P, et al. 2012. MicroRNA expression analysis in the cellulosic biofuel crop switchgrass (Panicum virgatum) under abiotic stress[J]. PLoS One, 7(3): e32017. DOI:10.1371/journal.pone.0032017 |
Tan H W, Song X M, Duan W K, et al. 2015. Genome-wide analysis of the SBP-box gene family in Chinese cabbage (Brassica rapa subsp. pekinensis)[J]. Genome, 58(11): 463-477. DOI:10.1139/gen-2015-0074 |
Teotia S, Tang G L. 2015. To bloom or not to bloom:role of microRNAs in plant flowering[J]. Molecular Plant, 8(3): 359-377. DOI:10.1016/j.molp.2014.12.018 |
Unte U S, Sorensen A M, Pesaresi P, et al. 2003. SPL8, an SBP-box gene that affects pollen sac development in Arabidopsis[J]. Plant Cell, 15(4): 1009-1019. DOI:10.1105/tpc.010678 |
Wang J W, Schwab R, Czech B, et al. 2008. Dual effects of miR156-targeted SPL genes and CYP78A5/KLUH on plastochron length and organ size in Arabidopsis thaliana[J]. Plant Cell, 20(5): 1231-1243. DOI:10.1105/tpc.108.058180 |
Wei S, Gruber M Y, Yu B Y, et al. 2012. Arabidopsis mutant sk156 reveals complex regulation of SPL15 in a miR156-controlled gene network[J]. BMC Plant Biology, 12(1): 49-54. DOI:10.1186/1471-2229-12-49 |
Wu G, Park M Y, Conway S R, et al. 2009. The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis[J]. Cell, 138(4): 750-759. DOI:10.1016/j.cell.2009.06.031 |
Wu G, Poethig R S. 2006. Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its target SPL3[J]. Development, 133(18): 3539-3547. DOI:10.1242/dev.02521 |
Wu H, Lv H, Li L, et al. 2015. Genome-wide analysis of the AP2/ERF transcription factors family and the expression patterns of DREB genes in Moso bamboo(Phyllostachys edulis)[J]. PLoS One, 10(5): e0126657. DOI:10.1371/journal.pone.0126657 |
Xie K B, Wu C Q, Xiong L Z. 2006. Genomic organization, differential expression, and interaction of SQUAMOSA promoter-binding-like transcription factors and microRNA156 in rice[J]. Plant Physiology, 142(1): 280-293. DOI:10.1104/pp.106.084475 |
Xing S, Salinas M, Garcia-Molina A, et al. 2013. SPL8 and miR156-targeted SPL genes redundantly regulate Arabidopsis gynoecium differential patterning[J]. Plant Journal, 75(4): 566-577. DOI:10.1111/tpj.2013.75.issue-4 |
Xu Z D, Sun L D, Zhou Y Z, et al. 2015. Identification and expression analysis of the SQUAMOSA promoter-binding protein (SBP)-box gene family in Prunus mume[J]. Mol Genet Genomics, 290(5): 1701-1715. DOI:10.1007/s00438-015-1029-3 |
Yamasaki K, Kigawa T, Inoue M, et al. 2004. A novel zinc-binding motif revealed by solution structures of DNA-binding domains of Arabidopsis, SBP-family transcription factors[J]. Journal of Molecular Biology, 337(1): 49-63. DOI:10.1016/j.jmb.2004.01.015 |
Yamasaki K, Kigawa T, Inoue M, et al. 2006. An Arabidopsis SBP-domain fragment with a disrupted C-terminal zinc-binding site retains its tertiary structure[J]. FEBS Letters, 580(8): 2109-2116. DOI:10.1016/j.febslet.2006.03.014 |
Yu N, Cai W J, Wang S C, et al. 2010. Temporal control of trichome distribution by microRNA156-targeted SPL genes in Arabidopsis thaliana[J]. Plant Cell, 22(7): 2322-2335. DOI:10.1105/tpc.109.072579 |
Zhang W, Li B, Yu B. 2016. Genome-wide identification, phylogeny and expression analysis of the SBP-box gene family in maize (Zea mays)[J]. Journal of Integrative Agriculture, 15(1): 29-41. DOI:10.1016/S2095-3119(14)60955-2 |
 2017, Vol. 53
2017, Vol. 53

