文章信息
- 毛秀红, 郑勇奇, 孙百友, 张元帅, 韩丛聪, 位晓, 荀守华
- Mao Xiuhong, Zheng Yongqi, Sun Baiyou, Zhang Yuanshuai, Han Congcong, Wei Xiao, Xun Shouhua
- 基于SSR的刺槐无性系遗传多样性分析和指纹图谱构建
- Genetic Diversity and Fingerprints of Robinia pseudoacacia Clones Based on SSR Markers
- 林业科学, 2017, 53(10): 80-89.
- Scientia Silvae Sinicae, 2017, 53(10): 80-89.
- DOI: 10.11707/j.1001-7488.20171009
-
文章历史
- 收稿日期:2017-03-14
- 修回日期:2017-07-20
-
作者相关文章
2. 山东省林业科学研究院 山东省林木遗传改良重点实验室 济南 250014;
3. 山东费县大青山林场 费县 273402;
4. 胜利油田胜大集团 东营 257083
2. Shandong Provincial Key Laboratory of Forest Tree Genetic Improvement Shandong Academy of Forestry Jinan 250014;
3. Daqingshan Forest Farm, Feixian, Shandong Feixian 273402;
4. Shengli Oilfield Shengda Group, Shandong Dongying 257083
刺槐(Robinia pseudoacacia)为豆科(Leguminosae)刺槐属(Robinia)阔叶大乔木(2n=22),原产美国,速生、抗旱、耐瘠薄、耐轻度盐碱,适宜多种土壤和气候条件,具有很高的观赏、材用、生态防护、蜜源和饲用价值。1601年被引入法国,至18世纪遍布欧洲各国(Keresztesi, 1980);1897年由德国人引入中国青岛(陈一山等,2005),这是据文献记载最早引入中国的刺槐遗传资源。此后的100年里,又有来自美国、朝鲜等刺槐遗传资源引入山东省(顾万春等,1991),部分优树无性系现在收集保存在山东省费县大青山林场刺槐种质资源库。山东在中国引种刺槐历史最早,栽培也很广泛,但是各地栽培的刺槐来源记载不清,遗传关系不明,遗传多样性状况信息甚少,急需开展刺槐遗传多样性评价,为刺槐新品种选育或者遗传改良奠定基础。
植物遗传多样性的研究方法主要有形态学标记、细胞学标记、生理生化标记以及分子标记4大类,它们分别从宏观和微观角度,以及个体、细胞和分子3个水平为植物的遗传多样性研究提供有价值的信息。前人在刺槐形态性状(Keresztesi,1980;李善文等,1997)、蛋白(Surles et al., 1989;Major et al., 1998;杨敏生等,2004)和分子(Major et al., 1998;Bindiya et al., 2003;韩宏伟,2007;孙芳等,2009;王东升等,2012)3个不同水平上分别做了相关研究,发现刺槐变异类型很多,遗传多样性丰富。相对于显性标记,SSR是共显性标记,具有多态性高、稳定性高、分布广泛等优点,被视为是检测植物遗传多样性和遗传分化的最有效标记(Ellegren,2004),在国内被广泛应用于植物遗传多样性评价或品种鉴定(冯锦霞等,2011;李美芹等,2016;Sun et al., 2016)。SSR分子标记也已经被应用于红花刺槐(R. pseudoacacia var. decaisneana)和香花槐(R. pseudoacacia ‘Idaho’)的品种鉴定(方志达,2006)、航天诱变刺槐群体遗传多样性分析(袁存权等,2010)。赵克奇等(2014)进行了刺槐EST-SSR标记PCR反应体系的优化,为以后应用于刺槐遗传多样性评价奠定了很好的基础。因此,应用SSR分子标记分析山东省刺槐无性系遗传多样性和构建指纹图谱,有助于了解我国刺槐多样性分布,鉴别无性系和制定杂交育种策略等。
本研究采用SSR分子标记对来自胶东半岛(青岛、威海、烟台)、鲁南(临沂、日照)、鲁中(济南、泰安、莱芜、淄博、潍坊)、鲁西南(济宁、菏泽、枣庄)和鲁西北(德州、滨州、聊城、东营)5个不同刺槐栽培区的49份刺槐无性系进行遗传多样性评价和指纹图谱构建,以期为刺槐新品种选育、遗传改良和品种鉴定提供可靠的依据,为中国其他省市刺槐遗传资源保存、评价与利用提供参考。
1 材料和方法 1.1 试验材料和采样地点试验材料为1973—2014年山东省不同地市从刺槐实生群体中筛选出的49株优树(表 1),通过嫁接保存到山东省禹城市刺槐属植物种质资源库和山东省费县大青山林场刺槐种子园。从以上2个地点采集幼嫩叶片,硅胶干燥,备用。
|
|
所有无性系的叶片DNA均采用英芮诚生化科技(上海)有限公司植物组织基因组DNA提取试剂盒(磁珠法)(货号PTED-6030)按照说明书进行提取,并采用多功能酶标仪(Spectra Max i3,上海)检测所提DNA浓度和OD260/OD280值,检测结果在1.6~1.9之间的-20 ℃保存备用。
根据文献收集到的刺槐SSR引物,选取9对用于本研究(赵克奇等,2014;Lian et al., 2004)。引物由北京市睿博兴科生物技术有限公司合成,纯化方式PAGE。引物的编号、重复单元、引物序列、片段长度、退火温度以及所用荧光见表 2。
|
|
荧光引物PCR扩增体系:2×Taq PCR MasterMix由北京市睿博兴科生物技术有限公司生产。PCR选用10 μL体系:DNA(20 ng·μL-1)0.5 μL,灭菌去离子水4.3 μL,2×Taq PCR MasterMix 5 μL,正反引物各0.1 μL。
荧光引物PCR扩增程序采用Touchdown模式:95 ℃预变性5 min;95 ℃变性30 s,57 ℃退火30 s(每个循环降低0.5 ℃),72 ℃延伸40 s,14个循环;95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸40 s,20个循环;72 ℃延伸10 min。
1.3 毛细管电泳上机检测用毛细管电泳仪ABI 3730XL检测PCR产物等位基因数目。取PCR产物各0.3 μL、分子量内标(生产商ABI)0.5 μL和去离子甲酰胺(生产商ABI)9.5 μL混合加入PCR板,95 ℃变性5 min,4 ℃冷却后离心,1×Buffer缓冲液上机检测。基因分析仪生产厂家是ABI公司,型号3730xl DNA analyzer,所用参数如下:工作电压15.0 kV,进样电压1.6 kV,喷射持续时间15 s,稳压电流30.0 μA。
1.4 数据分析采用Genemarker 2.2.0软件进行数据整理分析;用GenALEx软件(Peakall et al., 2012)计算平均等位基因数Na、有效等位基因数Ne和Shannon信息指数I;用CERVUS version 3.0(Kalinowski et al., 2007)计算多态性信息量PIC。
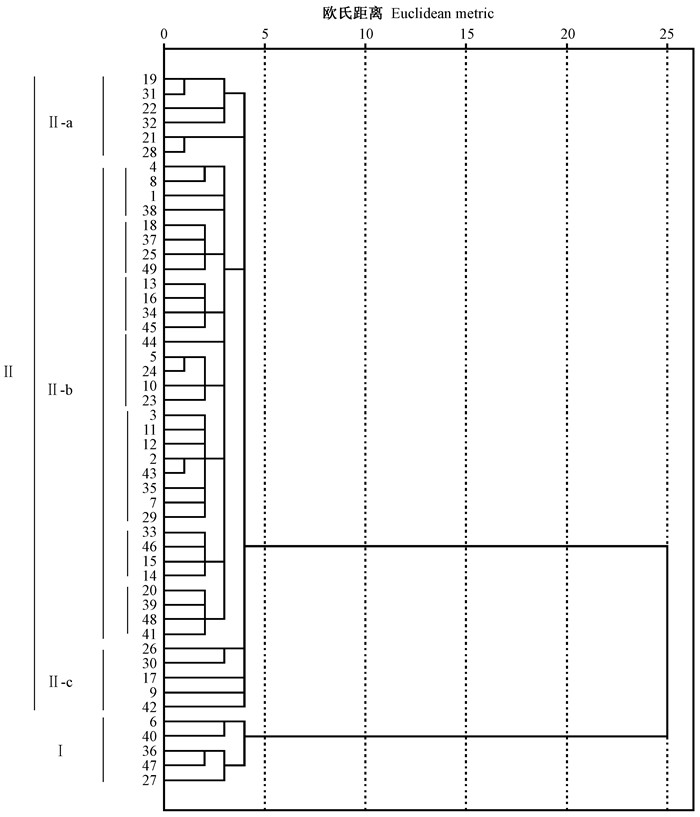
使用组间平均数联结法进行聚类分析,得到山东省49份无性系的UPGMA亲缘关系图。
1.5 指纹图谱构建与无性系鉴定根据每对SSR引物的带型结果,组合形成每个无性系的SSR指纹图谱,根据指纹图谱的差异,再区分无性系。
2 结果与分析 2.1 刺槐遗传多样性分析9个SSR位点中有1个在个别样品中得不到PCR扩增产物(GenAlEx软件不识别),故采用8个位点进行计算。在49个样本中共扩增出51个等位基因,每个位点的等位基因数为2~15个,平均每个位点扩增出6.375个等位基因,最多的rops16有15个等位基因,Rply109次之,有10个等位基因,最少的是Rply31,只有2个等位基因(图 1)。不同位点的PIC值变化范围很大,在0.092~0.879之间,平均PIC值为0.509 8(表 3)。根据Botstein(1980)的理论,有1个标记为低度多态位点(PIC < 0.25),3个标记为中度多态位点(0.25 < PIC < 0.5), 其他4个为高度多态位点(PIC>0.5)。以上遗传多样性参数表明山东省49份刺槐无性系具有较高的遗传多样性。
|
图 1 部分刺槐无性系在Rply31位点的等位基因变异 Figure 1 Allelic phenotype of Robinia pseudoacacia clones at Rply31 locus |
|
|
使用组间平均数联结法对山东省49份刺槐无性系进行聚类分析,得到的亲缘关系如图 2所示。来自临沂的4号(‘鲁刺8’)和8号(‘鲁刺13’)、来自青岛的14号(‘鲁刺40’)和15号(‘鲁刺42’)以及来自日照的6号(‘鲁刺10’)和40号(‘鲁刺86’)亲缘关系最近,最先聚在一起,说明它们遗传基础相近,可能来自同一种源。全部无性系聚为2大类:第Ⅰ类共有5份,包含了沿海地区青岛1份(36号)、日照3份(6、40和27号)和潍坊1份(47号);第Ⅱ类共有44份,占89.80%,包含的无性系非常丰富,涵盖的区域广。第Ⅱ类又可以分为3个亚类:Ⅱ-a共有6份,分别是鲁西南2份(19号和21号)、鲁南2份(32号和28号)和鲁中2份(31号和22号);Ⅱ-b共有33份,包含胶东半岛13份(烟台的3号、威海的38号和青岛的1、37、13、34、45、44、12、2、35、15、14号)、鲁西北3份(东营的24和23号以及德州的46号)、鲁南9份(日照的25号和临沂的4、8、18、49、5、7、33、39号)、鲁西南4份(菏泽的29号、济宁的41号和枣庄的16、43号)和鲁中4份(10、11、20和48号);Ⅱ-c共有5份,包含鲁南2份(26号和9号)、鲁西南2份(17号和42号)和青岛1份(30号)。由上可以看出这49份无性系聚类结果与地理区域并不完全相符,无性系分组与现有栽培区没有明显的规律,说明来自相同地区的刺槐无性系之间遗传差异较大。这与王东升等(2012)使用AFLP标记分析得出的结论“山东省刺槐无性系的聚类与地理位置不完全相关”一致。
利用毛细管电泳检测荧光PCR产物,共获得49个无性系9个SSR位点的DNA指纹图谱(表 4)。所有无性系都不具有特征谱带,无法仅用1对引物将某个无性系与其他无性系区分开,因此需要利用引物组合进行区分。所使用的9对引物中,其中2对Rply109和rops16除了区分不开‘鲁刺62’和‘鲁刺90’、‘鲁刺57’和‘鲁刺103’这4份无性系外,可以区分开其他45份无性系,鉴定率达到91.84%,而其他7对引物,也不能将以上4份无性系区分开,因此后续研究需要开发更有效的SSR引物或采用更多的引物对。
|
|
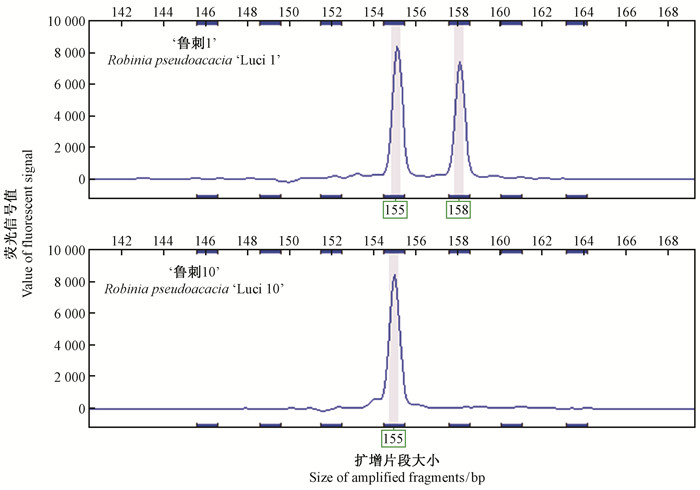
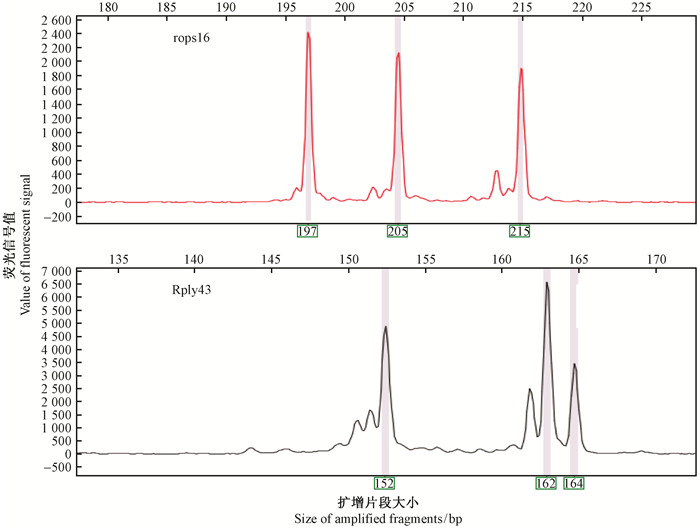
另外,本研究使用9对SSR引物对49份无性系进行分析时发现‘鲁刺2’、‘鲁刺10’、‘鲁刺14’、‘鲁刺28’、‘鲁刺36’、‘鲁刺38’、‘鲁刺40’、‘鲁刺47’、‘鲁刺57’、‘鲁刺61’、‘鲁刺62’、‘鲁刺68’、‘鲁刺84’、‘鲁刺87’、‘鲁刺90’、‘鲁刺102’、‘鲁刺103’、‘鲁刺155’、‘胶29’、‘鲁刺229’、‘壮美青山’和‘青山纽姿’共22份无性系在1个位点有3个等位基因,尤其是其中的‘鲁刺28’(图 3)、‘鲁刺40’和‘鲁刺87’这3份无性系在2个位点均得到3个等位基因(表 4),表明这22份无性系可能是自然变异的多倍体。
|
图 3 ‘鲁刺28’在rops16和Rply43位点的等位基因变异 Figure 3 Allelic phenotype of Robinia pseudoacacia 'Luci 28' at rops16 and Rply31 locus |
每个位点上等位基因的数目是最直接度量遗传多样性丰富度的参数之一(徐刚标,2009)。袁存权等(2010)利用筛选到的8对多态性好、条带清晰的SSR引物对刺槐航天诱变和地面对照共计48个植株组成的群体进行SSR分析,平均每个位点扩增出3.125个等位基因。本研究也是利用8对SSR引物对49个刺槐样本分析得到了每个位点上等位基因的数目2~15个,平均数目是6.375个,是袁存权等(2010)所得的等位基因数目2.04倍。分析原因可能有2个:一是所用的引物不完全一样,引物的多态性不一样;二是所用刺槐材料不同,袁存权等(2010)所用材料是来自河南林场的2 000粒刺槐种子,其中1 000粒进行航天诱变,另1 000粒作对照,播种育苗后共取48个植株进行SSR检测,各材料亲缘关系较近。另外洪丕征(2011)利用9对AFLP引物得到所有位点的等位基因数目平均为1.551 4。本研究得到的等位基因数目明显高于洪丕征(2011)的研究结果,主要是由所用的分子标记不同和所分析的刺槐材料不同导致的。
据Keresztesi(1980)记载,刺槐在美国的天然分布区分为2部分:东部分布区从宾夕法尼亚州中部延伸到亚拉巴马和佐治亚州北部的阿帕拉契安山脉,其中包括西弗吉尼亚州的一部分、弗吉尼亚、马里兰德、肯塔基、田纳西和南、北卡罗来纳州,在佐治亚州中部的南俄亥俄和印第安纳西南部也有刺槐分布;西部分布区包括密苏里南部的奥扎克高原,阿尔堪萨斯与东奥克拉荷马的北部和东部,在伊利诺斯南部和印第安纳西南部也有零星分布。本研究发现49份无性系聚为2大类(图 2),推测这2大类无性系有可能分别来自2个不同的区域,即美国的东部分布区和西部分布区。
在构建指纹图谱时发现使用9对引物区分不开‘鲁刺62’和‘鲁刺90’、‘鲁刺57’和‘鲁刺103’这4份无性系,后来作者所在实验室又利用新开发的9对引物依然区分不开这4份无性系,推测它们可能是同物异名。查看亲缘关系图(图 2)发现,这4份无性系均在第2大类中的a类出现,且‘鲁刺62’(21号)和‘鲁刺90’(28号)、‘鲁刺57’(19号)和‘鲁刺103’(31号)亲缘关系最近,分别最先聚在一起。查阅这4份无性系档案材料,发现‘鲁刺62’来自枣庄滕县,‘鲁刺90’来自临沂费县;‘鲁刺57’来自菏泽曹县,‘鲁刺103’来自淄博沂源县,是山东省不同地市在引种过程中从种子实生苗中选育出的速生抗逆优株。这一证据排除了不同地市间互相引种造成的“同物异名”。具体是否是同物异名,后期应该加大引物使用对数,进一步做SSR检测,并结合表型性状才能下定论。但是不论它们是否是同物异名,鉴于亲缘关系太近,在选择杂交亲本时最好避免同时用作父母本。
韩国辉(2012)在利用EST-SSR对柑橘(Citrus reticulata)多倍体进行遗传分析后,认为SSR分子标记可以作为不同倍性植株染色体倍性确认的一个参考;贾会霞等(2015)在对24份杨树无性系进行SSR指纹图谱构建时发现,5份无性系均扩增出3个不同等位基因,其余19份只出现1个或2个等位基因,FCM检测结果证实这5份无性系均为三倍体,因此认为SSR标记能够准确地反映植物的倍性。本研究检测到‘鲁刺2’、‘鲁刺10’等22份无性系在1个位点或者2个位点有3个等位基因,说明这些无性系可能是多倍体。北京林业大学1997年从韩国引入我国的饲料型四倍体刺槐K1-K5,是由人工诱导二倍体刺槐体细胞加倍而育成。目前国内外尚没有其他发现或者培育四倍体刺槐的报道。本实验室前期对这5个四倍体刺槐无性系K1-K5也做了上述9对引物的SSR分析,发现4个无性系K1-K4只在rops16位点出现3个等位基因,K5除在Rply43位点出现缺失外,其余8个位点各出现1个或者2个等位基因。后来本实验室又利用新开发的9对EST-SSR引物进一步验证,发现K5在RP7和RP13两个位点分别有3个和4个等位基因。本研究从分子水平验证了K1-K5是多倍体。同时韩国人对K1-K5做的核型分析结果从染色体水平验证了这5个无性系确实是多倍体(Kim,1973)。
本研究检测到49份无性系中‘鲁刺2’、‘鲁刺10’等共22份可能是发生自然变异的多倍体。随后经过查阅大青山林场提供的林木种质资源共性描述表,发现除了‘青山纽姿’外,其余21份无性系均具有典型的多倍体植株形态学特点,如高产(速生)、优质和抗逆。具体确认这22个无性系是几倍体,需要进一步做核型分析,观察染色体组成,才能下定论。
4 结论山东省刺槐具有较高的遗传多样性,无性系分组与现有栽培区没有明显的规律;引物Rply109和rops16对49份无性系的分子鉴定率为91.84%,可作为指纹图谱构建、分子鉴定的高效分子标记;利用SSR标记构建的指纹图谱可为刺槐遗传资源管理、品种鉴定和知识产权保护奠定坚实的基础,为刺槐的引种和遗传育种选择亲本提供科学的理论依据。
陈一山, 王凤喜, 董平, 等. 2005. 刺槐观赏种的引种与选育//王豁然, 江泽平, 李延峻, 等. 格局在变化——树木引种与植物地理. 北京: 中国林业出版社, 119-121. (Chen Y S, Wang F X, Dong P, et al. 2005. Introduction and selection of Robinia pseudoacacia L. for landscape planting//Wang H R, Jiang Z P, Li Y J, et al. Changing patterns-Tree introduction and phytogeography. Beijing:China Forestry Publishing House, 119-121. ) |
方志达. 2006. 刺槐无性系引种的初步研究. 保定: 河北农业大学硕士学位论文. (Fang Z D. 2006. Primary studies on introduction of black locust clone. Baoding:MS thesis of Agricultural University of Hebei. http://d.wanfangdata.com.cn/Thesis/Y933694) |
冯锦霞, 张川红, 郑勇奇, 等. 2011. 利用荧光SSR标记鉴别杨树品种[J]. 林业科学, 47(6): 167-174. (Feng J X, Zhang C H, Zheng Y Q, et al. 2011. Identification of poplar varieties by SSR markers using capillary electrophoresis with fluorescence detection[J]. Scientia Silvae Sinicae, 47(6): 167-174. DOI:10.11707/j.1001-7488.20110625 [in Chinese]) |
顾万春, 王金元, 张英脱, 等. 1991. 刺槐次生种源遗传差异及其选择评价//涂忠虞, 黄敏仁. 阔叶树遗传改良——"七·五"国家科技攻关主要速生丰产树种良种选育文集. 北京: 科学技术文献出版社, 231-237. (Gu W C, Wang J Y, Zhang Y T, et al. 1991. Genetic differences and selection evaluation of secondary provenance of Robinia pseudoacacia//Tu Z Y, Huang M R. Genetic improvement on broadleaf trees-Sympsium on selection and breeding of main fast-growing species with high yield in the "seventh five-year" National Technology Research and Development Program. Beijing:Scientific and Technical Documents Publishing House, 231-237. ) |
韩国辉. 2012. 基于EST-SSR、Genomic-SSR和SCoT标记的柑橘连锁图谱构建及杂种和多倍体遗传分析. 重庆: 西南大学博士学位论文. (Han G H. 2012. Construction of molecular linkage map and genetic analysis of hybrids and polyploidy of Citrus based on EST-SSR, Genomic-SSR and SCoT markers. Chongqing:PhD thesis of Southwest University. http://d.wanfangdata.com.cn/Thesis/Y2149423) |
韩宏伟. 2007. 中国刺槐遗传多样性和抗寒性地理变异规律的研究. 保定: 河北农业大学硕士学位论文. (Han H W. 2007. Genetic diversity and geographic variation of cold resistance of Robinia pseudoacacia populations from China. Baoding:MS thesis of Agricultural University of Hebei. http://cdmd.cnki.com.cn/Article/CDMD-11920-2007163333.htm) |
洪丕征. 2011. 刺槐耐盐优良无性系初步选育及AFLP遗传多样性分析. 泰安: 山东农业大学硕士学位论文. (Hong P Z. 2011. The preliminary selection of the salt-tolerant superior clones of Robinia pseudoacacia and AFLP analysis of genetic diversity. Tai'an:MS thesis of Shandong Agricultural University. http://cdmd.cnki.com.cn/article/cdmd-10434-1011098135.htm) |
贾会霞, 姬慧娟, 胡建军, 等. 2015. 杨树新品种的SSR指纹图谱构建和倍性检测[J]. 林业科学, 51(2): 69-79. (Jia H X, Ji H J, Hu J J, et al. 2015. Fingerprints of SSR markers and ploidy detection for new Populus varieties[J]. Scientia Silvae Sinicae, 51(2): 69-79. [in Chinese]) |
李美芹, 潘叶羽, 钱萍仙, 等. 2016. 杜鹃花EST-SSR标记的开发及遗传多样性分析[J]. 植物生理学报, 52(3): 356-364. (Li M Q, Pan Y Y, Qian P X, et al. 2016. Development of EST-SSR primers for azalea and genetic analysis of cultivars[J]. Plant Physiology Journal, 52(3): 356-364. [in Chinese]) |
李善文, 范泽成, 李安礼. 1997. 刺槐无性系生根性状的遗传变异分析[J]. 山东林业科技, (增): 29-32. (Li S W, Fan Z C, Li A L. 1997. Genetic variation analysis of black locust clones by rooting character[J]. Journal of Shandong Forestry Science and Technology, (supplementary issue): 29-32. [in Chinese]) |
孙芳, 杨敏生, 张军, 等. 2009. 刺槐不同居群遗传多样性的ISSR分析[J]. 植物遗传资源学报, 10(1): 91-96. (Sun F, Yang M S, Zhang J, et al. 2009. ISSR analysis of genetic diversity of Robinia pseudoacacia populations[J]. Journal of Plant Genetic Resources, 10(1): 91-96. [in Chinese]) |
王东升, 周继磊, 解荷锋, 等. 2012. 刺槐无性系遗传多样性的AFLP分析[J]. 西南林业大学学报, 32(6): 25-29. (Wang D S, Zhou J L, Xie H F, et al. 2012. AFLP genetic diversity analysis of Robinia pseudoacacia clones[J]. Journal of Southwest Forestry University, 32(6): 25-29. [in Chinese]) |
徐刚标. 2009. 植物群体遗传学[M]. 北京: 科学出版社: 193-200. (Xu G B. 2009. Plant population genetics[M]. Beijing: Science Press: 193-200. [in Chinese]) |
杨敏生, HertelH, SchneckV. 2004. 欧洲中部刺槐种源群体等位酶变异研究[J]. 遗传学报, 31(12): 1439-1447. (Yang M S, Hertel H, Schneck V. 2004. Allozyme variability of provenance populations of Robinia pseudoacacia from Middle Europe[J]. Acta Genetica Sinica, 31(12): 1439-1447. [in Chinese]) |
袁存权, 李云, 路超, 等. 2010. 刺槐种子航天诱变生物学效应研究[J]. 核农学报, 24(6): 1141-1147. (Yuan C Q, Li Y, Lu C, et al. 2010. Biological effects of space-induced mutation on Robinia pseudoacacia L[J]. Acta Agriculturae Nucleatae Sinica, 24(6): 1141-1147. DOI:10.11869/hnxb.2010.06.1141 [in Chinese]) |
赵克奇, 董黎, 王少明, 等. 2014. 刺槐EST-SSR标记PCR反应体系的优化[J]. 中国农学通报, 30(22): 45-52. (Zhao K Q, Dong L, Wang S M, et al. 2014. The optimization of EST-SSR PCR reaction system for Robinia pseudoacacia L[J]. Chinese Agricultural Science Bulletin, 30(22): 45-52. DOI:10.11924/j.issn.1000-6850.2014-0325 [in Chinese]) |
(Bindiya K, Kanwar K. 2003. Random amplified polymorphic DNA (RAPDs) markers for genetic analysis in micro-propagated plants of Robinia pseudoacacia L[J]. Euphytica, 132: 41-47. DOI:10.1023/A:1024623517374)
|
(Botstein D. 1980. A theory of modular evolution for bacteriophages[J]. Annals of the New York Academy of Sciences, 354(1): 484-491.)
|
(Ellegren H. 2004. Microsatellites:simple sequences with complex evolution[J]. Nature Reviews Genetics, 5(6): 435-445. DOI:10.1038/nrg1348)
|
(Kalinowski S T, Taper M L, Marshall T C. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J]. Mol Ecol, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x)
|
(Keresztesi B. 1980. The black locust[J]. Unasylva, 32(127): 23-33.)
|
(Kim C S. 1973. Studies on the colchitetraploids of Robinia pseudoacacia L[J]. The Research Report of the Institute of Forest Genetics, 12: 1-108.)
|
(Lian C L, Ryuya Oishi, Naoya Miyashita. 2004. High somatic instability of a microsatellite locus in a clonal tree, Robinia pseudoacacia[J]. Theor Appl Genet, 108: 836-841. DOI:10.1007/s00122-003-1500-0)
|
(Major A, Malvolti M E, Cannata F. 1998. Comparison of isozyme and RAPD variability of black locust (Robinia pseudoacacia) clones selected for silvicultural objectives[J]. Genet & Breed, 52: 49-62.)
|
(Peakall R, Smouse P E. 2012. GenAlEx6.5[J]. Bioinformatics, 28: 2537-2539. DOI:10.1093/bioinformatics/bts460)
|
(Sun R X, Lin F R, Huang P, et al. 2016. Moderate genetic diversity and genetic differentiation in the relict tree Liquidambar formosana Hance revealed by genic simple sequence repeat markers[J]. Frontiers in Plant Science, 7: 1411-1421.)
|
(Surles S E, Hamrick J L, Bongarten B C. 1989. Allozyme variation in black locust (Robinia pseudoacacia)[J]. Canadian Journal of Forest Research, 19(19): 471-479.)
|
 2017, Vol. 53
2017, Vol. 53