文章信息
- 孙丽娟, 王晓荣, 倪晓详, 程龙军
- Sun Lijuan, Wang Xiaorong, Ni Xiaoxiang, Cheng Longjun
- 巨桉非生物逆境响应基因EgrNAC1的基因结构和表达分析
- The Structure and Expression of EgrNAC1 Gene Associated with Stress Response in Eucalyptus grandis
- 林业科学, 2017, 53(10): 60-69.
- Scientia Silvae Sinicae, 2017, 53(10): 60-69.
- DOI: 10.11707/j.1001-7488.20171007
-
文章历史
- 收稿日期:2017-01-25
- 修回日期:2017-03-13
-
作者相关文章
植物容易遭受各种环境胁迫危害,仅干旱、盐、高温和低温等非生物逆境因子造成的全球范围内主要作物减产就达到50%以上(Rodriguez et al., 2005)。而且,随全球气候急剧变化,这一现象日趋严重(Lobell et al., 2011)。因此,研究植物应对各种逆境因子的分子生物学机制,通过遗传工程和分子辅助育种手段提高植物对逆境胁迫的抗性,对农林业生产有非常重要的意义。
植物本身为应对各种逆境因子,在长期进化过程中形成了一系列逆境响应机制。植物细胞通过信号传递效应接收逆境信号后,发生一系列基因调控过程,进而产生适应逆境环境的生理和代谢变化,如脯氨酸、甜菜碱等渗透调节物质积累等,以提高对逆境的适应性(Xiong et al., 2001; Shulaev et al., 2008)。
在基因调控阶段,转录因子作为效应基因控制开关,发挥极为重要的作用。重要转录因子(如WRKY,bZIP,MYB,AP2和NAC等)家族都参与了逆境响应调控过程(Agarwal et al., 2010; Cramer et al., 2011)。其中NAC(NAM,ATAF和CUC2蛋白)转录因子家族成员广泛参与植物对不同逆境因子的响应,并在其中发挥非常重要的作用(Nakashima et al., 2012; Shao et al., 2015)。
NAC转录因子基因为植物所特有,其成员众多,是一个庞大的基因家族。在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)和毛果杨(Populus trichocarpa)中分别有117,151和163个成员(Ooka et al., 2003; Hu et al., 2010; Nuruzzaman et al., 2010)。NAC基因最早是从矮牵牛(Petunia hybrida)中分离的NAM(no apical meristem,无顶端分生组织),该基因缺失影响根尖分生组织和叶片发育(Souer et al., 1997)。但近些年来的研究表明,NAC类基因的生物学功能非常广泛,几乎涉及植物生长发育各个方面,包括植物生殖、营养发育、次生生长和激素信号转导以及生物和非生物逆境胁迫响应等(Olsen et al., 2005)。植物NAC基因家族中相当比例的成员参与非生物逆境胁迫响应,如拟南芥中有33个NAC基因表达在盐胁迫下发生非常大的变化(Jiang et al., 2006);水稻中40个NAC基因响应干旱和盐胁迫(Fang et al., 2008);大豆(Glycine max)中也有38个NAC基因参与干旱响应(Le et al., 2011)。因此,植物NAC基因在非生物逆境胁迫中发挥的作用越来越被重视(Shao et al., 2015)。
桉树(Eucalyptus)作为一个重要工业用材树种,其生长过程中往往受低温、缺水和盐胁迫等非生物逆境限制,研究桉树逆境响应相关的重要基因对其分子辅助育种意义重大。本研究在巨桉(Eucalyptus grandis)低温处理的数字表达谱(digital gene expression,DGE)中发现1个受低温诱导的NAC类基因,该基因具有典型的NAC结构域,命名为EgrNAC1。对该基因结构特征和不同非生物逆境条件下的表达变化进行了分析,旨在为桉树抗逆分子辅助育种提供研究基础。
1 材料与方法 1.1 试验材料选取定植3个月、生长一致的巨桉无性系幼苗作为试验材料。所有试验处理均在Snijders微气候控制生长箱(MC1000,荷兰)中进行,培养条件:白天25 ℃ 14 h,夜间22 ℃ 10 h,光照强度150 μmol·m-2s-1,相对湿度70%。相应非生物逆境处理结束后,收集植株叶片迅速冻存于液氮,进行后续RNA提取。
1.2 EgrNAC1编码蛋白结构和启动子分析EgrNAC1(Eucgr.I00058)基因、蛋白序列从巨桉基因组数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Egrandis)下载,利用在线软件Protparam(http://www.expasy.ch/tools)预测EgrNAC1基因编码蛋白分子量和等电点。蛋白质二级结构、跨膜结构域和核定位序列预测分别用PSIPRE(http://bionf.cs.ucl.ac.uk/index.php),Tmhmm2.0(http://www.cbs.dtu.dk/services/TMHMM)和cNLS Mapper(http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)进行。根据Ooka等(2003)对植物中NAC家族的分类,拟南芥不同NAC子组成员各选取2个作为该子组代表,将它们的蛋白序列与EgrNAC1蛋白序列用Clustalx软件进行多序列联配,再用MEGA4.0软件构建1 000个自举重复的进化树(Ooka et al., 2003),以此为依据对EgrNAC1进行分类。EgrNAC1与其他植物中同源蛋白进化树构建,则在NCBI(http://www.ncbi.nlm.nih.gov)上,利用blast程序筛选与序列相似程度较高的蛋白序列,进行多序列联配后,同样用MEGA4.0软件构建进化树。启动子上顺式作用元件分析则是截取EgrNAC1基因起始密码子上游1.5 kb序列,在线利用MatInspector软件(http://www.genomatix.de/cgi-bin//matinspector_prof)进行分析。
1.3 EgrNAC1蛋白亚细胞定位在插入35S∷sGFP的载体pCAMBIA1300中,利用多克隆位点处BamHⅠ和XbaⅡ酶切位点连入去掉终止密码子的EgrNAC1编码序列PCR扩增产物(表 1),构建EgrNAC1∷sGFP融合蛋白表达载体。进行DH5α转化后,抽提质粒,金粉(1 μm,Bio-Rad)包埋,用PDS-1000/He型基因枪轰击洋葱(Allium cepa)表皮细胞,然后用激光共聚焦显微镜扫描成像。
|
|
3个月苗龄巨桉幼苗,4 ℃低温下,采用先后间隔0,24,36,42,46 h依次放入生长箱,作为48,24,12,6,2 h的低温处理,以25 ℃生长条件下的幼苗作为0 h(对照)处理,处理完毕后一起收获叶片(摘取枝条顶芽下面第3-5片完全展开的叶片,下同),提取RNA进行数字表达谱(DGE)测序(诺禾致源)。结果中的差异表达基因(>2倍)用WGCNA计算与EgrNAC1具有共表达关系基因的Pearson系数(cor), 根据cor的大小进行基因排序。
1.5 低温、高温、干旱、高盐、ABA和MeJA处理下的EgrNAC1基因表达分析参考魏晓玲等(2015)的方法,在生长箱中以25 ℃生长条件下幼苗作为对照(CK)。-8,-4,0,4,8 ℃作为低温处理温度,42 ℃作为高温处理温度,处理2 h收获叶片提取RNA备用。4 ℃低温不同时间试验处理方法同1.4,处理结束一起提取叶片RNA备用。高盐处理则将植株置于不同塑料容器(60 L)内,处理组塑料容器内保持植株栽培盆1/3高度的200 mmol·L-1NaCl溶液,对照组则用清水保持同样液面高度;同样采用间隔0,24,48,60,66 h的方法依次放入处理苗作为72,48,24,12,6 h处理。干旱处理用5,3,2,1天不浇水植株作为处理组,正常浇水的作为对照。100 μmol·L-1 ABA(脱落酸)和100 μmol·L-1MeJA(茉莉酸甲酯)处理采用植株叶面喷施的方法,以喷施激素溶液后2,6,24 h植株作为处理组,未喷施植株作为对照组,试验结束后,统一收获叶片,提取RNA。以上试验均为每处理组3株植株,进行3次重复。
1.6 RNA提取、cDNA合成及基因定量表达分析根据王亚红等(2010)的方法提取处理叶片RNA。利用PrimeScript® RT reagent Kit(TaKaRa,大连,中国)试剂盒完成反转录。实时荧光定量RT-PCR试验中采用SYBR-Green染料(Takara,大连,中国)和BIO-RAD CFX96实时PCR系统(Bio-Rad,USA)。以Egr18SrRNA作为内参基因,设计引物进行实时荧光定量RT-PCR,3次重复试验(表 1)。用系统自带的Bio-Rad CFX Manager(Version 1.5.5.34)软件进行结果分析,并用GraphPad (ver 4.0)作图。
2 结果与分析 2.1 EgrNAC1基因编码蛋白结构分析EgrNAC1是4 ℃不同时间(0,2,6,12,24,48 h)处理下,数字表达谱中被强烈诱导表达的基因。巨桉基因组数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Egrandis)中该基因编号为:Eucgr.I00058,数据库中该基因注释为1个编码含NAC结构域的蛋白。该基因全长1 968 bp,含3个外显子、2个内含子。开放阅读框长885 bp,编码蛋白氨基酸残基数量为294个。Protparam软件预测该蛋白的等电点为5.86,分子量为33.46 kDa。
所有NAC基因编码蛋白在N末端都含有1个约150个氨基酸残基组成的NAC结构域,该结构域高度保守,而且不同于目前已知的经典结构域,是由数个α螺旋环绕反向平行的β折叠构成,分为A,B,C,D,E 5个亚结构域,DNA结合结构域就位于其中。而C末端序列高度多样化,为转录激活区(Puranik et al., 2012)。通过蛋白结构域分析发现EgrNAC1的NAC结构域为8-158位氨基酸,包含A,B,C,D,E 5个典型NAC亚结构域,结构域中有2个α螺旋和5个β折叠(图 1a),结构域中含有核定位序列:KALVFYAGKAPKGVKTNWI。TMHMM软件分析表明EgrNAC1蛋白不具有跨膜结构域。不同植物NAC基因编码蛋白的进化树结果表明EgrNAC1与杨树、大豆中同源蛋白亲源关系比较近,氨基酸序列相似程度最高达到67%,而与单子叶植物中水稻亲源关系较远,同水稻中SNAC基因编码蛋白氨基酸序列相似程度仅为49%(图 1b)。
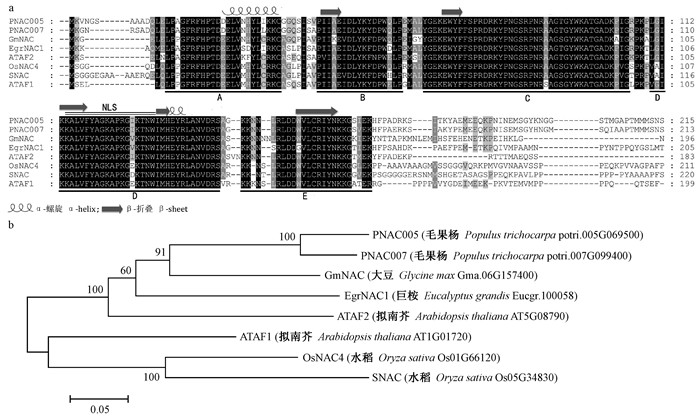
|
图 1 EgrNAC1蛋白与其他植物同源蛋白序列的比对(a)及系统进化树(b) Figure 1 Mutiple alignment (a) and phylogenetic tree (b) of EgrNAC1 in Eucalyptus grandis and its homologus proteins from other plants A, B, C, D和E划线处为NAC结构域的5个亚结构域; NLS:核定位序列,用双划线表示。 The amino acid sequences underlined with A, B, C, D and E are subdomain of NAC domain, and nuclear localization sequence(NLS) is indicated with double lines. |
由于NAC基因家族成员众多,根据Ooka等(2003)对拟南芥和水稻NAC家族的分类标准,NAC蛋白分为2个亚家族Ⅰ和Ⅱ,共18个子组;其中,亚家族Ⅰ含14个子组,亚家族Ⅱ含4个子组。将EgrNAC1与不同子组的拟南芥NAC蛋白序列联配后构建进化树进行聚类分析,结果表明EgrNAC1属于亚家族Ⅰ类ATAF子组(图 2)。Nakashima等(2012)则对不同植物中响应逆境胁迫的NAC类转录因子SNAC(stress-responsive NAC)基于进化关系进行了分类,按照这种分类模式,EgrNAC1属于SNAC-A亚类(Nakashima et al., 2012)。

|
图 2 EgrNAC1在不同NAC子类中的分类 Figure 2 Classification of EgrNAC1 in NAC subgroups |
MatInspector软件对EgrNAC1启动子序列分析结果表明,在其启动子序列上含有大量参与植物生长发育、代谢调控以及逆境响应相关的顺式作用元件。其中,可能与逆境响应相关的有脱落酸响应元件(ABRE)、MYB转录因子结合序列(MBS)、MYC蛋白结合序列(MCS)、WRKY转录因子结合元件(W-BOX)、热击蛋白结合元件(HSE)、NAC蛋白结合序列(NACR)、干旱应答元件(DREB)、乙烯响应元件(ERE)及茉莉酸甲酯响应序列(JARE)等,而且部分元件数目众多(表 2)。
|
|
尽管大部分NAC蛋白都属于转录因子,为核定位蛋白,但仍有部分NAC基因家族成员,其编码产物含有跨膜域,具有膜定位的属性(Kim et al., 2010)。EgrNAC1融合GFP序列构建表达载体,用基因枪轰击洋葱表皮细胞的结果发现,EgrNAC1蛋白仅在细胞核中表达(图 3),与其不具备跨膜结构域的预测相呼应,其应该仅参与核内基因表达调控作用。

|
图 3 EgrNAC1的亚细胞定位 Figure 3 Subcellular localization of EgrNAC1 |
4 ℃不同处理时间(0,2,6,12,24,48 h)数字表达谱中,随处理时间延长,EgrNAC1基因在叶片中的表达受到强烈诱导,0,2,6,12,24,48 h的RPKM(Reads Per Kilobase per Million mapped reads,每百万reads中来自于某基因每千碱基长度的reads数)值分别为10.15,18.89,215.28,1 094.90,1 295.44,1 755.42,48 h处理的植株叶片RPKM值为对照(0 h)的173倍。将差异表达基因(11 458个)与EgrNAC1进行共表达分析,获得与EgrNAC1表达高度相关(Pearson系数cor>0.9)基因有128个,占差异表达基因总数的1.1%。表 3中为相关系数最高的20个基因,其中包括6个转录因子基因,即5个NAC转录因子基因和1个AP2基因。
|
|
4 ℃不同处理时间(0,2,6,12,24,48 h)下EgrNAC1基因实时荧光定量RT-PCR分析结果也表明,该基因随4 ℃处理时间延长,诱导水平不断提高,处理48 h后基因表达量是对照处理(0 h)的163倍(图 4b),与数字表达谱数据相吻合。同时,不同低温(-8,-4,0,4,8 ℃)和高温(42 ℃)处理中,相对于25 ℃正常生长温度(CK),不同低温条件下EgrNAC1基因表达水平都大幅度增强,4 ℃处理和8 ℃处理2 h叶片中EgrNAC1表达水平分别达到了对照(25 ℃)的9.1倍和11.1倍;而高温(42 ℃)处理却能抑制EgrNAC1基因表达,处理2 h后,基因表达水平仅为对照的0.13倍(图 4a)。
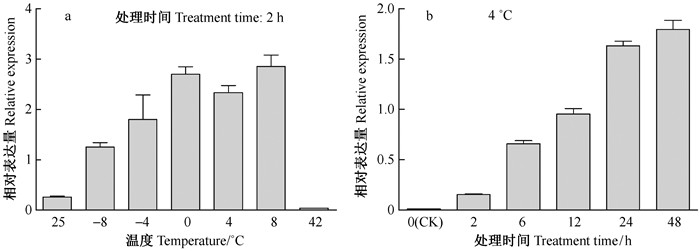
|
图 4 不同温度(a)和4 ℃不同时间(b)处理下EgrNAC1的相对表达差异 Figure 4 Relative expression of EgrNAC1 under different temperatures (a) and time course treatment at 4 ℃ (b) |
干旱处理1天后,EgrNAC1表达水平为未进行干旱处理的7.2倍,而随着处理时间延长,其表达水平又有所下降,处理第5天时,下降为对照的2.68倍,但仍处于诱导状态。盐胁迫(200 mmol·L-1NaCl)对EgrNAC1的诱导作用需要较长时间,处理48 h后,EgrNAC1表达水平才有较为明显的上升,在72 h处理时诱导效应表现强烈。EgrNAC1对100 μmol·L-1脱落酸(ABA)的诱导效应也需要24 h才能体现出来;而100 μmol·L-1茉莉酸甲酯(MeJA)对EgrNAC1的诱导在2 h后就已发生,而且这种诱导作用比较稳定,24 h内没有随时间延长而发生较大幅度变动(图 5)。
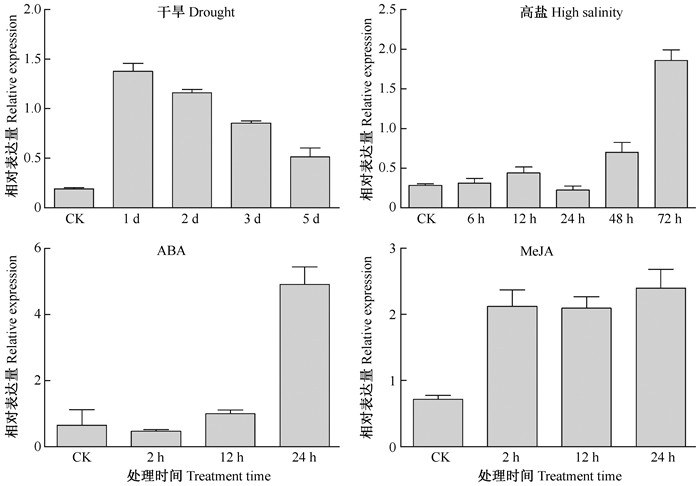
|
图 5 干旱、高盐、ABA和MeJA处理下EgrNAC1的相对表达差异 Figure 5 elative expression of EgrNAC1 under drought, high salinity, ABA and MeJA treatments |
NAC基因家族中,有约20%~25%的成员参与至少1种以上逆境因子响应(Puranik et al., 2012)。而且,基于蛋白质序列的系统进化分类中,参与逆境响应的NAC类转录因子往往同属于一个子类(Fang et al., 2008; Le et al., 2011; Wang et al., 2016)。在Ooka等(2003)的分类系统中EgrNAC1属于Ⅰ类亚家族ATAF子组,同时也属于基于逆境胁迫NAC分类中的SNAC-A亚类。而拟南芥、水稻ATAF子组中基因基本都属于SNAC-A亚类(Ooka et al., 2003; Nakashima et al., 2012)。ATAF子组中的代表基因ATAF1在拟南芥中是逆境胁迫响应中的一个负调控因子,其表达水平被干旱、高盐和机械损伤所诱导,拟南芥超表达ATAF1会提高植株对干旱、盐、ABA和氧化胁迫的敏感性(Lu et al., 2007; Wu et al., 2009)。但是,ATAF1超表达转基因水稻植株则能提高其对盐胁迫的耐受性(Liu et al., 2016)。暗示了该NAC转录因子在调控植物应对逆境胁迫响应中的复杂性。ATAF子组中水稻NAC转录因子基因OsNAC5和OsNAC6超表达转基因植株和对照相比,抗旱、耐低温和耐盐程度则都得到不同程度提升(Takasaki et al., 2010; Song et al., 2011)。由此可见,进化上即使亲缘关系较近的NAC转录因子在不同物种中,参与逆境响应的功能也有很大差异。基于其在逆境胁迫响应中的重要性,对不同植物中逆境相关的NAC转录因子进行深入研究是非常必要的。
EgrNAC1蛋白序列具有典型NAC结构域,结构域中含有DNA结合结构域和核定位序列,这些都是转录因子的明显特征。植物中部分NAC蛋白在C末端还存在跨膜结构域,负责蛋白的质膜和内质网膜锚定作用,它们参与转录调控的同时,还参与环境信号响应和细胞分裂调控作用(Kim et al., 2010; Li et al., 2016)。但无论跨膜域预测结果还是亚细胞定位试验,都表明EgrNAC1不具备跨膜结构域,其应该是一个在细胞核内发挥基因调控作用的转录因子。
干旱、低温和高盐都对EgrNAC1基因表达具有诱导作用。2 h处理条件下,不同低温(-8,-4,0,4,8 ℃)相对于正常温度对EgrNAC1都有明显诱导作用,而且在较高低温(8 ℃)下即产生明显响应,当温度降至0 ℃以下时,诱导水平反而有所下降,暗示EgrNAC1基因表达对低温有比较强的敏感性,这对植物提前感知低温,在生理上提高低温适应能力,降低低温伤害具有重要意义。4 ℃不同时间(0,2,6,12,24,48 h)处理下,随时间延长,EgrNAC1基因诱导水平持续增强;盐胁迫条件下,EgrNAC1基因响应速度则比较慢,处理72 h后,诱导效应才有明显体现;而干旱处理下,EgrNAC1的诱导表现出先强后弱的特点。另外,高温(42 ℃)处理对EgrNAC1有抑制作用。这些都说明EgrNAC1广泛参与了非生物逆境胁迫响应过程,但不同逆境中,其参与调控的途径和方式可能有很大不同。
非生物逆境响应中,ABA和MeJA 2种激素具有重要作用,很多逆境响应相关基因都与它们有密切关系(Turner et al., 2002; Wasternack, 2007; Agarwal et al., 2010),NAC类转录因子也不例外(Chen et al., 2014)。ATAF1,OsNAC5和OsNAC6基因表达都受ABA和MeJA诱导(Lu et al., 2007; Takasaki et al., 2010)。EgrNAC1表达在MeJA处理2 h后就受到稳定诱导,而ABA对基因的诱导则需要24 h。结合其他非生物逆境处理下EgrNAC1的表达情况,说明不同逆境响应中,该基因对这2种激素信号的反应可能也是不同的。
另外,EgrNAC1启动子上的顺式作用元件在一定程度上也说明该基因与非生物逆境胁迫关系密切。DREB上结合的转录因子往往参与干旱和低温逆境响应(Agarwal et al., 2006);植物中相当数量的MYB和MYC蛋白参与干旱、低温和ABA调控基因的表达(Abe et al., 1997),而EgrNAC1启动子上的MBS和MCS元件分别有7个和5个;与WRKY转录因子结合的W-BOX序列也有5个,WRKY在非生物逆境因子和生物逆境因子响应的基因表达调控中都发挥重要作用(Eulgem et al., 2007; Chen et al., 2012);与ERE相结合的乙烯响应蛋白则可能从正、负2个方向调控下游逆境相关基因表达(Fujimoto et al., 2000);ABRE和JARE元件的存在则呼应了EgrNAC1基因在ABA和MeJA处理下的表达情况。另外,其他NAC类转录因子也可能参与EgrNAC1调控,因为在EgrNAC1启动子上也存在NACR元件。
基因共表达分析在一定程度上可以揭示基因间的关系,对研究基因功能和调控机制具有重要意义(Aoki et al., 2007; Mao et al., 2009)。在低温不同处理中,参与叶绿体淀粉降解、LEA蛋白积累、果胶分解、多囊泡体转运等的基因,以及有5个NAC和1个AP2转录因子基因与EgrNAC1基因协同表达关系非常强。这些基因涉及的代谢途径都与非生物逆境胁迫有密切关系,如叶绿体淀粉酶在冷胁迫下可促进叶绿体淀粉降解,提高叶片中可溶性糖含量,增强植物抗寒性(Nagao et al., 2005)。LEA在低温、干旱等逆境下发挥清除氧自由基、稳定膜结构的功能(Shao et al., 2005)。ESCRT(endosomal sorting complex required for transport,内体蛋白分选复合物)则在多囊泡体蛋白转运尤其是泛素化蛋白分拣中有重要功能,与逆境下细胞代谢平衡关系密切(Reyes et al., 2011)。CBS结构域蛋白具有AMP(腺苷一磷酸)激活的蛋白激酶活性,参与盐胁迫的SOS信号转导途径(Kushwaha et al., 2009)。ARM重复蛋白在植物激素信号传递和抗病反应中发挥重要功能(Samuel et al., 2006)。此外,巨桉中多个其他NAC转录因子和AP2转录因子基因与EgrNAC1的共表达关系,暗示了NAC类转录因子在逆境胁迫中参与基因调控的复杂性。这些信息为EgrNAC1基因在低温逆境下参与的基因和代谢途径调控研究提供了线索,为进一步对其逆境条件下在桉树中所发挥功能的研究指明了方向。
4 结论EgrNAC1属于典型NAC类转录因子,基于蛋白序列的聚类分析表明其属于参与逆境胁迫响应的子类成员。低温逆境下EgrNAC1基因共表达分析,不同非生物逆境及ABA、MeJA处理下的表达变化,都暗示EgrNAC1参与了低温、干旱和高盐胁迫响应,并可能与ABA、MeJA信号转导有交叉互作效应。
王亚红, 刘缙, 王玉国. 2010. 高质量提取银杏种仁总RNA的改良方法[J]. 中国农学通报, 26(15): 48-52. (Wang Y H, Liu J, Wang Y G. 2010. An improved method of RNA isolation from seeds of Ginkgo biloba L[J]. Chinese Agricultural Science Bulletin, 26(15): 48-52. [in Chinese]) |
魏晓玲, 程龙军, 窦锦青, 等. 2015. 巨桉EgrDREB2A基因结构及表达特性分析[J]. 林业科学, 51(2): 80-89. (Wei X L, Cheng L J, Dou J Q, et al. 2015. The structure and expression characteristics of EgrDREB2A gene in Eucalyptus grandis[J]. Scientia Silvae Sinicae, 51(2): 80-89. [in Chinese]) |
(Abe H, Yamaguchi-Shinozaki K, Urao T, et al. 1997. Role of Arabidopsis MYC and MYB homologs in drought-and abscisic acid-regulated gene expression[J]. The Plant Cell, 9(10): 1859-1868.)
|
(Agarwal P K, Agarwal P, Reddy M K, et al. 2006. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants[J]. Plant Cell Reports, 25(12): 1263-1274. DOI:10.1007/s00299-006-0204-8)
|
(Agarwal P K, Jha B. 2010. Transcription factors in plants and ABA dependent and independent abiotic stress signalling[J]. Biologia Plantarum, 54(2): 201-212. DOI:10.1007/s10535-010-0038-7)
|
(Aoki K, Ogata Y, Shibata D. 2007. Approaches for extracting practical information from gene co-expression networks in plant biology[J]. Plant and Cell Physiology, 48(3): 381-390. DOI:10.1093/pcp/pcm013)
|
(Chen L, Song Y, Li S, et al. 2012. The role of WRKY transcription factors in plant abiotic stresses[J]. Biochimicaet Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 1819(2): 120-128. DOI:10.1016/j.bbagrm.2011.09.002)
|
(Chen X, Wang Y, Lv B, et al. 2014. The NAC family transcription factor OsNAP confers abiotic stress response through the ABA pathway[J]. Plant and Cell Physiology, 55(3): 604-619. DOI:10.1093/pcp/pct204)
|
(Cheong J J, Do Choi Y. 2003. Methyl jasmonate as a vital substance in plants[J]. TRENDS in Genetics, 19(7): 409-413. DOI:10.1016/S0168-9525(03)00138-0)
|
(Cramer G R, Urano K, Delrot S, et al. 2011. Effects of abiotic stress on plants:a systems biology perspective[J]. BMC Plant Biology, 11(1): 1-14. DOI:10.1186/1471-2229-11-1)
|
(Eulgem T, Somssich I E. 2007. Networks of WRKY transcription factors in defense signaling[J]. Current Opinion in Plant Biology, 10(4): 366-371. DOI:10.1016/j.pbi.2007.04.020)
|
(Fang Y, You J, Xie K, et al. 2008. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice[J]. Molecular Genetics and Genomics, 280(6): 547-563. DOI:10.1007/s00438-008-0386-6)
|
(Fujimoto S Y, Ohta M, Usui A, et al. 2000. Arabidopsis ethylene-responsive element binding factors act as transcriptional activators or repressors of GCC box-mediated gene expression[J]. The Plant Cell, 12(3): 393-404. DOI:10.1105/tpc.12.3.393)
|
(Hu R, Qi G, Kong Y, et al. 2010. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa[J]. BMC Plant Biology, 10(1): 1-23. DOI:10.1186/1471-2229-10-1)
|
(Jiang Y, Deyholos M K. 2006. Comprehensive transcriptional profiling of NaCl-stressed Arabidopsis roots reveals novel classes of responsive genes[J]. BMC Plant Biology, 6(1): 1-20. DOI:10.1186/1471-2229-6-1)
|
(Kim S G, Lee S, Seo P J, et al. 2010. Genome-scale screening and molecular characterization of membrane-bound transcription factors in Arabidopsis and rice[J]. Genomics, 95(1): 56-65. DOI:10.1016/j.ygeno.2009.09.003)
|
(Kushwaha H R, Singh A K, Sopory S K, et al. 2009. Genome wide expression analysis of CBS domain containing proteins in Arabidopsis thaliana (L.) Heynh and Oryza sativa L. reveals their developmental and stress regulation[J]. BMC Genomics, 10(1): 1-22. DOI:10.1186/1471-2164-10-1)
|
(Le D T, Nishiyama R, Watanabe Y, et al. 2011. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J]. DNA Research, 18(4): 263-276. DOI:10.1093/dnares/dsr015)
|
(Li S, Wang N, Ji D, et al. 2016. Evolutionary and functional analysis of membrane-bound NAC transcription factor genes in soybean[J]. Plant Physiology, 172(3): 1804-1820. DOI:10.1104/pp.16.01132)
|
(Liu Q, Zhao N, Yamaguch-Shinozaki K, et al. 2000. Regulatory role of DREB transcription factors in plant drought, salt and cold tolerance[J]. Chinese Science Bulletin, 45(11): 970-975. DOI:10.1007/BF02884972)
|
(Liu Y, Jie S, Wu Y. 2016. Arabidopsis ATAF1 enhances the tolerance to salt stress and ABA in transgenic rice[J]. Journal of Plant Research, 129(5): 1-8.)
|
(Lobell D B, Schlenker W, Costaroberts J. 2011. Climate trends and global crop production since 1980[J]. Science, 333(6042): 616-620. DOI:10.1126/science.1204531)
|
(Lu P L, Chen N Z, An R, et al. 2007. A novel drought-inducible gene, ATAF1, encodes a NAC family protein that negatively regulates the expression of stress-responsive genes in Arabidopsis[J]. Plant Molecular Biology, 63(2): 289-305.)
|
(Mao L, Van Hemert J L, Dash S, et al. 2009. Arabidopsis gene co-expression network and its functional modules[J]. BMC Bioinformatics, 10(1): 1-24. DOI:10.1186/1471-2105-10-1)
|
(Nagao M, Minami A, Arakawa K, et al. 2005. Rapid degradation of starch in chloroplasts and concomitant accumulation of soluble sugars associated with ABA-induced freezing tolerance in the moss Physcomitrella patens[J]. Journal of Plant Physiology, 162(2): 169-180. DOI:10.1016/j.jplph.2004.06.012)
|
(Nakashima K, Yamaguchi-Shinozaki K. 2005. Molecular studies on stress-responsive gene expression in Arabidopsis and improvement of stress tolerance in crop plants by regulon biotechnology[J]. Japan Agricultural Research Quarterly, 39(4): 221-229. DOI:10.6090/jarq.39.221)
|
(Nakashima K, Takasaki H, Mizoi J, et al. 2012. NAC transcription factors in plant abiotic stress responses[J]. Biochimica et Biophysica Acta, 1819(2): 2260-2290.)
|
(Narusaka Y, Nakashima K, Shinwari Z K, et al. 2003. Interaction between two cis-cting elements, ABRE and DRE, in ABA-dependent expression of Arabidopsis rd29A gene in response to dehydration and high salinity stresses[J]. The Plant Journal, 34(2): 137-148. DOI:10.1046/j.1365-313X.2003.01708.x)
|
(Nuruzzaman M, Manimekalai R, Sharoni A M, et al. 2010. Genome-wide analysis of NAC transcription factor family in rice[J]. Gene, 465(1/2): 30-44.)
|
(OlsenA N, Ernst H A, Leggio L L, et al. 2005. NAC transcription factors:structurally distinct, functionally diverse[J]. Trends in Plant Science, 10(2): 79-87. DOI:10.1016/j.tplants.2004.12.010)
|
(Ooka H, Satoh K, Doi K, et al. 2003. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research, 10(6): 239-247. DOI:10.1093/dnares/10.6.239)
|
(Puranik S, Sahu P P, Srivastava P S, et al. 2012. NAC proteins:regulation and role in stress tolerance[J]. Trends in Plant Science, 17(6): 369-381. DOI:10.1016/j.tplants.2012.02.004)
|
(Reyes F C, Buono R, Otegui M S. 2011. Plant endosomal trafficking pathways[J]. Current Opinion in Plant Biology, 14(6): 666-673. DOI:10.1016/j.pbi.2011.07.009)
|
(Rodríguez M, Canales E, Borras-Hidalgo O. 2005. Molecular aspects of abiotic stress in plants[J]. Biotecnologia Aplicada, 22(1): 1-10.)
|
(Samuel M A, Salt J N, Shiu S H, et al. 2006. Multifunctional ARM repeat domains in plants[J]. International Review of Cytology, 253: 1-26. DOI:10.1016/S0074-7696(06)53001-3)
|
(Shao H, Wang H, Tang X. 2015. NAC transcription factors in plant multiple abiotic stress responses:progress and prospects[J]. Frontiers in Plant Science, 6: 902.)
|
(Shao H B, Liang Z S, Shao M A. 2005. LEA proteins in higher plants:structure, function, gene expression and regulation[J]. Colloids & Surfaces B:Biointerfaces, 45(3/4): 131-135.)
|
(Shulaev V, Cortes D, Miller G, et al. 2008. Metabolomics for plant stress response[J]. Physiologia Plantarum, 132(2): 199-208. DOI:10.1111/j.1399-3054.2007.01025.x)
|
(Song S Y, Chen Y, Chen J, et al. 2011. Physiological mechanisms underlying OsNAC5-dependent tolerance of rice plants to abiotic stress[J]. Planta, 234(2): 331-345. DOI:10.1007/s00425-011-1403-2)
|
(Souer E, van Houwelingen A, Kloos D, et al. 1997. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 85(2): 159-170.)
|
(Takasaki H, Maruyama K, Kidokoro S, et al. 2010. The abiotic stress-responsive NAC-type transcription factor OsNAC5 regulates stress-inducible genes and stress tolerance in rice[J]. Molecular Genetics and Genomics, 284(3): 173-183. DOI:10.1007/s00438-010-0557-0)
|
(Turner J G, Ellis C, Devoto A. 2002. The jasmonate signal pathway[J]. The Plant Cell, 14: S153-S164. DOI:10.1105/tpc.000679)
|
(von Koskull-Dörinring P, Scharf K D, Nover L. 2007. The diversity of plant heat stress transcription factors[J]. Trends in Plant Science, 12(10): 452-457. DOI:10.1016/j.tplants.2007.08.014)
|
(Wang Y X, Liu Z W, Wu Z J, et al. 2016. Transcriptome-wide identification and expression analysis of the NAC gene family in tea plant[Camellia sinensis (L.) O. Kuntze][J]. PLoS One, 11(11): e0166727. DOI:10.1371/journal.pone.0166727)
|
(Wasternack C. 2007. Jasmonates:an update on biosynthesis, signal transduction and action in plant stress response, growth and development[J]. Annals of Botany, 100: 681-697. DOI:10.1093/aob/mcm079)
|
(Wu Y, Deng Z, Lai J, et al. 2009. Dual function of Arabidopsis ATAF1 in abiotic and biotic stress responses[J]. Cell Research, 19(11): 1279-1290. DOI:10.1038/cr.2009.108)
|
(Xiong L, Zhu J K. 2001. Abiotic stress signal transduction in plants:Molecular and genetic perspectives[J]. Physiologia Plantarum, 112(2): 152-166. DOI:10.1034/j.1399-3054.2001.1120202.x)
|
(Yoshida T, Fujita Y, Sayama H, et al. 2010. AREB1, AREB2, and ABF3 are master transcription factors that cooperatively regulate ABRE-dependent ABA signaling involved in drought stress tolerance and require ABA for full activation[J]. The Plant Journal, 61(4): 672-685. DOI:10.1111/tpj.2010.61.issue-4)
|
(Zhou Q Y, Tian A G, Zou H F, et al. 2008. Soybean WRKY-type transcription factor genes, GmWRKY13, GmWRKY21, and GmWRKY54, confer differential tolerance to abiotic stresses in transgenic Arabidopsis plants[J]. Plant Biotechnology Journal, 6(5): 486-503. DOI:10.1111/pbi.2008.6.issue-5)
|
 2017, Vol. 53
2017, Vol. 53

