文章信息
- 贾芳信, 周明兵, 陈荣, 杨海芸, 高培军, 徐川梅
- Jia Fangxin, Zhou Mingbing, Chen Rong, Yang Haiyun, Gao Peijun, Xu Chuanmei
- 4种竹子的核型及其基因组大小
- Karyotype and Genome Size in Four Bamboo Species
- 林业科学, 2016, 52(9): 57-66
- Scientia Silvae Sinicae, 2016, 52(9): 57-66.
- DOI: 10.11707/j.1001-7488.20160907
-
文章历史
- 收稿日期:2015-09-16
- 修回日期:2015-12-04
-
作者相关文章
竹类植物属于禾本科(Gramineae)竹亚科(Bambusoideae),是陆地森林生态系统的重要组成部分,具有良好的生态、经济和社会效益(江泽慧,2012)。竹类植物资源丰富,在中国分布的约有40余属500余种,变种变型100余种(Zhang et al.,2011)。竹类植物繁殖方式比较特殊,主要以营养体繁殖为主,也可进行有性繁殖(开花周期不确定,约30~120年),这种特殊的繁殖方式使得竹类植物既可以通过有性生殖产生各种变异,形成多倍体,也可通过无性生殖将一些多倍体、非整倍体、混倍体及各类染色体结构变异保持下来,并进行大量繁殖(李秀兰等,2001; 林树燕等,2010),因此,目前竹类植物无论是在形态特征上还是在染色体数目及结构上均表现出非常丰富的变异。
在植物进化过程中,其染色体数目、形态及大小会发生一定的改变,称为核型进化,进而引起植物基因组大小改变(Ingo,2007),植物核型进化的总体趋势是由对称向不对称方向发展(陈丙义等,2012),因此可以通过比较不同物种的核型不对称性来确定植物的进化顺序。C值指的是基因组未复制时单倍体细胞核的DNA含量,单位为pg,可以通过流式细胞仪进行测定,C值大小可以直接反映植物基因组的大小(Kumar et al.,2011)。目前,大量植物的基因组大小通过流式细胞仪被测定,并且建立了专门的C-值数据库(Bennett et al.,2005; Bennett et al.,2012)。在竹类植物基因组大小研究中,Gielis等(1997)首次利用流式细胞仪测定了在比利时(Belgium)分布的26个不同类型竹种的DNA含量,Kumar等(2011)利用流式细胞仪测定了在新加坡分布的37个竹种的DNA含量,在中国分布的竹种中,仅毛竹(Phyllostachys edulis)基因组大小被测定(李潞滨等,2008; Gui et al.,2007; Peng et al.,2013),除此之外,大部分竹种(约96%以上)的基因组大小信息还处于未知状态。
根据竹鞭的延伸性质,竹类植物可以分成散生竹种、混生竹种和丛生竹种3种类型,部分竹种的细胞学研究结果表明,散生竹种和混生竹种染色体数目基本为2n=48,丛生竹种染色体数目变化比较大,多数竹种染色体数目为2n=70±2(68,69,70,71,72),另外还有少数竹种染色体数目为2n=64,96,104等(陈瑞阳等,2002; 李秀兰等,1999; 2001)。目前,虽然一些研究者在一些竹种的染色体核型及基因组大小等方面做了一定的研究(陈瑞阳等,2002; Gielis et al.,1997; Kumar et al.,2011),但是这些研究彼此是孤立的,不同竹种染色体核型与其基因组大小之间缺乏直接的对应关系,特别是繁殖方式非常特殊的竹类植物,同一竹种不同个体间都有可能存在极大差异,因此有必要将同一株竹种的染色体核型分析数据与其基因组大小数据结合起来进行分析,来探索不同竹种染色体核型与其基因组大小之间的具体关系。另外,散生竹种、混生竹种及丛生竹种在基因组大小上的表现形式如何,目前也依然是未知状态。为此,本研究选取经济价值较高的散生竹种雷竹(Phyllostachys praecox cv. Prevernalis)、黄杆乌哺鸡竹(P. vivax f. aureocaulis)、混生竹种日本矮竹(Sasa pygmea)和丛生竹种鱼肚腩竹(Bambusa gibboides),采用染色体核型分析技术对这4个竹种的染色体核型进行研究,同时利用流式细胞仪对这4个竹种的基因组大小进行测定。通过该研究,揭示不同竹种染色体核型与其基因组大小之间的关系,从核型进化角度,判断不同竹种的进化顺序,以期为今后竹类植物分子系统学及基因组分析等研究提供一些理论依据及参考。
1 材料与方法 1.1 试验材料竹种DNA含量测定过程中,以水稻(Oryza sativa)(品种为‘日本晴’)作为对照,从浙江农林大学翠竹园和福建华安竹种园采集雷竹、黄杆乌哺鸡竹、日本矮竹及鱼肚楠竹幼嫩的叶片用于DNA含量测定,同时采集当年母竹鞭上抽出的新根根尖作为染色体制备材料。
1.2 试验方法 1.2.1 染色体标本制备及染色将采集的幼嫩的根尖用0.002 mol·L-1的8-羟基喹啉溶液室温条件下预处理5 h,用卡诺固定液固定(甲醇∶乙酸=3∶1)24 h以上,之后参照陈瑞阳等(2002)的方法进行染色体标本制备。相差显微镜下(Olympus BX 51)选取染色体分散较开、形态较好的中期染色体制片,利用Giemsa或 PI染色液进行染色,染色后的制片在荧光显微镜(Olympus BX 60)100倍油镜下拍照,染色体放大倍数为200倍。
1.2.2 染色体核型分析根据染色体形态大小等特征,采用Adobe Photo Shop 7.0图像处理软件对染色体进行剪贴、排列和同源染色体配对分析,并利用Motic Images Advanced 3.2高级图像处理软件进行染色体数据测量。核型分析参照陈瑞阳等(2002)和李懋学等(1985)的方法,核型分类参照Stebbins(1971)的分类标准。核型分析过程中,根据各染色体的相对长度系数和臂比,将竹类植物染色体组分依次分为A组(染色体着丝粒类型为m型,染色体的相对长度系数>1.4)、B组(染色体着丝粒类型为sm型)、C组(染色体着丝粒类型为t,st型)和D组(染色体着丝粒类型为m型,染色体的相对长度系数<1.4)4个组,每组染色体排列顺序按照由短到长的顺序进行排列,并依次命名为1~35。
1.2.3 流式细胞仪样品制备取新鲜幼嫩的尚未展开的竹子叶片,用锋利的刀片切取1段(长度约2 cm左右)置于干净的培养皿中,加入500 μL 细胞核提取液(PARTEC,cystain UV precise P Nuclei Extraction Buffer)于样品上,用双面刀片将样品快速剁碎,静置2 min提取细胞核。2 min后再加入2 mL染色液(PARTEC,cystain UV precise P stain Buffer)进行细胞核染色,4 min后将染色后的样品通过80目的滤膜过滤到3 mL样品管中,上样进行测量,测量过程中将流式细胞仪的电压、阈值等参数调整至最佳状态,样品变异系数均控制在5%以内,每个竹种采集3个不同单株,设置3个不同重复。
以已测序的水稻作为参照,登录C值数据库网站(Plant DNA C-values Database,http://data.kew.org/cvalues/),查出对照水稻2C DNA含量为1.0 pg,根据以下公式推算出不同竹种的2C DNA含量。待测竹种2C DNA含量(pg)=(待测竹种峰值/水稻峰值)×1.0 pg。
2 结果与分析 2.1 不同竹种的核型分析 2.1.1 雷竹染色体核型分析雷竹在分类上属于刚竹属(Phyllostachys),散生竹种,其染色体核型参数见表 1,主要核型特征见表 2。其核型公式为2n=48=40 m+6 sm+2 st,主要由20对中部着丝粒染色体(m)、3对近中部着丝粒染色体(sm)和1对近端部着丝粒染色体(st)组成,染色体相对长度变化范围为2.464%~6.269%,臂比大于2的染色体比例为12.50%,最长染色体与最短染色体之比为2.554,核型不对称系数为60.107,在核型分类属于2B类型(表 2)。图 1A1-A3 分别是雷竹的C-分带图、核型图和核型模式图。
|
|
|
|

|
图 1 雷竹的染色体制片(A1)、核型(A2)及核型模式(A3) Fig.1 Photomicrographs of metaphase chromosomes(A1),karyotypes(A2) and ideograms(A3) of P. praecox cv. Prevernalis |
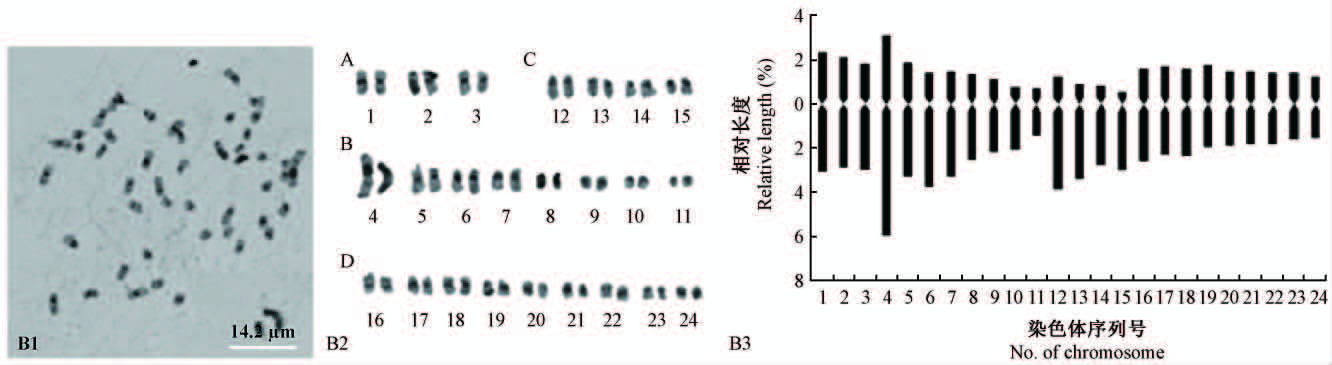
黄杆乌哺鸡竹在分类上属于刚竹属,散生竹种,其染色体核型参数见表 3,主要核型特征见表 2。其核型公式为2n=48=24m+16sm+8st,主要由12对中部着丝粒染色体(m)、8对近中部着丝粒染色体(sm)和4对近端部着丝粒染色体(st)组成,染色体相对长度变化范围为2.185%~9.091%,臂比大于2的染色体比例为29.17%,最长染色体与最短染色体之比为4.161,核型不对称系数为64.49,核型分类属于2C类型(表 2)。图 2B1-B3 B1-B3分别是黄杆乌哺鸡竹的C-分带图、核型图和核型模式图。
|
|
|
图 2 黄秆乌哺鸡竹的染色体制片(B1)、核型(B2)及核型模式(B3) Fig.2 Photomicrographs of metaphase chromosomes( B1),karyotypes( B2) and ideograms( B3) of P. vivax f. aureocaulis |
日本矮竹在分类上属于赤竹属(Sasa),为混生竹种,其染色体核型参数见表 4,主要核型特征见表 2。其核型公式为2n=48=28m+18sm+2st,主要由14对中部着丝粒染色体(m)、9对近中部着丝粒染色体(sm)和1对近端部着丝粒染色体(st)组成,染色体相对长度变化范围为2.080%~7.881%,臂比大于2的染色体比例为20.83%,最长染色体与最短染色体之比为3.789,核型不对称系数为61.505,核型分类属于2B类型(表 2)。图 3C1-C3 分别是日本矮竹的C-分带图、核型图和核型模式图。
|
|

|
图 3 日本矮竹的染色体制片(C1)、核型(C2)及核型模式(C3) Fig.3 Photomicrographs of metaphase chromosomes( C1),karyotypes( C2) and ideograms( C3) of S. pygmea |
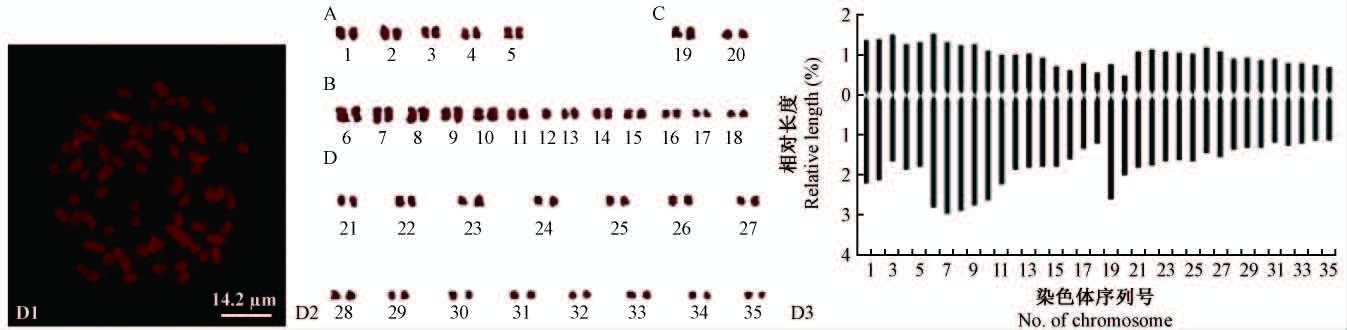
鱼肚腩竹在分类上属于簕竹属(Bambusa)孝顺竹亚属(Subgen. Leleba),属于丛生竹种,其染色体核型参数见表 5,主要核型特征见表 2。其核型公式为2n=69=40m+25sm+4st,主要由40条中部着丝粒染色体(m)、25条近中部着丝粒染色体(sm)和4条近端部着丝粒染色体(st)组成,染色体相对长度变化范围为1.790%~4.382%,臂比大于2的染色体比例为28.57%,最长染色体与最短染色体之比为2.448,核型不对称系数为64.076,核型分类属于2B类型(表 2)。图 4D-图 1-D3分别是鱼肚腩竹的C-分带图、核型图和核型模式图。
|
|
|
图 4 鱼肚腩竹染色体制片(D1)、核型(D2)及核型模式(D3) Fig.4 Photomicrographs of metaphase chromosomes(D1),karyotypes(D2) and ideograms(D3) of B. gibboides |
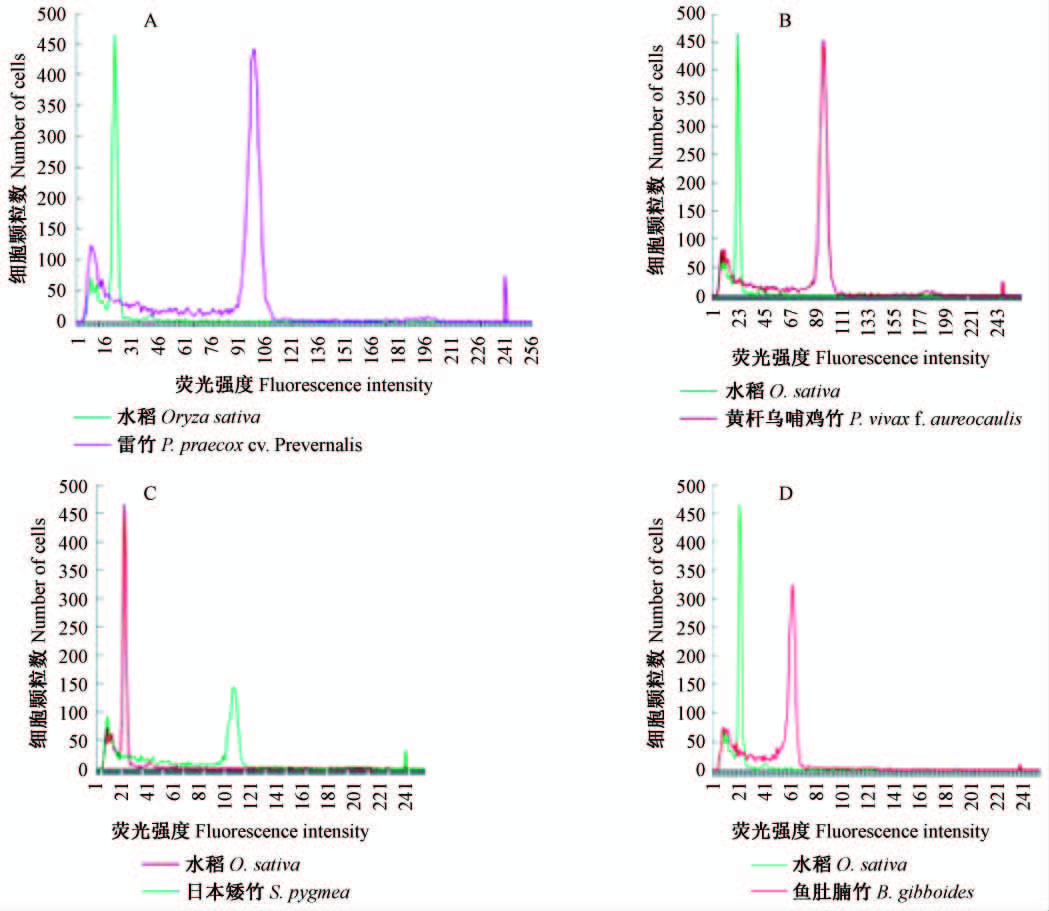
流式细胞仪可以快速精确地测定植物的DNA含量(Sapci et al.,2015)。本研究利用Partec 公司的Cyflow Ploidy Analyser 对不同竹种的DNA含量进行测定,采用DAPI荧光染料对DNA进行特异染色,荧光强度的大小代表所测物种的DNA含量的 多少。试验以已经测序的水稻作为对照,为了确保试验结果的可靠性,还选取了已经测序的毛竹来进行验证,测样结果表明,毛竹基因组大小为4.24 pg,这一结果与毛竹测序结果(Peng et al.,2013)基本吻合。图 5 A-D分别是每个竹种测样结果图,左侧是水稻基因组测样所出的峰值,右侧是不同竹种基因组测样所出的峰值,由图可以看出,水稻与所测竹种的峰值分散较开,没有重叠,而且所出的峰尖而细,说明所测结果的准确性较高。所测4个竹种中(表 6),除鱼肚腩竹体细胞染色体数目为69外,其余3个竹种染色体数目均为2n=48。但是在DNA含量上,日本矮竹基因组最大,其DNA含量为(4.67±0.52)pg;其次为黄秆乌哺鸡竹,DNA含量为(3.93±0.85)pg;雷竹DNA含量比黄秆乌哺鸡竹略小,为(3.71±0.10)pg;鱼肚腩竹基因组最小,仅为(2.92±0.38)pg。所测4个竹种DNA含量变化范围相差约1.6倍。
|
图 5 4 个竹种的DNA 含量测定结果 Fig.5 The DNA amount peak value of four bamboo species |
|
|
自然界中,伴随植物的进化,其染色体核型对称性也会发生相应改变,总体趋势是由对称向不对称方向发展,也即进化程度越高的植物,其染色体核型不对称性程度将增加,通常情况下,染色体长度比、臂比、核型不对称系数等是衡量核型对称与不对称 的主要参数,这些数值越大,植物的进化程度越高(陈瑞阳等,2002; Sousa et al.,2012)。本研究所分析的4个竹种中,黄杆乌哺鸡竹核型不对称系数为64.49,是4个竹种中最大的,后面依次为鱼肚腩竹、日本矮竹和雷竹,核型不对称系数分别为64.08,61.51和60.11。根据核型不对称系数大小,4个竹种的核型不对称性从强到弱依次排序为黄杆乌哺鸡竹>鱼肚腩竹>日本矮竹>雷竹;据此推断,4种竹子的进化程度(从进化到原始顺序)为黄杆乌哺鸡竹>鱼肚腩竹>日本矮竹>雷竹。
核型分析中,为了区分核型的对称和不对称程度,根据染色体长度比和臂比2个主要参数,核型分为12类,从对称到不对称依次为1A,2A,3A,4A,1B,2B,3B,4B,1C,2C,3C,4C(Stebbins,1971)。所分析的4个竹种,臂比大于2的染色体所占的百分比均在1%~50%之间;最长染色体与最短染色体长度之比参数中,雷竹、日本矮竹、鱼肚腩竹这3个竹种均介于2.0~4.0之间,因此这3个竹种在核型分类上属于2B类型,黄杆乌哺鸡竹的最长染色体与最短染色体长度比参数为4.161,大于4.0,因此在核型分类上属于2C类型。
陈瑞阳等(2002)对鱼肚腩竹进行核型分析,鱼肚腩竹有2种染色体核型,核型公式分别为2n=106=52m+18sm+12st(2SAT)+22t和2n=96=52m+16sm+6st+22t。本研究中鱼肚腩竹核型公式为2n=69=40m+25sm+4st,与陈瑞阳等(2002)公布的结果存在较大差异。作者曾根据陈瑞阳等(2002)公布的一些竹种的染色体信息,选择了染色体数目较多的吊丝单竹(Bambusa variostriata)和牛角竹(B. cornigera)(染色体数目均为2n=96,104),同时还选择了染色体数目与本研究中鱼肚腩竹相似的孝顺竹(B. multiplex)(染色体数目为70)、马甲竹(B. tulda)(染色体数目为68),利用流式细胞仪对这4个竹种基因组大小进行测定。结果表明,吊丝单竹、牛角竹、孝顺竹和马甲竹的2C DNA含量分别为4.14,4.19,2.78,2.86 pg(尚未发表),吊丝单竹和牛角竹2C DNA含量远远大于鱼肚腩竹(2.92 pg),鱼肚腩竹、 孝顺竹及马甲竹3个竹种2C DNA含量相差不大。结合以上几个竹种的基因组大小数据可推断,除了陈瑞阳等(2002)公布的2n=96,104这2种染色体数目的染色体核型外,鱼肚腩竹还存在染色体数目为69的第3种染色体核型。陈瑞阳等(2002)认为,丛生竹种分类学种内普遍存在着多倍体跌落现象(染色体在多倍体水平上的非整倍性减少),丛生竹种多倍体跌落形成的机制不是染色体丢失,而是由于着丝粒并合或Robertson并合所致,例如在染色体数为68,69,70,71,72的细胞型中,它们的臂指数(nombre fondamental,N.F.,核型分析中,各类染色体的染色体臂数总和称为臂指数)均为122,即染色体数目发生改变,但是染色体臂指数并没有发生改变,这可能是同一丛生竹种存在多种染色体数目及核型的主要原因。
一般情况下,植物DNA含量与其倍性水平呈正相关,即倍性水平越高,基因组越大,例如Attaleinae属中的一些植物(Gunn et al.,2015)。本研究表明,染色体数目较多的鱼肚腩竹与其余3个染色体数目相对较少的散生竹种和混生竹种间并不遵循这一规律。丛生竹种鱼肚腩竹染色体数目为69,在倍性水平上远远高于以上3个竹种,但是在DNA含量上鱼肚腩竹却是这4个竹种中最小的,这一结果与Gielis等(1997)及Kumar等(2011)测定的在其他一些国家分布的竹种情况类似。染色体数目较多的鱼肚腩竹为什么在基因组大小上反而会小于染色体数目较少的散生竹种和混生竹种呢?为此,对4个竹种的染色体相对长度范围进行了分析,鱼肚腩竹染色体相对长度范围为1.790%~4.382%,即最短染色体长度为1.79 μm,最长染色体长度为4.382 μm,其他3个竹种最小染色体长度均在2.0 μm以上,而在最大染色体长度上,其余3个竹种的长度均在6.269 μm以上,远远大于鱼肚腩竹的最长染色体。据此推断,虽然鱼肚腩竹染色体数目较多,但是在总体体积上较小,而以上3个竹种,虽然染色体数目较少,但是,染色体总体积较大,因此在DNA含量上占了优势,这可能是4个竹种基因组大小与染色体数目不呈正相关的主要原因。
Leitch等(1998)根据植物基因组大小,将植物基因组分为5类:极小基因组(2C≤2.8 pg)、小基因组(2.8 pg <2C≤7.0 pg)、中基因组(7.0 pg <2C≤28.0 pg)、大基因组(28.0 pg <2C≤70.0 pg)和极大基因组(2C> 70.0 pg)。现存植物中,89.8%的植物2C DNA含量小于2.8 pg,8.5%的植物的2C DNA含量介于2.8~7.0 pg之间,仅1.7%的植物2C DNA含量介于7.0~28.0 pg之间,极少植物(少量裸子植物)基因组2C DNA含量介于28.0~70.0 pg之间(Castro et al.,2012)。本研究所分析的4个竹种,2C DNA含量均介于2.8~7.0 pg之间,根据该基因组大小分类标准,雷竹、黄秆乌哺鸡竹、日本矮竹及鱼肚腩竹这4个竹种在基因组类型上均属于小基因组类型,而在现存植物中,这4个竹种基因组属于比较大的,特别是日本矮竹,基因组更大。
4 结论散生竹种雷竹和黄杆乌哺鸡竹、混生竹种日本矮竹及丛生竹种鱼肚腩竹这3类竹种在核型特征及基因组大小方面存在明显差异。在核型方面,雷竹和黄杆乌哺鸡竹的核型不对称系数分别为60.107和64.49,日本矮竹的核型不对称系数为61.505,鱼肚腩竹的核型不对称系数为64.076,因此这4个竹种的进化程度从原始到进化依次为:雷竹、日本矮竹、鱼肚腩竹、黄杆乌哺鸡竹。在基因组大小方面,混生竹种日本矮竹基因组最大,其次为散生竹种黄杆乌哺鸡和雷竹,丛生竹种鱼肚腩竹基因组最小。另外,所研究的 4个竹种中,丛生竹种鱼肚腩竹染色体数目最多,但是其2C DNA含量却是4个竹种中最小的,由此可见,所测竹种基因组大小与其染色体数目并不是成正相关的关系。
| [1] |
陈瑞阳, 李秀兰, 宋文芹. 2002. 中国主要经济植物基因组染色体图谱Ⅳ. 北京: 科学出版社 : 114 -272.
( Chen R Y, Li X L, Song W Q. 2002. Chromosome atlas of major economic plants genome in China Ⅳ. Beijing: Science Press : 114 -272. [in Chinese] ) |
| [2] |
陈丙义, 黄金凤, 高志红, 等. 2012. 6种野生草莓的核型分析. 西北植物学报 , 32 (8) : 1567–1572.
( Chen B Y, Huang J F, Gao Z H, et al.2012. Karyotype analysis of six wild strawberry species. Acta Botanica Boreali-Occidentalia Sinica , 32 (8) : 1567–1572. [in Chinese] ) (  0) 0)
|
| [3] |
李潞滨, 武静宇, 胡陶, 等. 2008. 毛竹基因组大小测定. 植物学通报 , 25 (5) : 574–578.
( Li L B, Wu J Y, Hu T, et al.2008. Estimation of genome size of Moso bamboo (Phyllostachys edulis). Chinese Bulletin of Botany , 25 (5) : 574–578. [in Chinese] ) (  0) 0)
|
| [4] |
李懋学, 陈瑞阳. 1985. 关于植物核型分析的标准化问题. 武汉植物学研究 , 3 (4) : 297–302.
( Li M X, Chen R Y.1985. Problem about standardization of plant karyotype analysis. Journal of Wuhan Botanical Research , 3 (4) : 297–302. [in Chinese] ) (  0) 0)
|
| [5] |
李秀兰, 林汝顺, 冯学琳, 等. 2001. 中国部分丛生竹类染色体数目报道. 植物分类学报 , 39 (5) : 433–442.
( Li X L, Lin R S, Fung X L, et al.2001. Chromosome numbers of some caespitose bamboos native in or introduced to China. Acta Phytotaxonomica Sinica , 39 (5) : 433–442. [in Chinese] ) (  0) 0)
|
| [6] |
李秀兰, 刘松, 宋文芹, 等. 1999. 40种散生竹的染色体数目. 植物分类学报 , 37 (6) : 541–544.
( Li X L, Liu S, Song W Q, et al.1999. Chromosome number of forty species of scattered bamboos. Acta Phytotaxonomica Sinica , 37 (6) : 541–544. [in Chinese] ) (  0) 0)
|
| [7] |
林树燕, 石文文, 缪彬彬, 等. 2010. 竹类植物生殖生物学研究进展. 世界竹藤通讯 , 8 (2) : 1–6.
( Lin S Y, Shi W W, Miu B B, et al.2010. Research advances in reproduction biology of bamboos. World Bamboo and Rattan , 8 (2) : 1–6. [in Chinese] ) (  0) 0)
|
| [8] |
江泽慧. 2012. 植物基因组学研究进展. 林业科学 , 48 (1) : 159–166.
( Jiang Z H.2012. Progress in bamboo genomics research. Scientia Silvae Sinicae , 48 (1) : 159–166. [in Chinese] ) (  0) 0)
|
| [9] |
Bennett M D, Leitch I J.2005. Nuclear DNA amounts in angiosperms:progress,problems and prospects. Ann Bot , 95 (1) : 45–90.
DOI:10.1093/aob/mci003 ( 0) 0)
|
| [10] | Bennett M D,Leitch I J.2012.Plant DNA C-values database (release6.0,Dec.2012).http://data.kew.org/cvalues/. |
| [11] |
Castro M, Castro S, Loureiro J.2012. Genome size variation and incidence of polyploidy in Scrophulariaceae sensu lato from the Iberian Peninsula. Aob Plants (1) : 1–14.
( 0) 0)
|
| [12] |
Gielis J, Valente P, Bridts C, et al.1997. Estimation of DNA content of bamboos using flow cytometry and confocal laser scanning microscopy//Chapman G. The Bamboos.London:Academic Press : 215–223.
( 0) 0)
|
| [13] |
Gui Y J, Wang S, Quan L Y, et al.2007. Genome size and sequence composition of moso bamboo:a comparative study. Sci China C Life Sci , 50 (5) : 700–705.
DOI:10.1007/s11427-007-0081-6 ( 0) 0)
|
| [14] |
Gunn B F, Baudouin L, BeuléT, et al.2015. Ploidy and domestication are associated with genome size variation in palms. American Journal of Botany , 102 (10) : 1625–1633.
DOI:10.3732/ajb.1500164 ( 0) 0)
|
| [15] |
Ingo S.2007. Chromosome evolution. Curr Opin Plant Biol , 10 (2) : 109–115.
DOI:10.1016/j.pbi.2007.01.001 ( 0) 0)
|
| [16] |
Kumar P P, Turner I M, Rao A N, et al.2011. Estimation of nuclear DNA content of various bamboo and rattan species. Plant Biotechnol Rep , 5 : 317–322.
DOI:10.1007/s11816-011-0185-0 ( 0) 0)
|
| [17] |
Leitch I J, Chase M W, Bennett M D.1998. Phylogenetic analysis of DNA C-values provides evidence for a small ancestral genome size in flowering plants. Annals of Botany , 82 : 85–94.
DOI:10.1006/anbo.1998.0783 ( 0) 0)
|
| [18] |
Peng Z H, Lu Y, Li L B, et al.2013. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla). Nature Genetics , 45 (4) : 456–463.
DOI:10.1038/ng.2569 ( 0) 0)
|
| [19] |
Sapci H D, Rewers M, Vural C, et al.2015. Flow cytometric estimation of the nuclear genome size of 22 Echinops (Asteraceae) taxa from Turkey. Turk J Bot , 39 : 580–587.
DOI:10.3906/bot-1406-30 ( 0) 0)
|
| [20] |
Sousa S M, Torres G A, Viccini L F.2012. Karyological studies in Brazilian species of Lippia L. (Verbenaceae).An Acad Bras Cienc , 84 (4) : 1029–1037.
DOI:10.1590/S0001-37652012005000068 ( 0) 0)
|
| [21] |
Stebbins G L.1971. Chromosomal evolution in higher plants. UK London:Edward Arnold Ltd : 85–104.
( 0) 0)
|
| [22] |
Zhang Y J, Ma P F, Li D Z.2011. High-throughput sequencing of six bamboo chloroplast genomes:phylogenetic implications for temperate woody bamboos (Poaceae:Bambusoideae). PLoS One , 6 (5) : e20596.
DOI:10.1371/journal.pone.0020596 ( 0) 0)
|
 2016, Vol. 52
2016, Vol. 52

