文章信息
- 刘文哲, 牛明月, 李秀云, 林二培, 黄华宏, 童再康
- Liu Wenzhe, Niu Mingyue, Li Xiuyun, Lin Erpei, Huang Huahong, Tong Zaikang
- 光皮桦实时荧光定量PCR内参基因的筛选
- The Selection of Reference Genes for Quantitative PCR in Betula luminifera
- 林业科学, 2016, 52(8): 29-37
- Scientia Silvae Sinicae, 2016, 52(8): 29-37.
- DOI: 10.11707/j.1001-7488.20160804
-
文章历史
- 收稿日期:2016-01-29
- 修回日期:2016-04-20
-
作者相关文章
实时荧光定量PCR是在普通PCR技术基础上发展起来的核酸定量技术,因其定量准确、特异性强、灵敏度高及高通量的优点被广泛应用于基因表达分析(Nolan et al., 2006; Schmid et al., 2003; Wong et al., 2005)。然而,由于受到RNA提取质量、逆转录效率等因素的影响,目的基因表达结果的准确性会受到一定程度的影响。为了使试验结果更加准确可靠,稳定表达的内参基因会被用来进行校正和标准化,以消除背景误差(Sinha et al., 2015)。理想的内参基因应该在各种环境条件下、在不同的组织和细胞中都能恒定或相对稳定地表达(Brunner et al., 2004; Udvardi et al., 2008);但是近几年的研究表明,内参基因在不同的试验对象、试验环境下,表达的稳定性差异很大,甚至可能截然相反,盲目地使用一种内参基因会影响试验结果的准确性(Bustin,2002; de Oliveira et al., 2012)。因此,选择何种内参基因对于定量PCR结果是否准确显得尤为重要。
光皮桦(Betula luminifera)系桦木科(Betulaceae)落叶大乔木,为我国特有优良速生用材树种,分布于秦岭、淮河流域以南各省区。光皮桦材质细腻坚韧,纹理精美雅致,是高级家具、地板等的理想原材料。同时,光皮桦具有童期短、天然变异丰富等生物学特性,是林木遗传研究的优良材料(陈伟等,2006; 胡晓媛等,2006; 李玉岭等,2013)。由于其重要的经济价值和研究价值,光皮桦的分子生物学研究逐渐受到重视。但目前缺乏对其内参基因的系统研究,使得基于定量PCR的基因表达分析准确性受到影响。因此,本研究以光皮桦16个不同组织为材料,选取16个常用的候选内参基因(Actin, 18S rRNA, GAPDH, TUBα, TUBβ, DnaJ-lp, UBi4, UBi-ep, UBi-lp, UBC, UBi10, CYP, HIS3, TATA, RPL3, EF1α),通过geNorm,NormFinder和BestKeeper 3个内参基因稳定性分析软件,分析候选内参基因的荧光定量数据,来筛选出不同组织器官中最为稳定的内参基因,并通过目的基因进行验证,以期为光皮桦的分子生物学研究提供参考。
1 材料与方法 1.1 试验材料采用的植物材料为种植于浙江农林大学林木种质资源圃的光皮桦无性系植株(7年生)。从3月初至5月中旬,取无性系植株不同时期的叶片(小叶、中叶、大叶)、雌花序(雌花芽、中期、盛花期)、雄花序(雄花芽、中期、盛花期)、茎(茎尖、幼嫩的茎),此外还有芽、韧皮部、木质部、种子及其水培苗的根共16种材料。试样经液氮速冻后于-80 ℃保存,用于RNA的提取。
1.2 总RNA的提取及反转录cDNA的合成利用PureLinkTM Plant RNA Reagent(Invitrogen)试剂提取材料的总RNA,操作步骤参考使用说明书进行,RNA的完整性通过琼脂糖凝胶电泳检测,RNA的浓度用Nanodrop ND-1000测定。全长cDNA第1链的合成根据PrimeScriptTM RT reagent Kit with gDNA Eraser(TAKARA)的操作方法进行,每个样品取1 000 ng总RNA为模板将各个样品反转录合成cDNA,反应获得的产物于-20 ℃储藏备用。
1.3 候选内参基因及定量引物设计根据转录组序列(登录号:SRX290467,SRX665078),选取16个功能不同的看家基因:Actin,18S rRNA,GAPDH,TUBα,TUBβ,DnaJ-lp,UBi4,UBC,UBi10,CYP,HIS3,TATA,RPL3,UBi-ep,UBi-lp,EF1α作为候选内参基因,所有候选内参基因序列均通过克隆测序确认(表 1)。利用Premier 5设计16个内参基因及目的基因CSLD和KOR的定量PCR引物,引物信息参见表 1。
|
|
将各个组织样品的cDNA等量混合为模板,对16个候选内参基因进行PCR扩增。PCR反应采用TaKaRa LA Taq,反应体系为cDNA模板1.0 μL,上下游引物(10 μmol·L-1)1.0 μL,反应Mix12.5 μL,加ddH2O至25.0 μL。反应条件: 95 ℃预变性3 min; 95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,共35个循环; 72 ℃延伸5 min。最后用2%的琼脂糖凝胶电泳检测引物特异性。
1.5 荧光定量PCR分析及扩增效率检测逆转录产物cDNA稀释10倍,使用SYBR Premix Ex TaqTM(TAKARA)定量试剂盒,在CFX-96荧光定量PCR仪(Bio-Rad)上进行定量PCR。反应体系为10 μL,其中SYBR mix 5 μL,上下游引物(10 μmol·L-1)各0.4 μL,cDNA模板0.5 μL,并加3.7 μL ddH2O,每个样本3次重复。PCR扩增反应程序:95 ℃预变性30 s,95 ℃变性5 s,60 ℃退火20 s,共40个循环。qRT-PCR扩增反应结束后进行65~95 ℃熔解曲线分析。
引物扩增效率分析中将所有样品cDNA等量混合,按照10倍梯度稀释产物,共5个浓度梯度进行定量PCR,制作标准曲线,并利用公式E(%)=(10-1/slope-1)× 100计算候选内参基因引物的扩增效率,式中,E为基因扩增效率,slope为标准曲线的斜率。R2=(总平方和-残差平方和)/总平方和,总平方和为标准曲线中相应浓度Ct值的实际值与平均值的平方差之和,残差平方和为相应浓度下Ct值的估计值与实际值的平方差之和;R2的大小表示了标准曲线回归方程估测可靠程度的高低。
1.6 数据分析和处理利用geNorm(Vandesompele et al., 2002),NormFinder(Andersen et al., 2004)和BestKeeper(Pfaffl et al., 2004)3个软件分别对16个候选内参基因在光皮桦不同组织器官中表达的稳定性进行分析,从而筛选出表达相对稳定的内参基因。
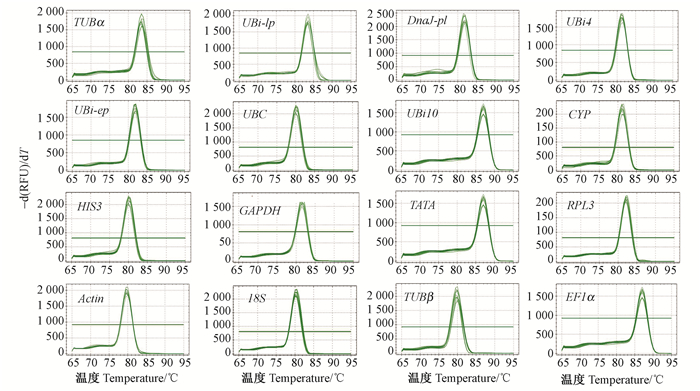
2 结果与分析 2.1 引物特异性和扩增效率检测将16个不同组织的cDNA等量混合作为模板,进行普通PCR和定量PCR。电泳检测结果显示,所有引物的产物均只有单一条带,也不存在引物二聚体(图 1),且所有溶解曲线都是明显的单一峰(图 2),这说明定量引物的特异性良好。进一步计算扩增效率显示,18S的扩增效率最低(91.2%),Actin的扩增效率最高(111.3%),而相关系数R2在0.983~0.999之间(表 2)。

|
图 1 候选内参基因PCR产物电泳检测 Fig.1 Electrophoresis of the PCR products of candidate reference genes M: DL 2000 marker. 1: TUBα; 2: UBi-lp; 3: DnaJ-lp; 4: UBi4; 5: UBC; 6: UBi-ep; 7: UBi10; 8: CYP; 9: HIS3; 10: GAPDH; 11: RPL3; 12: TATA; 13: Actin; 14: 18S; 15: TUBβ; 16: EF1α. |
|
图 2 候选内参基因PCR产物的溶解曲线 Fig.2 The melting curve of the candidate reference genes -d(RFU)/dT:荧光信号比温度的负导数Negative derivative ratio of fluorescent signal to temperature. |
|
|
利用比较Ct值的方法评估内参基因在不同组织样品中的表达水平。由图 3可知,不同组织中,18S的Ct值最小,为12.16,平均表达水平最高;UBC的Ct值最高,其平均表达水平最低;其他各候选内参基因的Ct值在25~30之间,表达水平相差不大。

|
图 3 候选内参基因在不同组织中的Ct值 Fig.3 Ct values of the candidate reference genes in different tissues |
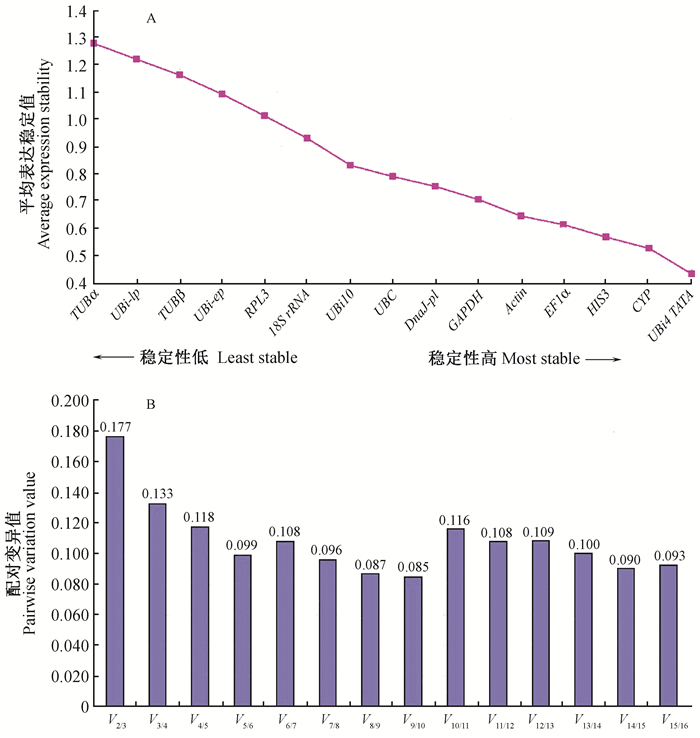
利用geNorm, Normfinder与BestKeeper 3个软件对候选内参基因在16个不同组织样品中的表达稳定性进行分析。geNorm软件通过计算得到所有候选内参基因的M值,进行稳定性排序,M值越大表明稳定性越差,相反则稳定性越高。作为筛选内参的标准,M值小于1.5被视为稳定表达的理想内参。图 4A显示,在16种不同组织样品中,所有候选内参的M值均小于1.5,达到作为内参基因的标准,其中TATA和 UBi4最为稳定,而TUBα表达最不稳定。此外,geNorm还根据候选内参基因标准化因子的配对差异分析(Vn/n+1)得到内参基因的最佳数目,软件默认Vn/n+1=0.15,当Vn/n+1 < 0.15时表明n个内参基因已经稳定,没有必要引入第n+1基因。通过图 4B显示,V2/3=0.177 > V3/4=0.133,说明加入第3个基因明显提高了组合的稳定性,而除了V2/3>0.15外,其他Vn/n+1均小于0.15,说明以3个内参基因组合就可以使结果稳定可靠。
|
图 4 geNorm软件分析内参基因表达稳定性及最适数目 Fig.4 Analysis of expression stability and optimal number of reference genes for normalization by geNorm |
NormFinder软件基于组内方差和组间方差,计算表达稳定值来验证内参基因的稳定性,表达稳定值越小表明基因越稳定。如表 3显示,由NormFinder分析得到不同组织中候选内参基因的表达稳定值和稳定性排名,其中EF1α和Actin排在前2位,稳定值十分接近,表达最稳定;UBC和TATA排在第3和第4;UBi-lp的稳定值相对较高(0.301),表达稳定性最差。
|
|
BestKeeper软件基于相关系数(r)变量来评价内参基因的稳定性,相关系数越高表明基因的表达越稳定。表 4显示,在不同组织中,EF1α的相关系数最大(r=0.982),说明基因表达的稳定性最高,排在后面的依次是TATA,UBi4和UBC。UBi-ep则由于相关系数值最小(r=0.161)排在最后。
|
|
为了进一步验证筛选得到的内参基因的稳定性,选取稳定性较好的3个候选内参基因(EF1α,TATA和UBi4)来分析CSLD和KOR这2个目的基因在不同组织中的表达模式;同时选取表达不稳定的内参基因UBi-lp作为对照。
CSLD和KOR基因是纤维素合酶类的基因,在细胞壁合成中发挥重要作用,一般认为其在茎中和木质部中表达量较高(Hamann et al., 2004; Maloney et al., 2012; Yu et al., 2014)。如图 5所示,根据3个候选内参基因进行相对表达量的量化,CSLD在木质部中的表达量最高,而在叶子中表达量很低(图 5A);KOR在茎中的表达量最高,而在其他组织中的表达较为平均(图 5B)。同时,以这3个内参基因的组合进行数据标准化,目的基因也显示出一致的相对表达水平(图 5),进一步表明了这3个内参基因的可靠性。此外,分析结果也显示不稳定的内参基因UBi-lp并没有对表达数据进行有效的标准化,出现了目的基因表达水平过高或过低的现象,如CSLD在雌花中表达水平偏低,在木质部中表达却偏高(图 5A)。

|
图 5 CSLD和KOR在光皮桦不同组织中的表达分析 Fig.5 Expression analysis for CSLD and KOR in different tissues of Betula luminifera B:芽; L1-L3:不同发育时期的叶片; M1-M3:不同发育时期的雄花; F1-F3:不同发育时期的雌花; P:韧皮部; X:木质部; St1,St2:茎; R:根; S:种子。 B: Bud; L1-L3: Different developmental stages of leaves; M1-M3: Different developmental stages of male flowers; F1-F3: Different developmental stages of female flowers; P: Phloem; X: Xylem; St1, St2: Stems; R: Root; S: Seed. |
荧光定量PCR由于精确性好、灵敏度高和易于操作的优点,已经成为一种量化转录表达和揭示基因表达规律的实用有效的方法。但是,在特定条件下稳定表达的内参基因是定量结果准确的必要条件(Bustin et al., 2010)。在很多植物中,看家基因如ACT,GAPDH,UBC等,常常被作为基因表达研究的内参基因(Kanakachari et al., 2015; Galli et al., 2015; Galeano et al., 2014)。然而,很多研究表明很多看家基因在不同的试验条件下实际的表达差异很大,并不一定适合作为内参基因(Expósito-Rodríguez et al., 2008; Jian et al., 2008)。传统的看家基因在不同物种或是相同物种不同的组织器官中表达并不一直稳定,这要求根据试验要求选取表达相对稳定的内参基因。
研究发现理想内参基因的表达水平既不能过高也不能过低,应是中度表达,Ct值在15~30之间最佳(Lilly et al., 2011; Wan et al., 2010)。Pettengill等(2012)对2种不同基因型杨树的不同组织进行长日照、短日照及短日照低温条件处理,最终得到的8个候选内参基因的Ct值分布在15~30之间。本研究通过Ct值比较分析发现,在16个不同的组织样品中,除18S以外,所有候选内参基因都中度表达,而18S的Ct值低于15,在定量分析中基线值对其影响过大(Vandesompele et al., 2002; 王金星等,2014),其不适合作为内参基因应用于基因表达分析。
经过3种软件计算得到的结果,可以看出,在光皮桦16种不同组织当中,NormFinder和BestKeeper显示EF1α的稳定性最好,而geNorm计算得出最稳定的是TATA,EF1α排到第5位。有研究显示,geNorm,NormFinder和BestKeeper软件得出的结果会有较小差异(Artico et al., 2010; Chen et al., 2011; De Spiegelaere et al., 2015)。Galeano等(2014)利用不同软件计算9个候选内参基因在柚木(Tectona grandis)不同组织中的稳定性,结果显示3个软件得出的稳定性排名均有差异。Fan等(2013)在毛竹(Phyllostachys edulis)中对14个候选内参基因进行稳定性评价,发现在不同发育时期的6个组织中,3种软件得出的稳定性排名也不一致。造成这种结果的原因可能是3种不同软件的算法不同。所以,由每个软件得到的内参基因稳定性排名靠前的都可能是可用内参,如NormFinder中排名第2位的actin,虽然其在geNorm和BestKeeper中排在第6和第8,但是其稳定值符合稳定内参的要求,也可以用作内参基因。当然,这些排名靠前的内参基因稳定性需要进一步的验证。
有研究结果显示,在基因定量表达研究中,单一内参基因可能会使试验结果有偏差,而选择2个或2个以上的内参基因可以增加结果的准确性(Schmid et al., 2003; Cui et al., 2016)。Park等(2012)对在胁迫作用下的4种番薯(Ipomoea batatas)品种进行10个候选内参基因的筛选,并使用单个内参(包括稳定内参ARF和不稳定内参TUB)及2个最佳内参组合(ARF/COX)对2个基因IbLEA14和swpa2进行表达验证,结果显示单个最佳内参ARF与组合ARF/COX的表达水平有微小差异,而以TUB为内参的表达水平则呈现相反的结果。本试验中,3个内参基因组合可以使定量结果准确可靠,为了进一步验证本研究筛选的3个最佳的内参基因(EF1α,TATA和UBi4)的可行性,对CSLD和KOR 2个目的基因在16个不同组织中进行了表达分析,结果显示3个内参基因与其组合定量标准化有较小差异,但是由最不稳定的内参基因(UBi-lp)得到的标准化定量数据明显不准确。
4 结论在16个不同候选基因中,筛选得到EF1α,TATA和UBi4等在光皮桦不同组织中表达稳定性高的内参基因,对EF1α,TATA和UBi4的稳定性进行了验证,明确了这些基因作为光皮桦荧光实时定量PCR内参的可靠性。因此,本研究为光皮桦不同组织器官基因表达分析的内参选择提供了依据。
| [] |
陈伟, 施季森, 刘希华. 2006. 光皮桦研究现状及遗传改良策略. 南京林业大学学报:自然科学版 , 30 (1) : 119–122.
( Chen W, Shi J S, Liu X H.2006. Present research and tactics for genetic improvement of Betula luminifera Winkl. Journal of Nanjing Forestry University:Natural Sciences Edition , 30 (1) : 119–122. [in Chinese] ) (  0) 0)
|
| [] |
胡晓媛, 李志真, 梁一池. 2006. 优良速生树种光皮桦研究进展. 福建林业科技 , 33 (2) : 159–163.
( Hu X Y, Li Z Z, Liang Y C.2006. The progress of the studies on the excellent fast-growing tree Betula luminifera. Journal of Fujian Forestry Science and Technology , 33 (2) : 159–163. [in Chinese] ) (  0) 0)
|
| [] |
李玉岭, 周厚君, 林二培, 等. 2013. 光皮桦BlSPL1转录因子基因的克隆、表达及单核苷酸多态性分析. 林业科学 , 49 (9) : 52–61.
( Li Y L, Zhou H J, Lin E P, et al.2013. Isolation, expression and single nucleotide polymorphisms analysis of transcription factor (BlSPL1) from Betula luminifera. Scientia Silvae Sinicae , 49 (9) : 52–61. [in Chinese] ) (  0) 0)
|
| [] |
王金星, 张利军, 廖资亿, 等. 2014. 刺槐实时定量PCR分析中内参基因的选择. 林业科学 , 50 (9) : 167–172.
( Wang J X, Zhang L J, Liao Z Y, et al.2014. The selection of reference genes for real-time quantitative PCR normalization in Black Locust (Robinia pseudoacacia). Scientia Silvae Sinicae , 50 (9) : 167–172. [in Chinese] ) (  0) 0)
|
| [] |
Andersen C L, Jensen J L, Orntoft T F.2004. Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Research , 64 (15) : 5245–5250.
DOI:10.1158/0008-5472.CAN-04-0496 ( 0) 0)
|
| [] |
Artico S, Nardeli S M, Brilhante O, et al.2010. Identification and evaluation of new reference genes in Gossypium hirsutum for accurate normalization of real-time quantitative RT-PCR data. BMC Molecular Biology , 10 (1) : 1–12.
( 0) 0)
|
| [] |
Brunner A M, Yakovlev I A, Strauss S H.2004. Validating internal controls for quantitative plant gene expression studies. BMC Plant Biology , 4 (1) : 1–7.
DOI:10.1186/1471-2229-4-1 ( 0) 0)
|
| [] |
Bustin S A.2002. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR):trends and problems. Journal of Molecular Endocrinology , 29 (1) : 23–39.
DOI:10.1677/jme.0.0290023 ( 0) 0)
|
| [] |
Bustin S A, Beaulieu J F, Huggett J, et al.2010. MIQE précis:Practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments. BMC Molecular Biology , 11 (1) : 1–5.
DOI:10.1186/1471-2199-11-1 ( 0) 0)
|
| [] |
Chen L, Zhong H Y, Kuang J F, et al.2011. Validation of reference genes for RT-qPCR studies of gene expression in banana fruit under different experimental conditions. Planta , 234 (2) : 377–390.
DOI:10.1007/s00425-011-1410-3 ( 0) 0)
|
| [] |
Cui B, Smooker P M, Rouch D A, et al.2016. Selection of suitable reference genes for gene expression studies in Staphylococcus capitis during growth under erythromycin stress. Molecular Genetics and Genomics : 1–17.
( 0) 0)
|
| [] |
De Oliveira L A, Breton M C, Bastolla F M, et al.2012. Reference genes for the normalization of gene expression in Eucalyptus species. Plant Cell Physiology , 53 (2) : 405–422.
DOI:10.1093/pcp/pcr187 ( 0) 0)
|
| [] |
De Spiegelaere W, Dern-Wieloch J, Weigel R, et al.2015. Reference gene validation for RT-qPCR, a note on different available software packages. PLoS One , 10 (3) : e0122515.
DOI:10.1371/journal.pone.0122515 ( 0) 0)
|
| [] |
Expósito-Rodríguez M, Borges A A, Borges-Pérez A, et al.2008. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process. BMC Molecular Biology , 8 (1) : 1–12.
( 0) 0)
|
| [] |
Fan C, Ma J, Guo Q, et al.2013. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis). PLoS One , 8 (2) : e56573.
DOI:10.1371/journal.pone.0056573 ( 0) 0)
|
| [] |
Galli V, Borowski J M, Perin E C, et al.2015. Validation of reference genes for accurate normalization of gene expression for real time-quantitative PCR in strawberry fruits using different cultivars and osmotic stresses. Gene , 554 (2) : 205–214.
DOI:10.1016/j.gene.2014.10.049 ( 0) 0)
|
| [] |
Galeano E, Vasconcelos T S, Ramiro D A, et al.2014. Identification and validation of quantitative real-time reverse transcription PCR reference genes for gene expression analysis in teak (Tectona grandis Lf.). BMC Research Notes , 7 (1) : 1–13.
DOI:10.1186/1756-0500-7-1 ( 0) 0)
|
| [] |
Hamann T, Osborne E, Youngs H L, et al.2004. Global expression analysis of CESA and CSL genes in Arabidopsis. Cellulose , 11 (3/4) : 279–286.
DOI:10.1023/B:CELL.0000046340.99925.57 ( 0) 0)
|
| [] |
Jian B, Liu B, Bi Y, et al.2008. Validation of internal control for gene expression study in soybean by quantitative real-time PCR. BMC Molecular Biology , 9 (9) : 59.
( 0) 0)
|
| [] |
Kanakachari M, Solanke A U, Prabhakaran N, et al.2016. Evaluation of suitable reference genes for normalization of qPCR gene expression studies in Brinjal (Solanum melongena L. ) during fruit developmental stages.Applied Biochemistry and Biotechnology , 178 (3) : 433–450.
( 0) 0)
|
| [] |
Lilly S T, Drummond R S, Pearson M N, et al.2011. Identification and validation of reference genes for normalization of transcripts from virus-infected Arabidopsis thaliana. Molecular Plant-Microbe Interactions , 24 (3) : 294–304.
DOI:10.1094/MPMI-10-10-0236 ( 0) 0)
|
| [] |
Maloney V J, Samuels A L, Mansfield S D.2012. The endo-1, 4-β-glucanase Korrigan exhibits functional conservation between gymnosperms and angiosperms and is required for proper cell wall formation in gymnosperms. New Phytologist , 193 (4) : 1076–1087.
DOI:10.1111/j.1469-8137.2011.03998.x ( 0) 0)
|
| [] |
Nolan T, Hands R E, Bustin S A.2006. Quantification of mRNA using real-time RT-PCR. Nature Protocols , 1 (3) : 1559–1582.
DOI:10.1038/nprot.2006.236 ( 0) 0)
|
| [] |
Park S C, Kim Y H, Ji C Y, et al.2012. Stable internal reference genes for the normalization of real-time PCR in different sweetpotato cultivars subjected to abiotic stress conditions. PLoS One , 7 (12) : e51502.
DOI:10.1371/journal.pone.0051502 ( 0) 0)
|
| [] |
Pettengill E A, Parmentier-Line C, Coleman G D, et al.2012. Evaluation of qPCR reference genes in two genotypes of Populus for use in photoperiod and low-temperature studies. BMC Research Notes , 5 (1) : 1–12.
( 0) 0)
|
| [] |
Pfaffl M W, Tichopad A, Prgomet C, et al.2004. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity:BestKeeper-Excel-based tool using pair-wise correlations. Biotechnology Letters , 26 (6) : 509–515.
DOI:10.1023/B:BILE.0000019559.84305.47 ( 0) 0)
|
| [] |
Schmid H, Cohen C D, Henger A, et al.2003. Validation of endogenous controls for gene expression analysis in microdissected human renal biopsies. Kidney International , 64 (1) : 356–360.
DOI:10.1046/j.1523-1755.2003.00074.x ( 0) 0)
|
| [] |
Sinha P, Singh V, Suryanarayana V, et al.2015. Evaluation and validation of housekeeping genes as reference for gene expression studies in Pigeonpea (Cajanus cajan) under drought stress conditions. PLoS One , 10 (4) : e0122847.
DOI:10.1371/journal.pone.0122847 ( 0) 0)
|
| [] |
Udvardi M K, Czechowski T, Scheible W R.2008. Eleven golden rules of quantitative RT-PCR. Plant Cell , 20 (7) : 1736–1737.
DOI:10.1105/tpc.108.061143 ( 0) 0)
|
| [] |
Vandesompele J, De Preter K, Pattyn F, et al.2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biology , 3 (7) : research0034.
( 0) 0)
|
| [] |
Wan H, Zhao Z, Qian C, et al.2010. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber. Analytical Biochemistry , 399 (2) : 257–261.
DOI:10.1016/j.ab.2009.12.008 ( 0) 0)
|
| [] |
Wong M L, Medrano J F.2005. Real-time PCR for mRNA quantitation. Biotechniques , 39 (1) : 75–85.
DOI:10.2144/05391RV01 ( 0) 0)
|
| [] |
Yu L, Chen H, Sun J, et al.2014. PtrKOR1 is required for secondary cell wall cellulose biosynthesis in Populus. Tree Physiology , 34 (11) : 1289–1300.
DOI:10.1093/treephys/tpu020 ( 0) 0)
|
 2016, Vol. 52
2016, Vol. 52

