文章信息
- 董丽莉, 赵韩生, 王丽丽, 孙化雨, 娄永峰, 高志民
- Dong Lili, Zhao Hansheng, Wang Lili, Sun Huayu, Lou Yongfeng, Gao Zhimin
- 毛竹PeSCR基因的表达与功能
- Expression and Function of PeSCR Gene from Phyllostachys edulis
- 林业科学, 2016, 52(6): 35-42
- Scientia Silvae Sinicae, 2016, 52(6): 35-42.
- DOI: 10.11707/j.1001-7488.20160605
-
文章历史
- 收稿日期:2015-06-01
- 修回日期:2015-08-19
-
作者相关文章
细胞分裂不均等产生1对命运不同的子细胞,是细胞功能和命运多样化的一种很重要的机制(Horvitz et al.,1992),一些研究已经证实了植物发育过程的细胞不均等分裂,高等植物中合子的第1次分裂是属于细胞横向的不均等分裂,分裂后产生1个小的顶端细胞和1个大的基底细胞。细胞分裂、细胞伸长和细胞特化之间高度的协调调控产生最终的有机体形态(Gallagher et al.,1997; Scheres et al.,1999)。植物的生长发育贯穿于整个生命过程,而位于茎段和根尖分生组织的干细胞调控这些过程。研究发现,一些分子途径在茎段分生组织和根端分生组织中是相同的,例如根端和茎端的表皮细胞发育是受一种途径调控,这种途径中TRANSPARENT TESTA GLABRA和GLABRA2基因发挥相似的作用(Galway et al.,1994; Hung et al.,1998; Masucci et al.,1999; Rerie et al.,1994)。
SCARECROW(SCR)和SHORT-ROOT(SHR)是2个与根和茎端组织发育相关的重要基因(Wysocka-Diller et al.,2000; Helariutta et al.,2000),其中SCR基因首先在拟南芥(Arabidopsis thaliana)中被发现,该基因在皮层和表皮原始细胞表达,scr突变体由于不均等分裂的破坏而丢失1层细胞层,表明SCR基因在拟南芥根的发育中起着重要的调控作用(Di Laurenzio et al.,1996)。SCR基因的突变不仅使得根部出现辐射类型缺失,而且下胚轴和茎段花梗也出现相同的缺失症状,表明SCR基因调控拟南芥胚根和胚芽的辐射发育(Wysocka-Diller et al.,2000)。有研究发现SCR还参与拟南芥根部的干细胞巢的定位(Sabatini et al.,2003)。除此之外,SCR在拟南芥中还参与糖响应和ABA调节途径(Cui et al.,2012)。在玉米(Zea mays)中,ZmSCR参与玉米根部的定位信息,这种定位信息主要作用是决定根部结构类型的产生(Lim et al.,2000);与拟南芥中的SCR基因相比,ZmSCR基因可以恢复SCR突变缺失的功能,ZmSCR和SCR在功能上是保守的(Lim et al.,2005);另外还发现ZmSCR基因参与C4植物花环结构的形成(Slewinski et al.,2012)。水稻(Oryza sativa)OsSCR不仅参与皮层和内皮层原始细胞的不均等分裂,还参与气孔和舌叶的形成(Kamiya et al.,2003)。白羽扇豆(Lupinus albus)LaSCR 1 基因影响其排根的生长(Sbabou et al.,2010)。最新研究发现,拟南芥SCR和辐射松(Pinus radiata)PrSCR与成熟细胞形成不定根有关(Della Rovere et al.,2015; Abarca et al.,2014),SCR通过调控生长素影响其他基因的表达方式调控干细胞的分裂与分化,从而协调根的生长(Moubayidin et al.,2013)。
毛竹(Phyllostachys edulis)是最具特色的竹种之一,在生长季节2个月内完成高生长和粗生长,其速生的特点与其根系的生长发育密切关联。毛竹基因组草图(Peng et al.,2013)的发布为从分子水平揭示毛竹分生组织的发育与调控机制奠定了基础。本研究以毛竹为材料,获得毛竹SCR同源基因PeSCR,在充分分析基因结构特点的基础上,研究其组织表达特异性以及不同激素和非生物胁迫下的表达特性,通过在模式植物拟南芥中表达,鉴定其功能,以期为深入研究PeSCR在毛竹根系生长发育中的调控作用提供参考。
1 材料和方法 1.1 试验材料及处理方法毛竹半年生实生苗,实验室温室盆栽培养(腐殖土与蛭石的体积比为3 ∶1),温度25 ℃,光照强度150 μmol ·m-2s-1,光照周期为光/暗=16 h/8 h。
激素处理方法(Ma et al.,2010):用200 μmol ·m-2s-1 GA3溶液喷洒毛竹叶片,分别在处理后0,0.25,0.5,1,3,5 h取毛竹根系;用200 μmol ·m-2s-1ABA溶液喷洒毛竹叶片,分别在处理后0,1,3,6,12,24 h取毛竹根系。NaCl和干旱处理方法(杨丽,2011):用200 mmol ·m-2s-1 NaCl溶液浇灌毛竹,分别在处理后0,2,5,9,14,20天取毛竹根系;对毛竹进行控水干旱处理,分别在处理后0,2,5,9,14,20天取毛竹根系。取样后,所有样品用液氮速冻处理,置于-80 ℃超低温冰箱保存备用。
1.2 基因组DNA提取及cDNA合成采用改良CTAB法提取毛竹叶片的基因组DNA(高志民等,2006),用紫外分光光度计测量A260和A280的值,检测提取的DNA的质量。将DNA放置-20 ℃保存备用。
采用Trizol法(Gao et al.,2006)分别提取毛竹的根、茎、叶和鞘的RNA,以及不同处理根的RNA,按照Promega公司的反转录试剂盒操作说明合成cDNA,存-20 ℃备用。
1.3 基因的分离、测序和分析用水稻转录因子OsSCR2蛋白序列(Os12g02870)在毛竹基因组数据库BambooGDB(http://www.bamboogdb.org)中查找同源基因序列及其上游调控序列,采用Spidey(http://www.ncbi.nlm.nih.gov/spidey/)分析基因结构特点,利用在线平台Plant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析上游调控序列所含作用元件,用MEGA 6.0软件构建基于目的基因编码蛋白的系统进化树。
根据目的基因序列的ORF设计引物(SCR-F: 5′-A T G G G C T C C T C C T C C C T C C T C-3′; SCR-R: 5′-T C A A C A G C C T G A A G T C T G A A T C G G-3′),由生工生物工程(上海)股份有限公司合成。PCR反应体系(20 μL):5×Primer STARTM Buffer 4.0 μL,dNTP Mixture 1.6 μL,引物SCR-F 1.0 μL,引物SCR-R 1.0 μL,cDNA 2.0 μL,Primer STARTM HS DNA polymerase 0.2 μL,DMSO 1.0 μL,超纯水9.2 μL。PCR程序:98 ℃预变性10 min;98 ℃变性30 s,66 ℃退火30 s,72 ℃延伸2 min,35个循环;72 ℃延伸10 min。PCR产物用BioMIGE公司回收试剂盒,回收后加A连接到pGEM-T easy载体上,转化大肠杆菌(Escherichia coli)Trans5α,筛选出阳性克隆,送生工生物工程股份有限公司测序。
1.4 基因的表达分析根据基因序列的非保守区设计定量引物qSCR-F( 5′-C C A T G G C G G C T T C T T G A T G-3′)和qSCR-R( 5′-C T C C T C C T C C T C C G A C C T-3′)。选取毛竹NTB基因作为内参(Fan et al.,2013),引物为NTB-F( 5′-T C T T G T T T G A C A C C G A A G A G G A G-3′)和NTB-R( 5′-A A T A G C T G T C C C T G G A G G A G T T T-3′),分别以毛竹的根、茎、叶和鞘4种组织的cDNA为模板,进行组织特异性表达分析。以不同处理毛竹根系的cDNA为模板,进行不同处理对基因表达的影响检测。RT-PCR反应体系(20 μL):10×PCR buffer 2.0 μL,dNTP Mixture 2.0 μL,引物qSCR-F 1.0 μL,引物qSCR-R 1.0 μL,cDNA 1.0 μL,rTaq 0.2 μL,超纯水12.8 μL。PCR程序:95 ℃预变性10 min;95 ℃变性30 s,62 ℃退火30 s,72 ℃延伸30 s,28个循环;72 ℃延伸10 min。采用Roche LightCycler480 SYBR GreenⅠMaster试剂盒在耶拿公司QTower仪器上进行qPCR反应。qPCR反应体系(10 μL):5.0 μL Mix,0.8 μL cDNA,qSCR-F和qSCR-R各0.2 μL(10 μmol ·L-1),超纯水3.8 μL。qPCR程序:95 ℃ 10min;95 ℃ 30 s,62 ℃ 30 s,50个循环。利用2-△△Ct法(Livak et al.,2001)分析数据,用Excel作图。
1.5 植物表达载体构建根据植物表达载体pCAMBIA1301的多克隆位点及目的基因序列的酶切位点,分别设计添加酶切位点的引物,合成正义扩增引物SCR-S-F(5′-CCatcgatA T G G G C T C C T C C T C C C T C C T C-3′)和SCR-S-R(5′-CGggatccT C A A C A G C C T G A A G T C T G A A T C G G-3′),末端分别添加ClaⅠ和BamHⅠ;反义扩增引物SCR-A-F(5′-GCtctagaA T G G G C T C C T C C T C C C T C C T C-3′)和SCR-A-R(5′-CGggatccT C A A C A G C C T G A A G T C T G A A T C G G-3′),末端分别添加BamHⅠ和XbaⅠ。采用Pyrobest 高保真酶进行PCR扩增,产物后加A连接到pGEM-Teasy 载体上,转化大肠杆菌,选择阳性单克隆测序。将测序正确的片段分别连接到pCAMBIA1301的多克隆位点,形成目的基因的正义、反义植物表达载体。
1.6 拟南芥转化、转基因植株检测与表型观察采用电击法将含有目的基因的植物表达载体质粒转入农杆菌(Agrobacterium tumefaciens)GV3101,采用浸花法转化野生型拟南芥(Clo-0)(Clough et al.,1998)。筛选抗性(Hgy50 mg ·L-1)植株,观察分析表型并拍照。选择3株正义植株和2株反义植株,分别提取RNA,将提取的RNA反转录成cDNA。分别用引物SCR-S-F和SCR-S-R、SCR-A-F和SCR-A-R检测目的基因在转基因植株中的表达情况,同时以野生型拟南芥作对照。
2 结果与分析 2.1 基因克隆与序列分析从毛竹基因组数据库中,查找到与水稻OsSCR 2 同源基因序列FP094510,用DNAStar软件分析该基因全长2 301 bp,包含5′UTR为238 bp,3′UTR为134 bp和ORF为1 929 bp。将该基因命名为PeSCR。PeSCR上游调控序列为1 820 bp,除含有TATA、CAAT-box启动子基本元件外,还包含生长素应答元件AuxRR-core、生长素调控元件TGA-element、ABA响应元件Motif Ⅱb、低温应答顺式作用元件LTR、干旱诱导MYB结合位点MBS、节律控制顺式作用元件circadian以及光应答元件和顺势作用元件,这意味着PeSCR可能受到激素、低温、干旱等的调控。
用引物SCR-F和SCR-R,分别以毛竹的cDNA和基因组DNA为模板,进行PCR扩增,电泳检测发现对应的目的条带分别为2 000 bp和2 600 bp左右,与预测的片段大小相符。测序结果显示,cDNA对应的目的片段为1 926 bp,除比原有ORF序列中少了3个碱基(第130—132位:TCC)外,获得序列与FP094510完全一致;基因组DNA对应的片段为2 598 bp。用软件在线分析PeSCR基因的结构,发现该基因含有1个长度673 bp内含子,位于1484—2156 bp(图 1),内含子完全符合GT-AG剪接原则(Moore et al.,1993)。
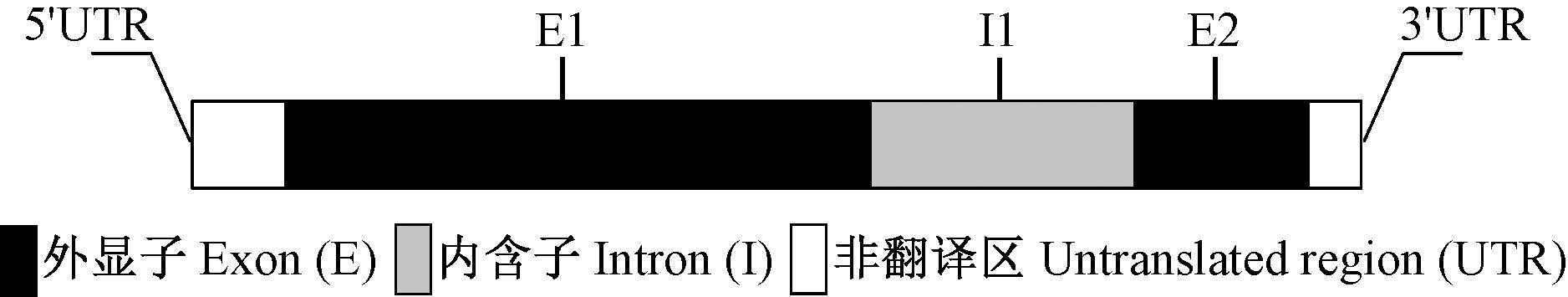
|
图 1 PeSCR基因结构示意 Fig.1 Structural diagram of PeSCR |
克隆得到的PeSCR编码641个氨基酸的蛋白,该蛋白除了包含GRAS家族的5个保守基序(LRⅠ,VHIID,LRⅡ,PFYRE和SAW)外,还含有AtSCR亚家族的特有基序Ⅰ,Ⅱ,进一步说明它属于GRAS转录因子家族的AtSCR亚家族。同源性分析结果表明,PeSCR与拟南芥的AtSCR的相似度为54.9%,与水稻的OsSCR1和OsSCR2相似度分别为82.1%,84.9%,与玉米的相似度为80.5%。构建基于SCR同源序列的系统进化树,结果显示 PeSCR与单子叶植物水稻、高粱(Sorghum bicolor)、玉米等的SCR聚在一起,而双子叶植物拟南芥、黄瓜(Cucumis sativus)、野大豆(Glycine soja)和蒺藜苜蓿(Medicago truncatula)的SCR聚类在一起,这与植物的进化关系相一致(图 2)。
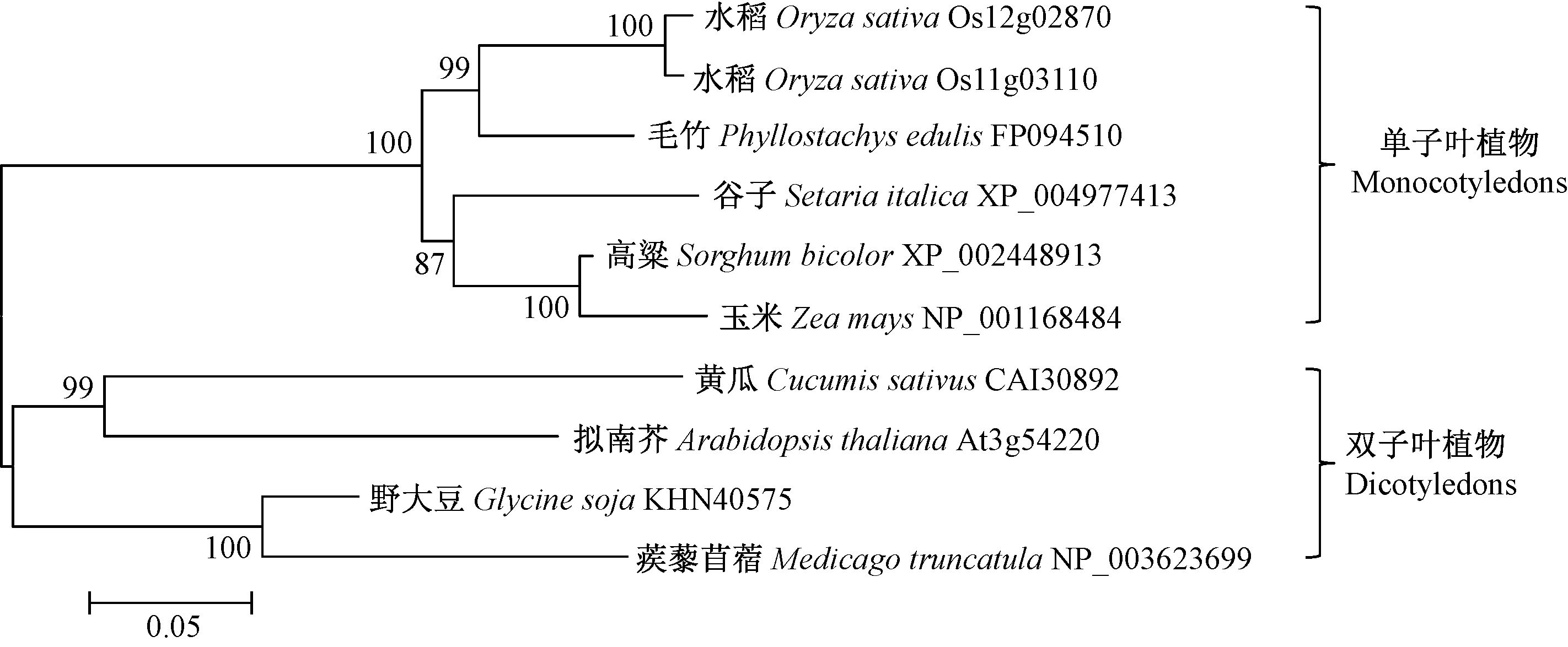
|
图 2 基于SCR序列构建的系统进化树 Fig.2 Phylogenetic tree based on the sequence of SCR 每个分支上的数字表示1 000次重复搜索的靴带值。 Numbers on major branches indicate bootstrap estimates for 1 000 replicate analysis. |
qPCR检测结果显示,PeSCR基因在毛竹的根、茎、叶和叶鞘中均有表达,其中在叶中表达的丰度最高,其次分别是在根和茎中,而在鞘中的表达丰度最低(图 3)。说明PeSCR基因属于组成型表达,可能对毛竹根、茎、叶和鞘的发育都有影响。
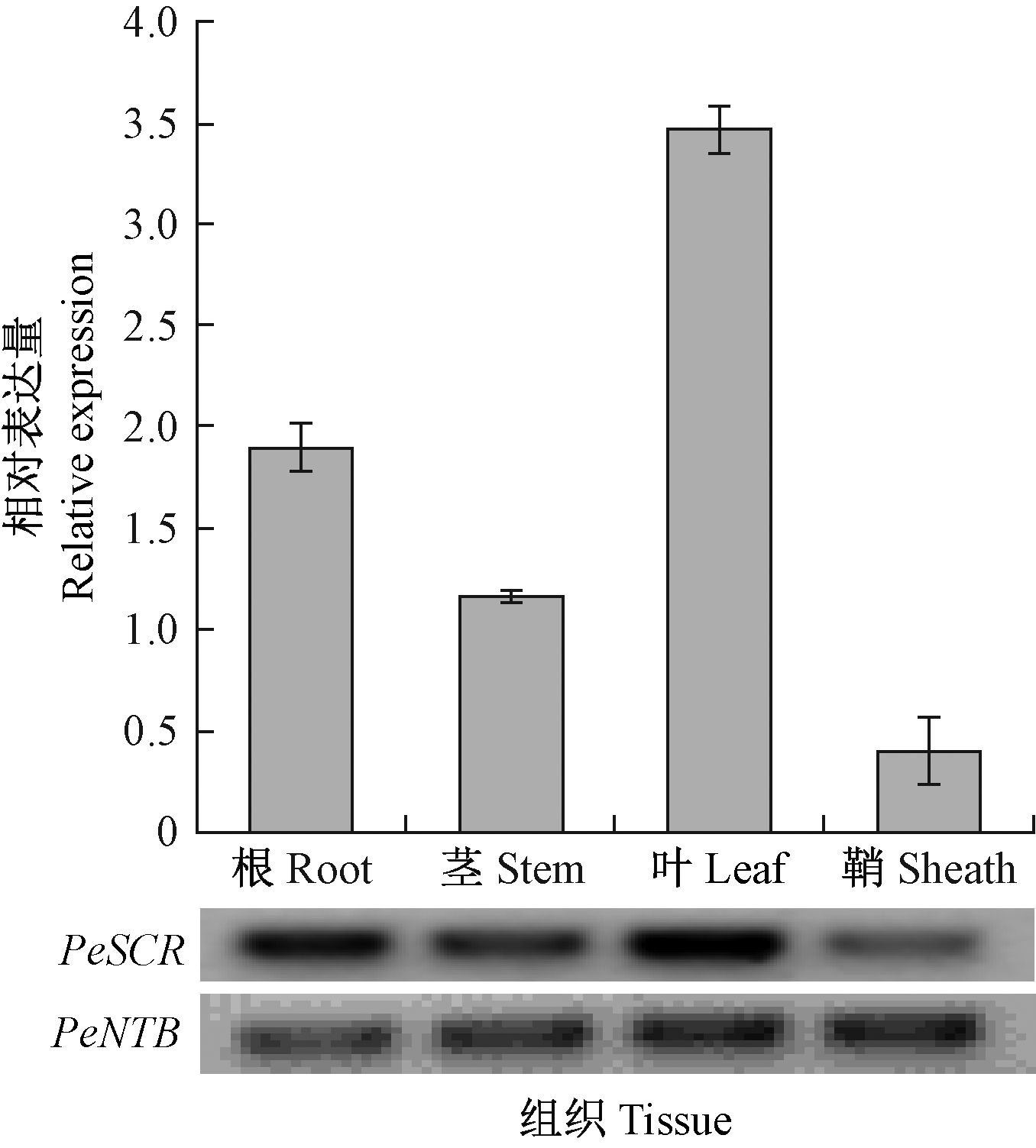
|
图 3 PeSCR在毛竹不同组织的表达 Fig.3 Expression of PeSCR in the different tissues of P. edulis |
GA3和ABA是调控植物根系生长发育的2类重要激素,其中GA3调控主根发育(Fu et al.,2003),ABA抑制侧根生长(De Smet et al.,2003)。GA3处理后,PeSCR基因表达量在1 h内逐渐降低,至1 h时表达量降低了80%,随着处理时间的增加,该基因的表达量开始上升,至5 h 表达量为0 h的2.5倍(图 4),表明PeSCR的表达短时间受GA3的抑制,随着处理时间的增加,该基因的表达受到诱导。在ABA处理6 h内PeSCR基因表达量逐渐下降,6 h时仅为0 h的10%,
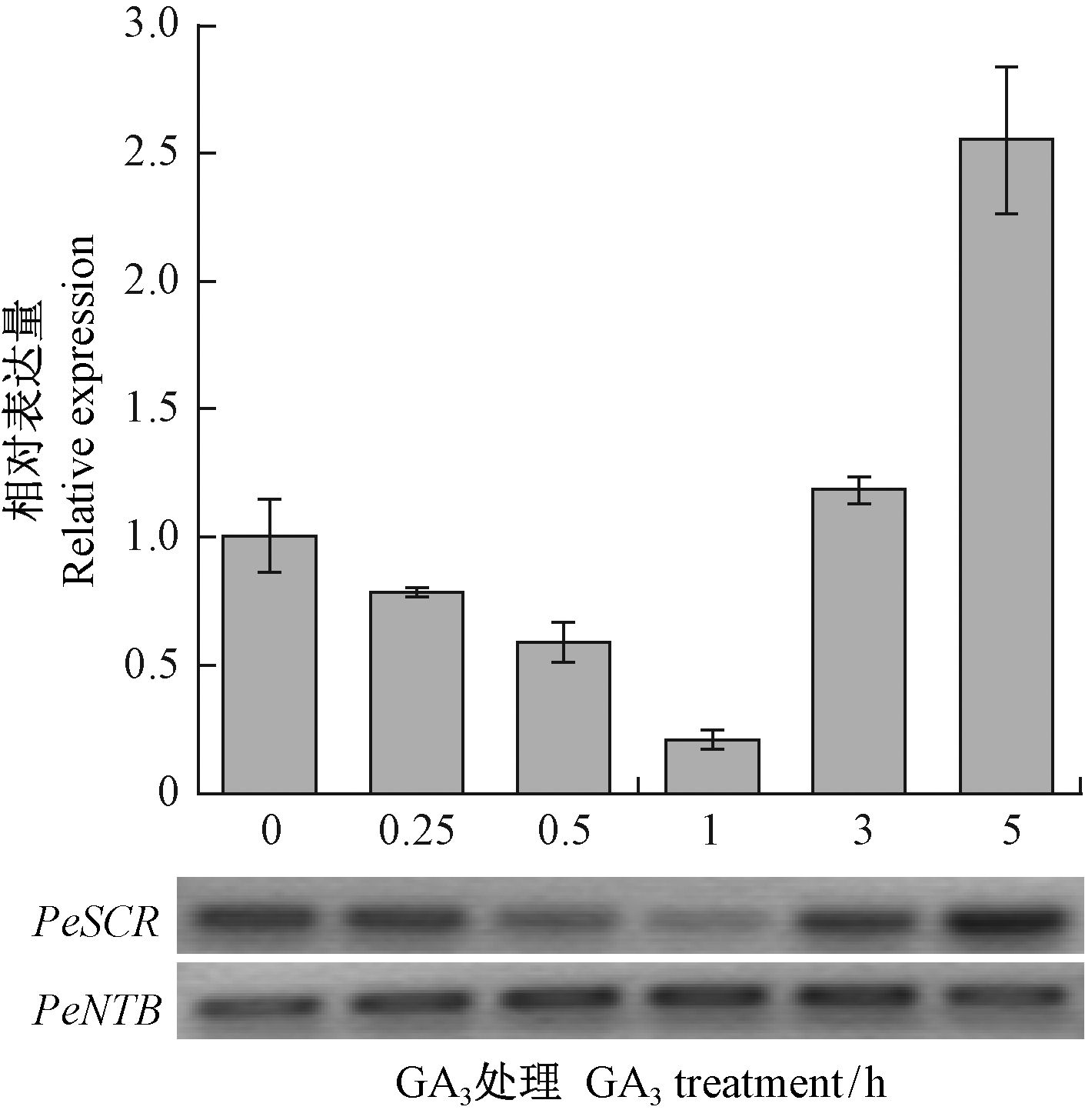
|
图 4 GA3处理下PeSCR基因的表达分析 Fig.4 Expression analysis of PeSCR treated by GA3 |
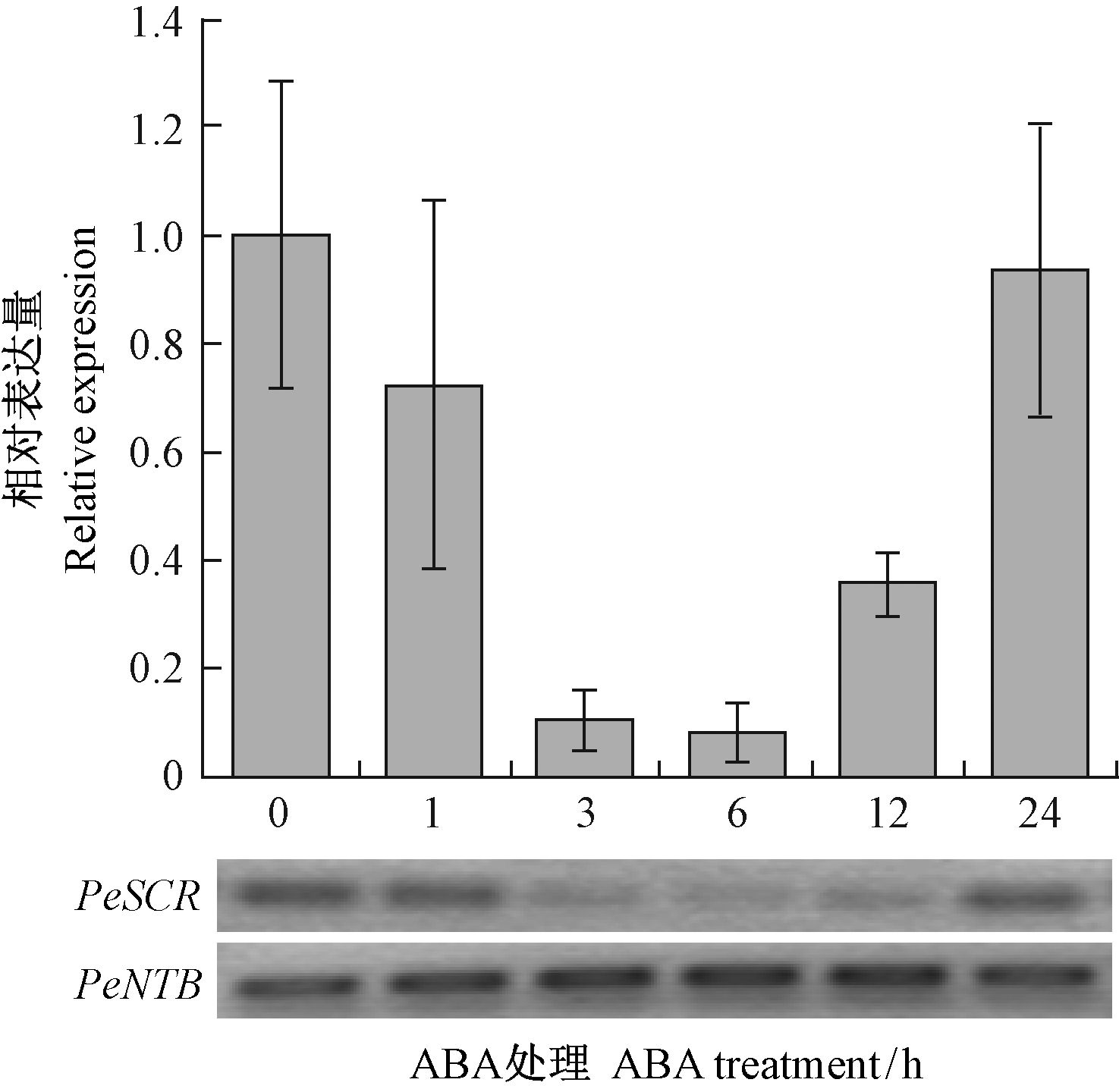
随后该基因表达逐渐恢复,至24 h时该基因的表达量与0 h处理相接近,但仍低于0 h处理(图 5),表明PeSCR的表达总体是受到ABA抑制的,自身具有一定的调节能力。GA3和ABA是2种完全不同类型的激素,处理毛竹后最终导致其根系中PeSCR的表达分别受到诱导和抑制。由此可以推测,外源GA3和ABA可能通过调控PeSCR的表达来影响毛竹根系生长发育。
|
图 5 ABA处理下PeSCR基因的表达分析 Fig.5 Expression analysis of PeSCR treated by ABA |
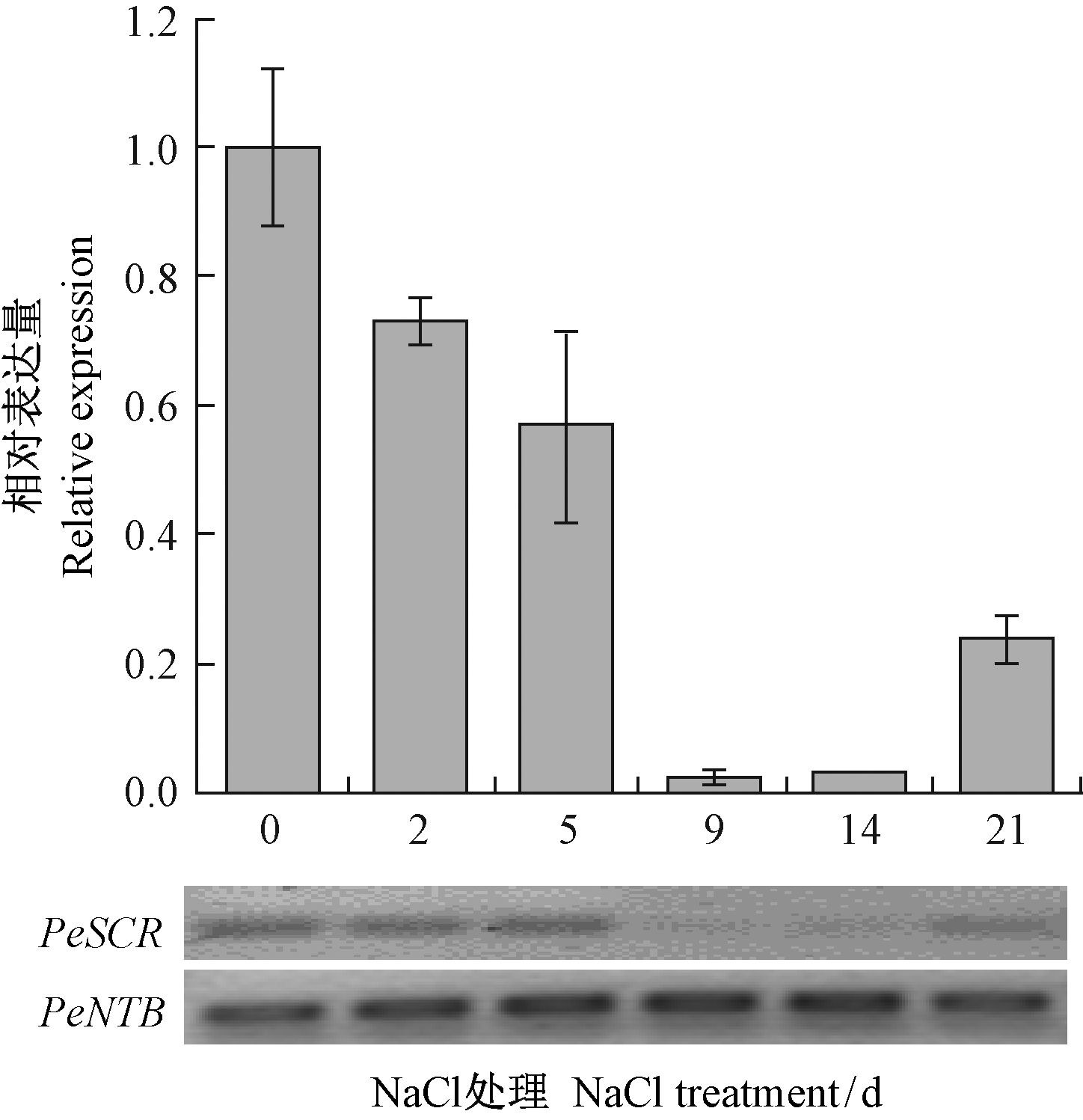
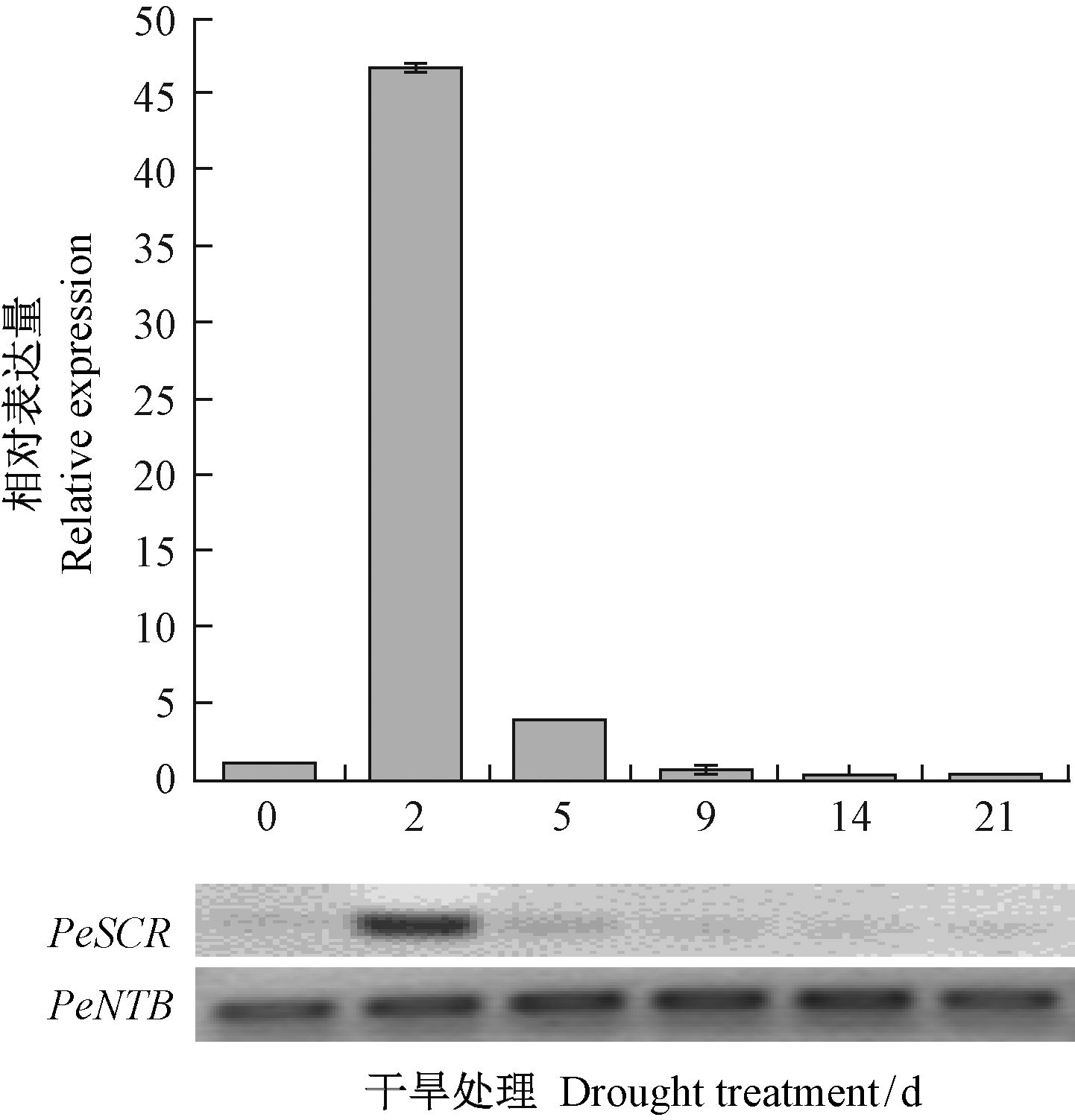
干旱和盐渍化是影响植物生长的非生物胁迫因子,根系最先感受到胁迫信号作出反应,并通过信号传导对有关基因的表达进行调整,最终影响根系生长。在NaCl处理0~9天内,PeSCR的表达量不断下降,至9天表达量相对于0天降低了98%,随后随处理时间的增加,该基因表达量有所恢复,至21天时其表达量约是0天的24%(图 6)。干旱处理条件下,PeSCR基因整体呈先上升后下降的趋势,干旱处理2天时表达量急剧增加,约是0天的46.6倍,随后表达量急剧下降,至21天时其表达量约是0天的20%(图 7)。由此表明,NaCl处理抑制PeSCR的表达,而干旱处理在短期内(5天)诱导PeSCR表达,但随干旱时间延长(9 天之后),其表达量受到抑制,这可能与NaCl和干旱胁迫的信号传导机制密切关联,导致基因表达的差异。
|
图 6 NaCl处理下PeSCR基因的表达分析 Fig.6 Expression analysis of PeSCR treated by NaCl |
|
图 7 干旱处理下PeSCR基因的表达分析 Fig.7 Expression analysis of PeSCR treated by drought |
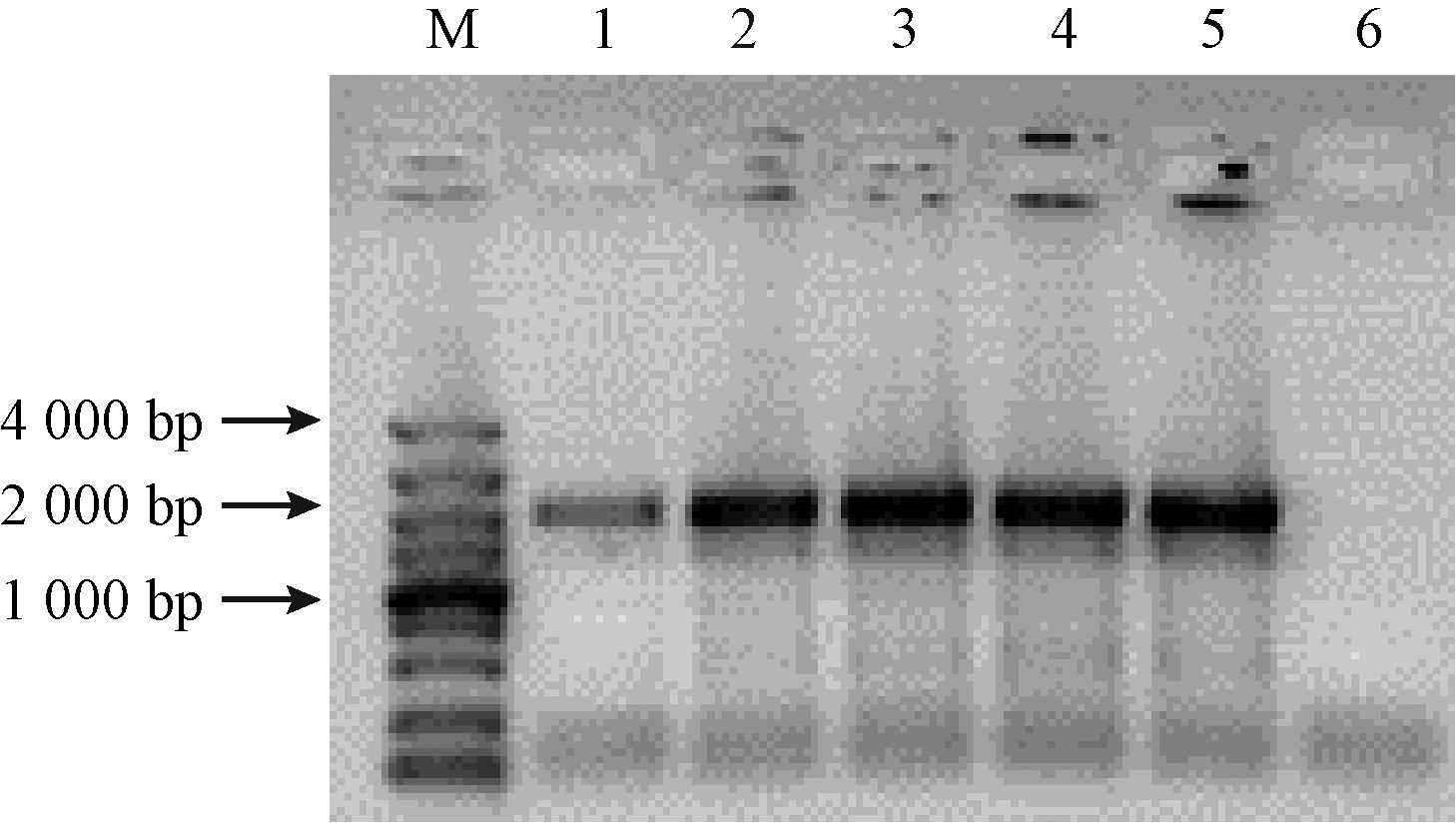
利用PeSCR基因正、反义表达载体转化野生型拟南芥,收获种子并进行抗性(Hgy)筛选,获得正义抗性植株7个,反义抗性植株5个。经过连续的抗性筛选获得纯合的T3株系,做后续的试验。挑选3株正义植株和2株反义植株进行分子检测,结果显示目的基因PeSCR均在转基因植株中得到表达,而野生型拟南芥中未检测到(图 8)。
|
图 8 转PeSCR基因植株RT-PCR检测 Fig.8 Transgenic plants of PeSCR checked by RT-PCR 1 -3:正义;3-5:反义;6:野生型。 1-3: Sense; 3-5: Antisense; 6: Wild type. |
表型观察发现,正义转基因植株与野生型表型类似,且生长健壮;而反义转基因植株则矮小,生长缓慢,叶片很小(图 9A)。将转基因植株在垂直平板上培养并观察根系生长情况,与野生型相比,10天后正义转基因植株的根系正常,且主根较长(图 9B),而反义转基因植株的根系,生长较慢,主根不明显且相对较短。14天后测量根系长度发现,正义转基因植株根系比野生型的偏长,反义转基因植株根系比野生型偏短,均达到显著水平(P<0.05);同时,正义转基因植株根系的侧根数目多于野生型植株,而反义转基因植株的根系侧根明显少于野生型植株(图 9C),均达到显著水平(P<0.05)。
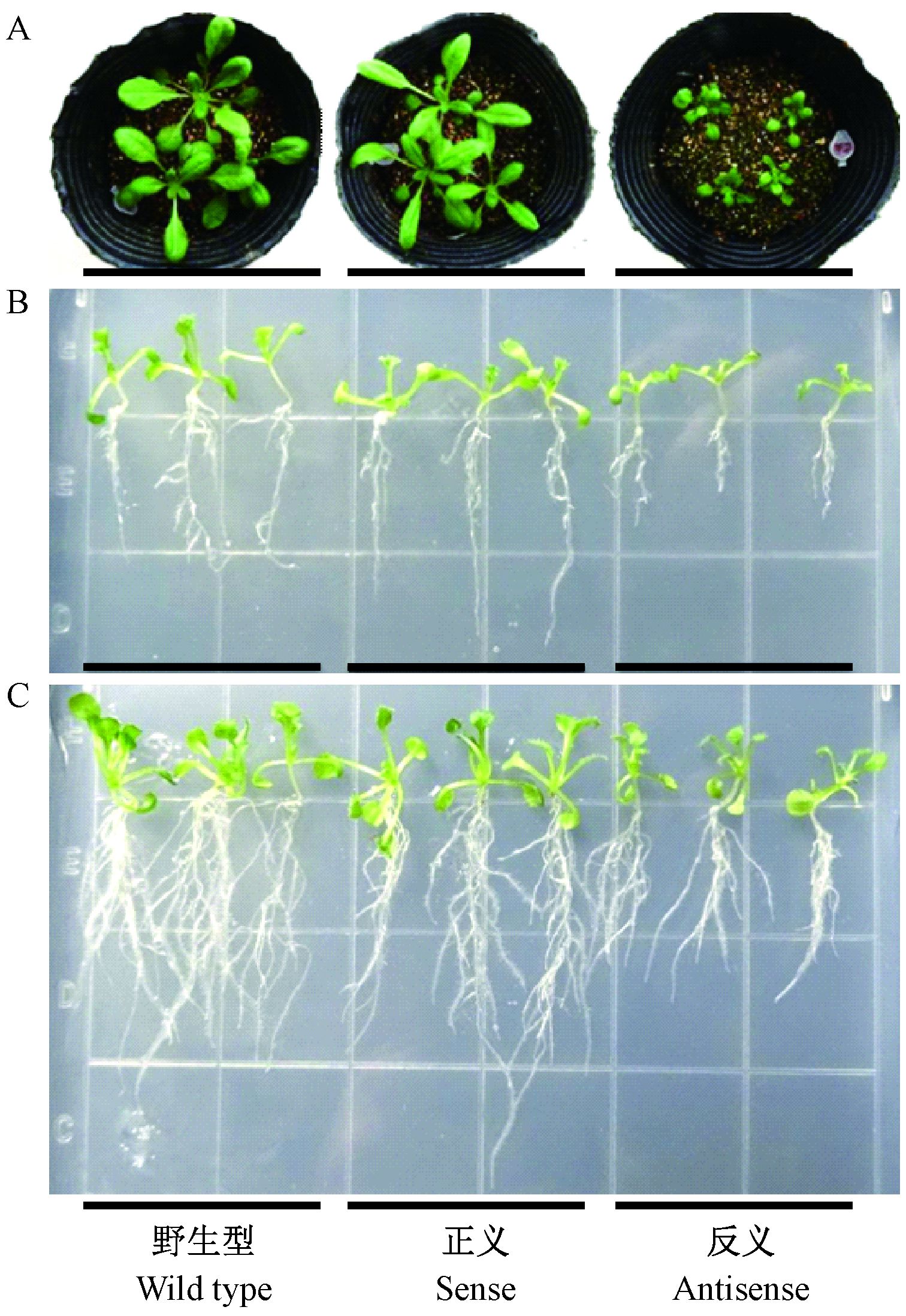
|
图 9 转基因植株表型 Fig.9 Root phenotype of transgenic plants A:盆栽25天;B:垂直板10天;C:垂直板15天。 A: Potted 25 days; B: 10 days on vertical plate; C: 15 days on vertical plate. |
植物的生长发育依赖于根系不断地从土壤吸收水分和必要的矿质养分(Bassani et al.,2004),竹子的快速生长意味着高固碳效率,这就需要大量的水分和多种必需的矿物质营养素的供给,因此研究竹子根系的生长发育调控对揭示其快速生长具有重要价值。
GRAS家族是植物特有的一类转录因子(Bolle,2004),其中的AtSCR亚家族成员主要参与根的发育调控(Di Laurenzio et al.,1996; Wysocka-Diller et al.,2000)。本研究克隆的毛竹PeSCR基因编码的转录因子属于AtSCR亚家族,PeSCR和水稻OsSCR及玉米ZmSCR的差异主要在于N端以及AtSCR亚家族特有基序Ⅰ,Ⅱ,所含LRⅠ,VHIID,LRⅡ,PFYRE和SAW 结构域非常保守,结构上的保守意味着功能上的相似。PeSCR基因在根、茎、叶和鞘中的表达存在明显的差异,在根中的表达丰度仅次于叶片,这可能意味着该基因在毛竹中除了参与根的发育调控外,也参与叶的发育调控,转基因拟南芥植株表型进一步证明了这一点。与野生型拟南芥相比,正义PeSCR转基因植株的主根较长,侧根数目较多,而反义转基因植株矮小,生长较慢,叶片偏小,主根较短,侧根数目较少,这与水稻OsSCR和玉米ZmSCR基因的功能相类似(Kamiya et al.,2003; Lim et al.,2005)。然而,在毛竹中PeSCR具体是如何影响根系发育的,是否参与其他组织的发育调控,有待进一步研究。
植物根系发育调控涉及大量的激素信号转导途径,生长素、细胞分裂素、乙烯、油菜素内酯和赤霉素等均参与根器官发生和形成等方面的调控(Benková et al.,2009)。同时,非生物胁迫也会通过引起细胞破坏和扰乱细胞的渗透平衡来影响植物根系的生长和发育(Mahajan et al.,2005)。本研究中对PeSCR上游调控序列的分析表明,其中包含生长素、ABA、干旱等多种应答作用元件,qPCR结果进一步证明PeSCR在根中的表达受到外源GA3、ABA的调控,以及非生物胁迫NaCl和干旱处理的影响。其原因可能是在处理后,激活了某些转录因子,使其与PeSCR基因的上游启动子区域的作用元件结合,从而调控该基因的表达。但目前尚未见关于GA3、ABA以及NaCl、干旱等胁迫处理条件下对SCR基因表达影响的报道,其具体的调控与响应机制有待深入研究。
PeSCR基因编码的蛋白作为转录因子可能具有多方面的功能,基因结构特点决定了功能,但是功能的实现除了会受到来自外界因素的影响外,其表达还受到内源激素、其他转录因子、miRNA、糖浓度等多种因素的调控。研究表明,拟南芥中AtSCR和AtSHR参与调控根的生长发育和激活内皮层中miR165/166的转录(Khan et al.,2011),而且SCL3-DELLA互作调节的GA信号途径与SHR/SCR途径联合共同调控根的分裂区基本组织分裂的时间和程度(Heo et al.,2011),另外研究表明,JACKDAW通过结合SCR启动子序列来直接调控SCR(Ogasawara et al.,2011),而SCR对糖应答具有独立调节作用(Cui et al.,2012),所以推测毛竹的PeSCR也可能存在复杂的网络调控途径。因此,要全面揭示PeSCR基因在毛竹根系生长发育中的功能尚需从调控与被调控的多个角度进行全面研究。
4 结论毛竹PeSCR基因在GA3、ABA、NaCl和干旱处理条件下的表达变化说明其参与了毛竹对激素和非生物胁迫的响应,对毛竹适应环境具有一定的作用。该基因的异源表达影响了转基因植株根系的生长发育,表明该基因可能通过参与植物根系的发育调控,进而在胁迫中发挥一定的功能。
| [1] | Gao Z M, Fan S H, Gao J, et al. 2006. Extract genomic DNA from Phyllostachys edulis by CTAB-based method. Forest Research 19(6):725-728.[in Chinese] |
| [2] |
杨丽. 2011.毛竹抗逆相关基因PeZFP和PeMYB基因的克隆及PeZFP基因功能验证.杭州:浙江农林大学硕士学位论文. ( Yang L. 2011. Cloning of PeZFP and PeMYB genes and function analysis of PeZFP gene in Phyllostachys edulis. Hangzhou:MS thesis of Zhejiang Agriculture and Forestry University.[in Chinese][in Chinese]) |
| [3] | Abarca D, Pizarro A, Hernández I, et al. 2014. The GRAS gene family in pine:transcript expression patterns associated with the maturation-related decline of competence to form adventitious roots. BMC Plant Biol 14:354. |
| [4] | Bassani M, Neumann P M, Gepstein S. 2004. Differential expression profiles of growth-related genes in the elongation zone of maize primary roots. Plant Mol Biol 56(3):367-380. |
| [5] | Benková E, Hejátko J. 2009. Hormone interaction at the root apical meristem. Plant Mol Biol 69(4):383-396. |
| [6] | Bolle C. 2004. The role of GRAS proteins in plant signal transduction and development. Planta 218(5):683-692. |
| [7] | Clough S J, Bent A F. 1998. Floral dip:a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J 16(6):735-743. |
| [8] | Cui H, Hao Y, Kong D. 2012. SCARECROW has a SHORT-ROOT-independent role on modulating the sugar response. Plant Physiol 158(4):1769-1778. |
| [9] | De Smet I, Signora L, Beeckman T, et al. 2003. An abscisic acid-sensitive checkpoint in lateral root development of Arabidopsis. Plant J 33(3):543-55. |
| [10] | Della Rovere F, Fattorini L, D'Angeli S, et al. 2015. Arabidopsis SHR and SCR transcription factors and AUX1 auxin influx carrier control the switch between adventitious rooting and xylogenesis in planta and in in vitro cultured thin cell layers. Ann Bot 115(4):617-628. |
| [11] | Di Laurenzio L, Wysocka-Diller J, Malamy J E, et al. 1996. The SCARECROW gene regulates an asymmetric cell division that is essential for generating the radial organization of the Arabidopsis root. Cell 86(3):423-433. |
| [12] | Fan C J, Ma J, Guo Q, et al. 2013. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis). PLoS One 8(2):e56573. |
| [13] | Fu X, Harberd N P. 2003. Auxin promotes Arabidopsis root growth by modulating gibberellin response. Nature 421(6924):740-743. |
| [14] | Gallagher K, Smith L G. 1997. Asymmetric cell division and cell fate in plants. Curr Opin Cell Biol 9(6):842-848. |
| [15] | Galway M E, Masucci J D, Lloyd A M, et al. 1994. The TTG gene is required to specify epidermal cell fate and cell patterning in the Arabidopsis root. Dev Biol 166(2):740-754. |
| [16] | Gao Z, Li X, Li L, et al. 2006. An effective method for total RNA isolation from bamboo. Chinese For Sci Tech 5(3):52-54. |
| [17] | Helariutta Y, Fukaki H, Wysocka-Diller J, et al. 2000. The SHORT-ROOT gene controls radial patterning of the Arabidopsis root through radial signaling. Cell 101(5):555-567. |
| [18] | Heo J O, Chang K S, Kim I L, et al. 2011. Funneling of gibberellin signaling by the GRAS transcription regulator SCARECROW-LIKE3 in the Arabidopsis root. Proc Natl Acad Sci USA 108(5):2166-2171. |
| [19] | Horvitz H R, Herskowitz I. 1992. Mechanisms of asymmetric cell division:two Bs or not two Bs, that is the question. Cell 68(2):237-255. |
| [20] | Hung C Y, Lin Y, Zhang M, et al. 1998. A common position-dependent mechanism controls cell-type patterning and GLABRA2 regulation in the root and hypocotyl epidermis of Arabidopsis. Plant Physiol 117(1):73-84. |
| [21] | Kamiya N, Itoh J, Morikami A, et al. 2003. The SCARECROW gene's role in asymmetric cell division in rice plants. Plant J 36(1):45-54. |
| [22] | Khan G A, Declerck M, Sorin C, et al. 2011. MicroRNAs as regulators of root development and architecture. Plant Mol Biol 77(1/2):47-58. |
| [23] | Lim J, Helariutta Y, Specht C D, et al. 2000. Molecular analysis of the SCARECROW gene in maize reveals a common basis for radial patterning in diverse meristems. Plant Cell 12(8):1307-1318. |
| [24] | Lim J, Jung J W, Lim C E, et al. 2005. Conservation and diversification of SCARECROW in maize. Plant Mol Biol 59(4):619-630. |
| [25] | Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct method. Methods 25(4):402-408. |
| [26] | Ma H S, Liang D, Shuai P, et al. 2010. The salt- and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana. J Exp Bot 61(14):4011-4019. |
| [27] | Mahajan S, Tuteja N. 2005. Cold, salinity and dorught stresses:an overview. Arch Biochem Biophys 444(2):139-158. |
| [28] | Masucci J D, Rerie W G, Foreman D R, et al. 1999. The homeobox gene GLABRA2 is required for position-dependent cell differentiation in the root epidermis of Arabidopsis thaliana. Development 122(4):1253-1260. |
| [29] | Moore M J, Query C C, Sharp P A. 1993. Splicing of precursors to mRNA by the spliceosome//Gesteland R F, Atkins J F. The RNA world. Cold Spring Harbor:Cold Spring Harbor Laboratory Press 303-357. |
| [30] | Moubayidin L, Di Mambro R, Sozzani R, et al. 2013. Spatial coordination between stem cell activity and cell differentiation in the root meristem. Dev Cell 26(4):405-415. |
| [31] | Ogasawara H, Kaimi R, Colasanti J, et al. 2011. Activity of transcription factor JACKDAW is essential for SHR/SCR-dependent activation of SCARECROW and MAGPIE and is modulated by reciprocal interactions with MAGPIE, SCARECROW and SHORT ROOT. Plant Mol Biol 77(4/5):489-499 |
| [32] | Peng Z, Lu Y, Li L, et al. 2013. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla). Nat Genet 45(4):456-561. |
| [33] | Rerie W G, Feldmann K A, Marks M D. 1994. The GLABRA2 gene encodes a homo domain protein required for normal trichome development in Arabidopsis. Genes Dev 8(12):1388-1399. |
| [34] | Sabatini S, Heidstra R, Wildwater M, et al. 2003. SCARECROW is involved in positioning the stem cell niche in the Arabidopsis root meristem. Genes Dev 17(3):354-358. |
| [35] | Sbabou L, Bucciarelli B, Miller S, et al. 2010. Molecular analysis of SCARECROW genes expressed in white lupin cluster roots. J Exp Bot 61(5):1351-1363. |
| [36] | Scheres B, Benfey P N. 1999. Asymmetric cell division in plants. Annu Rev Plant Physiol Plant Mol Biol 50:505-537. |
| [37] | Slewinski T L, Anderson A A, Zhang C, et al. 2012. Scarecrow plays a role in establishing Karnz anatomy in maize leaves. Plant Cell Physiol 53(12):2030-2037. |
| [38] | Wysocka-Diller J W, Helariutta Y, Fukaki H, et al. 2000. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot. Development 127(3):595-603. |
 2016, Vol. 52
2016, Vol. 52

