文章信息
- 廖柏勇, 王芳, 陈丽君, 刘明骞, 欧阳昆唏, 阙青敏, 惠文凯, 李培, 陈晓阳
- Liao Boyong, Wang Fang, Chen Lijun, Liu Mingqian, Ouyang Kunxi, Que Qingmin, Xi Wenkai, Li Pei, Chen Xiaoyang
- 基于SRAP分子标记的苦楝种质资源遗传多样性分析
- Genetic Diversity of Germplasm Resources of Melia azedarach Revealed by SRAP Markers
- 林业科学, 2016, 52(4): 48-58
- Scientia Silvae Sinicae, 2016, 52(4): 48-58.
- DOI: 10.11707/j.1001-7488.20160406
-
文章历史
- 收稿日期:2015-05-04
- 修回日期:2015-06-10
-
作者相关文章
2. 北京林业大学生物科学与技术学院 北京 100083
2. College of Biological Sciences and Technology, Beijing Forestry University Beijing 100083
苦楝(Melia azedarach)不仅生长快、材质好,是优良的建筑用材和家具用材,而且用途广,是植物源杀虫剂原料树种(Xu et al., 2009; 2010; 2011)。苦楝广泛分布于我国18°—40°N、海拔2 100 m以下的常绿阔叶林、落叶林,以及溪谷、田地的边缘和道路两旁(Peng et al., 2008)。从北方的河北保定、山西运城和甘肃陇南到南方的海南崖县,从东部的台湾、福建沿海到西部的四川成都和云南保山,分布范围覆盖中国陆地的三分之一(程诗明等,2006)。苦楝也分布于热带和亚热带其他的国家和地区,如斯里兰卡、泰国、越南、土耳其、菲律宾、不丹、尼泊尔、巴布亚新几内亚、印度、印度尼西亚、老挝、澳大利亚的热带地区以及太平洋群岛。由于苦楝对环境适应性强以及广泛的栽培,其起源中心尚不清楚(Peng et al., 2008)。
国内对苦楝表型遗传变异研究较多(程诗明等,2006; 陈丽君等,2014; 廖柏勇等,2014),而遗传多样性研究报道仅限于少数引物组合和有限的群体数量(夏海涛,2009; 程诗明,2005)。然而,苦楝分布相当广泛,只有广泛搜集种质资源,才有可能准确地开展遗传多样性分析。
SRAP分子标记基于PCR技术,利用长度为17~18个核苷酸的引物针对开放阅读框进行扩增分析(Li et al., 2001),常用于基因连锁作图和基因标记,具有简便、高重复率的特性。相对于其他分子标记方法,SRAP分子标记在植物物种的遗传多样性和亲缘关系分析中将能获得更多的信息。本研究采用SRAP分子标记,对中国17个省份的31个苦楝种源进行遗传多样性分析,为苦楝种质资源的保存和育种计划的制订提供依据。
1 材料与方法 1.1 试验材料试验选取苦楝主要地理分布区的31个种源(表 1),分别来自广东、广西、贵州、海南、江西、湖南、四川、安徽、河北、湖北、河南、山东、山西、福建、浙江、甘肃和云南17个省(区)。2014年2月,在华南农业大学播种育苗。从每个种源中抽取15个或者14个(GS,HANI,HEN和YN3种源)不同家系,记录家系编号,每个家系中随机抽取1株苦楝幼苗代表该家系,共计461个单株参与试验。按单株采集叶片样品,保存在-70 ℃冰箱,用于DNA提取。
|
|
从保存的每份样品中取约150 mg叶片,采用EZNA high-performance DNA mini kit 试剂盒(Omega Bio-Tek,Norcross,GA,USA)提取基因组DNA,用1%的琼脂糖凝胶电泳检测其质量。并用NanoDrop 1000分光光度计(Thermo Fisher Scientific,Waltham,MA,USA)检测每份DNA的浓度,再将其浓度调整为50 ng·μL-1,-20 ℃低温保存备用。
1.3 SRAP分析SRAP分子标记操作方法参照Li等(2001),试验中所用试剂由宝生物工程有限公司(Otsu,Japan)提供,PCR扩增采用25 μL反应体系,体系包括: 50 ng基因组DNA、200 μmol·L-1 dNTPs、2.75 mmol·L-1 MgCl2、0.4 μmol·L-1上游引物和下游引物、2.5 μLPCR缓冲液、0.75单位Taq DNA 酶和16.7 μL双蒸水。PCR程序在东胜热循环仪(EDC-810,苏州,中国)中进行,热循环体系程序如下: 94 ℃预变性5 min;前5个循环为94 ℃ 1 min,35 ℃ 1 min,72 ℃ 1 min;随后进行的35个循环为94 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min;最后72 ℃延伸5 min,于4 ℃保存。
用8个不同种源的基因组DNA对783对引物组合(27条上游引物,29条下游引物,表 2)进行初筛选,再用16个不同种源的苦楝基因组DNA进行引物复筛,最后得到20对引物(12条上游引物,13条下游引物),可以扩增可重复、清晰的多态带。PCR产物采用6%的聚丙烯酰胺凝胶电泳进行分析,随后以12.5 V·cm-1的速度电泳1.5 h,再用银染法显色(Bassam et al., 1993)。将可靠、清晰、明显的扩增条带记录为1,没有条带扩增出来的位点记录为0,统计100~1 500 bp 区间的条带,构建SRAP数据1/0矩阵。
|
|
采用 POPGEN 32 软件分析优选引物的总条带数、多态性条带数、多态带百分率(PPB)、多态性信息指数(PIC),分析种源的多态位点百分率(Pp)、Nei’s遗传多样性指数(H)以及Shannon’s信息指数(I)(Nei,1978)。
多态带百分率(PPB)计算公式如下: PPB=(K/N)×100%。式中: K为多态带的条数;N为总条带数。多态性信息指数(PIC)作为评价引物鉴别能力的标记指标,计算公式如下: PICi =2fi(1-fi)。式中: PICi为第i个标记的PIC值;fi 为第i个标记片段扩增出来的频率。
用POPGEN 32计算31个种源的遗传距离矩阵,然后采用Mega6.06软件中的邻接聚类法(Neighbour-Joining)建立种源亲缘关系聚类树状图(Saitou et al., 1987;Tamura et al., 2013)。采用GenAIEx 6.5(Peakall et al., 2012)软件,进行分子方差分析(AMOVA),采用PCoA法进一步分析不同种源间、种源内个体间的亲缘关系远近。采用Mantel检验法估算种源间遗传距离和地理距离的相关性(Mantel,1967)。
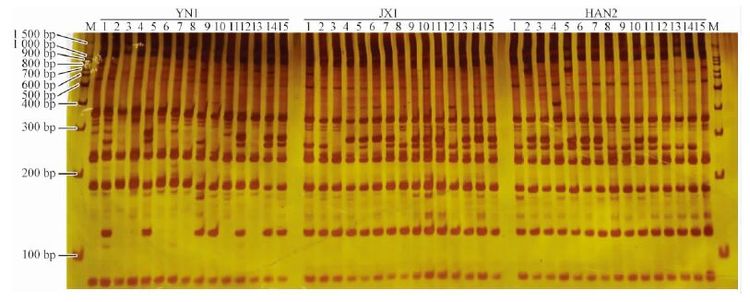
2 结果与分析 2.1 引物扩增结果20对引物在461个DNA样本中共扩增出257条清晰的条带,其中145条为多态带(58.24%)。Me6/Em10引物对的扩增效果如图 1所示。每对引物所扩增的总条带数、多态带条数、多态带百分率(PPB)、多态性信息指数值(PIC)列于表 3中。
|
图1 引物Me6/Em10扩增产物的聚丙烯酰胺凝胶电泳 Fig.1 Amplified pattern on polyacrylamide gel with primer combinations of Me6/Em10 M: Marker 100 bp DNA ladder.其他泳道分别为YN1、JX1和HAN2种源各自15个单株。 YN1,JX1, and HAN2 represent their each 15 plants of provenance respectively. |
|
|
20对引物的扩增条带数为4(Me6/Em29)~26(Me17/Em29)条,平均为12.85条。多态性条带数为2~14条,平均有7.25条。PPB值最高的是Me11/Em29引物组合(100%),其次是Me28/Em15(90.91%)和Me24/Em14(88.89%)。引物组合Me1/Em17和Me2/Em12的PPB值最低,分别为23.08%和16.67%。PIC平均值为0.385,PIC值最高和最低的引物组合分别是Me1/Em17(0.478)和Me24/Em14(0.262),表明引物组合Me1/Em17能提供的各种源遗传多样性信息最多。
2.2 遗传多样性分析31个种源的主要遗传多样性参数列于表 4。POPGEN 32 软件统计结果表明,样本的总遗传多样性(Ht)为0.37±0.01,种源遗传多样性(Hs)为0.20±0.01。多态位点百分率(Pp)变化范围为35.17%~76.55%,均值是58.24%。Nei’s遗传多样性指数为0.13~0.31,平均值为0.20。来自贵州册亨GZ2种源Nei’s遗传多样性指数最高。Shannon’s信息指数为0.18~0.45,均值为0.30。AMOVA(表 5)分析表明,在总遗传变异中,38.96%来自于种源间变异,61.04%来自于种源内变异,说明变异主要来源于种源内。
|
|
|
|
对31个苦楝种源Nei’s遗传距离进行邻接法聚类分析(图 2)。Nei’s遗传距离在31个种源的变动范围为0.039 5~0.532 3。邻接法根据Nei’s遗传距离计算各类群之间的聚类分支距离,31个种源紧密相关,分支距离最低为0.003,最高为0.076。根据分支距离从大到小顺序,将聚类群分为2类,2类的分支距离为 0.076。从图 2可以看出,第Ⅰ类群以贵州遵义(GZ4)、贵州兴义(GZ1)、四川成都(SC1)和云南楚雄(YN3)分支距离最小,联系最为紧密,其次是四川达州(SC2)、云南勐腊(YN1)、甘肃陇南(GS)以及贵州册亨(GZ2),主要为我国西南、西北地区高原、盆地种源。第Ⅱ类群包括其他23个种源。根据聚类分支距离大小,第Ⅱ类群中又细分为5个聚类中心,其中济南(SD1)、保定(HEB)、泰安(SD2)、渭南(SX)、许昌(HEN)种源聚为Ⅱ1类;临安(ZJ)、滁州(AH)和瑞昌(JX2)聚为Ⅱ2类;荆门(HUB)和永安(FJ)种源聚为Ⅱ3类;屯昌(HAN1)、五指山(HAN2)、钦州(GX2)和仁化(GD2)种源聚为Ⅱ4类;于都(JX1)和开平(GD1)种源分支距离更接近Ⅱ3类;东安(HUN1)和浏阳(HUN3)聚为Ⅱ5类;都安(GX3)种源分支距离介于Ⅱ4类与Ⅱ5类之间,但与Ⅱ4类更加紧密,桂林(GX1)则与Ⅱ5类更加接近。麻栗坡(YN2)、炎陵(HUN2)和黎平(GZ3)种源归于类群Ⅱ,但是与类群Ⅱ其他种源分支距离相距较远。

|
图2 31个苦楝种源邻接聚类系统树 Fig.2 Neighbour-Joining dendrogram of the 31 M. azedarach provenances |
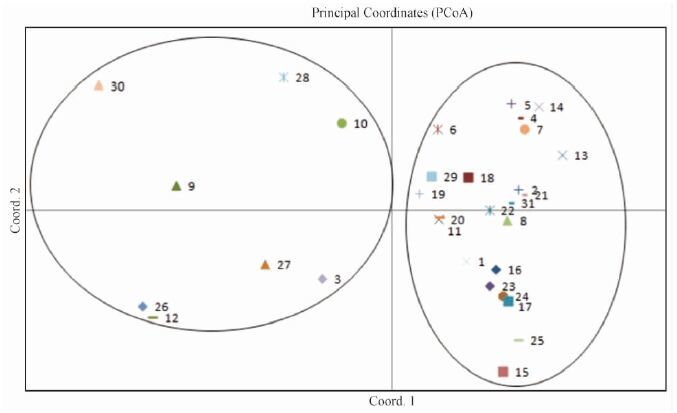
遗传距离是遗传变异在种源之间的分布,它是检测种源间及其种源内个体间遗传差异相关性的指标。种源间遗传距离的PCoA分析表明前3个主分量在总变异中所占的比例分别为71.79%,6.33%,4.06%(合计82.17%)。主分量1和主分量2的二维双标图(图 3)明显将31个苦楝种源聚为2类,与邻接聚类的分析结果基本保持一致。种源内个体间遗传距离矩阵PCoA分析(图 4)表明,主分量1和主分量2将整体试验样本大致分为2类,相同种源内的个体基本都聚集在一起。其分析结果与种源间主分量分析结果大致相同,细节有差异。
|
图3 种源间主分量分析 Fig.3 Biplots of PCoA1 and PCoA2 for the 31 M. azedarach provenances 图中数字为表 1中的序号,分别代表 31个不同的种源。Numbers in the figure above for the serial number in Tab. 1,representing 31 different provenances,respectively. |

|
图4 种源内主分量分析 Fig.4 Biplots of PCoA1 and PCoA2 within 31 M. azedarach provenances |
利用Mantel检验计算种群的地理距离和遗传距离相关性,当 P< 0.05 时,认为地理距离和遗传距离具有显著相关性。Mantel检验经过1 000次随机置换计算,运算结果表明遗传距离矩阵和地理距离矩阵呈极显著相关,相关系数为0.256(P=0.003,图 5)。以上结果证明苦楝种源存在地理变异,也为进一步研究苦楝空间遗传结构以及地理变异模型提供理论依据。

|
图5 遗传距离和地理距离相关性 Fig.5 Correlation between the geographic distance and Nei’s genetic distance |
本研究将SRAP分析标记用于苦楝的遗传多样性研究。相比于AFLP,RAPD以及ISSR分子标记,SRAP分子标记可以提供更多的信息(Youssef et al., 2011; Liu et al., 2008; Shao et al., 2010; Budak et al., 2004a)。研究结果表明,SRAP分子标记是分析苦楝种源间和种源内个体间遗传多样性的有效工具(Li et al., 2001; Budak et al., 2004b; Valdez-Ojeda et al., 2014; Jehan et al., 2014)。
从783对SRAP引物中共筛选出20对引物,可以从31个苦楝种源中扩增出清晰的特异条带。PIC是用于遗传多样性研究的重要指标,是引物鉴别能力和信息可靠性的反映值(Talebi et al., 2012; Barkley et al., 2006; Milla-Lewis et al., 2012; Uzun et al., 2011)。一般来说,当PIC>0.5 时,位点多态性非常好,遗传标记提供的信息度高;当 0.5>PIC>0.25 时,遗传标记可以提供较合理的信息;当PIC< 0.25 时,遗传标记可提供的信息较少(Vaiman et al., 1994; Xie et al., 2010)。本研究中PIC平均值为0.385,表明苦楝SRAP分子标记可以较好地反映苦楝不同种源多样性,可用于不同种源苦楝的分类和鉴定。其中Me1/Em17,Me2/Em12,Me19/Em5,Me6/Em10,Me27/Em18,Me19/Em7,Me6/Em29,Me28/Em15,Me4/Em5以及Me1/Em9,共10对引物扩增的56条多态带,具有较高的PIC值,可以区分所有的种源。这些引物可以构建一种苦楝种源快速鉴定的SRAP方法。
SRAP标记AMOVA分析表明,种源内变异61.04%,38.96%来自于种源间变异。而ISSR的AMOVA分析结果也显示种源内变异为65.4%,种源间变异为34.6%;AFLP的AMOVA结果认为72.74%的变异来源于种源内,27.26%的变异来源于种源间(夏海涛,2009;程诗明,2005)。多种不同的分子标记都表明苦楝遗传多样性变异的主要来源是种源内,种源间的基因交流受到某些因素的限制。因此,在选择高遗传多样性种源的基础上,侧重苦楝种源内家系和单株选择,将更有利于苦楝遗传改良。
树种的遗传多样性主要受繁育系统、种源地理分布以及种源迁移历史的影响。苦楝种源遗传多样性偏低可能是种子扩散途径有限的结果。有些树种种子容易被动物消化,通过排泄分散到其他地方,这是林木树种提高遗传多样性最为重要的机制之一(Hamrick et al., 1992)。然而,与麻疯树(Jatropha curcas)及其他树种种子类似,苦楝种子含有相当多的三萜类有毒物质。这些树种的种子被消化过程中常具有致命性,因此,食果动物和大部分鸟类以及其他动物都避免食用它,这样就会导致苦楝种子扩散的几率降低(Hamrick et al., 1992; Ferreiro et al., 2010),从而造成苦楝低水平的种源遗传多样性。
准确估算一个树种在其分布区的遗传多样性对于基因保存和优树选择非常重要。尽管苦楝的整体遗传多样性不高,但Nei’s 遗传多样性和Shannon’s 信息指数2项遗传多样性指标表明31个种源中有10个种源的遗传多样性高于ISSR和AFLP的结果(夏海涛,2009;程诗明,2005)。它们分别是贵州的册亨和黎平,云南的勐腊和麻栗坡,湖南的东安、炎陵和浏阳,四川达州,河北保定,安徽滁州种源。这10个种源Nei’s遗传多样性和Shannon’s信息指数值相对高于本研究中其他种源。而除了河北保定和安徽滁州以外,其他几个种源都来自容易形成局部地理生境气候的复杂山地地域。这种复杂山地地域也许是它们具有高遗传多样性的原因之一。
邻接聚类法将31个种源聚为两大类(图 2)。PCoA和Mantel 检验结果也证实该分析结果的正确性。聚类结果依中国地形及气候分布具有一定的规律。类群Ⅰ的遵义、兴义、成都、楚雄、达州、勐腊、陇南以及册亨种源都来自西南、西北地区的云贵高原、黄土高原以及四川盆地,这些地区降雨少、海拔高,具有气候一致性。类群Ⅱ跨度比较大,南北方种源都有,但是聚类小群从北往南,相同地理气候条件种源有聚为一类的趋势。例如,济南、保定、泰安、渭南和许昌种源都是典型的北方种源,聚为Ⅱ1类;华南地区的屯昌、五指山、钦州和仁化种源聚为Ⅱ4类;大致的趋势是从北往南相邻或相近的种源聚为一类。Mantel检验证实遗传距离和地理距离呈极显著相关(Liu et al., 2012),从侧面证明了聚类结果随地理变化具有一定可靠性。该聚类结果与AFLP和ISSR的苦楝种源遗传多样性聚类分析的结果有相似的结论。AFLP分子标记UPGMA聚类法将8个种源中的渭南、保定、武都群体聚为一类,其他的聚为另一类(程诗明,2005)。ISSR分子标记UPGMA聚类法将云南保山种源单独聚为一类(夏海涛,2009)。这些种源的聚类结果都与SRAP分子标记类群Ⅰ结果保持一致。而苦楝果核、种子表型变异欧氏距离离差平方和聚类也显示,四川、陕西、甘肃的种源都聚为一类(陈丽君等,2014)。这种将大多数种源都聚为2类,而且存在明显的地域特点的现象表明,苦楝的遗传多样性起源中心或许存在于2类种源的过渡地区。本研究中云南和贵州种源大多聚于类群I,而麻栗坡(YN2)和黎平(GZ3)种源聚归于类群Ⅱ,或许它们之间就是两大苦楝类群生态过渡区存在的体现。
对苦楝的过度开发和人为的破环,将降低苦楝遗传多样性。因此,应建立种质基因库或种子园,加强对苦楝优质种质的保护。
4 结论SRAP分子标记可以准确、有效地用于苦楝遗传多样性分析。苦楝遗传变异来源于种源内,而种源间基因交流有限。种源遗传多样性整体偏低,而部分山区种源遗传多样性较高。在选择高遗传多样性种源的基础上,苦楝选育应侧重种源内家系和单株选择。苦楝种源特征明显,地理环境差异对其遗传多样性具有一定的影响。31个种源大致可分为东、西2类,我国苦楝遗传多样性起源中心或许存在于2类不同种源的生态过渡区。
| [1] |
程诗明, 顾万春.2006. 苦楝表型性状梯度变异的研究. 林业科学, 42(5):29-35. (Cheng S M, Gu W C. 2006. Studies on phenotypical characteristics gradient variation of Melia azedarach. Scientia Silvae Sinicae, 42(5):29-35.[in Chinese])(  2) 2)
|
| [2] |
程诗明. 2005. 苦楝聚合群体遗传多样性研究与核心种质构建. 北京:中国林业科学研究院博士学位论文.(Cheng S M. 2005. Study on genetic diversity of multitudinous populations and construction of core germplasm in Melia azedarach. Beijing:PhD thesis of Chinese Academy of Forestry.[in Chinese])( 4) 4)
|
| [3] |
陈丽君, 邓小梅, 丁美美, 等. 2014. 苦楝种源果核及种子性状地理变异的研究. 北京林业大学学报, 36(1):15-20. (Chen L J, Deng X M, Ding M M, et al. 2014. Geographic variation in traits of fruit stones and seeds of Melia azedarach. Journal of Beijing Forestry University, 36(1):15-20.[in Chinese])(  2) 2)
|
| [4] |
廖柏勇, 陈晓阳, 陈丽君, 等.2014. 苦楝种源间种子发芽变异的观测. 广东农业科学, (11):43-47, 52. (Liao B Y, Chen X Y, Chen L J, et al. 2014. Observation of Melia azedarach seed germination variation among provenances. Guangdong Agriculture Sciences, (11):43-47, 52.[in Chinese])(  1) 1)
|
| [5] |
夏海涛. 2009. 药用苦楝遗传多样性ISSR分析和遗传变异规律研究. 福州:福建农林大学硕士学位论文.(Xia H T. 2009. The research on genetic diversity by ISSR analysis and genetic variation laws of medicinal Melia azedarach. Fuzhou:MS thesis of Fujian Agriculture and Forestry University.[in Chinese])( 4) 4)
|
| [6] |
Barkley N A, Roose M L, Krueger R R, et al. 2006. Assessing genetic diversity and population structure in a citrus germplasm collection utilizing simple sequence repeat markers(SSRs). Theoretical and Applied Genetics, 112(8):1519-1531.( 1) 1)
|
| [7] |
Bassam B J, Anolles G C. 1993. Silver staining of DNA in polyacrylamide gels. Applied Biochemistry and Biotechnology, 42(2):181-188.( 1) 1)
|
| [8] |
Budak H, Shearman R C, Parmaksiz I, et al. 2004a. Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs, SSRs, RAPDs, and SRAPs. Theoretical and Applied Genetics, 109(2):280-288.( 1) 1)
|
| [9] |
Budak H, Shearman R C, Parmaksiz I, et al. 2004b. Molecular characterization of buffalograss germplasm using sequence-related amplified polymorphism markers. Theoretical and Applied Genetics, 108(2):328-334.( 1) 1)
|
| [10] |
Ferreiro D, Orozco J P, Mirón C, et al.. 2010. Chinaberry tree(Melia azedarach) poisoning in dog:a case report. Topics in Companion Animal Medicine, 25(1):64-67.( 1) 1)
|
| [11] |
Hamrick J L, Godt M J W, Sherman-Broyles S L. 1992. Factors influencing levels of genetic diversity in woody plant species. New Forests, 6(1-4):95-124.( 2) 2)
|
| [12] |
Jehan T, Vashishtha A, Yadav S R, et al. 2014. Genetic diversity and genetic relationships in Hyacinthaceae in India using RAPD and SRAP markers. Physiology and Molecular Biology of Plants, 20(1):103-114.( 1) 1)
|
| [13] |
Li G, Quiros C. 2001. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics, 103(2):455-461.( 3) 3)
|
| [14] |
Liu L W, Zhao L P, Gong Y Q, et al. 2008. DNA fingerprinting and genetic diversity analysis of late-bolting radish cultivars with RAPD, ISSR and SRAP markers. Scientia Horticulturae, 116(3):240-247.( 1) 1)
|
| [15] |
Liu Z G, Shu Q Y, Wang L, et al. 2012. Genetic diversity of the endangered and medically important Lycium ruthenicum Murr. revealed by sequence-related amplified polymorphism(SRAP) markers. Biochemical Systematics and Ecology, 45:86-97.( 1) 1)
|
| [16] |
Mantel N. 1967. The detection of disease clustering and a generalized regression approach. Cancer Research, 27(2):209-220.( 1) 1)
|
| [17] |
Milla-Lewis S R, Kimball J A, Zuleta M C, et al. 2012. Use of sequence-related amplified polymorphism(SRAP) markers for comparing levels of genetic diversity in centipedegrass(Eremochloa ophiuroides(Munro) Hack.) germplasm. Genetic Resources and Crop Evolution, 59(7):1517-1526.( 1) 1)
|
| [18] |
Nei M. 1978. The theory of genetic distance and evolution of human races. The Japanese Journal of Human Genetics, 23(4):341-369.( 1) 1)
|
| [19] |
Peng H, David J M. 2008. Melia Linnaeus//Wu Z Y, Raven P H. Flora of China:Volume 11. Beijing:Science Press; St. Louis:Missouri Botanical Garden, 130-131.( 2) 2)
|
| [20] |
Peakall R, Smouse P E. 2012. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 28(19):2537-2539.( 1) 1)
|
| [21] |
Saitou N, Nei M. 1987. The neighbor-joining method:A new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 4(4):406-425.( 1) 1)
|
| [22] |
Shao Q S, Guo Q S, Deng Y M, et al. 2010. A comparative analysis of genetic diversity in medicinal Chrysanthemum morifolium based on morphology, ISSR and SRAP markers. Biochemical Systematics and Ecology, 38(6):1160-1169.( 1) 1)
|
| [23] |
Talebi M, Kazemi M, Sayed-Tabatabaei B E. 2012. Molecular diversity and phylogenetic relationships of Pistacia vera, Pistacia atlantica subsp. mutica and Pistacia khinjuk using SRAP markers. Biochemical Systematics and Ecology, 44:179-185.( 1) 1)
|
| [24] |
Tamura K, Stecher G, Peterson D, et al. 2013. MEGA6:Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 30(12):2725-2729.( 1) 1)
|
| [25] |
Uzun A, Yesiloglu T, Polat I, et al. 2011. Evaluation of genetic diversity in lemons and some of their relatives based on SRAP and SSR markers. Plant Molecular Biology Reporter, 29(3):693-701.( 1) 1)
|
| [26] |
Vaiman D, Mercier D, Moazami-Goudarzi K, et al. 1994. A set of 99 cattle microsatellites:characterization, synteny mapping, and polymorphism. Mammalian Genome, 5(5):288-297.( 1) 1)
|
| [27] |
Valdez-Ojeda R, James-Kay A, Ku-Cauich J R, et al. 2014. Genetic relationships among a collection of Musa germplasm by fluorescent-labeled SRAP. Tree Genetics & Genomes, 10(3):465-476.( 1) 1)
|
| [28] |
Xie W G, Zhang X Q, Cai H W, et al.. 2010. Genetic diversity analysis and transferability of cereal EST-SSR markers to orchardgrass(Dactylis glomerata L.). Biochemical Systematics and Ecology, 38(4):740-749.( 1) 1)
|
| [29] |
Xu H, Xiao X. 2009. Natural products-based insecticidal agents 4. Semisynthesis and insecticidal activity of novel esters of 2-chloropodophyllotoxin against Mythimna separata Walker in vivo. Bioorganic & Medicinal Chemistry Letters, 19(18):5415-5418.( 1) 1)
|
| [30] |
Xu H, Xiao X, Wang Q. 2010. Natural products-based insecticidal agents 7. Semisynthesis and insecticidal activity of novel 4α-alkyloxy-2-chloropodophyllotoxin derivatives against Mythimna separata Walker in vivo. Bioorganic & Medicinal Chemistry Letters, 20(17):5009-5012.( 1) 1)
|
| [31] |
Xu H, Zhang J L. 2011. Natural products-based insecticidal agents 9. Design, semisynthesis and insecticidal activity of 28-acyloxy derivatives of toosendanin against Mythimna separata Walker in vivo. Bioorganic & Medicinal Chemistry Letters, 21(7):1974-1977.( 1) 1)
|
| [32] |
Youssef M, James A C, Rivera-Madrid R, et al. 2011. Musa genetic diversity revealed by SRAP and AFLP. Molecular Biotechnology, 47(3):189-199.( 1) 1)
|
 2016, Vol. 52
2016, Vol. 52

