文章信息
- 丁昌俊, 张伟溪, 高暝, 黄秦军, 褚延广, 苏晓华
- Ding Changjun, Zhang Weixi, Gao Ming, Huang Qinjun, Chu Yanguang, Su Xiaohua
- 不同生长势美洲黑杨转录组差异分析
- Analysis of Transcriptome Differences among Populus deltoides with Different Growth Potentials
- 林业科学, 2016, 52(3): 47-58
- Scientia Silvae Sinicae, 2016, 52(3): 47-58.
- DOI: 10.11707/j.1001-7488.20160306
-
文章历史
- 收稿日期:2015-09-29
- 修回日期:2015-12-26
-
作者相关文章
2. 中国林业科学研究院亚热带林业研究所 富阳 311400
2. Research Institute of Subtropical Forestry, Chinese Academy of Forestry Fuyang 311400
杂种优势是指由遗传结构差异的亲本杂交产生的杂种F1在生长势、适应性、抗逆性及产量等方面超过亲本的生物学现象(Birchler et al.,2003)。目前,已被广泛用于农作物和林木育种中,但其遗传机制研究主要集中在遗传差异与杂种优势的关系上,主要包括显性假说、超显性假说和上位假说,尚不能完全解释杂种优势的复杂现象(Shull,1914; Bruce,1910; Chen,2010)。随着分子生物学技术不断发展,利用分子标记、mRNA差异显示、基因芯片等技术对杂种优势分子机制研究表明,基因差异表达模式与杂种F1的表型存在一定的联系(Birchler et al.,2003; 2010),杂种的特定性状可能是父、母本特定位点的等位基因共同表达的结果。通过玉米(Zea mays)杂种F1及其亲本全基因组基因表达分析显示,1 367个差异显著EST序列中有22%的EST序列是非加性基因表达模式,其中181个是超亲表达模式,23个是低亲表达模式,44个EST序列是超显性或显性不足表达模式,78%的EST序列表现为加性基因表达模式(Swanson-Wagner et al.,2006); 进一步eQTL研究表明,杂种F1中超过1/3的差异基因的表达调控与父本等位基因表达调控一致(Swanson-Wagner et al.,2009)。对4个毛果杨(Populus trichocarpa)×美洲黑杨(P. deltoides)杂种F1中30个等位基因研究显示,17个等位基因在杂种F1间、杂种F1与亲本间均显著差异表达,其余13个基因与亲本显著差异表达,其中亲本中1个基因在杂种F1中沉默(Zhuang et al.,2007)。对落叶松(Larix)的杂种优势研究表明,杂交种F1与亲本之间有54个差异显著基因为非加性表达模式,且这些基因参与生理过程、应激反应及淀粉和蔗糖代谢等多种重要的生物学功能(Li et al.,2012)。因此,深入研究杂种F1基因差异表达模式,从分子水平上揭示杂种优势形成机制已经成为新的研究热点。
转录组学是在RNA水平上研究生物体中各基因转录情况及其调控规律,用以阐明生物表型与功能的一门科学。随着第2代深度测序技术的飞速发展,采用转录组测序技术更能精确、全面地深入研究杂种F1基因差异表达模式,揭示杂种优势形成的分子基础。利用RNA-Seq技术检测杂交水稻(Oryza sativa)的等位基因表达,发现杂种F1中存在基因依赖性表达现象。亲本和子代的差异表达主要表现在等位基因的特异性表达,在杂种F1中存在显著的等位基因互补效应(Song et al.,2013)。对异源六倍体小麦(Triticum aestivum)杂种优势研究显示,非加性表达的编码蛋白基因与生长活性相关,父母本中高水平表达的编码蛋白的优势基因及同源基因与后代表达水平相似,并可能参与发育或适应性(Li et al.,2014)。
杨树作为林木的模式树种,其杂种优势利用与遗传机制研究已被探索了1个多世纪,但其分子机制仍不明确。美洲黑杨是我国杨树杂交育种中重要的基因供体,目前通过种内杂交,已选育生长显著超过亲本,且水分、养分利用效率高的新品种(高暝等,2014; 2013)。本实验室前期通过甲基化免疫共沉淀测序技术(MeDIP-seq),在全基因组范围内比较亲本与杂种F1的DNA甲基化组差异,杂种F1与亲本之间具有显著不同的甲基化组; 超高亲杂种F1的甲基化水平高于双亲平均值,呈现出DNA甲基化非加性遗传(Gao et al.,2014)。本研究在实验室前期研究基础上,采用Illumina HiSeq 2000高通量测序技术进行转录组测序,通过比较杂种F1与亲本转录组间差异,获得大量差异表达基因,并对差异表达基因进行功能注释和代谢通路分析,为深入研究杂种F1的基因表达模式,从基因表达水平揭示杂种优势形成机制奠定分子基础。
1 材料与方法 1.1 试验材料采用5年生的美洲黑杨50号杨(P. deltoides cv. '55/65’)(母本,编号为Q1)和美洲黑杨10/17杨(P. deltoides cl.'10/17’)(高暝等,2013)(父本,编号为Q2)及其种内杂种F1代中3个生长超过父母本无性系(编号为H1,H2,H3)和2个生长低于父母本的无性系(编号为L1,L2)共7个美洲黑杨无性系为试验材料(表 1)。
|
|
2002年在中国林业科学研究院温室通过父母本花枝水培和人工控制授粉获得杂种F1代种子,2003年在中国林业科学研究院玉泉山苗圃对生长1年的实生苗进行扦插扩繁,2004年将获得的149个杂种F1及亲本引入河南省焦作市农林科学研究院进行苗期观察,2005年选取表现优良的18个杂种F1和亲本在河南省焦作市武陟县西峰村种植,2007年移至河南省焦作市武陟县阳城(35°8' N,113°17' E)栽植,采用随机区组设计,4次重复,每次重复4~8株不等,株行距为3 m×5 m。本研究于2011年8月美洲黑杨生长最旺盛期采集主干顶端幼嫩叶片(处于生长活跃中心,具代表性),每系号每次重复选取3株,混合制样后迅速放置液氮中速冻保存,以备RNA提取。
1.2 试验方法 1.2.1 cDNA文库准备及RNA-Seq测序采用Trizol法提取杨树叶片总RNA,按Dynabeads mRNA Purification Kit分离纯化mRNA(Invitrogen)。用破碎缓冲液将mRNA打断成短片段,用随机引物和逆转录酶将其反转成双链cDNA。末端修复、加'A’碱基及测序接头,电泳回收300 bp大小的DNA片段,PCR扩增富集测序样本。采用Illumina Hiseq2000测序平台的双端100 bp测序模式对2样本的测序文库进行高通量测序。
1.2.2 序列比对及差异表达基因分析 预处理后序列与毛果杨参考基因组数据库(http://genome.jgi-psf.org/Poptr1/Poptr1.home.html/)进行比对。根据与参考基因组比对情况计算序列的全基因组覆盖情况并计算RNA的表达量。RNA表达量参考基因区域内匹配read标准化模式(cufflink软件)采用"每百万比配序列中每千个碱基对长度上的片段"(Fragments Per Kilobase of exon model per Million mapped reads,FPKM)计算(Mortazavi et al.,2008)。对父本和母本、超亲杂种F1(H1,H2,H3)、低亲杂种F1(L1,L2)基因表达量进行相关性分析拟合,形成亲本、超亲杂种F1及低亲杂种F1 3组数据。差异分析对基因和基因异构体层面分别进行计算。将试验样本(条件)之间的log ratio与其中某个条件表达量的log值做比较,选择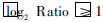 和FDR(false discovery rate)≤0.05作为显著性标准阈值。
和FDR(false discovery rate)≤0.05作为显著性标准阈值。
利用Gene Ontology数据库中毛果杨基因组注释数据库(http://genome.jgi-psf.org/programs/plants/index.jsf)及真核生物直系同源基因注释系统(KOG)中毛果杨基因组注释信息(http://www.phytozome.net/poplar)对差异基因进行功能注释及分类; 利用KEGG数据库对差异表达基因进行代谢通路注释分析,采用Fisher检验计算差异表达基因的富集度,classic Fisher≤0.01作为筛选阈值。
采用SwissProt与Uniprot非冗余蛋白质数据库(UniProt: http://www.uniprot.org/)针对差异表达基因进行同源蛋白的比对和注释分析,选择e value<1E-5,作为筛选阈值。
2 结果与分析 2.1 转录组测序及参考基因组比对使用Illumina HiSeq 2000高通量测序技术对7个美洲黑杨的cDNA文库测序,共产生171 154 127条Reads(平均长度200 bp,约31.32 Gb)(表 2)。每个样品平均产出24 450 000余条Reads,经引物、adaptor序列去除及测序片段3'端质量检验,最终获得Reads占原始数据量的99%以上。将处理后Reads与毛果杨参考基因组数据库(http://genome.jgi-psf.org/Poptr1/Poptr1.home.html)比对,同时对不能匹配Reads进行序列分割,充分利用可变剪接junction形成区域的测序片段与参考基因组比对,合计有61.89%的Reads可以与参考基因组匹配,37.11%的Reads与参考基因组不能匹配。
|
|
采用FPKM计算基因表达量,能消除基因长度和测序量差异对计算基因表达的影响。mRNA的表达具有异质性和冗余2个显著特点。当大多数mRNA的表达水平低时,仍有一小部分的mRNA高度表达。因此,样本内基因表达密度分布可用来评估RNA-Seq数据的正常性。由图 1可知,7个样品的RNA测序的基因表达密度分布相似,遵循双峰分布(Hebenstreit et al.,2011)。此外,对基因覆盖度统计(图 2)发现,7个样品RNA测序文库的基因覆盖度分布模式基本相似,超过20%的测序序列基因覆盖度超过50%,大约有80%的序列基因覆盖度范围相似。以上结果说明本试验测序数据正常,可用于下一步分析。
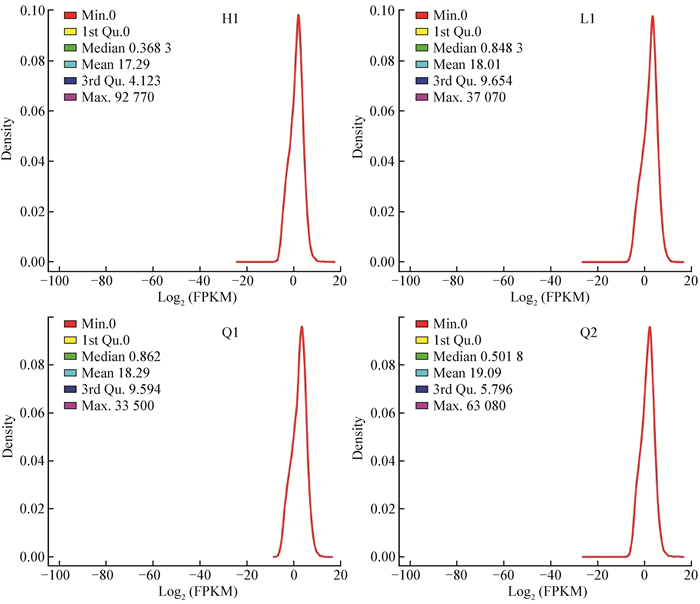 |
图 1 样品内基因表达密度分布
Fig. 1 The density distribution of gene expression among the samples
对各样本内表达量的对数(取2为底的对数,log2)的密度分布进行统计,并计算表达量分布参数(中位数、均值以及分位数等)。表达量为FPKM法计算所得数值。 The expression was represented by the FPKM value (log2). The median, mean and fractile numbers were counted. |
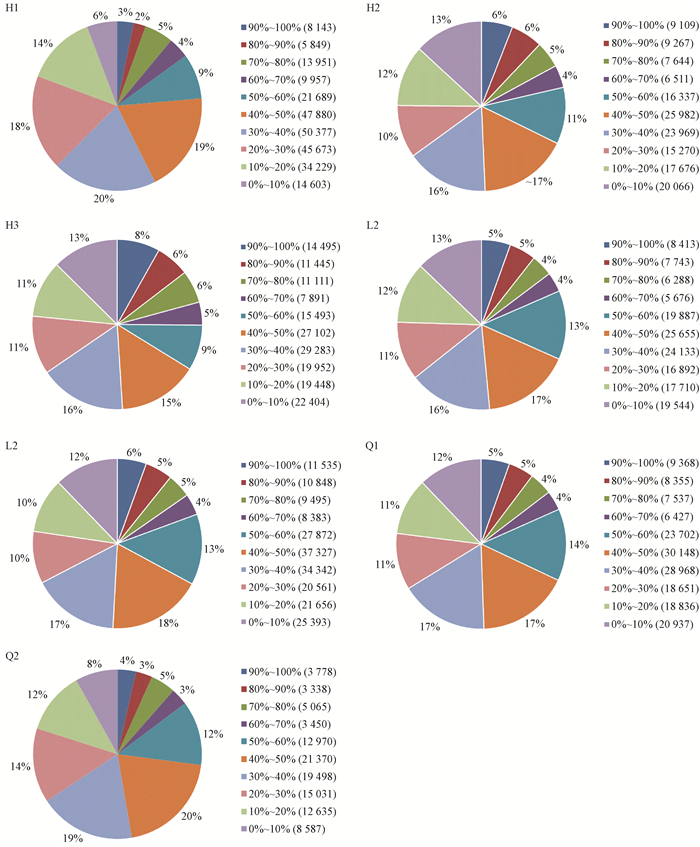 |
图 2 基因覆盖度统计
Fig. 2 Statistics for the gene coverages
基因覆盖度指每个基因被reads覆盖的百分比,其值等于基因中被唯一比对的reads覆盖的碱基数跟基因编码区所有碱基数的比值。图中不同颜色代表具有某一覆盖度范围的基因所占的比例。 Gene coverage is the percentage of the genes covered by the mapped reads, calculated as the ratio of the number of covered bases to the number of total bases in the coding regions. Different colors indicate the proportion of the coverage of specific coverage range. |
通过将测序序列进行拼接后,7样品中平均检出11 067条基因表达,参考KOG注释系统中毛果杨基因组注释信息(http://www.phytozome.net/poplar),对检出基因进行功能注释,发现7样品中平均有7 642个基因被注释,涉及2 500个基因功能(表 3)。通过进一步统计分析发现,在3个超双亲杂种F1中共表达基因有9 470个,在2个低双亲杂种F1中共表达基因9 582条,在亲本中共表达基因9 722条(图 3)。
|
|
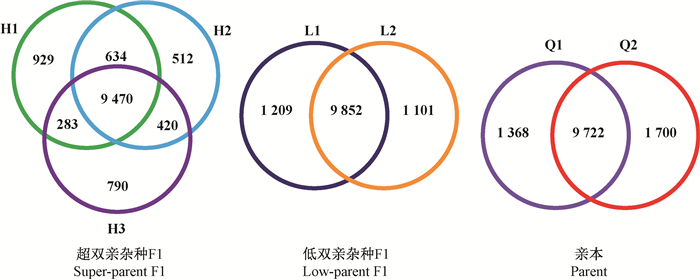 |
图 3 维恩图分析显示超双亲F1、低双亲F1及亲本中基因表达
Fig. 3 The expression profile among the super-parent hybrids, low-parent hybrids and parents illustrated by Venn diagram
|
以生长超双亲杂种F1代H1,H2,H3作为超亲杂种H,以低双亲杂种F1代L1,L2作为低双亲杂种L,以母本、父本作为亲本Q,根据基因FPKM值计算比较超亲杂种、低亲杂种及亲本间差异基因表达情况,选择差异倍数为2及FDR(false discovery rate)≤0.001作为显著性标准阈值。超亲杂种相对亲本筛选出342个基因显著差异表达,其中上调表达87个基因,下调表达255个基因; 低亲杂种相对亲本共筛选出577个基因差异表达,其中上调表达基因146个,下调表达基因431个; 超亲杂种相对低亲杂种共筛选出486个基因显著差异表达,其中上调表达基因200个,下调表达基因286个(图 4)。
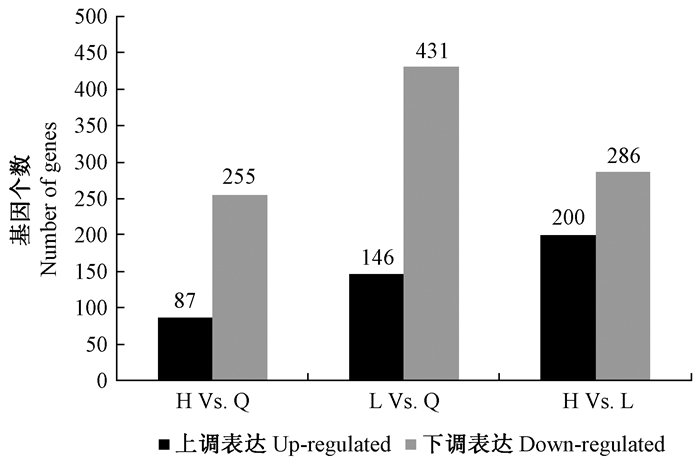 |
图 4 超双亲F1(H)、低双亲F1(L)及亲本
(Q)中基因表达差异
Fig. 4 Differential expressed gene among super-parent hybrids(H), low-parent hybrids(L) and parents(Q)
|
将3个对比组获得的差异显著基因进行统计分析(图 5)发现,与亲本相比,具有催化活性的糖基转移酶(Potri.008G101000)、单加氧酶(Potri.019G003500)、谷胱甘肽转移酶(Potri.016G083500.1)及抗病蛋白基因(Potri.T002100.1)在超亲F1及低亲F1中均为下调表达,且在超亲F1中这些基因下调表达倍数约为低亲F1的2倍左右; 磷脂酶基因(Potri.019G014900.1)超亲F1中为上调表达,但在低亲F1为下调表达(表 4)。有129个差异基因在超亲F1 Vs 亲本及超亲F1 Vs低亲F1 2个对比组中显著表达,其中19个差异基因显著上调表达,差异倍数为1倍左右(表 5),110个差异基因显著下调表达; 通过功能注释发现,这些基因主要为具有调控功能的生长调节因子(Potri.012G022600.1)、锌指蛋白(Potri.006G245400.1)、MADS-box(Potri.003G170000.1)等转录因子基因,植物防御功能的抗病蛋白基因(Potri.001G422800.2,Potri.015G025300.1等),具有信号传导、物质转运等功能的酯酶、磷酸酶(Potri.010G151000.1,Potri.013G133800.1,Potri.006G104100.1等)及材性改良相关的纤维素合成基因(Potri.019G057500.1)。此外,有72个差异基因在超亲F1 Vs亲本及低亲F1 Vs 亲本2个对比组中均显著差异表达,且各差异基因整体为显著下调表达,但这些基因在超亲F1中与低亲F1中差异不显著; 有177个基因仅在低亲F1 Vs 亲本及超亲F1 Vs低亲F1中显著差异表达。由此可以看出,杂种F1中基因差异表达来自亲本的非加性遗传。
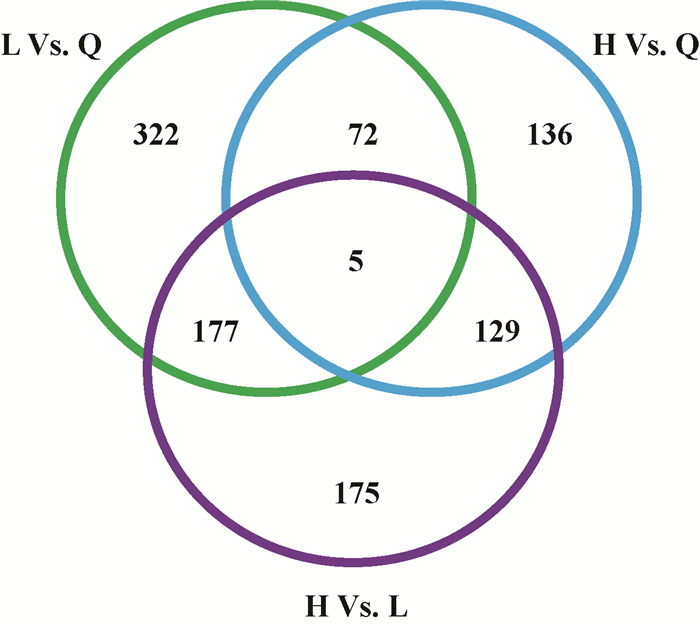 |
图 5 超亲F1(H)、低亲F1(L)及亲本(Q)中差异表达显著基因
Fig. 5 Significantly differentially expressed genes among super-parent hybrids(H), low-parent hybrids(L) and parents(Q)
|
|
|
|
|
使用Gene Ontology数据库以及毛果杨基因组注释数据库对3个对比组获得的差异显著基因功能富集及注释分析。分析显示(图 6)超亲杂种F1与亲本的显著差异表达基因中有167个基因能被功能注释且主要涉及46个生物学功能,在分子功能(molecular function,MF)、生物学过程(biological process,BP)、细胞组分(cellular component,CC)3种GO分类中,分别富集21,24和1种生物功能,其中在"催化活性"功能中富集基因最多(162个),具有"氧化还原酶活性"功能的基因数量次之(49个); 超亲杂种F1与低亲杂种F1的差异表达显著基因中有233个基因能被功能注释且主要涉及45个生物学功能,在3种GO(MF,BP,CC)分类中,分别富集15,26和4种生物功能,其中在"催化活性"功能中富集基因最多(223个),在"胺的代谢过程"及"氧化还原酶活性"功能中富集基因次之; 低亲杂种F1与亲本的差异表达显著基因中有280个基因能被功能注释且主要涉及51个生物学功能,在3种GO(MF,BP,CC)分类中,分别富集23,23和5种生物功能,其中在 "催化活性"功能中富集基因最多(218个),在"转移酶活性"、"核酸结合"及"磷酸盐代谢过程"功能中次之。
 |
图 6 差异表达基因GO分析结果
Fig. 6 GO analysis of the differentially expressed genes
A.1-15: 分子功能; 16-41: 生物学过程; 42-45: 细胞组分 1.水解酶活性,作用于糖基体; 2.催化活性; 3.水解酶活性,作用于糖基化合物; 4.裂解酶的活性; 5.结合模式; 6.几丁质酶活性; 7.β-葡糖苷酶活性; 8. 电子载体活动; 9.氧化还原酶活性,作用于CH—OH基团; 10.转移酶活性,转移烷基或芳基(不是甲基)基团; 11.碳-氮裂解酶的活性; 12.酸氨(或酰胺)连接酶活性; 13.多糖结合; 14.L-谷氨酸依赖性甲基转移酶活性; 15.苯丙氨酸氨-裂合酶活性; 16.细胞氨基酸代谢过程; 17.氨基聚糖分解代谢过程; 18.几丁质代谢过程; 19.有机酸代谢过程; 20.L-苯丙氨酸代谢过程; 21.细胞氨基酸生物合成过程; 22.细胞氨基酸分解代谢过程; 23.芳香族氨基酸家族代谢过程; 24.芳香族氨基酸家族分解代谢过程; 25.胺代谢过程; 26.胺合成过程; 27.胺分解代谢过程; 28.有机生物合成过程; 29.有机酸分解代谢过程; 30.芳香族化合物生物合成过程; 31.芳香族化合物分解代谢的过程; 32.光合作用,光反应; 33.羧酸的代谢过程; 34.细胞酮代谢过程; 35.含氧酸代谢过程; 36.二羧酸的代谢过程; 37.细胞胺代谢过程; 38.细胞氮化合物生物合成过程; 39.羧酸合成过程; 40. 羧酸分解代谢过程; 41.分支酸代谢过程; 42.光系统I; 43.类囊体; 44.光合膜; 45. 类囊体部分 1-15: Molecular function; 16-41: Biological process; 42-45:Cellular component 1. Hydrolase activity, acting on glycosyl bonds; 2. Catalytic activity; 3. Hydrolase activity, hydrolyzing O-glycosyl compounds; 4. Lyase activity; 5. Pattern binding; 6. Chitinase activity; 7. Beta-glucosidase activity; 8. Electron carrier activity; 9. Oxidoreductase activity, acting on CH—OH group of donors; 10. Transferase activity, transferring alkyl or aryl (other than methyl) groups; 11. Carbon-nitrogen lyase activity; 12. Acid-ammonia (or amide) ligase activity; 13. Polysaccharide binding; 14. 5-methyltetrahydropteroyltri-L-glutamate-dependent methyltransferase activity; 15. Phenylalanine ammonia-lyase activity; 16. Cellular amino acid metabolic process; 17. Aminoglycan catabolic process; 18. Chitin metabolic process; 19. Organic acid metabolic process; 20. L-phenylalanine catabolic process; 21. Cellular amino acid biosynthetic process; 22. Cellular amino acid catabolic process; 23. Aromatic amino acid family metabolic process; 24. Aromatic amino acid family catabolic process; 25. Amine metabolic process; 26. Amine biosynthetic process; 27. Amine catabolic process; 28. Organic acid biosynthetic process; 29. Organic acid catabolic process; 30. Aromatic compound biosynthetic process; 31. Aromatic compound catabolic process; 32. Photosynthesis, light reaction; 33. Carboxylic acid metabolic process; 34. Cellular ketone metabolic process; 35. Oxoacid metabolic process; 36. Dicarboxylic acid metabolic process; 37. Cellular amine metabolic process; 38. Cellular nitrogen compound biosynthetic process; 39. Carboxylic acid biosynthetic process; 40. Carboxylic acid catabolic process; 41. Chorismate metabolic process; 42. Photosystem I; 43. Thylakoid; 44. Photosynthetic membrane; 45. Thylakoid part B.1-23: 分子功能; 24-46: 生物学过程; 47-51: 细胞组分 1.水解酶活性,作用于糖基体; 2.腺嘌呤核苷酸结合; 3.氧化还原酶活性; 4.抗氧化活性; 5.磷酸转移酶活性,醇基团作为受体; 6.嘌呤核苷三磷酸结合; 7.激酶活性; 8.催化活性; 9.转移酶活性; 10.核苷酸结合; 11.环核苷酸依赖的蛋白激酶活性; 12.阴离子跨膜转运活动; 13.电子载体活动; 14.无机阴离子跨膜转运活动; 15.硫酸盐跨膜转运活动; 16.叶绿素结合; 17.氧化还原酶活性,作用于超氧自由基的受体; 18.转移酶活性,转移烷基或芳基(不是甲基)基团; 19.转移酶活性,转移含磷基团; 20.嘌呤核苷酸结合; 21.激酶调节活性; 22.核糖核苷酸结合; 23.嘌呤核苷酸结合; 24.磷酸化; 25.运输; 26.代前体的代谢物和能量; 27.离子转运; 28.磷代谢过程; 29.氨基聚糖分解代谢过程; 30.几丁质代谢过程; 31.磷代谢过程; 32.铁离子转运; 33.细胞离子平衡; 34.细胞金属离子平衡; 35.光系统Ⅱ光合电子传递; 36.无机阴离子转运; 37.光合作用,光反应; 38.电子传递链; 39.细胞阳离子平衡; 40.化学平衡; 41.离子平衡; 42.金属离子平衡; 43.铁离子平衡; 44.阳离子平衡; 45.细胞的化学平衡; 46.活性氧代谢过程; 47.内在膜; 48.类囊体; 49.光合膜; 50.膜部分; 51.类囊体部分 1-23: Molecular function; 24-46: Biological process; 47-51: Cellular component 1. Hydrolase activity, acting on glycosyl bonds; 2. Adenyl nucleotide binding; 3. Oxidoreductase activity; 4. Antioxidant activity; 5. Phosphotransferase activity, alcohol group as acceptor; 6. Purine ribonucleoside triphosphate binding; 7. Kinase activity; 8. Catalytic activity; 9. Transferase activity; 10. Nucleotide binding; 11. Cyclic nucleotide-dependent protein kinase activity; 12. Anion transmembrane transporter activity; 13. Electron carrier activity; 14. Inorganic anion transmembrane transporter activity; 15. Sulfate transmembrane transporter activity; 16. Chlorophyll binding; 17. Oxidoreductase activity, acting on superoxide radicals as acceptor; 18. Transferase activity, transferring alkyl or aryl (other than methyl) groups; 19. Transferase activity, transferring phosphorus-containing groups; 20. Purine nucleotide binding; 21. Kinase regulator activity; 22. Ribonucleotide binding; 23. Purine ribonucleotide binding; 24. Phosphorylation; 25. Transport; 26. Generation of precursor metabolites and energy; 27. Ion transport; 28. Phosphate metabolic process; 29. Aminoglycan catabolic process; 30. Chitin metabolic process; 31. Phosphorus metabolic process; 32. Iron ion transport; 33. Cellular ion homeostasis; 34. Cellular metal ion homeostasis; 35. Photosynthetic electron transport in photosystem Ⅱ; 36. Inorganic anion transport; 37. Photosynthesis, light reaction; 38. Electron transport chain; 39. Cellular cation homeostasis; 40. Chemical homeostasis; 41. Ion homeostasis; 42. Metal ion homeostasis; 43. Iron ion homeostasis; 44. Cation homeostasis; 45. Cellular chemical homeostasis; 46. Reactive oxygen species metabolic process; 47. Intrinsic to membrane; 48. Thylakoid; 49. Photosynthetic membrane; 50. Membrane part; 51. Thylakoid part C.1-21: 分子功能; 22-45: 生物学过程; 46: 细胞组分 1.氧化还原酶活性; 2.催化活性; 3.结合模式; 4. 3-脱氧-7-磷酸合酶活性; 5.谷胱甘肽转移酶活动; 6.水解酶活性,水解成糖基化合物; 7.几丁质酶的活性; 8. S-甲基转移酶活性; 9.辅酶A连接酶活性; 10.氧化还原酶活性,作用于CH—CH供体,NAD或NADP受体; 11.氧化还原酶活性,作用于硫基团供体; 12.氧化还原酶活性,作用于二酚及相关物质的供体,氧离子受体; 13.氧化还原酶活性,作用于配对供体,掺入或减少氧分子; 14.氧化还原酶活性,作用于配对供体,掺入1个氧原子或者还原分子氧,NADH或NADPH作为供体和参与氧还原; 15.水解酶活性,作用于糖基体; 16.碳-氮裂解酶活性; 17.连接酶活性,形成碳-硫键; 18.酸-硫醇连接酶活性; 19.酸氨(或酰胺)连接酶活性; 20.多糖结合; 21.苯丙氨酸解氨酶活性; 22.细胞氨基酸代谢过程; 23.多糖分解代谢过程; 24.氨基糖基代谢过程; 25.氨基糖基分解过程; 26.几丁质代谢过程; 27.有机酸代谢过程; 28. L-苯丙氨酸代谢过程; 29.细胞氨基酸分解代谢过程; 30.芳香族氨基酸的家族代谢过程; 31.芳香族氨基酸的家族分解代谢过程; 32.胺代谢过程; 33.胺分解代谢过程; 34.碳水化合物分解代谢过程; 35.有机酸分解代谢过程; 36.芳香族化合物分解代谢的过程; 37.羧酸的代谢过程; 38.细胞酮代谢过程; 39.含氧酸代谢过程; 40.细胞壁大分子代谢过程; 41.细胞胺代谢过程; 42.羧酸分解代谢过程; 43.分支酸代谢过程; 44.细胞壁组织或生物合成; 45.细胞组分组织或生物合成; 46.细胞器核糖体 1-21: Molecular function; 22-45:Biological process; 46: Cellular component 1. Oxidoreductase activity; 2. Catalytic activity; 3. Pattern binding; 4. 3-deoxy-7-phosphoheptulonate synthase activity; 5. Glutathione transferase activity; 6. Hydrolase activity, hydrolyzing O-glycosyl compounds; 7. Chitinase activity; 8. S-methyltransferase activity; 9. CoA-ligase activity; 10. Oxidoreductase activity, acting on the CH—CH group of donors, NAD or NADP as acceptor; 11. Oxidoreductase activity, acting on a sulfur group of donors; 12. Oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor; 13. Oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen; 14. Oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen, NADH or NADPH as one donor, and incorporation of one atom of oxygen; 15. Hydrolase activity, acting on glycosyl bonds; 16. Carbon-nitrogen lyase activity; 17. Ligase activity, forming carbon-sulfur bonds; 18. Acid-thiol ligase activity; 19. Acid-ammonia (or amide) ligase activity; 20. Polysaccharide binding; 21. Phenylalanine ammonia-lyase activity; 22. Cellular amino acid metabolic process; 23. Polysaccharide catabolic process; 24. Aminoglycan metabolic process; 25. Aminoglycan catabolic process; 26. Chitin metabolic process; 27. Organic acid metabolic process; 28. L-phenylalanine metabolic process; 29. Cellular amino acid catabolic process; 30. Aromatic amino acid family metabolic process; 31. Aromatic amino acid family catabolic process; 32. Amine metabolic process; 33. Amine catabolic process; 34. Carbohydrate catabolic process; 35. Organic acid catabolic process; 36. Aromatic compound catabolic process; 37. Carboxylic acid metabolic process; 38. Cellular ketone metabolic process; 39. Oxoacid metabolic process; 40. Cell wall macromolecule metabolic process; 41. Cellular amine metabolic process; 42. Carboxylic acid catabolic process; 43. Chorismate metabolic process; 44. Cell wall organization or biogenesis; 45. Cellular component organization or biogenesis; 46. Organellar ribosome |
为进一步了解差异表达显著基因参与代谢通路,将所有差异表达基因比对到KEGG数据库进行pathway富集分析。经比对发现,有64,46和136个基因分别富集到34(超亲F1 Vs亲本)、30(低亲F1 Vs亲本)和46(超亲F1 Vs低亲F1)个代谢途径。各对比组差异基因主要富集在碳水化合物代谢、氨基酸代谢、能量代谢途径及外来物质的降解和代谢,其中超亲F1 Vs低亲F1中差异表达基因参与各类代谢途径最多。此外,在超亲F1 Vs低亲F1与低亲F1 Vs亲本中差异表达基因还参与类脂(化合)物代谢、辅助因子和维生素代谢。
3 讨论本文基于Illumina Hiseq 2000高通量测序技术分析比较了5个不同生长势美洲黑杨及其亲本的转录组,在3个对比组超亲杂种F1 Vs亲本、超亲杂种F1 Vs低亲杂种F1、低亲杂种F1 Vs亲本分别筛选出342、486和577个差异表达基因,其中被功能注释的差异显著基因,均在"催化活性"功能中富集最多,此外还主要涉及"氧化还原酶活性"、"胺的代谢过程"、"转移酶活性"、"核酸结合"及"磷酸盐代谢过程"等功能。进一步分析发现差异基因主要富集在碳水化合物代谢、氨基酸代谢、能量代谢途径及外来物质的降解和代谢,其中超亲F1 Vs低亲F1中差异显著基因参与各类代谢途径最多。前期研究发现,超亲杂种F1的叶片δ13C值和碳、氮含量、水分利用效率均显著大于低亲杂种F1,其光合叶面积、光合生产率、叶绿素含量、RuBP羧化酶活性等光合特性指标均超过亲本(高暝等,2014; 2013),这可能与参与光合作用、物质代谢吸收等重要代谢途径相关基因显著差异表达有关,进而促进杂种优势的产生。
重要生物学功能基因的差异显著表达可能对杂种优势产生具有重要作用。在本研究中有129个差异基因在超亲F1 Vs亲本及超亲F1 Vs低亲F1 2个对比组中显著表达,其中显著上调表达1倍以上的19个差异基因主要对生长发育调控、信号传导、物质转运以及植物防御等生物过程具有重要作用,如锌指蛋白转录因子主要参与调控一些重要的生理过程,如花的发育、光形态建成以及对胁迫的响应等方面(Takatsuji,1998),MADS-box在植物中主要参与花、根、茎、叶等器官的生长发育调控并在信号传导中发挥着重要作用,扩展蛋白基因(expansin)在细胞壁扩张过程中起着重要作用,对于调控植物生长至关重要。这些基因在超亲F1中的上调表达可能对杂种优势的产生具有正向效应; 此外显著下调表达的110个差异基因可能对杂种优势具有负向效应。有研究表明,杂种优势产生过程中并不仅仅是一些基因表达增强是有利的,而同时某些基因受到抑制也是有利的(姜涛等,2013)。结合本研究结果,推测一些具有生长发育调控、信号转导、物质转运及植物防御等生物学功能基因的大量差异表达,对光合作用、物质代谢、氧化还原等与生长紧密联系的代谢途径中有效物质发生正向或负向效应,促使林木生长等性状杂种优势的形成,且差异基因的上调或下调表达可能对杂种优势产生都具有重要作用。这些差异基因的发掘及后续功能验证将为深入揭示杂种优势形成分子机制提供一定基础。
转录组是特定组织或细胞在某一发育阶段或功能状态下转录出来的所有RNA的集合,具有时间性和空间性(祁云霞等,2011)。如果采样期或部位不同,结果可能会有所差异,对于杂种优势研究而言,主要是选择好性状优势形成的关键时期和组织部位,如本研究选取对生长性状形成具有至关重要作用的"生长最旺盛期"以及关键生长中心"主干顶端幼嫩叶片"取样研究,结果具有代表性。当然,本文研究的仅是是美洲黑杨成龄大树的杂种优势表现,由于林木生长周期长,幼苗到成龄期间生长性状的杂种优势表现可能会发生波动,从而影响不同生长势子代与亲本间以及子代间转录组差异性,为此将进行苗期回溯研究和有目的的跟踪研究。
杂种优势是复杂的遗传现象,影响其产生的因素很多。目前对其形成机制的研究大多集中在亲本遗传差异与子代杂种优势的相关性上,而对子代杂合度与杂种优势的关系研究较少。从仅有的研究结果来看,杂种优势与子代杂合度存在一定的相关性(郑康乐等,1999; 程昕昕等,2007; 王晓阳等,2011)。因此,需要采用多种现代生物技术手段从基因组、转录组、蛋白组、代谢组等多个水平开展深入研究,以期更为全面地揭示杂种优势成因,从而更好地指导杂交育种亲本的选配和子代的选优,提高育种效率和效果。
4 结论为了解杨树杂种优势形成机制,本文进行了不同生长势美洲黑杨及其亲本的转录组测序和差异分析,结果显示不同生长势美洲黑杨及其亲本间存在着大量差异表达的基因,主要集中在参与与生长关系密切的重要代谢途径,据此认为杂种优势的形成,可能是由于相关基因的显著差异表达,调控光合作用、物质代谢吸收等与生长紧密联系的代谢活动,进而促进生长优势的产生。研究从转录组水平初步揭示了基因差异表达与杂种优势的关系,有关结果对深入研究杂种优势形成机制从而深度开发杨树杂种优势以创制超高产优良新品种具有参考价值。
| [1] |
程昕昕,耿广汉,刘正. 2007.过氧化物酶杂合性与玉米F1产量性状相关性分析.中国农学通报, 23(2):271-274. (Cheng X X, Geng G H, Liu Z. 2007. Relationship of peroxidase diversity with Fl yield in maize. Chinese Agricultural Science Bulletin, 23(2):271-274[in Chinese]).(  1) 1)
|
| [2] |
高暝,丁昌俊,苏晓华,等. 2014.美洲黑杨及其杂种F1无性系光合特性的研究.林业科学研究, 27(6):721-728. (Gao M, Ding C J, Su X H, et al. 2014. Comparison of photosynthetic characteristics of Populus deltoides and their hybrid clones. Forest Research, 27(6):721-728[in Chinese]).(  2) 2)
|
| [3] |
高暝,黄秦军,丁昌俊,等. 2013.美洲黑杨及其杂种F1不同生长势无性系叶片δ13C和氮素利用效率.林业科学, 49(8):51-57. (Gao M, Huang Q J, Ding C J, et al. 2013. Foliar δ13C and nitrogen use efficient of Populus deltoides and the different growth vigor F1 hybrid clones. Scientia Silvae Sinicae, 49(8):51-57[in Chinese]).(  3) 3)
|
| [4] |
姜涛,王丕武,张君. 2013.玉米mRNA差异显示与杂种优势关系的研究.玉米科学, 21(5):41-45. (Jiang T, Wang P W, Zhang J. 2013. Relationship between mRNA differential gene expression and heterosis in maize. Journal of Maize Sciences, 21(5):41-45[in Chinese]).(  1) 1)
|
| [5] |
祁云霞,刘永斌,荣威恒. 2011.转录组研究新技术:RNA-Seq及其应用.遗传, 33(11):1191-1202. (Qi Y X, Liu Y B, Rong W H. 2011. RNA-Seq and its applications:A new technology for transcriptomics. Hereditas(Beijing), 33(11):1191-1202[in Chinese]).(  1) 1)
|
| [6] |
王晓阳,李火根. 2011.鹅掌楸苗期生长杂种优势的SSR分析.林业科学, 47(4):57-62. (Wang X Y, Li H G. 2011. Possible mechanism analysis for heterosis of hybrid Liriodendron based on seedling growth and SSR markers. Scientia Silvae Sinicae, 47(4):57-62[in Chinese]).(  1) 1)
|
| [7] |
郑康乐,庄杰云,樊叶杨,等. 1999.杂交水稻产量性状的上位性效应及杂合性与杂种优势的相关性.云南大学学报:自然科学版, (S3):47-48. (Zheng K L, Zhuang J Y, Fan Y Y, et al. 1999. Correlation of epistatic effects and heterozygosity of yield traits with heterosis in hybrid rice. Journal of Yunnan University:Natural Sciences Edition, (S3):47-48[in Chinese]).(  1) 1)
|
| [8] |
Birchler J A, Auger D L, Riddle N C. 2003. In search of the molecular basis of heterosis. The Plant Cell, 15(10):2236-2239.( 2) 2)
|
| [9] |
Birchler J A, Yao H, Chudalayandi S, et al. 2010. Heterosis. Plant Cell, 22(7):2105-2112.( 1) 1)
|
| [10] |
Bruce A B. 1910. The Mendelian theory of heredity and the augmentation of vigor. Science, 32(827):627-628.( 1) 1)
|
| [11] |
Chen Z J. 2010. Molecular mechanisms of polyploidy and hybrid vigor. Trends in Plant Science, 15(2):57-71.( 1) 1)
|
| [12] |
Gao M, Huang Q J, Chu Y G, et al. 2014. Analysis of the leaf methylomes of parents and their hybrids provides new insight into hybrid vigor in Populus deltoides. BMC Genetics, 15(Suppl 1):S8.( 1) 1)
|
| [13] |
Hebenstreit D, Fang M, Gu M, et al. 2011. RNA sequencing reveals two major classes of gene expression levels in metazoan cells. Molecular Systems Biology, 7:497.( 1) 1)
|
| [14] |
Li A, Fang M, Song W, et al. 2012. Gene expression profiles of two intraspecific Larix lines and their reciprocal hybrids. Molecular Biology Reports, 39(4):3773-3784.( 1) 1)
|
| [15] |
Li A, Liu D, Wu J, et al. 2014. mRNA and small RNA transcriptomes reveal insights into dynamic homoeolog regulation of allopolyploid heterosis in nascent hexaploid wheat. Plant Cell, 26(5):1878-1900.( 1) 1)
|
| [16] |
Mortazavi A, Williams B A, Mccue K, et al. 2008. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 5(7):621-628.( 1) 1)
|
| [17] |
Shull G H. 1914. Duplicate genes for capsule-form in Bursa bursa-pastoris. Molecular and General Genetics, 12(1):97-149.( 1) 1)
|
| [18] |
Song G, Guo Z, Liu Z, et al. 2013. Global RNA sequencing reveals that genotype-dependent allele-specific expression contributes to differential expression in rice F1 hybrids. BMC Plant Biology, 13(1):221.( 1) 1)
|
| [19] |
Swanson-Wagner R A, Decook R, Jia Y, et al. 2009. Paternal dominance of trans-eQTL influences gene expression patterns in maize hybrids. Science, 326(5956):1118-1120.( 1) 1)
|
| [20] |
Swanson-Wagner R A, Jia Y, Decook R, et al. 2006. All possible modes of gene action are observed in a global comparison of gene expression in a maize F1 hybrid and its inbred parents. Proceedings of the National Academy of Sciences, 103(18):6805-6810.( 1) 1)
|
| [21] |
Zhuang Y, Adams K L. 2007. Extensive allelic variation in gene expression in Populus F1 hybrids. Genetics, 177(4):1987-1996.( 1) 1)
|
| [22] |
Takatsuji H. 1998. Zinc-finger transcription factors in plants. Cellular and Molecular Life Sciences, 54(6):582-596.( 1) 1)
|
 2016, Vol. 52
2016, Vol. 52

