文章信息
- 王杰, 李军, 仝婷婷, 邵香君, 宋丽丽, 吴蔼民
- Wang Jie, Li Jun, Tong Tingting, Shao Xiangjun, Song Lili, Wu Aimin
- 毛竹PeIRX10基因在木聚糖合成中的功能
- Functions of PeIRX10 Gene from Phyllostachys edulis in Xylan Synthesis
- 林业科学, 2016, 52(11): 79-87
- Scientia Silvae Sinicae, 2016, 52(11): 79-87.
- DOI: 10.11707/j.1001-7488.20161110
-
文章历史
- 收稿日期:2015-11-04
- 修回日期:2016-04-19
-
作者相关文章
2. 浙江农林大学 亚热带森林培育国家重点实验室培育基地 临安 311300;
3. 浙江省临海市林木种子苗木管理站 临海 317000;
4. 华南农业大学 广东省森林植物种质创新与利用重点实验室 广州 510642;
5. 浙江省临安市种苗管理总站 临安 311300
2. The Nurturing Station for the State Key Laboratory of Subtropical Silviculture Zhejiang A&F University Lin'an 311300 ;
3. Forestry Seed and Seedling Administration of Linhai, Zhejiang Province Linhai 317000 ;
4. Guangdong Key Laboratory for Innovative Development and Utilization of Forest Plant Germplasms South China Agricultural University Guangzhou 510642 ;
5. Forestry Seed and Seedling Administration of Lin'an, Zhejiang Province Lin'an 311300
由次生细胞壁构成的木质纤维素是森林木材的重要组成成分,也是非粮食类生物质的主要成分,是地球上重要的可再生植物生物质资源。木质纤维素的积累主要通过纤维素、半纤维素和木质素三大组分合成而导致次生细胞壁的次生加厚生长完成(Pauly et al., 2008)。纤维素和木质素合成机制已经有广泛深入的研究(Boerjan et al., 2003;Scheible et al., 2004;Lerouxel et al., 2006;Somerville, 2006;Pauly et al., 2010)。然而相对于纤维素和木质素来说,有关半纤维素合成的分子机制只在近年来才有揭示(Zhong et al., 2005;Hayden et al., 2007;Pena et al., 2007;Mortimer et al., 2015)。
木聚糖是双子叶植物和大多数单子叶植物次生细胞壁的主要半纤维素成分,它通过与纤维素的相互作用在植物细胞壁形成过程中起着重要作用。木聚糖主链由木糖以β-(1→4)糖苷键聚合而成,侧链由葡萄糖醛酸和/或甲基化的葡萄糖醛酸组成。其中主链中木糖基的个数在拟南芥(Arabidopsis thaliana)和小麦(Triticum aestivum)的胚乳中分别为X150(拟南芥次生壁中木聚糖主链的木糖基单元数)和X1 000~4 000(小麦胚乳中木聚糖主链木糖基单元数)(Freeman et al., 2015)。此外,一些参与合成木聚糖的糖基转移酶(GT)的相关基因已有报道(Brown et al., 2007)。目前已发现与木聚糖主链合成相关的基因主要包括GT43家族的4个成员[ IRX9(AT2g37090),IRX 9 L(AT1g27600),IRX 14(AT4g36890),IRX 14 L(AT5g67230)]和GT47家族的2个成员[IRX 10(AT1g27440),IRX 10 L(AT5g61840)](Lee et al., 2007;Pena et al., 2007;Keppler et al., 2010;Wu et al., 2010;Chiniquy et al., 2013;Hörnblad et al., 2013)。由于这些基因的突变体使得木质部出现异常而被命名为IRX (irregular xylem)。近年来,已经有一些研究表明在不同植物之间木聚糖合成基因间有很强的保守性和相似性。Zhou等(2007)研究发现毛白杨(Populus tomentosa)中PoGT 43 B为IRX 9在杨树中的同源基因,能够回补拟南芥irx 9突变体的表型。在拟南芥fra 8突变体中过量表达FRA 8的同源基因PoGT 47 C,能够回补拟南芥fra 8突变体的表型(Zhou et al., 2006;Lee et al., 2009)。
IRX 10基因作为参与半纤维素木聚糖主链延伸的关键基因,目前已有不少对该基因的分子生物学研究。已经发现,IRX 10和IRX 10 L参与了木聚糖主链的延伸,且对正常的次生壁加厚都是必要的(Brown et al., 2009;Wu et al., 2009)。Brown等(2005)通过与次生壁标记基因IRX 3的共表达分析识别了IRX 10(AT1g27440),并发现其突变体irx 10的花序和茎中木糖含量下降,木质部呈现一定程度的不规则而导致次生细胞壁缺失。Zhang等(2014)克隆分析了水稻(Oryza sativa)的1个OsGT 47 A(Os01g0926600/Os01g70190)基因,发现该基因与拟南芥IRX10和IRX10L的氨基酸序列同源性高达93%,同时过量表达OsGT 47 A能够回补irx10l(-/-)irx 10(-/-)双突植株的缺陷,表明OsGT 47 A与IRX 10具有相似功能,参与了GX (glucuronoxylan)的合成。木聚糖合成关键酶基因的体外酶活性测定很长一段时间并不成功,直到最近才有报道。Jensen等(2014)通过在毕赤酵母(Pichia pastoris)中单独异源表达拟南芥、小立碗藓(Physcomitrella patens)和洋车前草(Plantago ovata)的IRX10蛋白,在体外检测到木糖转移酶活性,并且来自小立碗藓的IRX10酶活性最高,其次是洋车前草,拟南芥的IRX10蛋白只检测到相对较弱的活性。与此同时,Urbanowicz等(2014)通过在人胚胎肾细胞系293中表达拟南芥的IRX10L蛋白,证明这个蛋白具有木糖转移酶活性,而且IRX10L比Jensen等(2014)报道的IRX10活性高很多;这可能是2个同源蛋白本身之间的差异(虽然二者之间具有86.65%相似度),也可能是由于不同的异源表达系统。
毛竹(Phyllostachys edulis)是我国种植面积最大的经济竹种,被广泛应用于食品、建材、纺织、生物质能源等领域。已经发现毛竹的快速生长过程中伴随着次生细胞壁生长,木质素和纤维素含量增加(Lin et al., 2002)。Luo等(2008)发现,苯丙氨酸解氨酶(PAL)、肉桂醇脱氢酶(CAD)、过氧化物酶(POD)等在毛竹笋木质素和纤维素的合成中起着重要的作用。但毛竹细胞壁生长过程中合成半纤维素木聚糖的相关基因研究仍未有涉及。本研究以毛竹为材料,利用RT-PCR的方法克隆得到1个类似拟南芥IRX 10(AtIRX 10)的基因,并对该基因进行了序列相似性分析;通过qRT-PCR技术分析了该基因在毛竹不同营养器官中的表达情况,并构建表达载体转化到拟南芥irx10l(-/-)irx 10(+/-)双突植株中,获得了使表型矮小、次生细胞壁生长不全、木聚糖含量减少或缺失这些缺陷明显减轻的具有irx10l(-/-)irx 10(-/-)双突背景下的转基因拟南芥植株。从而验证毛竹PeIRX 10的基因功能以及在半纤维素木聚糖合成中的作用,为进一步探讨毛竹次生细胞壁半纤维素的合成和调控机制奠定基础。
1 材料与方法 1.1 试验植物材料和种植条件本研究中所用的毛竹采自浙江临安,分别取根、茎、叶(1年生实生苗)以及花序、幼笋于液氮中速冻保存(-70 ℃),用于总RNA的提取。拟南芥用于评定毛竹中基因的生物功能。拟南芥种子的消毒、培养基和生长条件以及筛选鉴定方法参照Wu等(2009),具体方法如下:首先种子表面消毒,并播撒在MS培养基上(1%蔗糖和0.8%琼脂,用0.5 mol ·L-1 NaOH将pH调为5.8),然后在MS培养基平板上培育至发芽,最后拟南芥的种植(种植土壤为营养土:蛭石体积比3 :1)在植物培养室中进行。培养条件是:光照强度100~120 μmol ·m-2s-1,光周期为光照16 h/黑暗8 h;温度22 ℃,相对湿度60%。
1.2 毛竹PeIRX10基因表达模式检测用RNA提取试剂盒(OMEGA)提取毛竹各样品的总RNA。采用PrimeScriptⅡ1st Strand cDNA Synthesis Kit (Takara)试剂盒进行反转录,合成的样品cDNA稀释20倍后为模板用于qRT-PCR分析。根据毛竹PeIRX 10编码区序列设计引物(上游引物1: 5′-TCTTGTTTGACACCGAAGAGGAG-3′;下游引物2: 5′-AATAGCTGTCCCTGGAGGAGTTT-3′)。内参选用NTB基因(Fan et al., 2013)。qRT-PCR按照SYBR Premix Ex TaqTMⅡ(Takara)的要求配制20 μL体系,在Roche LightCyler 480 system (Roche)上进行qRT-PCR分析。具体PCR反应程序为: 95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环。采用Livak 2-ΔΔCt法(Livak, 2001)对数据进行分析。每个样品做3个生物学重复。
1.3 植物表达载体的构建与基因互补基于Gateway系统用PCR扩增毛竹PeIRX 10基因的ORF序列,扩增引物PeIRX 10-F: 5′-GGGGACAAGTTTGTACAAAAAAGCAGGCTTCATGA GGAGGTGGGTCTTGGCC-3′;PeIRX 10-R: 5′-GGGG ACCACTTTGTACAAGAAAGCTGGGTCCCAAGGCTTC AGGTCGCCCACCG-3′。将扩增产物与pDONR207连接,转化大肠杆菌(Escherichia coli) DH5α得到阳性克隆,提取质粒后进行质粒PCR鉴定并测序。将测序正确的重组质粒与Pearleygate101连接用于大肠杆菌DH5α转化,培养后提取质粒并测序。将测序正确的重组质粒转化发根农杆菌(Agrobacterium rhizogenes) C58,获得的阳性菌落用蘸花法转化到拟南芥irx10l(-/-)irx10(+/-)双突植株中[IRX10L(At5g61840, GABI_179G 11);IRX 10(At1g27440, SALK_046368)],因为拟南芥irx10l(-/-)irx 10(-/-)双突植株不育。用BASTA (250 mg ·mL-1)喷洒法筛选转基因植株。此外,由于拟南芥irx10l(-/-)irx10(+/-)双突植株的下一代会分离成3种基因型[双突纯合irx10l(-/-)irx 10(-/-)以及irx10l(-/-)irx 10(+/-)和irx10l(-/-),分离比为1 :2 :1],但只有双突纯合植株才有明显的缺陷表型,其他2种基因型的植株表型正常(Wu et al., 2009),因此用BASTA筛选到的转基因植株需鉴定植株的背景。取4周龄的转基因植株,通过PCR做表型鉴定。PCR引物IRX 10-F: 5′-CCACTCGGAGGACTTGGA-3′;IRX 10-R:5′-GGAAAAAGCCATTGAAAGGG-3′。T-DNA插入引物LBa1-F:5′-TGGTTCACGTAGTGG GCCATCG-3′(与下游引物IRX 10-R配合使用)。通过表型鉴定获得具有双突纯合背景的转基因植株。提取4周龄的3种基因型的拟南芥植株的RNA,反转录成cDNA,拟南芥的β-Actin(AT3G18780)作为内标(ACT2-F: 5′-TGCTGTTGACTACGAGCAGG-3′;ACT2-R: 5′-AACCACCGATCCAGACACTG-3′),通过半定量PCR来检测目的基因PeIRX 10在转基因植株中的过表达情况。
1.4 细胞壁单糖组分分析分别取生长8周的3种类型的拟南芥茎于液氮中充分研磨,用80%的乙醇处理之后冷冻干燥作为细胞壁组分分析的材料。细胞壁材料的处理以及单糖组分分析按照Wu等(2009)的方法。
1.5 切片及光学显微镜观察取生长8周的拟南芥基部的茎固定于FAA[38%(V/V)的甲醛5 mL、冰乙酸5 mL、70%(V/V)的乙醇90 mL]中,然后用3%(m/V)的琼脂糖包埋固定(Zhao et al., 2014),并在Leica VT1000S震动切片机上切成厚度为40 μm的横切片。接着用0.02%(m/V)的甲苯氨蓝对切片进行染色1~2 min,最后于光学显微镜下观察拍照。
1.6 木聚糖免疫定位以1.5中提到的拟南芥茎部横切切片为样本,利用多糖单克隆抗体LM10(Plantprobes http://www.plantprobes.net/)检测3种拟南芥植株细胞壁半纤维素木聚糖的分布。具体操作参照Zhao等(2014)和Zhang等(2014)的方法,步骤如下:用0.1 mol ·L-1磷酸盐缓冲液(pH7.2)洗涤切片5~10 min;用新鲜的3%脱脂牛乳浸泡切片1 h并不断吹打脱脂牛乳;去除脱脂牛乳,并用PBS缓冲液洗涤切片5 min;用大鼠抗木聚糖抗体LM10孵育切片2 h,然后用PBS缓冲液洗涤切片,以洗净未结合的一抗;用稀释50倍的FITC-羊抗大鼠抗体(Zomanbio,Cat.Z1319)孵育2 h,然后用PBS缓冲液洗涤切片10次,以洗净未结合的二抗;最后将切片固定于载玻片上,置于激光共聚焦电子显微镜(Zeiss LSM710,495 nm)下观察拍照。
1.7 数据处理与分析试验数据用Excel 2007和SPSS 17.0进行统计处理,所有数据为3次及以上重复的平均值和标准误差。采用SPSS 17.0进行邓肯氏多重差异分析。
2 结果与分析 2.1 毛竹PeIRX 10基因及编码蛋白的序列分析为了验证单子叶植物与双子叶植物半纤维素木聚糖的合成机制是否相似,克隆了毛竹PeIRX 10基因。利用植物已知的IRX 10基因序列及其编码的蛋白序列为探针,通过毛竹中已完成测序的基因序列数据库和其相应的蛋白序列数据库进行BLAST检索及比对,得到PeIRX 10(PH01004923G0080,http://www.bamboogdb.org/)基因的ORF序列,长度为1 251 bp,编码416个氨基酸,蛋白质的理论分子量为46 821 Da, 等电点为6.26。然后用Blastp程序分析PeIRX10基因编码的氨基酸,发现其与拟南芥IRX10(NP_174064.1)具有高度的相似性(83.89%),与水稻(NP_001045263.1)、玉米(Zea mays)(DAA56030.1)和高粱(Sorghum bicolor)(XP_002459016.1)等植物IRX10以及拟南芥IRX10L (NP_568941.1)氨基酸序列的相似性分别为83.41%,83.05%,84.17%和82.21%(图 1)。

|
图 1 毛竹IRX10与其他物种的IRX10和IRX10L的氨基酸序列比较 Fig.1 Multiple alignment of the deduced amino acid sequences of PeIRX10 with the sequences of related proteins from other species AtIRX10:拟南芥Arabidopsis thaliana (NP_174064.1);AtIRX10L:拟南芥Arabidopsis thaliana (NP_568941.1);PeIRX10:毛竹Phyllostachys edulis (PH01004923G0080);SbIRX10:高粱Sorghum bicolor (XP_002459016.1);OsIRX10:水稻Oryza sativa (NP_001045263.1);ZmIRX10:玉米Zea mays (DAA56030.1)。灰色阴影表示相似的氨基酸序列,黑色阴影表示相同的氨基酸序列。Grey shade showsthe similar amino acid sequence and black the same amino acid sequence. |
拟南芥IRX 10和IRX10L基因在木聚糖含量较高的导管和茎中高度表达(Wu et al., 2009)。为了研究毛竹PeIRX 10基因在不同器官中的表达水平,对毛竹根、茎、叶、花和幼笋5个部位进行表达分析,结果表明,PeIRX 10基因在毛竹的各个器官中均有表达,其中在笋中的表达水平最高,其次是茎,在叶中的表达水平最低(图 2)。表明在不同的器官中PeIRX 10基因的表达量不同。
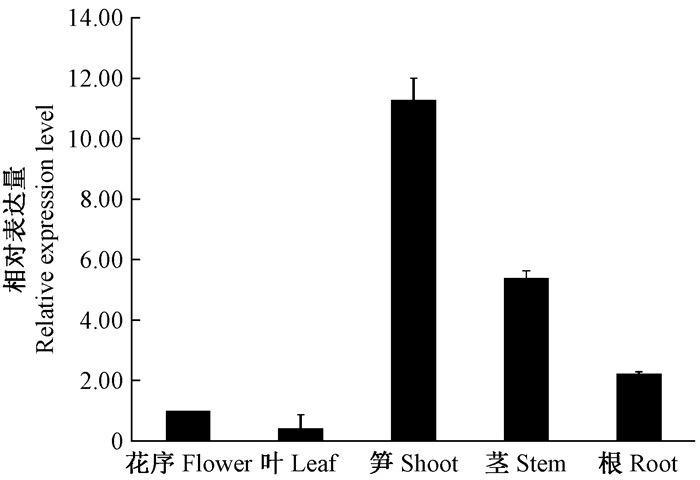
|
图 2 PeIRX 10基因的qRT-PCR分析 Fig.2 qRT-PCR analysis of PeIRX 10gene |
将PeIRX 10基因克隆后连入植物表达载体转入农杆菌,用蘸花法转化拟南芥并收取转化后的种子,经BASTA筛选后得到阳性苗,对其培养并收获种子再播种。然后分单株提取基因组DNA,分别做PCR检测(图略),以得到具有irx10l(-/-)irx10(-/-)双突背景的转基因互补植株。通过对4株转基因植株半定量检测,发现PeIRX 10基因已经转入拟南芥并表达(图 3)。在拟南芥的IRX 10和IRX10 L这组同源基因中,单基因突变植株表型不明显,但irx10l(-/-)irx10(-/-)双突植株却显示出明显的表型:植株极其矮小,生长发育迟缓,叶色浓绿,花粉完全败育(Wu et al., 2009)。PeIRX 10基因过表达的irx10l(-/-)irx10(-/-)双突拟南芥植株与野生型拟南芥相比,具有正常的株高、茎粗以及叶片大小和数量(表 1,图 4)。表明毛竹PeIRX 10基因与拟南芥的AtIRX 10基因具有相似的功能。
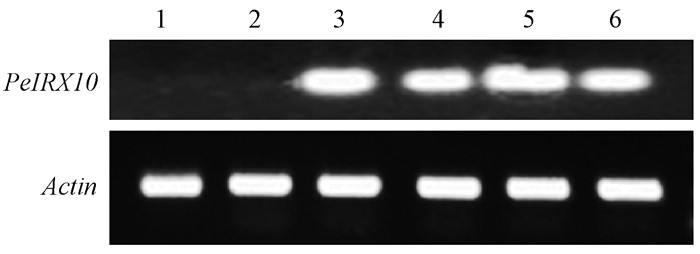
|
图 3 过表达PeIRX 10基因拟南芥植株RT-PCR检测 Fig.3 RT-PCR analysis of transformed plants of Arabidopsis thaliana for PeIRX 10 gene 1.野生型拟南芥;2.irx10l(-/ -)irx10(-/ -)双突植株;3-6.转PeIRX 10基因植株。 1. Wild type; 2.irx10l(-/ -)irx 10(-/ -) double mutant; 3-6. Transformed plants. |
|
|

|
图 4 野生型、irx10l(-/ -)irx10(-/ -)双突植株和互补型拟南芥植株的表型变化 Fig.4 Change of Arabidopsis thaliana phenotype for wild-type, irx 10 l(-/ -)irx 10(-/ -) double mutantsand complemented irx10l(-/ -)irx10(-/ -) double mutants withPeIRX 10 |
irx10l(-/-)irx10(-/-)双突植株次生细胞壁生长基本缺失(Wu et al., 2009),维管束间纤维和木质部导管细胞的细胞壁明显变薄(图 5B,E)。然而,PeIRX 10基因的互补植株中维管束间纤维和木质部导管细胞的细胞壁厚度(图 5C,F已经与野生型拟南芥(图 5A,D)相近。表明PeIRX 10基因能够互补拟南芥irx10l(-/-)irx10(-/-)双突植株细胞壁的次生加厚缺陷。
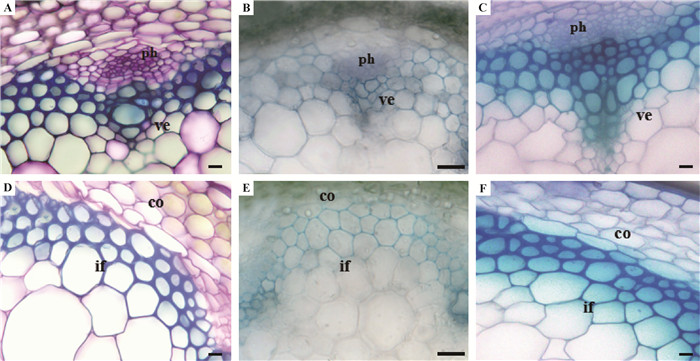
|
图 5 拟南芥野生型、irx10l(-/ -)irx10(-/ -)双突植株和互补植株茎的次生细胞壁变化
Fig.5 The changes of secondary cell wall in stem tissues of the wild type, irx10l(-/ -)irx10(-/ -) double mutants and complemented irx10l(-/ -)irx10(-/ -) double mutants withPeIRX 10
标尺Scale bars: 20 μm. ph:韧皮部;ve:导管;co:皮层;if:维管束间纤维。 A, D:野生型茎的横切面;B, E: irx10l (-/ -) irx10 (-/ -)双突植株茎的横切面;C, F: irx10l (-/ -) irx10 (-/ -)+ PeIRX 10 互补植株茎的横截面。 ph: Phloem; ve:Vessel; co:Cortex; if: Interfascicular fiber. A, D: Wild type; B, E: irx10l (-/ -) irx10 (-/ -) double mutants; C, F:Complemented irx10l (-/ -) irx10 (-/ -) double mutants with PeIRX 10 . |
irx10l(-/-)irx10(-/-)双突植株主要缺陷为木聚糖主链合成受阻,从而导致其细胞壁单糖组成及含量与野生型相比发生显著变化(Brown et al., 2009;Wu et al., 2009)。从表 2可以看出,与野生型相比,irx10l(-/-)irx10(-/-)双突植株中木糖含量减少至8%左右,而细胞壁中其他单糖(例如阿拉伯糖、半乳糖和葡萄糖等)的含量与野生型相比则有明显增加。而在互补植株中,木糖含量以及其他单糖的含量与野生型相近,这说明在irx10l(-/-)irx10(-/-)双突植株中,PeIRX 10基因的过表达可以使其细胞壁中的单糖含量恢复到与野生型相似。
|
|
为了进一步验证PeIRX 10基因的过表达在irx10l(-/-)irx10(-/-)双突植株中可以弥补植株中木聚糖合成不足,用单克隆抗体LM10进行标记,对互补植株做免疫荧光检测,发现LM10能够特异结合(4-O-甲基)葡萄糖醛酸木聚糖,但不能结合阿拉伯木聚糖或葡萄糖醛酸阿拉伯木聚糖(McCartney et al., 2005)。在野生型和互补型拟南芥茎的横切片中木质部具有强烈的免疫信号,而在irx10l(-/-)irx10(-/-)双突植株中没有检测到免疫信号(图 6),这表明PeIRX 10基因的过表达使irx10l(-/-)irx10(-/-)双突植株中木聚糖缺乏得到恢复。揭示了PeIRX 10基因的功能与拟南芥AtIRX 10基因相似,可能在毛竹木聚糖合成中起重要作用。
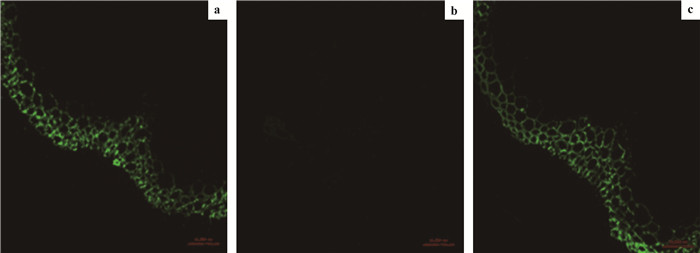
|
图 6 拟南芥野生型、 irx10l (-/ -) irx10 (-/ -)双突植株和互补植株茎部切片的LM10化学免疫观察
Fig.6 Immunolocalization of the LM10 epitope in stem tissues ofArabidopsis thaliana
标尺Scale bars: 50 μm. a.野生型;b. irx10l(-/ -)irx10(-/ -)双突植株;c. irx10l(-/ -)irx10(-/ -)+PeIRX 10互补植株。 a. Wild type; b. irx10l(-/ -)irx10(-/ -); c.irx 10 l(-/ -)irx 10(-/ -)+PeIRX 10. |
在某些单子叶禾本科(Gramineae)植物中,木聚糖主要以4-O-甲基葡萄糖醛酸-阿拉伯糖基木聚糖(glucuronoarabinoxylan, GAX)形式存在(Faik, 2010),它也以β-(1→4)-D-木糖为主链。IRX 10基因的体外活性已经通过一些生化方法检测到(Jensen et al., 2014;Urbanowicz et al., 2014),证实了IRX10蛋白具有β-(1→4)木聚糖糖基转移酶(xylosyltransferase,XylT)的活性, 主要功能涉及木聚糖主链的延伸。因此对IRX 10基因功能的研究为探究单子叶植物中木聚糖的合成机制提供理论基础。目前,Chen等(2013)研究发现将水稻中拟南芥AtIRX 10的同源基因OsIRX 10(Os01g70200/Os01g0926600)敲除后,突变体的木聚糖含量下降,特别是在水稻的茎秆中下降更为明显。Zhang等(2014)也通过对水稻中拟南芥另一个AtIRX 10的同源基因的克隆和互补分析,发现该基因能够基本修复拟南芥突变体中木聚糖合成缺陷。本试验发现毛竹PeIRX 10基因在irx10l(-/-)irx10(-/-)双突植株中过表达能够修复拟南芥双突纯合植株中由木聚糖合成缺失而造成的次生细胞壁缺陷。这与Chen等(2013)和Zhang等(2014)观点一致,表明在双子叶植物拟南芥和某些单子叶植物(如水稻、毛竹)中木聚糖主链的合成具有保守性(Wu et al., 2009)。PeIRX 10基因属于GT47类基因家族,其编码的蛋白与来自其他种属的IRX 10的一致性均在80%以上,充分显示了基因家族在进化上的保守性。因此可推测PeIRX 10基因与拟南芥中的IRX 10类似,参与木聚糖的生物合成,且对正常的次生壁加厚是必要的。
PeIRX 10基因的组织特异性表达结果表明,该基因在幼笋中高度表达,在根和叶片中的表达量相对较低。这可能与竹笋处于竹子生命活动初期阶段,具有快速的细胞伸长和次生细胞壁加厚过程有关。细胞壁多糖分析表明,与野生型拟南芥相比,irx10l(-/-)irx10(-/-)双突植株细胞壁中木聚糖含量显著降低,而拟南芥irx10l(-/-)irx10(-/-)双突植株中PeIRX 10基因的过表达使植株中木聚糖含量增加。说明PeIRX 10基因对细胞壁木聚糖的合成有重要作用。
4 结论本研究构建毛竹PeIRX 10基因过量表达载体并转化模式植物拟南芥,通过对拟南芥T1代植株的研究发现过表达PeIRX 10基因可使拟南芥双突纯合体表型恢复正常、细胞壁不正常的木质部消失,且细胞壁中木聚糖含量恢复到正常水平。同时,分子检测证实目的基因在转基因植株中得到表达。这表明毛竹的PeIRX 10基因可能与拟南芥AtIRX 10基因具有相同的生物学功能,通过参与木聚糖的生物合成而导致毛竹组织次生细胞壁的形成。
| [] | Boerjan W, Ralph J, Baucher M.2003. Lignin biosynthesis. Annu Rev Plant Biol , 54 : 519–546. DOI:10.1146/annurev.arplant.54.031902.134938 |
| [] | Brown D M, Zeef L A, Ellis J, et al.2005. Identification of novel genes in Arabidopsis involved in secondary cell wall formation using expression profiling and reverse genetics. Plant Cell , 17 (8) : 2281–2295. DOI:10.1105/tpc.105.031542 |
| [] | Brown D M, Goubet F, Wong V W, et al.2007. Comparison of five xylan synthesis mutants reveals new insight into the mechanisms of xylan synthesis. Plant J , 52 (6) : 1154–1168. DOI:10.1111/tpj.2007.52.issue-6 |
| [] | Brown D M, Zhang Z, Stephens E, et al.2009. Characterization of IRX10 and IRX10-like reveals an essential role in glucuronoxylan biosynthesis in Arabidopsis. Plant J , 57 (4) : 732–746. DOI:10.1111/tpj.2009.57.issue-4 |
| [] | Chen X, Vega-Sanchez M E, Verhertbruggen Y, et al.2013. Inactivation of OsIRX10 leads to decreased xylan content in rice culm cell walls and improved biomass saccharification. Mol Plant , 6 (2) : 570–573. DOI:10.1093/mp/sss135 |
| [] | Chiniquy D, Varanasi P, Oh T, et al.2013. Three novel rice genes closely related to the Arabidopsis IRX9, IRX9L, and IRX14 genes and their roles in xylan biosynthesis. Front Plant Sci , 4 (1) : 83. |
| [] | Fan C, Ma J, Guo Q, et al.2013. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis). PLoS One , 8 (2) : e56573. DOI:10.1371/journal.pone.0056573 |
| [] | Faik A.2010. Xylan biosynthesis: news from the grass. Plant Physiol , 153 (2) : 396–402. DOI:10.1104/pp.110.154237 |
| [] | Freeman J, Lovegrove A, Wilkinson M D, et al.2015. Effect of suppression of arabinoxylan synthetic genes in wheat endosperm on chain length of arabinoxylan and extract viscosity. Plant Biotechnol , 14 (1) : 109–116. |
| [] | Hayden C A, Jorgensen R A.2007. Identification of novel conserved peptideu ORF homology groups in Arabidopsis and rice reveals ancient eukaryotic origin of select groups and preferential association with transcription factor-encoding genes. BMC Biol , 5 (1) : 32. DOI:10.1186/1741-7007-5-32 |
| [] | Hörnblad E, Ulfstedt M, Ronne H, et al.2013. Partial functional conservation of IRX10 homologs in Physcomitrella patens and Arabidopsis thaliana indicates an evolutionary step contributing to vascular formation in land plants. BMC Plant Biol , 13 (1) : 3. DOI:10.1186/1471-2229-13-3 |
| [] | Jensen J K, Johnson N R, Wilkerson C G.2014. Arabidopsis thaliana IRX10 and two related proteins from psyllium and Physcomitrella patens are xylan xylosyltransferases. Plant J , 80 (2) : 207–215. DOI:10.1111/tpj.12641 |
| [] | Keppler B D, Showalter A M.2010. IRX14 and IRX14-LIKE, two glycosyltransferases involved in glucuronoxylan biosynthesis and drought tolerance in Arabidopsis. Mol Plant , 3 (5) : 834–841. DOI:10.1093/mp/ssq028 |
| [] | Lee C, O'Neill M A, Tsumuraya Y, et al.2007. The irregular xylem9 mutant is deficient in xylan xylosyltransferase activity. Plant Cell Physiol , 48 (11) : 1624–1634. DOI:10.1093/pcp/pcm135 |
| [] | Lee C, Teng Q, Huang W, et al.2009. Down-regulation of PoGT47C expression in poplar results in a reduced glucuronoxylan content and an increased wood digestibility by cellulase. Plant Cell Physiol , 50 (6) : 1075–1089. DOI:10.1093/pcp/pcp060 |
| [] | Lerouxel O, Cavalier D M, Liepman A H, et al.2006. Biosynthesis of plant cell wall polysaccharides: a complex process. Curr Opin Plant Biol , 9 (6) : 621–630. DOI:10.1016/j.pbi.2006.09.009 |
| [] | Lin J, He X, Hu Y, et al.2002. Lignification and lignin heterogeneity for various age classes of bamboo (Phyllostachys pubescens) stems. Physiol Plant , 114 (2) : 296–302. DOI:10.1034/j.1399-3054.2002.1140216.x |
| [] | Livak K J, Schmittgen T D.2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod. Methods , 25 (4) : 402–408. DOI:10.1006/meth.2001.1262 |
| [] | Luo Z S, Xu X L, Yan B F.2008. Accumulation of lignin and involvement of enzymes in bamboo shoot during storage. Eur Food Res Technol , 226 (4) : 635–640. DOI:10.1007/s00217-007-0595-y |
| [] | McCartney L, Marcus S E, Knox J P.2005. Monoclonal antibodies to plant cell wall xylans and arabinoxylans. J HistochemCytochem , 53 (4) : 543–546. |
| [] | Mortimer J C, Faria-Blanc N, Yu X, et al.2015. An unusual xylan in Arabidopsis primary cell walls is synthesised by GUX3, IRX9L, IRX10L and IRX14. Plant J , 83 (3) : 413–426. DOI:10.1111/tpj.2015.83.issue-3 |
| [] | Pauly M, Keegstra K.2008. Cell-wall carbohydrates and their modification as a resource for biofuels. Plant J , 54 (4) : 559–568. DOI:10.1111/j.1365-313X.2008.03463.x |
| [] | Pauly M, Keegstra K.2010. Plant cell wall polymers as precursors for biofuels. Curr Opin Plant Biol , 13 (3) : 305–312. |
| [] | Pena M J, Zhong R, Zhou G K, et al.2007. Arabidopsis irregular xylem8 and irregular xylem9: implications for the complexity of glucuronoxylan biosynthesis. Plant Cell , 19 (2) : 549–563. DOI:10.1105/tpc.106.049320 |
| [] | Scheible W R, Pauly M.2004. Glycosyltransferases and cell wall biosynthesis: novel players and insights. Curr Opin Plant Biol , 7 (3) : 285–295. DOI:10.1016/j.pbi.2004.03.006 |
| [] | Somerville C.2006. Cellulose synthesis in higher plants. Annu Rev Cell Dev Biol , 22 (1) : 53–78. DOI:10.1146/annurev.cellbio.22.022206.160206 |
| [] | Urbanowicz B R, Pena M J, Moniz H A, et al.2014. Two Arabidopsis proteins synthesize acetylated xylan in vitro. Plant J , 80 (2) : 197–206. DOI:10.1111/tpj.12643 |
| [] | Wu A M, Rihouey C, Seveno M, et al.2009. The Arabidopsis IRX10 and IRX10-L glycosyltransferases are critical for glucuronoxylan biosynthesis during secondary cell wall formation. Plant J , 57 (4) : 718–731. DOI:10.1111/tpj.2009.57.issue-4 |
| [] | Wu A M, Hornblad E, Voxeur A, et al.2010. Analysis of the Arabidopsis IRX9/IRX9-L and IRX14/IRX14-L pairs of glycosyltransferase genes reveals critical contributions to biosynthesis of the hemicellulose glucuronoxylan. Plant Physiol , 153 (2) : 542–554. DOI:10.1104/pp.110.154971 |
| [] | Zhang B L, Zhao T M, Yu W G, et al.2014. Functional conservation of the glycosyltransferase gene GT47A in the monocot rice. J Plant Res , 127 (3) : 423–432. DOI:10.1007/s10265-014-0631-5 |
| [] | Zhao X H, Ouyang K X, Gan S M, et al.2014. Biochemical and molecular changes associated with heteroxylan biosynthesis in Neolamarckia cadamba (Rubiaceae) during xylogenesis. Front Plant Sci , 5 : 602. |
| [] | Zhong R Q, Pena M J, Zhou G K, et al.2005. Arabidopsis fragile fiber8, which encodes a putative glucuronyltransferase, is essential for normal secondary wall synthesis. Plant Cell , 17 (12) : 3390–3408. DOI:10.1105/tpc.105.035501 |
| [] | Zhou G K, Zhong R, Richardson E A, et al.2006. The poplar glycosyltransferase GT47C is functionally conserved with Arabidopsis fragile fiber8. Plant Cell Physiol , 47 (9) : 1229–1240. DOI:10.1093/pcp/pcj093 |
| [] | Zhou G K, Zhong R Q, Himmelsbach D S, et al.2007. Molecular characterization of PoGT8D and PoGT43B, two secondary wall-associated glycosyltransferases in poplar. Plant Cell Physiol , 48 (5) : 689–699. DOI:10.1093/pcp/pcm037 |
 2016, Vol. 52
2016, Vol. 52

