文章信息
- 时小东, 朱学慧, 盛玉珍, 庄国庆, 陈放
- Shi Xiaodong, Zhu Xuehui, Sheng Yuzhen, Zhuang Guoqing, Chen Fang
- 基于转录组序列的楠木SSR分子标记开发
- Development of SSR Markers Based on Transcriptome Sequence of Phoebe zhennan
- 林业科学, 2016, 52(11): 71-78
- Scientia Silvae Sinicae, 2016, 52(11): 71-78.
- DOI: 10.11707/j.1001-7488.20161109
-
文章历史
- 收稿日期:2015-10-26
- 修回日期:2015-12-03
-
作者相关文章
2. 四川省雷波县林业局 雷波 616550;
3. 四川省林业科学研究院 成都 610081
2. Forestry Bureau of Leibo County, Sichuan Province Leibo 616550 ;
3. Sichuan Academy of Forestry Chengdu 610081
楠木(Phoebe zhennan)为我国特有樟科(Lauraceae)常绿大乔木,因其木材材质优良、树形优美而被广泛用于建筑和家具、庭院观赏及园林绿化(贺维等,2014;张炜等,2012)。由于长期的人为破坏和气候的变化,现存楠木群体规模较小,群体结构衰退日趋明显,使得楠木资源日趋濒危,被列为国家重点保护的Ⅱ级濒危树种,其种质资源保护和恢复性培育迫在眉睫。利用分子标记的手段对楠木遗传背景进行研究,可为楠木遗传多样性和分子标记辅助育种等奠定基础。
目前,国内外仍缺乏楠木全基因组和分子标记信息的研究,仅有张炜等(2011)建立了楠木RAPD体系。简单重复序列(simple sequence repeats,SSR),又称为微卫星,在基因组中是一类1~6个核苷酸组成的串联重复单元,可直接反映遗传多样性,并具有操作简单、稳定性好以及通用性好等优点,被广泛应用于品种鉴定、遗传多样性分析、进化研究以及分子育种等方面(Lu et al., 2015)。
基于新一代测序技术的转录组测序技术(RNA-seq),具有高效准确、快速和低成本等优势,因而在模式和非模式物种中得到广泛应用(Martin et al., 2013)。与传统的芯片技术相比,RNA-seq技术能够更加全面准确快速地获得组织中所有转录本信息,而且无需该物种基因组信息(Haas et al., 2010)。应用转录组测序的方法,获得大量的转录本信息,为在短时间内高效准确地开发SSR标记创造了极为便利的条件。目前,SSR标记技术已经在植物中得到广泛应用,如人参(Panax ginseng)、芒果(Mangifera indica)、中华猕猴桃(Actinidia chinensis)、桉树(Eucalyptus robusta)等(Li et al., 2013;刘果等,2013)。截止到2015年8月,美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库中仅公布了11条楠木DNA/RNA序列,远远无法进行SSR标记开发。
本研究基于楠木的转录组测序的结果进行全转录组SSR位点分析和挖掘,从而在RNA层面上揭示楠木SSR规律和特征,以期在分子水平上为楠木种质资源研究、亲缘关系鉴定以及分子辅助育种奠定基础。
1 材料与方法 1.1 转录组数据来源楠木种子采自四川省峨眉山,种植于四川省林业科学研究院基地,取幼苗全株,迅速置于液氮中,提取RNA后,构建测序文库,用Illumina HiSeqTM 2000平台进行测序。对原始测序数据进行过滤后采用Trinity (http://trinityrnaseq.sourceforge.net/)软件对经过过滤的高质量数据进行组装,共获得67 331条unigene。
1.2 转录组SSR位点筛选对楠木转录组高通量测序组装后获得的67 331条unigene运用MISA (http://pgrc.ipkgatersleben.de/misa/)软件进行SSR位点筛选和分析。其中筛选标准参数设置为:重复基元为1~6 bp,一、二、三、四、五、六核苷酸基元最小重复次数分别为10,6,5,5,5,5。
1.3 SSR引物设计及验证利用Primer 3.0进行引物批量设计,并从中随机挑选18对引物,由成都擎科梓熙生物技术有限公司进行合成,按照黄海燕等(2013)报道的方法进行DNA提取及PCR,程序为:94 ℃预变性4 min;35个循环(94 ℃变性30 s,60 ℃复性30 s,72 ℃延伸1 min);最后72 ℃延伸5 min。PCR产物用2%的琼脂糖凝胶电泳进行初步验证。
2 结果与分析 2.1 楠木转录组序列中SSR的数量对楠木转录组获得的67 331条unigene利用软件MISA进行搜索,从SSR分布(表 1)上看,共搜索得到9 405个SSR位点,SSR出现频率(搜索出的SSR个数与搜索序列数比值)为13.97%,平均每3.37 kb就会出现1个SSR位点(搜索序列总长度与SSR个数的比值)。所有SSR位点分布于6 667条unigene上,发生频率(搜索出的含有SSR的序列数与搜索序列数比值)为9.90%,其中,1 963条unigene含有1个以上SSR位点,含有复合型SSR的unigene数目为846条。
|
|
楠木转录组SSR种类较为丰富(表 2),共检索到166种重复基元种类,但各种不同重复类型基元之间数量差别较大,分布极不平衡。楠木转录组SSR种类中单核苷酸分布最多,SSR数目为3 890,占总SSR数目的41.36%,含有单核苷酸的unigene数目为3 614,发生频率为5.37%,平均长度为19 bp;其次是二核苷酸和三核苷酸,分别为30.68%(2 885个SSR位点,2 743条unigene)和26.46%(2 489个SSR位点,2 344条unigene),平均长度分别为23 bp和24 bp;而四、五、六核苷酸重复类型所占比例较小,SSR位点数量分别为102,22,15,SSR位点平均长度为30,34,45 bp,三者比例总和为1.5%。
|
|
在楠木转录组SSR中,三核苷酸类型最多,为60种;其次是四核苷酸基元类型,有58种;单、二、五、六核苷酸基元种类个数分别为4,11,18,15种(表 2)。在核苷酸重复类型中,A/T基元种类数目最多,所占比例高达40.67%,其次为二核苷酸重复类型中的AG/CT基元(2 595个,27.59%)和三核苷酸重复类型中的AAG/CTT (1 062个,11.29%)。四核苷酸重复类型中最多的基元种类为AAAG/CTTT (37,0.39%),五、六核苷酸基元种类所占比例较小( < 0.2%)。
三核苷酸重复类型的60个基元种类中,SSR位点数范围为1~222,最多的为GAA,其次是AGA (221个),AAG (203个),TCT (158个),TTC (149个)。从图 1可以看出,三核苷酸基元类型中AAG/CTT的SSR位点数目为1 062个,占总SSR位点数目的11.29%,出现频率为1.58%,远远高于其他三核苷酸基元类型;其次是AGC/CTG,AGG/CCT和ATC/ATG,分别占总SSR位点数目的3.75%,3.49%和2.49%。楠木转录组SSR中其他三核苷酸基元类型所占总SSR位点比例均不足2%。

|
图 1 SSR中三核苷酸基元类型分布 Fig.1 Distribution of trinucleotide motif in SSR |
SSR位点多态性主要是由基元重复次数的变化所引起的,从楠木SSR位点重复次数分布统计结果(图 2)可以看出,楠木转录组基元随着重复次数的增加,其数量和比例逐渐减少。楠木转录组SSR中重复次数主要集中在5~15次,占总数的91.21%,其中低重复次数(5~10次)的SSR位点为6 537,占总数的69.51%;一般重复次数(11~20次)中,11~15次的SSR位点为2 041个(21.70%),16~20次的分布有650个(6.91%) SSR位点;重复次数多于20次为较高重复次数,楠木转录组SSR较高重复次数中,分布有177个SSR位点,占总数的1.88%。

|
图 2 SSR位点数重复次数分布 Fig.2 Distribution of the number of repeats in SSRs |
对楠木转录组SSR中二、三核苷酸基元重复类型进行重复次数分析(图 3),2种核苷酸重复类型在重复次数方面与整体重复次数表现出一致的变化趋势,均随着重复次数的增加,转录组SSR位点数逐渐降低,低重复次数占主导。另外,二核苷酸重复次数普遍高于三核苷酸类型重复次数,其中二核苷酸类型重复6次的SSR数目最多,总数为763个,其次是重复7次,数目为556个;三核苷酸类型重复5次的SSR数目最多,总数为1 436个,其次是重复6次,数目为707个。一般重复类型中,三核苷酸类型仅出现4次;二核苷酸类型出现154次,主要集中在重复11次(132个),重复次数在12~20次的SSR数目仅为22个。三核苷酸类型中未检索到较高重复次数,二核苷酸中仅在GA基元类型中出现1次。
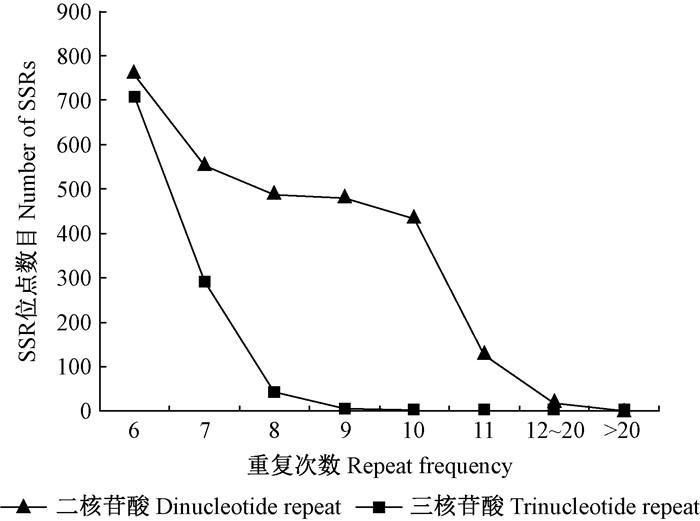
|
图 3 二、三核苷酸重复基元类型重复次数分布 Fig.3 Distribution of dinucleotide and trinucleotide repeat frequency |
除了基元重复次数之外,SSR长度是影响SSR分子标记多态性的另一个重要因素(Temnykh et al., 2001)。楠木转录组SSR中,以10 bp为筛选限制条件,SSR长度集中在10~24 bp (表 3)。低于12 bp的SSR数量为1 524个,占总SSR数量的17.24%,其中10 bp和11 bp的SSR数量分别为991个(11.21%)和533个(6.03%);12~20 bp的SSR数量为5 688个(64.33%);其他的SSR的长度为大于20 bp。其中楠木转录组SSR中长度在15 bp (1 378个,15.58%)最多,其次是18 bp (1 111,12.57%)。大多数研究表明,SSR长度能达到20 bp时,其多态性较高,是理想的标记位点;长度在12~20 bp时,其多态性中等;而低于12 bp时,其多态性极低。依照SSR长度和多态性研究,楠木SSR长度主要集中在中等多态性长度,长度低于12 bp的极低多态性SSR占17.24%,说明理论上具有中等以上的多态性。另有研究表明,高级基元SSR (四、五、六核苷酸重复)多态性普遍比低级基元(二、三核苷酸重复)的低(Dreisigacker et al., 2004),本研究筛选出的楠木SSR主要为低级基元,表明大部分楠木SSR位点具有较高的多态性潜能,预计其具有较高的利用价值和实用性。
|
|
选择不同产地的10份楠木核DNA混合样为模板,对合成的18对引物进行初步分析。由图 4可以看出,18对引物中有14对可以扩增出条带,其中的9对引物(表 4)可以扩增出预期大小的条带,扩增效率为50.0%;5对引物的扩增产物与预期大小不符,占验证引物的27.8%;剩余的4对引物扩增不出条带。
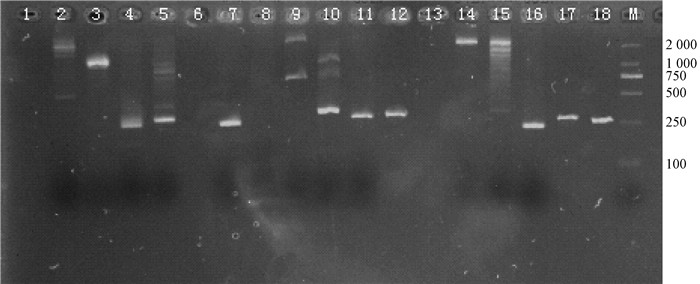
|
图 4 1-18号引物的扩增 Fig.4 Amplification of primer 1-18 |
|
|
全基因组测序受物种基因组大小、杂合度,以及成本等问题的限制,短时间内很难完成大量物种的测序。目前,楠木缺乏全基因组和遗传信息的研究,其分子标记研究、遗传多样性分析和分子育种等方面研究仍基本空白。而转录组测序不依赖于物种的全基因组信息,能够快速、准确、廉价地获得物种全转录本序列信息。近年来随着新一代测序技术的发展,基于转录组数据的SSR研究已经被广泛报道,如茶树(Camellia sinensis)(杨华等,2011)、橡胶树(Hevea brasiliensis)(甘霖等,2014)、油棕(Elaeis guineensis)(周丽霞等,2014)和芒果(罗纯等,2015)。与传统SSR标记方法相比,基于转录组的SSR分析表现出了一定的优越性。
本研究正是基于楠木转录组的67 331条转录本序列展开SSR分析和引物发掘,筛选出9 405个SSR位点,涉及6 667条unigene序列,分布频率为1/3.37 kb。楠木转录组SSR的分布频率高于红松(Pinus koraiensis)(1/17.38 kb)(张振等,2015)、毛果杨(Populus trichocarpa)(1/14.0 kb)(Fossati et al., 2005)、柑橘(Citrus)(1/5.2 kb)(Chen et al., 2006)等,这表明楠木转录组SSR数量上更加丰富;楠木SSR分布频率与茶树(1/3.68 kb)(杨华等,2011)相近,低于橡胶树(1/0.92 kb)(甘霖等,2014)等。SSR频率会出现差异,即使运用同种方法进行SSR分析也会出现不同,这是由多种因素造成的,如物种的基因组大小、搜索的重复数量和SSR的长度等(Varshney et al., 2005)。
从前人报道来看,多数植物SSR标记重复类型主要为二、三核苷酸重复,如茶树(刘振等,2008)、桃树(Prunus persica)(Jung et al., 2005)、橡胶树(Li et al., 2012)、芝麻(Sesamum indicum)(Wei et al., 2011)等为二核苷酸主导,而在毛果杨(Fossati et al., 2005)、番薯(Ipomoea batatas)(Wang et al., 2010)、拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)(Lawson et al., 2006)等为三核苷酸主导。本研究中发现,楠木转录组SSR二核苷酸重复类型数量(2 885个)多于三核苷酸重复类型数目(2 489个),其中二核苷酸中的AG/CT为优势重复基元,三核苷酸中优势重复基元为AAG/CCT,均与前人报道(Li et al., 2012)一致。
楠木转录组SSR标记二、三核苷酸类型重复次数多、跨度大,高级基元重复次数较少,而研究表明,重复次数与SSR标记的多态性具有正相关性(Gao et al., 2003),所以楠木转录组SSR中二核苷酸SSR理论上具备更高的多态性。此外,基于楠木转录组SSR重复序列长度的分析,也表明在理论上具有丰富的多态性,可作潜在的SSR重复基元序列进行有目的的引物设计和开发。
本研究基于楠木转录组的高通量测序数据,从总RNA水平上有针对性地进行了楠木特异性SSR位点的检索和评价,总的来说,楠木转录组SSR发生频率较高,类型丰富,可用性较高。因此,该研究结果能够为进一步的楠木SSR开发和筛选奠定基础,同时有助于在楠木分子数据匮乏的情况下,更好地开展楠木分子标记研究、遗传资源多态性评价、分子标记辅助育种等相关工作。
4 结论将楠木高通量转录组测序获得的67 331条unigene进行简单重复序列(SSR)位点挖掘,通过软件分析,共获得9 405个SSR位点,出现频率为13.97%,涉及序列数量为6 667条,发生频率为9.90%。SSR序列中包括166种重复基元类型,主导重复基元类型为A/T (40.67%),AG/CT (27.59%),AAG/CTT (11.29%)。SSR位点重复中10次比例最高,SSR位点数为1 621(17.24%),其次是5次(1 549,16.47%)和6次(1 495, 15.90%)。运用多态性分析的方式初步验证了SSR位点在楠木标记中的可行性。同时,利用Primer 3.0进行引物设计,随机筛选18对进行验证,9对引物可以扩增出预期大小的条带。本文通过对楠木高通量转录组序列的SSR信息的研究,为楠木遗传多样性分析和资源保护提供了方法。
| [] |
甘霖, 覃碧, 刘实忠, 等. 2014. 巴西橡胶树转录组中SSR位点的信息分析. 广东农业科学 , 41 (16) : 142–146.
( Gan L, Qin B, Liu S Z, et al.2014. Bioinformatic analysis of SSR markers in transcriptome of rubber tree Hevea brasiliensis Muell. Arg. Guangdong Agricultural Sciences , 41 (16) : 142–146. [in Chinese] ) |
| [] |
贺维, 胡庭兴, 王锐, 等. 2014. 施肥对桢楠幼苗光合生理及生长特性的影响. 西北植物学报 , 34 (6) : 1187–1197.
( He W, Hu T X, Wang R, et al.2014. Effect of fertilization on photosynthetic physiology and growth characteristics of Phoebe zhennan seedlings. Acta Botanica Boreali-Occidentalia Sinica , 34 (6) : 1187–1197. [in Chinese] ) |
| [] |
刘果, 张党权, 谢耀坚, 等. 2013. 桉树Genomic-SSR和EST-SSR引物的快速筛选与通用性研究. 林业科学 , 49 (2) : 127–133.
( Liu G, Zhang D Q, Xie Y J, et al.2013. Rapid screening and transferability analysis of genomic-SSR and EST-SSR primers in eucalypt. Scientia Silvae Sinicae , 49 (2) : 127–133. [in Chinese] ) |
| [] |
刘振, 王新超, 赵丽萍, 等. 2008. 基于EST-SSR的西南茶区茶树资源遗传多样性和亲缘关系分析. 分子植物育种 , 6 (1) : 100–110.
( Liu Z, Wang X C, Zhao L P, et al.2008. Genetic diversity and relationship analysis of tea germplasms originated from south western China based on EST-SSR. Molecular Plant Breeding , 6 (1) : 100–110. [in Chinese] ) |
| [] |
罗纯, 武红霞, 姚全胜, 等. 2015. 芒果转录组中SSR位点信息分析与引物筛选. 热带作物学报 , 36 (7) : 1261–1266.
( Luo C, Wu H X, Yao Q S, et al.2015. Data mining and primer screening of simple sequence repeats in mango transcriptome. Chinese Journal of Tropical Crops , 36 (7) : 1261–1266. [in Chinese] ) |
| [] |
黄海燕, 杜红岩, 乌云塔娜, 等. 2013. 基于杜仲转录组序列的SSR分子标记的开发. 林业科学 , 49 (5) : 176–181.
( Huang H Y, Du H Y, Wuyun T N, et al.2013. Development of SSR molecular markers based on transcriptome sequencing of Eucommia ulmoides. Scientia Silvae Sinicae , 49 (5) : 176–181. [in Chinese] ) |
| [] |
杨华, 陈琪, 韦朝领, 等. 2011. 茶树转录组中SSR位点的信息分析. 安徽农业大学学报 , 38 (6) : 882–886.
( Yang H, Chen Q, Wei C L, et al.2011. Analysis on SSR information in Camellia sinensis transcriptome. Journal of Anhui Agricultural University , 38 (6) : 882–886. [in Chinese] ) |
| [] |
张炜, 龙汉利, 贾廷彬, 等. 2011. 桢楠DNA提取和RAPD条件的优化. 四川林业科技 , 32 (4) : 55–57.
( Zhang W, Long H L, Jia T B, et al.2011. DNA extraction and optimization of RAPD reaction system for Phoebe zhennan. Journal of Sichuan Forestry Science and Technology , 32 (4) : 55–57. [in Chinese] ) |
| [] |
张炜, 何兴炳, 唐森强, 等. 2012. 四川桢楠生长特性与分布. 林业科技开发 , 26 (5) : 38–41.
( Zhang W, He X B, Tang S Q, et al.2012. Study on the growth characteristics and distribution of Phoebe zhennan in Sichuan. China Forestry Science and Technology , 26 (5) : 38–41. [in Chinese] ) |
| [] |
张振, 张含国, 莫迟, 等. 2015. 红松转录组SSR分析及EST-SSR标记开发. 林业科学 , 51 (8) : 114–119.
( Zhang Z, Zhang H G, Mo C, et al.2015. Transcriptome sequencing analysis and development of EST-SSR markers for Pinus koraiensis. Scientia Silvae Sinicae , 51 (8) : 114–119. [in Chinese] ) |
| [] |
周丽霞, 肖勇, 杨耀东. 2014. 油棕转录组SSR标记开发研究. 广东农业科学 , 41 (14) : 136–138.
( Zhou L X, Xiao Y, Yang Y D.2014. Development of SSR markers in oil palm (Elaeis guineensis) based on information from transcriptome sequencing. Guangdong Agricultural Sciences , 41 (14) : 136–138. [in Chinese] ) |
| [] | Chen C, Zhou P, Choi Y, et al.2006. Mining and characterizing microsatellites from citrus ESTs. Theor Appl Genet , 112 (7) : 1248–1257. DOI:10.1007/s00122-006-0226-1 |
| [] | Dreisigacker S, Zhang P, Warburton M L, et al.2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments. Crop Science , 44 (2) : 381–388. DOI:10.2135/cropsci2004.3810 |
| [] | Fossati Tiziana, Zapelli Ilaria, Bisoffi Stefano, et al.2005. Genetic relationships and clonal identity in a collection of commercially relevant poplar cultivars assessed by AFLP and SSR. Tree Genetics & Genomes , 1 (1) : 11–20. |
| [] | Gao L, Tang J, Li H, et al.2003. Analysis of microsatellites in major crops assessed by computational and experimental approaches. Mol Breed , 12 (3) : 245–261. DOI:10.1023/A:1026346121217 |
| [] | Haas B J, Zody M C.2010. Advancing RNA-Seq analysis. Nature Biotechnology , 28 (5) : 421–423. DOI:10.1038/nbt0510-421 |
| [] | Jung S, Abbott A, Jesudurai C, et al.2005. Frequency, type, distribution and annotation of simple sequence repeats in Rosaceae ESTs. Functional & Integrative Genomics , 5 (3) : 136–143. |
| [] | Lawson M J, Zhang L.2006. Distinct patterns of SSR distribution in the Arabidopsis thaliana and rice genomes. Genome Biology , 7 (2) : R14. DOI:10.1186/gb-2006-7-2-r14 |
| [] | Li C, Zhu Y, Guo X, et al.2013. Transcriptome analysis reveals ginsenosides biosynthetic genes, microRNAs and simple sequence repeats in Panax ginseng C. A. Meyer. BMC Genomics , 14 (1) : 1–11. DOI:10.1186/1471-2164-14-1 |
| [] | Li D, Deng Z, Qin B, et al.2012. De novo assembly and characterization of bark transcriptome using Illumina sequencing and development of EST-SSR markers in rubber tree (Hevea brasiliensis Muell. Arg.). BMC Genomics , 13 : 192–201. DOI:10.1186/1471-2164-13-192 |
| [] | Lu C, Zou C, Zhang Y, et al.2015. Development of chromosome-specific markers with high polymorphism for allotetraploid cotton based on genome-wide characterization of simple sequence repeats in diploid cottons (Gossypium arboreum L. and Gossypium raimondii Ulbrich). BMC Genomics , 16 (1) : 1–12. DOI:10.1186/1471-2164-16-1 |
| [] | Martin L B, Fei Z, Giovannoni J J, et al.2013. Catalyzing plant science research with RNA-seq. Frontiers in Plant Science , 4 (6) : 66. |
| [] | Temnykh S, Declerck G, Lukashova A, et al.2001. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.): Frequency, length variation, transposon associations, and genetic marker potential.. Genome Res , 11 (8) : 1441–1452. DOI:10.1101/gr.184001 |
| [] | Varshney R K, Graner A, Sorrells M E.2005. Genic microsatellite markers in plants: features and applications. Trends in Biotechnology , 23 (1) : 48–55. DOI:10.1016/j.tibtech.2004.11.005 |
| [] | Wang Z, Fang B, Chen J, et al.2010. De novo assembly and characterization of root transcriptome using Illumina paired-end sequencing and development of cSSR markers in sweetpotato (Ipomoea batatas). BMC Genomics , 11 : 726–739. DOI:10.1186/1471-2164-11-726 |
| [] | Wei W, Qi X, Wang L H, et al.2011. Characterization of the sesame (Sesamum indicum L.) global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers. BMC Genomics , 12 (1) : 451–463. DOI:10.1186/1471-2164-12-451 |
 2016, Vol. 52
2016, Vol. 52

